Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Phyton (Buenos Aires)
versión On-line ISSN 1851-5657
Phyton (B. Aires) vol.79 no.1 Vicente López ene./jun. 2010
ARTÍCULOS ORIGINALES
Variabilidad genética en 22 variedades híbridas de caña de azúcar (Saccharum spp. Híbrido)
Genetic variability in 22 sugarcane hybrid varieties (Saccharum spp. Hybrid)
Alejandre Rosas1 JA, ME Galindo Tovar2, HE Lee Espinosa2, OG Alvarado Gómez3
1 Universidad Veracruzana. Laboratorio de Alta Tecnología S.C. Orizaba, Veracruz. México. C.P. 94300.
2 Universidad Veracruzana. Facultad de Ciencias Biológicas y Agropecuarias. Peñuela, Amatlán de los Reyes, Veracruz. México. 94945.
3 Universidad Autónoma de Nuevo León. Facultad de Agronomía. Escobedo. Nuevo León. México. CP 66055.
Address Correspondence to: Jorge Alberto Alejandre Rosas. e-mail: jalejandre@uv.mx
Recibido - Received 25.V.2010.
Aceptado - Accepted 6.VII.2010.
Resumen. El desarrollo de nuevas variedades en caña de azúcar permite aumentar la producción de sacarosa, y enfrentar grandes desafíos como nuevas plagas y la pérdida de fertilidad de los suelos. El propósito del presente estudio fue aportar herramientas para la selección de progenitores útiles en los programas de cruzamiento. Para ello, se utilizaron marcadores moleculares RAPDs; se probaron 14 iniciadores y se seleccionaron los cuatro que generaron información suficiente para realizar el análisis genético de 22 variedades híbridas. Éstas fueron usadas como progenitores en los programas de hibridación en la Estación de Hibridación, Cámara Nacional de la Industria Azucarera y Alcoholera, Tapachula, Chiapas. En el análisis de distancia genética se obtuvieron tres grupos principales. El primer grupo incluyó variedades de origen mexicano y hawaiano. El segundo grupo fue una mezcla de variedades de México, de Barbados, Cuba e India, y el tercer grupo fue el más diverso incluyendo variedades de México, Cuba, Java, India y Brasil; esto sugiere un origen común y un alto grado de hibridación. Los grupos no guardaron una relación en cuanto a sus características agroindustriales, de origen o distribución geográfica. Esto sugiere a las variedades B 35187, POJ 2878 y Mex 68-P-23 como candidatos para cruzas dirigidas, ya que son los híbridos más separados en cuanto a la información genética obtenida.
Palabras clave: Marcadores moleculares; RAPDs; Caña de azúcar; Selección; Mejoramiento genético.
Abstract. Development of new sugarcane varieties allows to increase sucrose production and faces challenges as new pests and diseases, and soil fertility losses. The objective of this study was to contribute to expand molecular tools in selecting useful progenitors for hybridization programs. With this purpose, 14 RAPDs primers were pre-screened, and four of them which produced clearly resolved, reproducible and polymorphic fragment patterns were selected for genetic analysis of 22 sugarcane variety hybrids. These were used in the Estación de Hibridación at Tapachula, Chiapas, as progenitors in genetic improvement programs. Genetic distance analysis showed three main clusters. The first cluster contained sugarcane varieties from México and Hawaii. The second cluster included varieties from Barbados, Cuba and India, and the third cluster was the most diverse, with varieties from Mexico, Cuba, Java and Brazil; this suggests a common origin and a high hybridization degree. Cluster grouping was not related to agronomic characteristics, origin or geographic distribution. It is suggested that B 35187, POJ 2878 y Mex 68-P23 could be used as candidates for hybridization. This is because they are the most distant progenitors.
Keywords: Molecular markers; RAPDs; Sugarcane; Selection; Genetic improvement.
INTRODUCCIÓN
La caña de azúcar es un cultivo cuyo centro de origen es Nueva Guinea, desde donde se esparció a Asia, a las islas del Pacífico y posteriormente al mundo conocido de entonces. A fines del siglo XV fue traída a América por Cristóbal Colón. En México la producción de caña de azúcar y sus derivados es una de las principales agroindustrias. En 2005, México ocupó el sexto lugar como productor de caña de azúcar en el mundo con 45,126 millones de toneladas (FAO, 2008). El estado de Veracruz ha sido el principal productor de caña de azúcar en México (SIAP, 2008). Sin embargo, la producción de esta especie enfrenta grandes dificultades, y uno de los temas de mayor importancia es el desarrollo y caracterización de variedades mejoradas para la producción de sacarosa.
Tradicionalmente las variedades utilizadas como progenitores y las seleccionadas en los programas de mejoramiento genético han sido caracterizadas e identificadas mediante marcadores morfológicos y agronómicos. La obtención de nuevas variedades de caña de azúcar con dicha metodología ha sido un proceso largo y tedioso, que puede llevar hasta diez años.
Hoy en día, con el desarrollo de nuevas metodologías se han creado variedades híbridas adaptadas a diferentes suelos y climas, que además presentan resistencia a plagas y enfermedades. Si bien se tienen identificadas las variedades a sembrar, aún no se cuenta con la información genética actualizada del material sembrado. Además, la propagación por estacas no garantiza actualmente ni la calidad ni la pureza adecuada de este material de propagación. En la caña de azúcar, como resultado del proceso inter-específico de domesticación, las plantas comerciales son híbridos polianeuploides con números cromosómicos que exceden 100. Esto hace complejo su mejoramiento genético (Manners et al., 2004). Además, el desarrollo de cultivares está relacionado con el hecho de que la caña de azúcar es un monocultivo, que se mantiene sin rotación y sin barbecho por muchos años. Esto permite que las plagas y enfermedades se adapten y los daños a las plantaciones se acumulen. Por lo tanto, es necesario desarrollar nuevas variedades con alto rendimiento y con las ventajas necesarias para reemplazar a las variedades en cultivo (Heinz y Osgood, 1994). En caña de azúcar, como resultado de años de mejoramiento genético, las variedades comerciales que actualmente se encuentran en producción son de alto rendimiento, resistentes a enfermedades y buena producción en fábrica (Schenck et al., 2004). Las estrategias más empleadas por los mejoradores de caña de azúcar para la obtención de nuevas variedades han sido las cruzas biparentales que incluyen cruzas entre clones femeninos y masculinos, o policruzas en las que se utilizan más de un clon femenino y masculino (McIntyre y Jackson, 2001).
En la actualidad, no sólo es importante el desarrollo de nuevos híbridos. También es necesario conocer la pureza varietal y la estabilidad de estos híbridos. En estos temas, la biología molecular representa un apoyo importante.
En el mejoramiento genético, los marcadores moleculares tienen un gran potencial para mejorar la eficiencia de los procesos, no sólo en la marcación de las características seleccionadas, sino también en la precisión con que los genotipos pueden ser seleccionados (Soltis y Soltis, 1993). Ejemplo de ello son los RAPD (Polimorfismo del ADN amplificado al azar), que han sido utilizados en diversos estudios del germoplasma de la caña de azúcar (Alwala et al., 2006).
Los RAPDs son marcadores que proveen variación a nivel de loci múltiples, por lo que resultan de aplicación potencialmenteútil para recomendar genotipos de caña de azúcar para los programas de mejoramiento. La diversidad genética en caña de azúcar ha sido estudiada con RAPDs por Nair et al. (1999) y por Khan et al. (2009), y con microsatélites por Ríasco et al. (2003). En ambos casos se reporta una base genética limitada para este híbrido. En caña de azúcar también se han explorado mapas de ligamiento con RAPDs; Al-Janabi et al. (1993) estudiaron progenies de Saccharum spontaneum y estimaron el tamaño del mapa en aproximadamente 2550 centimorgans. Por otro lado, Mudge et al. (1996), desarrollaron un mapa de ligamiento genético y observaron una segregación de 1:1 y 50 grupos de ligamiento, que resultaron muy importantes para el uso de la selección asistida por marcadores moleculares.
El objetivo del presente trabajo fue evaluar la variación genética presente en 22 variedades híbridas de caña de azúcar y agruparlas según su similitud, tomando como base el análisis del ADN utilizando RAPDs. El conocimiento de la diversidad genética permite preservar y usar de manera dirigida los recursos fitogenéticos de una especie o grupo de especies. Por lo tanto, los estudios sobre diversidad genética e interrelaciones entre variedades permiten obtener información útil que facilita el desarrollo de materiales mejorados.
MATERIALES Y MÉTODOS
Se seleccionaron 22 variedades híbridas de caña de azúcar de la Estación de Hibridación perteneciente a la CNIAA (Cámara Nacional de la Industria Azucarera y Alcoholera), Tapachula, Chiapas, México. Sus características de origen se muestran en las Tablas 1 y 2. En base en sus características agronómicas (Tabla 3), se seleccionaron las variedades más utilizadas en los programas de mejoramiento en los últimos 10 años, especialmente las que ocupan una mayor área cultivada en las diferentes zonas cañeras de México.
Tabla 1. Progenitores de las 22 variedades híbridas de caña de azúcar utilizadas en este estudio
Table 1. Parents of the 22 sugarcane hybrid varieties used in this study.

Tabla 2. País de origen de las 22 variedades híbridas de caña de azúcar utilizadas en este estudio
Table 2. Country of origin for the 22 sugarcane hybrid varieties used in this study.

Table 3. Características agronómicas de las 22 variedades híbridas de caña de azúcar seleccionadas para este estudio.
Table 3. Agronomic characteristics of the 22 sugarcane hybrid varieties used in this study.
De cada variedad se recolectaron dos tallos, que fueron llevados al Laboratorio de Alta Tecnología de Orizaba (LATO). Se eligieron cinco yemas sanas de la parte media del tallo, que se sembraron para su brotación en un invernadero.
Para realizar la extracción de ADN se utilizaron cinco plántulas de 20 cm de altura por cada variedad. Se eligieron las hojas más jóvenes y sanas y de ellas se utilizó la parte media (eliminando la nervadura central). Se compararon tres métodos de extracción: el paquete comercial NucleoSpin® Plant de la casa Machery-Nagel, el método de Dellaporta et al. (1989), y el de Doyle y Doyle (1990). El ADN extraído se almacenó a -70 °C para su posterior uso. La calidad del ADN extraído se verificó en geles de integridad utilizando agarosa al 1%, teñida con bromuro de etidio, y se visualizó en un transiluminador con luz ultravioleta. La cantidad y pureza del ADN se estimó midiendo la absorción de luz de las muestras a longitudes de onda de 260 y 280 manómetros en un espectrofotómetro Perkin Elmer Lambda 12.
El ADN extraído se amplificó con la técnica de la reacción en cadena de la polimerasa (PCR), de acuerdo con el protocolo de Harvey et al. (1996). Se utilizó un termociclador Perkin Elmer, Amplitron I. La técnica se estandarizó para las condiciones del LATO, utilizando un volumen de reacción de 25 μl. Para determinar las condiciones óptimas para la amplificación, se estandarizaron los reactivos. Para ello, se tomaron cinco muestras al azar y se probaron diferentes concentraciones de ADN, MgCl2 y Taq ADN polimerasa. De acuerdo a esta prueba, la mezcla final de reacción consistió en (1) 1 U de enzima Taq Polimerasa (Promega); (2) buffer 1X (Tris-HCl 50mM; pH 8,0; KCl 100mM; EDTA 0,1mM; DTT 1mM; glicerol 50%; Tween® 20 0,5% y Nonidet®-P40 0,5%); (3) MgCl2 2,5 mM; (4) dNTP 200 μM para cada uno de ellos; (5) 20 ng del oligonucleótido iniciador de diez bases de longitud con secuencia aleatoria, y (6) 35 ng de ADN genómico de caña de azúcar.
Para cada una de las variedades se probaron 14 iniciadores RAPDs (MWG Inc. Biotech). De estos iniciadores se utilizaron los cuatro que mostraron mejor amplificación. Las condiciones de amplificación fueron las reportadas por Williams et al. (1990) para estos iniciadores.
Los productos de amplificación se separaron en un gel de agarosa al 1,5%, se tiñeron con bromuro de etidio, se observaron en un transiluminador de luz ultravioleta y se tomaron fotografías para su análisis.
Los productos de amplificación se capturaron en matrices de datos registrados como variables discretas (presencia-ausencia) y el análisis de los datos se efectuó usando el programa POPGENE versión 3.2 (Yeh y Boyle, 1997). La similitud genética se estimó con el índice de Dice; la diversidad genética de acuerdo a Nei (1978) y el índice de Shanon; la distancia genética como la función decreciente de similitud, y el dendograma se construyó usando el análisis de vecino más cercano (Saitou y Nei, 1987).
RESULTADOS Y DISCUSIÓN
Al comparar los tres métodos para la extracción de ADN, los mejores resultados para pureza del ADN, medida con la relación OD260/OD280, se obtuvieron con el paquete comercial NucleoSpin Plant. Los valores de pureza observados oscilaron entre 1,9 y 2,0, lo que indica una buena calidad de ADN. Por otro lado, la cantidad de ADN recuperado varió de los 12 hasta 19 μg/μL, y los productos de amplificación del ADN al separarse presentaron bandas nítidas y con excelente resolución, por lo que este método se consideró confiable.
En cuanto a la estandarización de las concentraciones de ADN, MgCl2 y Taq ADN polimerasa se encontró que la mejor cantidad de ADN para los oligos utilizados fue de 20 ng; la concentración de MgCl2 con la que se obtuvo la mejor resolución para la amplificación de ADN fue de 2,5 mM, y la concentración adecuada de Taq polimerasa para las condiciones de amplificación en el programa térmico utilizado fue de 1 unidad por reacción.
De los 14 iniciadores utilizados en el presente estudio, se eligieron para ser empleados en el análisis de diversidad genética utilizando PCR, los cuatro que mostraron consistencia en la presencia de bandas: A-01, B-05, B-19 y C-13. Para estos cuatro iniciadores, las bandas generadas presentaron un rango entre 150 y 1800 pares de bases, exhibiendo bajo polimorfismo.
Los mayores valores de diversidad genética (Tabla 4) fueron generados con el iniciador B-19 con valor de 0,49; con una media general de 0,22, lo que indica poca variación dentro de la población estudiada, y por lo tanto una limitada base genética para programas de mejoramiento genético. En cuanto al índice de Shannon, el valor más alto fue encontrado en los iniciadores A-01 y B-19, ambos con un valor de 0,68, con un promedio general de 0,35. Aunque la variabilidad fue baja, se sugiere que el iniciador B-19 es un buen indicador de la variabilidad genética en las variedades de caña de azúcar estudiadas.
Tabla 4. Valores de diversidad y distancia genética de Nei para las 22 variedades. En la parte superior e inferior de la diagonal se muestran la identidad genética y la distancia genética, respectivamente.
Table 4. Nei's measures of genetic identity and genetic distance for the 22 sugarcane hybrid varieties. Genetic identity and genetic distance are shown in the upper and lower part of the diagonal, respectively.


Los valores de diversidad genética indican que a través del proceso de domesticación y la introducción de muestras pequeñas de material genético se han producido cuellos de botella que han reducido la variabilidad. Esto ha redundado en la baja efectividad de las cruzas para obtener nuevos materiales genéticos con los requerimientos de calidad adecuados para la agroindustria azucarera.
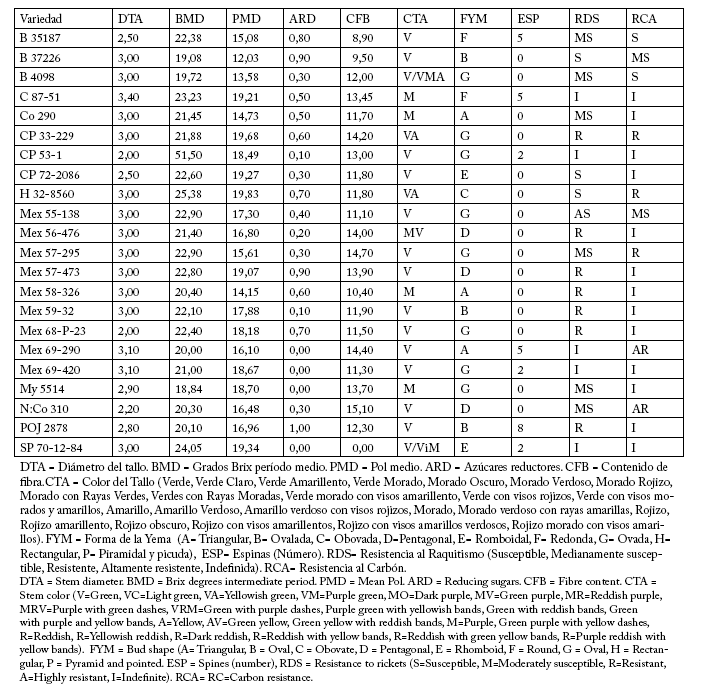
Los cuatro iniciadores generaron un total de 39 bandas, lo que indica que se debe probar una mayor cantidad de iniciadores para estudiar la diversidad genética en caña de azúcar con RAPDs. El promedio de bandas fue de 25 por variedad, siendo la variedad H 32-8560 la que generó una menor cantidad (18 bandas), y la CP 33-229 la de mayor número de bandas (30 bandas), como se muestra en la Figura 1.
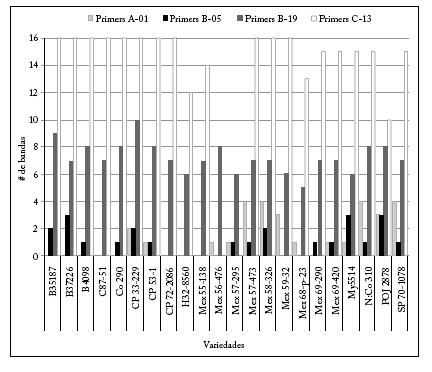
Fig. 1. Bandas generadas por 22 híbridos de caña de azúcar usando cuatro iniciadores RAPD.
Fig 1. Bands generated from 22 sugarcane hybrids using four RAPDs primers.
En las corridas electroforéticas, el iniciador B-05 presentó siete bandas polimórficas, que representan el 100% del total de los fragmentos registrados para este iniciador. El índice de afinidad es del 100%, lo que indica que todas las bandas son compartidas; sin embargo, para el iniciador B-05 se observaron perfiles de bandeo específicos para las variedades B 35187, B 37226, My 5514 y POJ 2878, lo que permitió distinguirlas de las otras, a pesar de la homogeneidad genética de las variedades analizadas.
El iniciador A-01 presentó cinco bandas, de las cuales todas fueron polimórficas, con un índice de afinidad del 100%. Esto indica que todas las bandas son compartidas. En este caso no se observaron patrones de bandeo específicos para ninguna variedad.
El iniciador B-19 tuvo un total de 11 bandas, cinco de ellas fueron monomórficas y seis polimórficas, lo que representó el 54,55% de bandas polimórficas. Aunque este iniciador generó 159 fragmentos, representando el 29,28% del total de fragmentos de los cuatro iniciadores, el índice de afinidad fue del 100% y no se observaron patrones de bandeo específicos. Debido a esto, este iniciador no se recomienda para estudios de diversidad en estas variedades.
El iniciador C-13 es el que mostró mayor número de bandas (16), con 9 bandas polimórficas y 7 monomórficas. Ninguna de estas bandas fue específica para las variedades probadas, lo que indica una cercana relación genética.
Estos resultados muestran la limitada diversidad genética de las variedades estudiadas, lo que coincide con lo encontrado por Nair et al. (1999). Estos autores encontraron un bajo nivel de diversidad genética para Saccharum. Esto probablemente se debe a que en la búsqueda de características que permitan una mejor producción de azúcar y manejo agronómico, la presión de selección ha significado un cuello de botella en la reducción de la diversidad genética. Además, siendo la caña de azúcar una especie domesticada hace mucho tiempo, es de esperarse una reducción de su diversidad genética (Gepts, 2004; Zeder et al., 2006).
Por otro lado, para mejorar el proceso de identificación de variedades es necesario incorporar otro tipo de marcadores moleculares que permitan detectar mayor diversidad genética.
La distribución de la frecuencia de las bandas polimórficas para las distintas variedades no fue homogénea (Fig. 2). Sin embargo, el marcador C-13 no fue sólo el que presentó el mayor número de bandas polimórficas (13 a 19), sino que además presentó polimorfismo en todas las variedades estudiadas. B-09, aunque presentó un menor número de bandas (7 a 5) también presentó polimorfismo en todas las variedades. Esto no sucede con los marcadores A-01 y B-05, que además de su baja proporción de polimorfismo, sólo estuvieron presentes en 17 variedades. La distribución irregular de los marcadores utilizados indica variantes alélicas entre el material genético analizado. Este resultado coincide con la investigación realizada por Janoo et al. (1999). Además, el gran número de bandas comunes sugiere asociaciones muy antiguas entre las variedades estudiadas.
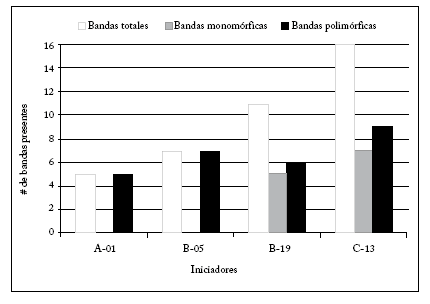
Fig. 2. Frecuencia del número de bandas obtenidas en la amplificación del ADN con los cuatro iniciadores RAPD.
Fig. 2. Band frequency obtained in the DNA amplification using the four most resolved RAPD primers.
En el análisis por distancia genética (Fig. 3) se observaron dos grupos principales. En el primer grupo, se observaron tres subgrupos: (1) uno que incluye variedades de origen mexicano y de Barbados; (2) el segundo subgrupo, muy diverso, incluye variedades de Hawaii, México, India y Cuba, y (3) en el tercer subgrupo se agrupan tres variedades mexicanas. En el segundo grupo se incluyeron variedades mexicanas, sudafricanas y brasileñas. Por último, las variedades Mex 68-P-23 (México) y POJ 2878 (Java) se hallaron separadas de los dos grupos. Esta mezcla de variedades sugiere un origen común y un alto grado de hibridación. Si bien con el uso de estos iniciadores encontramos información para separar grupos, éstos no guardan una relación en cuanto a sus características agroindustriales, ni de origen o geográficas. Esto probablemente se deba al intercambio de material entre las diferentes zonas geográficas, o a que las condiciones de clima y edafológicas fueron similares, y la selección no favoreció la formación de clones diferentes. Estos resultados coinciden con lo encontrado por Khan et al. (2009) respecto a que la diversidad genética entre los clones utilizados en mejoramiento genético es baja. Sin embargo, los datos obtenidos permiten sugerir que las variedades Mex 68-P-23 y POJ 2878, que se encuentran separadas de los dos grupos, y la variedad B 35187, que es la más distante, son candidatos apropiados para realizar cruzas dirigidas.
Fig. 3. Dendrograma para las 22 variedades híbridas de caña de azúcar con base a las distancias genéticas, usando la distancia UPGMA y datos de los iniciadores A-01, B-05, B-19 y C-13.
Fig. 3. UPGMA dendrogram of 22 sugarcane hybrids from four RAPD primers data (A-01, B-05, B-19 y C-13).
CONCLUSIONES
El uso de los marcadores moleculares RAPDs constituyó una herramienta útil; ya que mostró alta reproducibilidad y permitió identificar las similitudes y diferencias entre los híbridos de la caña de azúcar.
La mayor distancia genética se observó entre las variedades híbridas B-35187 y Mex 68-P-23 con POJ-2878, por lo queéstos son candidatos como progenitores en cruzas dirigidas. Sin embargo, debido a la homogeneidad genética observada, se sugiere ampliar la base de germoplasma para los programas de mejoramiento en caña de azúcar.
AGRADECIMIENTOS
Los autores agradecen a la estación de Hibridación de Tapachula, Chiapas, por el material genético aportado para esta investigación; al Dr. Eloy Camacho Díaz, Director del LATO, por el apoyo otorgado, y los subsidios aportados por el COVECyT fondo 37161 y FPV 300686.
REFERENCIAS
1. Al-Janabi, S.M., R.J. Honeycutt, M. McClelland y B.W.S. Sobral (1993). Genetic linkage map of Saccharum spontaneum L. 'SES 208'. Genetics 134: 1249-1260. [ Links ]
2. Alwala, S., A. Suman, J.A. Arro, J.C. Veremis y C.A. Kimbeng (2006). Target region amplification polymorphism (TRAP) for assessing genetic diversity in sugarcane germplasm collections. Crop Science 46: 448-455. [ Links ]
3. Dellaporta, S.L., J. Wood y J.B. Hicks (1989). A plant DNA minipreparation: version II. Plant Molecular Biology Reporter 1: 19-21. [ Links ]
4. Doyle, J.J. y J.L. Doyle (1990). Isolation of plant DNA from tissue. Focus 12: 13. [ Links ]
5. FAO (2008). Principales productores de alimentos y productos agrícolas - Países por producto básico. Disponible en línea. http:// www.fao.org/es/ess/top/commodity.html?lang=es. Consultada el 4 de enero de 2009. [ Links ]
6. Gepts, P. (2004). Crop domestication as a long-term selection experiment. En: J. Janick (ed). Plant Breeding Reviews 24: 1-44. [ Links ]
7. Harvey, M. y E.C. Botha (1996). Use of PCR-based methodologies for the determination of DNA diversity between Saccharum varieties. Euphytica 89: 257-265. [ Links ]
8. Heinz, D.J. y R. Osgood (1994). Sugarcane. En: Encyclopedia of Agricultural Science. C.J. Arntzen y E.M. Ritter (ed). Academic Press. USA. [ Links ]
9. Jannoo, N., L. Grivet, M. Seguin, F. Paulet, R. Domaingue, P.S. Rao, A. Dookun, A. D'Hont y J.C. Glaszmann (1999). Molecular investigation of the genetic base of sugarcane cultivars. Theoretical Applied Genetics 99: 171-184. [ Links ]
10. Khan, F.A., A. Khan, F.M. Azhar y S. Rauf (2009). Genetic diversity of Saccharum officinarum accessions in Pakistan as revealed by random amplified polymorphic DNA. Genetics and Molecular Research 8: 1376-1382. [ Links ]
11. Manners, J., L. McIntyre, R. Casu, G. Cordeiro, M. Jackson, K. Aitken, P. Jackson, G. Bonnett, S. Lee y R. Henry (2004). Can genomics revolutionize genetics and breeding in sugarcane? Proceedings of the 4th International Crop Science Congress. Brisbane, Australia, 26 Sep - 1 Oct 2004. [ Links ]
12. McIntyre, C.L. y P.A. Jackson (2001). Low level of selfing found in a sample of crosses in Australian sugarcane breeding programs. Euphytica 117: 245-249. [ Links ]
13. Mudge, J., W.R. Andersen, R.L. Kehrer y D.J. Fairbanks (1996). A RAPD genetic map of Saccharum officinarum. Crop Science 36: 1362-1366. [ Links ]
14. Nair, N.V., S. Nair, T.V. Sreenivasan y M. Mohan. (1999). Analysis of genetic diversity and phylogeny in Saccharum and related genera using RAPD markers. Genetic Resources and Crop Evolution 46: 73-79. [ Links ]
15. Nei, M. (1987). Molecular evolutionary genetics. Columbia University Press. [ Links ]
16. Ríasco, J.J., J.I. Victoria y F. Ángel (2003). Diversidad genética en variedades de caña de azúcar (Saccharum spp.) usando marcadores moleculares. Revista Colombiana de Biotecnología V: 6-15. [ Links ]
17. Saitou, N. y M. Nei (1987). The neighbor-joining method: a new method for reconstructing phylogenetic trees. Molecular Biology and Evolution 4: 406-425. [ Links ]
18. Schenck, S., M.W. Crepeau, K.K. Wu, P.H. Moore, Q. Yu y R. Ming (2004). Genetic diversity and relationships in native Hawaiian Saccharum officinarum sugarcane. Journal of Heredity 95: 327-331. [ Links ]
19. SIAP (2008). Sistema de Información Agroalimentaria y Pesquera, SAGARPA. Disponible en línea http://reportes.siap.gob.mx/ Agricola_siap/ResumenProducto. Consultada el 8 de noviembre del 2008. [ Links ]
20. Soltis, D.E. y P.S. Soltis (1993). Molecular data and the dynamic nature of polyploidy. Critical Reviews in Plant Sciences 12: 243-273. [ Links ]
21. Williams, J.G.K., A.R. Kubelik, K.J. Livak, J.A. Rafalski y S.V. Tingey (1990). DNA polymorphisms amplified by arbitrary primers are useful as genetic markers. Nucleic Acids Research 18: 6531-6535. [ Links ]
22. Yeh, F.C. y T.J.B. Boyle (1997). Population genetic analysis of codominant and dominant markers and quantitative traits. Belgian Journal of Botany 129: 157. [ Links ]
23. Zeder, M.A., E. Emshwiller, B.D. Smith y D.G. Bradley (2006). Documenting domestication: the intersection of genetics and archeology. Trends in Genetics 22: 138-155. [ Links ]














