Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Darwiniana, nueva serie
versión impresa ISSN 0011-6793versión On-line ISSN 1850-1699
Darwiniana v.42 n.1-4 San Isidro ene./dic. 2004
Variabilidad genética en Prosopis ferox (Mimosaceae)
Alicia D. Burghardt1,3, Shirley M. Espert1 & Rolando H. Braun Wilke2
1 Laboratorio de Plantas Vasculares, Dpto. de Biodiversidad y Biología Experimental, Universidad de Buenos Aires, C1428EHA Buenos Aires, Argentina. E-mail: alibu@bg.fcen.uba.ar
2 Facultad de Ciencias Agrarias, Universidad Nacional de Jujuy, San Salvador de Jujuy, Argentina.
3 Miembro de la Carrera del Investigador, CONICET.
RESUMEN: Burghardt, A. D., Espert, S. M. & Braun Wilke, R. H. 2004. Variabilidad genética en Prosopis ferox (Mimosaceae). Darwiniana 42(1-4): 31-36.
Prosopis ferox (Mimosaceae) es una especie arbustiva o arbórea espinosa que se distribuye desde el Sur de Bolivia hasta el noroeste de la Argentina. En la provincia de Jujuy se encuentra a grandes alturas (entre los 2400 y los 3700 m s.m.). Existe una gran variabilidad morfológica, especialmente en cuanto a las dimensiones del fruto y la cantidad de semillas por fruto, ambas características importantes debido al uso de esta planta como forraje. Con el objeto de verificar si existe además variabilidad genética, se realizó un estudio electroforético de proteínas seminales de árboles procedentes de distintas localidades de la provincia de Jujuy. Los patrones polipeptídicos obtenidos por SDS-PAGE presentaron en total 26 bandas. Cada población se caracterizó por sus patrones de presencia-ausencia de bandas, habiéndose encontrado variabilidad intrapoblacional (polimorfismo) en algunas de ellas, siendo otras genéticamente homogéneas. Los índices polimórficos en poblaciones de P. ferox son comparables a los obtenidos previamente en P. ruscifolia. La variabilidad genética interpoblacional hallada por medio del estudio electroforético de las proteínas seminales hace suponer la existencia de ecotipos.
Palabras clave: Prosopis ferox; Mimosaceae; "Churqui"; Variabilidad genética; Proteínas seminales.
ABSTRACT: Burghardt, A. D., Espert, S. M. & Braun Wilke, R. H. 2004. Genetic variation in Prosopis ferox (Mimosaceae). Darwiniana 42(1-4): 31-36.
Prosopis ferox (Mimosaceae) is a spiny bush or tree, distributed in southern Bolivia and northwestern Argentina. Populations of P. ferox from Jujuy grow at high elevations (2400 to 3700 m). There is in this species a great morphological variability, especially in fruit dimensions and the number of seeds per fruit. These characteristics are important because of its use as forage. With the aim of analyzing the genetic variability among and within populations, a seed protein electrophoretic study of different accessions from the province of Jujuy was performed. The polipeptidic patterns obtained by SDS-PAGE showed a total of 26 bands. The analyzed populations could be distinguished by their protein profiles, and intrapopulation variability (polymorphism) was found in some of them, while others were monomorphic. The polymorphism indexes found in P. ferox were comparable to those previously found in P. ruscifolia. The genetic variability found among populations of P. ferox, using seed proteins analysis, suggests the existence of ecotypes.
Key words: Prosopis ferox; Mimosaceae; "Churqui"; Genetic variability; Seed proteins.
Original recibido el 19 de diciembre de 2003;
aceptado el 14 de junio de 2004.
INTRODUCCIÓN
Prosopis ferox Griseb. es una especie arbustiva o arbórea de tronco corto, espinosa, perteneciente a la Sección Strombocarpa (Serie Cavenicarpae), conocida con la denominación vernácula "Churqui" en la mayor parte de su área de distribución. Ésta se extiende desde el Sur de Bolivia hasta el NO de la Argentina, llegando a los valles secos de la prepuna salteña. En la provincia de Jujuy se encuentra a grandes alturas (entre los 2400 y los 3700 m s.m.).
La distribución de P. ferox abarca una diversidad de ambientes, que resulta de la variación altitudinal, edáfica, pluvial y térmica (Braun Wilke, 1991).
La gran variabilidad morfológica encontrada, en especial en lo referente a la forma y el tamaño de los frutos ("choloncas"), así como a la cantidad de semillas por fruto, ha hecho suponer la existencia de ecotipos. Es posible que la intensa radiación solar, así como la amplitud térmica diaria resultante, hayan sido los principales factores causantes de las adaptaciones de esta especie al ambiente, que condujeron a dicha variabilidad (Braun Wilke et al., 1999).
Debe tenerse en cuenta que los ecosistemas que integra P. ferox han sido afectados por la acción del hombre. Cabe señalar que las cuencas hidrográficas donde se encuentran sus rodales ya eran, en tiempos prehispánicos, importantes vías de tránsito y sitios de asentamientos indígenas. La especie es utilizada como forrajera, melífera, protectora de suelos, refugio, y para la obtención de madera, la cual es aprovechada como combustible y material para artesanías desde tiempos precolombinos, llegando a ser de tal magnitud su explotación que la puso en peligro de extinción (Braun Wilke, 1991).
Los patrones polipeptídicos de proteínas seminales, obtenidos mediante electroforesis, han demostrado ser de utilidad para el estudio de la variabilidad intraespecífica en varias familias de plantas y en especial en Leguminosas (Thome et al., 1995; Przybylska & Zimniak-Przybylska, 1997; Burghardt & Palacios, 1998). Para verificar si existe variabilidad genética entre y dentro de las poblaciones, se realizó un estudio electroforético de proteínas seminales. Con el fin de llevar a cabo este estudio, se analizaron colecciones de distintas localidades de la provincia de Jujuy.
MATERIALES Y MÉTODOS
Ejemplares analizados
Los ejemplares de Prosopis ferox aquí estudiados fueron coleccionados por Rolando Braun Wilke (RBW) en las siguientes localidades de la provincia de Jujuy, República Argentina.
Puna: Dpto. Cochinoca: Cerro Negro, límite entre Salta y Jujuy, 24-V-1989, RBW 98-99-101-102- 103 (JUA); Encrucijada, en el cruce de Rutas 16 y 40, Salinas Grandes, 24-V-1989, RBW 104-105-106- 107-108 (JUA). Dpto. Yavi: Yavi, cerca del límite con Bolivia, 24-V-1989, RBW 126-127-128-129- 130 (JUA).
Prepuna: Dpto. Humahuaca: Chorrillos, 24-V- 1989, RBW 109-110-111-112-113 (JUA); Humahuaca, 24-V-1989, RBW 114-115-116-117-118 (JUA); Mocote, 24-V-1989, RBW 119-120-121-122-123 (JUA); Ovara, 24-V-1989, RBW 131-132-133-134- 135 (JUA); Peñas Blancas 24-V-1989, RBW 93-94-95- 96-97 (JUA); Sapagua, 24-V-1989, RBW 136-137- 138-139-140 (JUA). Dpto. Tilcara: Juella, 24-V-1989, RBW 141-142-143-144-145 (JUA).
En la Figura 1 se indica la ubicación de las localidades estudiadas. En cada una de ellas se realizó un muestreo al azar de los árboles, recolectándose los frutos individualmente. Se analizaron como mínimo 10 semillas por árbol, totalizando un mínimo de 50 individuos por población.

Fig. 1.- Ubicación de las procedencias estudiadas.
Análisis de las proteínas seminales
Los patrones polipeptídicos de proteínas provenientes de semillas individuales fueron obtenidos por electroforesis vertical en geles de poliacrilamida, en condiciones desnaturalizantes (SDSPAGE) y posterior tinción con Azul de Coomasie, siguiendo la técnica descripta por Espert & Burghardt (2003). Se determinó el perfil proteico de cada individuo, considerándose a semillas distintas como individuos genéticamente diferentes, con el objeto de analizar la variabilidad intrapoblacional y también la variación entre poblaciones. Las bandas fueron numeradas de acuerdo a su movilidad y la relación de frente fue calculada para cada una.
Análisis numérico
Las bandas presentes en todos los individuos fueron consideradas "constantes", asignándoles una frecuencia de 1. Para las bandas halladas sólo en algunos de los individuos de la población, se calculó la frecuencia de aparición. La variabilidad intrapoblacional fue analizada mediante el cálculo del índice polimórfico (IP) (Marshall & Allard, 1970), donde pi es la frecuencia de la iésima banda y n es el número de bandas para cada población. El IP varía de 0 a 0,25.

Sobre la base de la matriz de frecuencias de aparición de las fracciones proteicas en cada población se confeccionó una matriz de distancias Manhattan (Cain & Harrison, 1958). Ésta fue utilizada para el estudio de la variabilidad interpoblacional mediante un análisis de agrupamientos, siguiendo el método de medias no ponderadas (UPGMA) (Sneath & Sokal, 1973).
Con el mismo objetivo, a partir de la matriz básica de datos, se calcularon los coeficientes de correlación de Pearson (Michener & Sokal, 1957) entre poblaciones para realizar un análisis de componentes principales (Crisci & López Armengol, 1983).
Para todos los análisis llevados a cabo, fue utilizado el paquete de programas STATISTICA 5.1 (1998).
RESULTADOS
Los patrones polipeptídicos obtenidos por SDSPAGE están esquematizados en la Figura 2. Se encontró un total de 26 bandas que fueron numeradas según el orden de movilidad creciente. Los patrones característicos de cada población se construyeron considerando como presentes tanto a bandas constantes, como a aquéllas que presentaron variabilidad intrapoblacional. Las bandas 6 y 11 pueden considerarse características de la especie ya que fueron constantes en todas las poblaciones estudiadas. Las procedencias se diferenciaron por sus patrones de presencia-ausencia de bandas.

Fig. 2.- Representación esquemática de los patrones polipeptídicos característicos de cada población de P. ferox analizada. Se indica el número de las bandas y la relación de frente (Rf).![]() bandas con frecuencia 1,
bandas con frecuencia 1, ![]() bandas con frecuencias menores a 1,
bandas con frecuencias menores a 1, ![]() bandas que aparecen como rastros. Abreviaturas: C= Cerro Negro; PB= Peñas Blancas; MO= Mocote; OV= Ovara; R40= Encrucijada; HU= Humahuaca; JU= Juella; SA= Sapagua; YA= Yavi; CH= Chorrillos.
bandas que aparecen como rastros. Abreviaturas: C= Cerro Negro; PB= Peñas Blancas; MO= Mocote; OV= Ovara; R40= Encrucijada; HU= Humahuaca; JU= Juella; SA= Sapagua; YA= Yavi; CH= Chorrillos.
La Tabla 1 muestra la frecuencia de cada banda en las distintas procedencias. Se observó variabilidad intrapoblacional en algunas de ellas, siendo otras genéticamente homogéneas.
Tabla 1.- Matriz de frecuencias observadas de cada una de las bandas en las procedencias de P. ferox analizadas (se indica la altura sobre el nivel del mar de las distintas poblaciones).

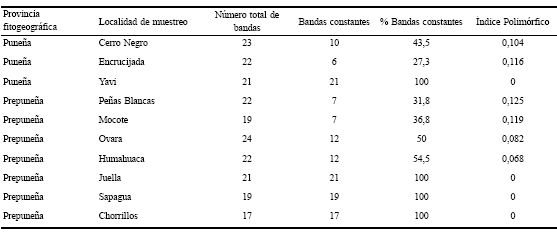
En la Tabla 2 se indica el índice polimórfico para cada procedencia. Es posible observar que las poblaciones prepuneñas de Juella, Sapagua y Chorrillos, así como la puneña de Yavi, no presentan variabilidad, mientras que en el resto de las localidades se encontraron bandas variantes y constantes en distintas proporciones.
Tabla 2.- Variabilidad dentro de cada procedencia de Prosopis ferox.
El ordenamiento de las procedencias sobre un plano delimitado por los dos primeros componentes resultantes del análisis de componentes principales se representa en la Figura 3. En ella se puede observar que no existe agrupación diferencial de procedencias puneñas y prepuneñas.

Fig. 3.- Distribución de las poblaciones analizadas con respecto a los dos primeros componentes principales. Los círculos negros corresponden a procedencias de la región prepuneña y los blancos a la región puneña.
La Figura 4 muestra el fenograma obtenido mediante el análisis de agrupamiento (UPGMA). En el fenograma se observan dos grupos. En ambos existe asociación de poblaciones puneñas y prepuneñas.

Fig. 4.- Fenograma obtenido a partir de una matriz de distancias Manhattan, por el método de medias aritméticas no ponderadas (UPGMA). Abreviaturas: P, región puneña; PP, región prepuneña.
DISCUSIÓN
Los resultados aquí obtenidos demuestran la existencia de diferencias genéticas entre las distintas procedencias analizadas. La variabilidad encontrada entre ellas se evidencia tanto en la composición, como en el número y en la frecuencia de las bandas. Algunas poblaciones presentaron polimorfismo para ciertas fracciones polipeptídicas, mientras que otras fueron monomórficas para todas ellas. En cada uno de los grupos generados por el análisis de agrupamiento UPGMA se puede observar la relación entre localidades prepuneñas y puneñas, lo cual refuerza la hipótesis de que estas últimas habrían sido originadas debido al traslado de animales alimentados en la prepuna (Kiesling, 1989). En particular, el material de Yavi (en la puna, limitando con Bolivia) puede vincularse con el de Sapagua y otras localidades prepuneñas, confirmando las observaciones de Kiesling (1989). Sorprende la aparente afinidad de los materiales de Juella y Sapagua (ambas en la prepuneña Quebrada de Humahuaca) con 800 m de diferencia en altitud.
Además, los índices polimórficos en poblaciones de P. ferox son comparables con los obtenidos en P. ruscifolia por Burghardt & Palacios (1998). Aunque la variación genética hallada sobre la base de los patrones electroforéticos puede ser considerada insuficiente evidencia para la fundación de entidades taxonómicas infraespecíficas, señala al menos, una incipiente diferenciación racial y constituye un elemento de juicio que, sumado a la variación morfológica observada (Braun Wilke et al., 1999), indicaría la presencia de ecotipos en P. ferox.
AGRADECIMIENTOS
Los autores agradecen a la Universidad de Buenos Aires por los subsidios otorgados, con parte de los cuales fue financiada la presente investigación. Asimismo, desean expresar su gratitud a los revisores anónimos por sus valiosas sugerencias.
BIBLIOGRAFÍA
1. Braun Wilke, R. H. 1991. Principales especies forestales nativas de la puna y prepuna argentina, pp. 39-43, en Agroforestería: Sistemas agroforestales y silvopastoriles para las zonas montañosas del noroeste. Desarrollo Forestal Participativo de los Andes/ FAO. Buenos Aires. [ Links ]
2. Braun Wilke, R. H., Picchetti, L. P. E., Guzmán, G. F. & Massié, A. I. 1999. Estudios Genecológicos de Prosopis ferox Gris. Revista Agraria 1: 33-41. [ Links ]
3. Burghardt, A. D. & Palacios, R. A. 1998. Variabilidad intraespecífica en Prosopis ruscifolia Griseb. (Leguminosae). Physis. Secc C, 55: 49-57. [ Links ]
4. Cain, A. J. & Harrison, G. A. 1958. An analysis of the taxonomist's judgement of affinity. Proc. Zool. Soc. Lond. 131: 85-89. [ Links ]
5. Crisci, J. V. & López Armengol, M. F. 1983. Introducción a la Teoría y Práctica de la Taxonomía Numérica. Serie Biología. Monografía No 26. O.E.A. [ Links ]
6. Espert, S. M. & Burghardt, A. D. 2003. Electrophoretic analysis of seed proteins in Argentinean species of Phaseolinae (Fabaceae). Bol. Soc. Argent. Bot. 38: 311-317. [ Links ]
7. Kiesling, R. 1989. Vegetación del NOA. Conferencia. Actas XIV Reunión Argentina de Ecología, ASAE/ UNJu, San Salvador de Jujuy: 7. [ Links ]
8. Marshall, D. R. & Allard, R. W. 1970. Isozyme polymorphisms in natural populations of Avena fatua and A. barbata. Heredity 25: 373-382. [ Links ]
9. Michener, C. D. & Sokal, R. R. 1957. A quantitative approach to a problem in classification. Evolution 11: 130-136. [ Links ]
10. Przybylska, J. & Zimniak-Przybylska, Z. 1997. Electrophoretic seed albumin patterns in Vicia species of sectt. Hypechusa and Peregrinae (Fabaceae). Plant Syst. Evol. 208: 3-4. [ Links ]
11. Sneath, P. H. A. & Sokal, R. R. 1973. Numerical Taxonomy: The Principles and Practice of Numerical Classification. Ed. Freeman. San Francisco. [ Links ]
12. StatSoft, Inc. 1998. STATISTICA 5.1 for Windows [Computer program manual]. Tulsa, Oklahoma. [ Links ]
13. Thome, J., Toro, O., Vargas, J. & Debouck, D. G. 1995. Variability in Andean Nuña Common Beans (Phaseolus vulgaris, Fabaceae). Econ. Bot. 49: 78-95. [ Links ]














