INTRODUCCIÓN
El género Paspalum L. es muy diverso y está compuesto por aproximadamente 330 especies que están distribuidas principalmente en América (Zuloaga et al., 2014). La importancia económica de este género se debe principalmente a la producción de forraje de algunas especies, como Paspalum notatum Flüggé, P. dilatatum Poir., P. atratum Swallen, o de césped, como P. vaginatum Sw., las cuales han sido introducidas en regiones subtropicales de todo el mundo (Zuloaga & Morrone, 2005; Marcón et al., 2018).
Las especies de Paspalum se distribuyen desde Norteamérica hasta los campos de la pampa en Argentina (Chase, 1929; Nicora & Rúgolo de Agrasar, 1987). El centro de diversidad del género se halla en la región tropical de América, y es particularmente abundante en Brasil (Chase, 1929; Pohl, 1980), en donde se han descripto aproximadamente 220 taxones (Valls, 1987). En Paraguay se han descripto 80 especies y 3 variedades (Zuloaga et al., 2014). En Argentina viven 77 especies (Morrone & Zuloaga, 2012) y la Mesopotamia es una de las regiones más biodiversas. Existe una gran representación de las especies de Paspalum en la biodiversidad de los ecosistemas en Sudamérica, los cuales están constantemente amenazados por la expansión de la agricultura, forestación y el desarrollo urbanístico. Una comprensión de la evolución y taxonomía íntegra del género ayudaría a comprender la riqueza en estos ecosistemas, y a diseñar mejores estrategias para conservar la biodiversidad y a su vez facilitar la domesticación de las especies con potencial forrajero (Honfi et al., 2021).
Las especies de Paspalum poseen citotipos diploides y poliploides, siendo el número básico más frecuentemente encontrado x=10 (Honfi et al., 1990; Morrone et al., 2006; Hojsgaard et al., 2009; Honfi et al., 2021). Entre las especies de Paspalum con datos citogenéticos, la poliploidía tiene una incidencia del 75,6 % (Honfi et al., 2021). A nivel intraespecífico, la presencia de 2 citotipos es una situación común, y la existencia de diploides de reproducción sexual y contrapartes conespecíficas tetraploides apomícticas facultativas, constituye una combinación de sistemas genéticos presente en numerosas especies (Ortiz et al., 2013). Existe un gran desconocimiento alrededor de la constitución cariotípica de las especies del género, y los pocos estudios que existen son incipientes o fragmentarios y se utilizan en apreciaciones de valor taxonómico y evolutivo (Speranza et al., 2003; Vaio et al., 2005; Reutemann et al., 2017). Los cromosomas de las especies de Paspalum tienen la ventaja que normalmente, en metafase mitótica, presentan una condensación homogénea importante para estudios comparativos. Sin embargo, solo unos pocos cariogramas han sido reportados para especies de Paspalum, usualmente sin una descripción de la constitución del complemento cromosómico o de la morfometría de los cromosomas. Como ejemplo, podemos mencionar los cariogramas de P. notatum tetraploide (Powell, 1968) y de P. almum Chase (el citotipo diploide, P. hexastachyum Parodi, Quarin, 1974, y el tetraploide, Quarin & Hanna, 1980).
Paspalum cromyorrhizon Trin. ex Döll pertenece al grupo informal Notata del género Paspalum. Este grupo está caracterizado por la presencia de 2 (-3) racimos por inflorescencia y espiguillas solitarias normalmente glabras, dispuestas en dos series a lo largo del raquis de la inflorescencia (Zuloaga et al., 2004). En estudios filogenéticos basados en marcadores plastidiales y marcadores morfológicos, P. cromyorrhizon se encuentra dentro del clado “NBTP” (Notata- Bertoniana-Thrasyopsis-Plicatula) y ha sido relacionada con P. equitans Mez, una especie alternativamente excluida (Zuloaga et al., 2004) o incluida (Souza-Chies et al., 2006) dentro del grupo Notata (Rua et al., 2010). Morfológicamente, P. cromyorrhizon se caracteriza por formar matas cespitosas con rizomas cortos, láminas glabras con márgenes membranosos y ciliados hacia el ápice, inflorescencias terminales, con 2 racimos conjugados terminados en una espiguilla bien desarrollada, espiguillas ovoides, plano-convexas, con gluma superior de márgenes alados, terminando en dos dientes en la región subapical, formando una especie de corona, de extensión variable (Zuloaga et al., 2004). Este taxón representa un endemismo regional distribuido por el sur de Brasil, centro-oeste de Uruguay y nordeste de Argentina, donde habita generalmente los campos bajos de las provincias de Corrientes y Entre Ríos (Zuloaga et al., 2004; Reutemann, 2020). Esta especie es un recurso forrajero de calidad, muy apetecido por el ganado en los pastizales del subtrópico.
El número básico de esta especie es x = 10 y se corresponde con el número haploide n = 10 (Reutemann, 2020). A la fecha, se conocen solo citotipos diploides (2n = 2x = 20) y tetraploides (2n = 4x = 40) en sus poblaciones naturales (Fernandes-Moraes et al., 1974; Quarin, 1977, Quarin et al., 1982; 1984; Quarin & Norrmann, 1987; Honfi et al., 1990; Reutemann, 2020). El citotipo diploide se reproduce sexualmente, aunque el desarrollo de sacos embrionarios apospóricos adicionales indicaría la potencialidad para la reproducción apomíctica (Quarin et al., 1982; Reutemann, 2020). Si bien el citotipo tetraploide se reproduce por apomixis apospórica pseudógama (Bashaw et al., 1970; Burson & Bennet, 1971; Reutemann, 2020), se han reportado algunos tetraploides capaces de generar progenie vía sexualidad (Quarin et al., 1982; Reutemann, 2020). Los dos citotipos se diferencian morfológicamente en los caracteres vegetativos, presentando las plantas tetraploides un mayor porte (Quarin et al., 1984, Reutemann, 2020). A través de cruzamientos con otras especies del grupo Notata, se ha determinado que esta especie posee un genoma NN modificado, y que los tetraploides son posiblemente autopoliploides (Quarin et al., 1984; Quarin & Norrmann, 1987).
Las descripciones clásicas de cariotipos, basados en la medición morfométrica individual de cromosomas y su posterior ordenamiento con base en la posición del centrómero y la longitud de cromosomas, han sido de gran utilidad comparativa en numerosos géneros de plantas (Solis Neffa & Fernández, 2002; Acosta et al., 2005; Honfi et al., 2017). Sin embargo, estudios similares no han sido aplicados comúnmente en especies de Paspalum. De acuerdo con los antecedentes presentados, los objetivos de este trabajo fueron analizar la composición y simetría del cariotipo de los citotipos diploide y tetraploide de P. cromyorrhizon y obtener información que soporte el origen de los poliploides y que brinde una caracterización genética básica preliminar para programas de mejoramiento de esta especie de interés forrajero de los campos naturales del subtrópico de Sudamérica.
MATERIAL Y MÉTODOS
Material Vegetal
Para la determinación de la constitución de los cariotipos, se utilizaron 19 individuos diploides y 39 tetraploides provenientes de cuatro poblaciones naturales de Corrientes, Argentina (Fig. 1).

Fig. 1 Poblaciones naturales de Paspalum cromyorrhizon estudiadas (Corrientes, Argentina). A, población H1733. B, detalle de las matas de la población H1733. C, población H1732. D, población H1735. Nótese en C y D los campos uniformes y pastoreados. Figura en color en la versión en línea http://www.ojs.darwin.edu.ar/index.php/darwiniana/article/view/987/1237
Las poblaciones analizadas fueron: Honfi 1732, Dto. Curuzú Cuatiá, colectada entre la Ruta Nacional (RN) 123 y Río Miriñay (18-II-2015; 29,55815º S 57,50227º O); Honfi 1733, Dto. Paso de los Libres, colectada sobre la Ruta Provincial 126, entre Curuzú Cuatiá y Paso de los Libres, en márgenes del Río Miriñay (19-II-2015; 29,84539º S 57,66850º O); Honfi 1735 Ruta provincial 23, entre Sauce y Perugorría (20-II-2015; 29,95637º S 58,81866º O) y Honfi 1955 Dto. Monte Caseros, colectada entre la RN 14 y el arroyo Mocoretá (18- IV-2015; 30,62889º S 57,98386º O). Los ejemplares de herbario correspondientes a cada accesión están depositados en el Herbario de la Universidad Nacional de Misiones (MNES). Los individuos diploides analizados provienen de la población Honfi 1732, mientras que los tetraploides provienen de las 4 poblaciones mencionadas.
Mitosis
Se realizaron preparaciones de cromosomas mitóticos a partir de ápices radiculares previamente tratados con solución saturada de α-bromonaftaleno por 3 h, fijados en etanol absoluto: ácido acético glacial en proporción 3:1, y conservados en etanol al 70% a 4 ºC hasta su utilización. En el momento de los análisis, en los ápices radiculares se llevó a cabo la tinción convencional de Feulgen y para ello, se hidrolizaron las raicillas en ácido clorhídrico 1N por 10 min a 60 ºC y luego se colorearon con fucsina básica (reactivo de Schiff) en cámara oscura. Los ápices radiculares se maceraron con orceína acética al 2% en un portaobjetos, se les aplicó un cubreobjetos y se analizaron al microscopio óptico Leica DM LS (Leica, Wetzlar, Alemania). Algunos preparados se hicieron permanentes por el método de congelación con nitrógeno líquido y posterior montaje en Euparal o Depex. Durante el análisis de ploidía se tomaron fotomicrografías de metafases selectas usando una cámara Leica MPS 30 (Leica, Wetzlar, Alemania) adosada al microscopio óptico Leica DM LS.
Cariotipos
A partir de un mínimo de 10 metafases óptimas (i.e. aquellas con los cromosomas separados y condensados) se realizó un análisis morfométrico de los cromosomas, de acuerdo a la nomenclatura propuesta por Levan et al. (1964). Los respectivos idiogramas se confeccionaron a partir de las fotomicrografías tomadas con un microscopio Leica DM LS con cámara Leica MPS 30 y se basaron en los valores medios calculados para cada parámetro cromosómico medido a través del programa MicroMeasure 3.3 (Reeves, 2001). Las mediciones y cálculos de los parámetros morfométricos de los cromosomas que se consideraron son los siguientes: longitud brazo corto (s) y largo (l), longitud total de cada cromosoma (c), donde c = s + l, longitud total del complemento cromosómico (LTC), el índice centromérico (i), donde i=100*s/c, relación de brazos (r), donde r = l / s. En cada citotipo, los cromosomas se ordenaron de mayor a menor según la longitud total y de acuerdo a los cuatro tipos cromosómicos definidos por Levan et al. (1964).
Para analizar el agrupamiento de los cromosomas homólogos en el idiograma del cariotipo, se consideró como base el trabajo de Solís Neffa & Fernández (2002) sobre cariotipos de un complejo autopoliploide, donde se espera que los cromosomas de los poliploides reflejen un múltiplo del cariotipo de los diploides. La racionalidad empleada en este trabajo, considera que como en el citotipo diploide de P. cromyorrhizon (2n = 2x = 20) los cromosomas se aparean formando bivalentes en la meiosis (Quarin et al., 1982), para analizar el cariotipo y confeccionar su idiograma, los cromosomas fueron tomados como pares homólogos.
En los tetraploides de P. cromyorrhizon (2n = 4x = 40), los cromosomas se asocian formando bivalentes, cuadrivalentes y octovalentes durante la meiosis, sugiriendo evidencias a favor de un número básico ancestral de x = 5 para Paspalum (Quarin et al., 1982) y por tal razón, se analizó el cariotipo considerando 3 posibles agrupamientos de cromosomas homólogos, i) como cuartetos (grupo homólogo de 4 cromosomas) asumiendo un número básico x = 10 para Paspalum; alternativamente, ii) como octetos (grupos de 8 cromosomas con cierta homología ancestral o genoma básico ancestral) presumiendo x = 5 como posible número básico ancestral del género y iii) como pares homólogos. Se calcularon los coeficientes de variación (CV) de la longitud cromosómica media (c), agrupando los cromosomas del citotipo tetraploide en pares homólogos y alternativamente en grupos de cuatro u ocho cromosomas homólogos (Solís Neffa & Fernández, 2002), para detectar evidencias a favor del origen autopoliploide y posibles polimorfismos.
Se realizó una prueba de igualdad de medias (prueba t para diferencias de medias en muestras independientes) entre el LTC esperado para los tetraploides (LTC de los diploides por duplicado) y el valor observado (LTC de los tetraploides) para testear el origen autopoliploide. Los idiogramas representativos del cariotipo se confeccionaron utilizando el programa gráfico AutoCAD 2006.
Asimetría del cariotipo
La asimetría cariotípica se estimó mediante las categorías de asimetría de Stebbins (1971) y los índices A1 y A2 de Romero Zarco (1986). Un cariotipo simétrico se caracteriza por la predominancia de cromosomas metacéntricos (m) o submetacéntricos (sm) del mismo tamaño (Paszko, 2006). La asimetría puede presentarse por dos motivos: variación en la posición del centrómero entre una localización medial / submedial a subterminal / terminal; o por variación en el tamaño de los cromosomas dentro del cariotipo (Stebbins, 1971; Paszko, 2006). El método de Stebbins (1971) y los índices A1 y A2 de Romero Zarco (1986) tienen en cuenta la heterogeneidad producida por ambos tipos de variaciones en el cariotipo. Para estimar la asimetría de los cariotipos según la categoría de Stebbins (1971), se consideró el valor de la relación entre los cromosomas de mayor y menor tamaño y la proporción de cromosomas con r > a 2:1. El sistema de clasificación consiste en establecer una de 12 categorías posibles para cada especie (1A-4A, 1B-4B, 1C-4C; Stebbins, 1971).
El índice A1 se utilizó para medir la asimetría intra-cromosómica, aplicando la siguiente fórmula: A1= 1- [(Ʃsi/li)/n], siendo si y li las longitudes promedio de los brazos cortos y largos de cada par de cromosomas homólogos, respectivamente, y n el número de pares de cromosomas homólogos. El índice A1 varía de 0 a 1 y se acerca a 0 cuando los cromosomas tienden a ser metacéntricos (Romero Zarco, 1986). El índice A2 se estimó usando la relación entre el desvío estándar y la media del largo de los cromosomas de la muestra, siendo A2 = S/, en donde S corresponde al desvío estándar y a la media de la longitud de todos los cromosomas del complemento haploide (Romero Zarco, 1986).
RESULTADOS
A los fines de testear el posible origen del citotipo poliploide, el cariotipo de P. cromyorrhizon se describe separadamente para cada nivel de ploidía. En cada caso, no se observó variación en la constitución de la fórmula del cariotipo entre individuos de un mismo citotipo ni entre individuos provenientes de distintas poblaciones, i.e. los diploides presentaron 20 cromosomas metacéntricos (20 m) y los tetraploides presentaron 40 cromosomas metacéntricos (40 m).
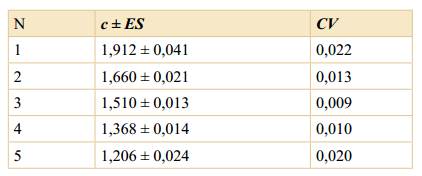
El resumen de los parámetros morfométricos del citotipo diploide se presentan en la Tabla 1.
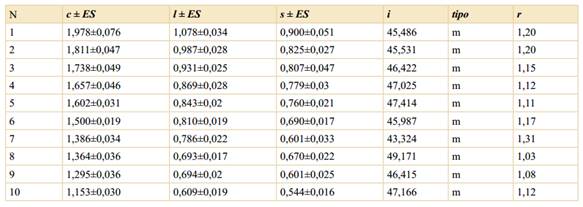
Tabla 1 Promedio de los parámetros morfométricos del set cromosómico haploide del citotipo 2n = 2x = 20 de P. cromyorrhizon. Abreviaturas: N, número del par cromosómico; c ± ES, largo total del cromosoma y su error estándar; l ± ES, longitud del brazo largo y su error estándar; s ± ES, longitud del brazo corto y su error estándar; i, índice centromérico; tipo, morfología cromosómica; m, metacéntrico; r, razón de brazos.
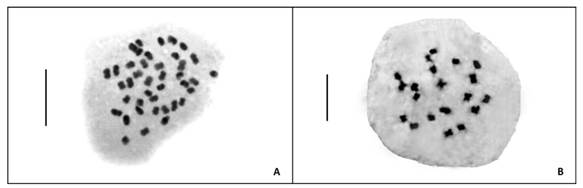
Fig. 2 Cromosomas mitóticos de P. cromyorrhizon. A, Metafase del citotipo tetraploide, 2n = 4x = 40. B, Metafase del citotipo diploide, 2n = 2x = 20. Escala: 10 µm.
Este citotipo presenta un cariotipo compuesto por 20 cromosomas metacéntricos (20 m; Fig. 2A) y un idiograma homogéneo (Fig. 3A) con una longitud promedio de los cromosomas de 1,55 µm, y un rango de variación entre 1,15 - 1,98 µm. La longitud total del complemento (LTC) diploide es de 30,96 µm. El índice centromérico (i) promedio es de 46,39. La relación entre la longitud del par cromosómico de mayor y menor tamaño dentro del complemento fue de 1,72. La ausencia de cromosomas con una relación de brazos mayor o igual a 2:1 ubica al cariotipo dentro de la categoría 1A de asimetría de Stebbins. Los índices de asimetría de Romero Zarco A1= 0,132 y A2= 0,164, indican también que se trata de un cariotipo simétrico.
Los parámetros morfométricos de cada cromosoma del citotipo tetraploide se presentan en la Tabla 2. El cariotipo tetraploide está compuesto por 40 cromosomas metacéntricos (40 m, Fig. 2B) y un idiograma poliploide homogéneo (Fig. 3B), con una longitud promedio de los cromosomas de 1,53 µm y una variación entre un valor mínimo de 1,08 µm y uno máximo de 2,1 µm. La LTC tetraploide es de 60,84 µm y el i promedio es de 46,71. La relación entre la longitud del par cromosómico mayor y menor del complemento tetraploide fue de 1,95.
Los individuos tetraploides evaluados, al igual que los diploides, tienen un cariotipo sin cromosomas con una relación de brazos mayor o igual a 2:1 y se ubican en la categoría 1A de Stebbins. Los índices de asimetría de Romero Zarco del citotipo tetraploide fueron similares a los observados en el cariotipo diploide, donde A1= 0,123 y A2= 0,166, indican que se trata de un cariotipo simétrico.

Fig. 3 Idiogramas de P. cromyorrhizon. A, Diploide (2n = 2x = 20). Se representa el set haploide (10m). B, Tetraploide (2n = 4x = 40). Se presenta el complemento completo. Escala: 1 µm. Figura en color en la versión en línea http://www.ojs.darwin.edu.ar/index.php/darwiniana/ article/view/987/1237
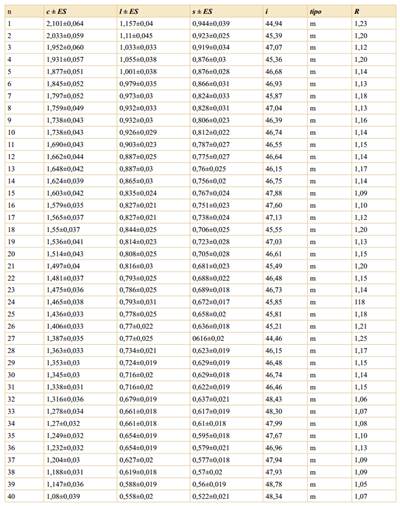
Tabla 2 Promedio de los parámetros morfométricos del set cromosómico del citotipo 2n = 4x = 40 de P. cromyorrhizon. Abreviaturas: N, número del par cromosómico; c ± ES, largo total del cromosoma y su error estándar; l ± ES, longitud del brazo largo y su error estándar; s ± ES, longitud del brazo corto y su error estándar; i, índice centromérico; tipo, morfología cromosómica; m, metacéntrico; r, razón de brazos.
En cuanto al origen del citotipo poliploide, los datos de LTC no muestran diferencias significativas entre el valor esperado de un diploide duplicado y el valor observado del tetraploide natural (t = 1,2142; g.l. = 32; p = 0,233). La longitud del complemento cromosómico del diploide duplicado esperado por autopoliploidización asciende a 30,96 µm x 2 = 61,92 µm, apenas diferente al valor estimado en el tetraploide. Comparativamente, la longitud cromosómica total por genoma monoploide fue de 15,48 µm en los diploides y 15,21 µm en tetraploides.
Teniendo en cuenta que todos los cromosomas son metacéntricos en ambos citotipos, para el análisis del cariotipo de los tetraploides, se realizó un agrupamiento de los cromosomas homólogos teniendo en cuenta solo su similitud en tamaño, en 3 situaciones distintas: a) cuartetos homólogos; b) octetos homólogos y c) pares homólogos. En la Tabla 3, se detallan los valores promedio de la longitud total del cromosoma, considerando la existencia de cuartetos homólogos en los tetraploides y el respectivo coeficiente de variación (CV) estimado para cada cuarteto. Los valores de CV estimados han sido bajos y oscilaron entre 0,005 - 0,024. Alternativamente, se calcularon los valores medios de longitud cromosómica y CV del agrupamiento de los cromosomas como genoma básico ancestral (octetos homólogos) y el rango de variación en la longitud dentro de cada octeto es aún menor (0,009 - 0,022; Tabla 4) al encontrado para un agrupamiento de cuatro cromosomas homólogos. Por otro lado, los valores medios de longitud cromosómica y el CV estimados reuniendo en pares homólogos a los cromosomas de los 4x, presentaron un rango de variación mayor (0,0 - 0,043; Tabla 5) a los observados en los otros agrupamientos (cuartetos y octetos).
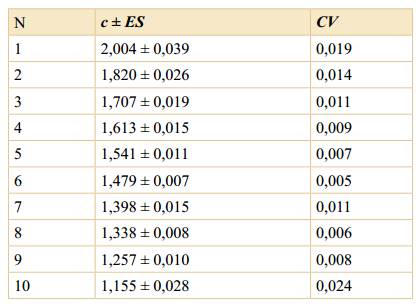
Tabla 3 Promedio de los parámetros morfométricos de los agrupamientos en cuartetos del citotipo 2n = 4x = 40 de P. cromyorrhizon. Abreviaturas: N, número del cuarteto cromosómico; c ± ES, largo total del cromosoma y su error estándar; CV, coeficiente de variación de cada cuarteto de cromosomas.
DISCUSIÓN
En el presente trabajo, describimos de modo clásico el cariotipo de los dos niveles de ploidía encontrados en P. cromyorrhizon. Todos los parámetros e índices cariomorfométricos calculados muestran que el cariotipo de las accesiones diploides y tetraploides de P. cromyorrhizon es unimodal y simétrico. No se observaron variaciones intra- e interpoblacionales en los parámetros morfométricos del cariotipo entre las diferentes accesiones tetraploides.
La caracterización de los cariotipos en Paspalum es particularmente desafiante. Sus especies presentan cromosomas extremadamente pequeños (~1 - 2 μm) y morfologías uniformes que dificultan su análisis. Esta es probablemente la principal razón por la que, tradicionalmente, los estudios citológicos solo han informado los niveles de ploidía y/o el comportamiento meiótico de los cromosomas en muchas de las especies de Paspalum, pero han dejado de lado los análisis del cariotipo.
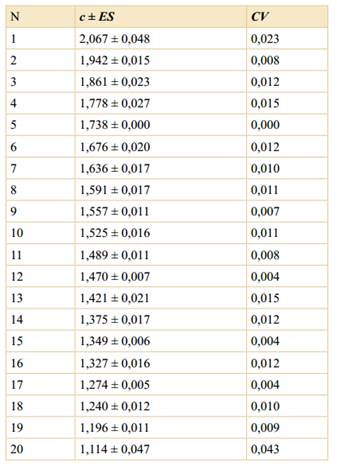
Tabla 5 Promedio de los parámetros morfométricos del agrupamiento a pares de los cromosomas del citotipo 2n = 4x = 40 de P. cromyorrhizon. Abreviaturas: N, número del octeto cromosómico; c ± ES, largo total del cromosoma y su error estándar; CV, coeficiente de variación de cada octeto de cromosomas.
Para entender estas dificultades metodológicas, basta mencionar que, de las aproximadamente 330 especies de Paspalum, solo el 52,85% posee un registro cromosómico, de las cuales el 75,6% es poliploide (Honfi et al., 2021). Por estas mismas razones los resultados presentados aquí resultan pioneros con respecto al análisis morfométrico pormenorizado de los atributos del cariotipo en el género Paspalum, e incluso también sobre autopoliploides en general porque todavía sabemos muy poco sobre las consecuencias genéticas y genómicas de la autopoliploidía (Soltis et al., 2016).
La morfología de los cromosomas en metafase mitótica se ha utilizado a menudo como criterio para distinguir entre autopoliploides y alopoliploides (Stebbins, 1971; Solís Neffa & Fernández, 2002). En complejos poliploides, el cariotipo y el comportamiento meiótico permiten orientar la identificación del origen poliploide y en autopoliploides su cariotipo refleja en mayor dosis al cariotipo de los diploides conespecíficos (Stebbins, 1971; Solís Neffa & Fernández, 2002). El análisis de la morfología y tamaño de los cromosomas del cariotipo tetraploide de P. cromyorrhizon sugiere que el camino más parsimonioso para llegar al poliploide es a través de una duplicación cromosómica a nivel intraespecífico sin influencia de cromosomas interespecíficos. El cariotipo se muestra como aditivo de los cariotipos de sus contrapartes diploides. Esto confirma el origen autopoliploide propuesto previamente sobre la base de las cantidades variables de asociaciones multivalentes observadas durante el comportamiento cromosómico meiótico en metafase I (Quarin et al., 1984; Quarin & Norrmann, 1987). Adicionalmente, el análisis de la simetría cariotípica basada en numerosos individuos y poblaciones diferentes, muestra gran similitud entre los complementos cromosómicos diploide y tetraploide, tanto respecto de las categorías de Stebbins como en la asimetría intracromosómica e intercromosómica. Intraespecíficamente, existe poca variación en los tamaños cromosómicos de ambos citotipos, puesto que apenas difieren los valores del índice A2 de asimetría intercromosómica y además, el índice de asimetría intracromosómico medio (A1) tampoco muestra diferencias marcadas entre los brazos cromosómicos de ambos citotipos. La evidencia obtenida sugiere que los tetraploides se han originado recientemente y aún conservan los rasgos morfométricos de los diploides conespecíficos ancestrales.
El número básico de cromosomas más frecuentemente encontrado en Paspalum es x = 10, aunque se han registrado otros como x = 6, 9, 16 (Quarin 1974, Selva 1976; Honfi et al., 1990, 2021). La cariología de P. cromyorrhizon, particularmente el comportamiento de los cromosomas en meiosis, ha sido motivo para argumentar a favor de la hipótesis de x = 5 como número básico ancestral o primitivo para el género Paspalum (Quarin et al., 1982; 1984). Al estudiar el comportamiento meiótico de una planta individual diploide de P. cromyorrhizon, Quarin et al. (1982) encontraron inesperadamente la presencia de cuadrivalentes en baja frecuencia (0,01%) y asociación secundaria de bivalentes. Esta observación, sugiere la presencia de una translocación en heterocigosis en dicho material, o bien, la existencia de homología vestigial en un segmento de pequeño tamaño, debido a que la formación del cuadrivalente en el diploide es ocasional. En todas las plantas diploides estudiadas aquí se observó el mismo cariotipo de 20 m sin presencia de pares heteromórficos, razón que, aunque circunstancial, apoyaría la segunda explicación. Además, en la meiosis irregular de 3 plantas tetraploides de P. cromyorrhizon analizadas por estos autores (Quarin et al., 1982; 1984), se observaron principalmente bivalentes (10,56; rango 1-17) y en menor frecuencia multivalentes, como III (0,03; rango 0-2), IV (3,56; rango 1-8), VI (0,34; rango 0-2) y VIII (0,25; rango 0-2). Basados en estos datos, Quarin et al., (1982) proponen que tales asociaciones homólogas son evidencia a favor de x = 5. En los tetraploides estudiados aquí, es notable la estabilidad cariotípica entre individuos de poblaciones distantes geográficamente (entre 45,4 -174 km). Los cromosomas son pequeños, metacéntricos y con escasa diferenciación tanto en el agrupamiento en cuartetos como en octetos, y no existe una distinción clara en términos de morfometría del cariotipo a favor de la hipótesis de x = 5 propuesta para el género.
El proceso de autopoliploidización consiste en la multiplicación del genoma diploide y posteriormente al evento, continúan procesos de modificación del genoma en su conjunto, que en algunas especies se expresa como una disminución o aumento del tamaño genómico esperado por multiplicación simple del genoma diploide parental, cambios estructurales, entre otros (Parisod et al., 2010; Poggio et al., 2014; Soltis et al., 2016). La longitud total del complemento diploide y tetraploide de P. cromyorrhizon como una expresión morfométrica de autopoliploidía, muestra que el citotipo tetraploide conserva el valor esperado. Además, la longitud cromosómica media del complemento monoploide es similar en ambos niveles de ploidía.
La disminución del contenido de ADN por genoma básico (monoploide) en poliploides puede detectarse si se observa una disminución de la LTC por genoma básico, comoocurreporejemploen Santolinapectinata Lag. que muestra una disminución significativa en la longitud de los cromosomas (Rivero-Guerra, 2008). La reducción del tamaño genómico ha sido observada en varios autopoliploides naturales, por ejemplo Biscutella laevigata L. (König & Müllner, 2005) y poliploides de Hippeastrum (Poggio et al., 2014). También ocurre en autopoliploides sintéticos, por ejemplo en un autotetraploide de Paspalum notatum que exhibió una eliminación de secuencias de ADN del 9,5% en relación con su progenitor diploide (Martelotto et al., 2007). Inicialmente nuestra hipótesis de trabajo fue asumir que si el rango de variación de la longitud cromosómica media (CV) en la agrupación de a pares de los cromosomas del tetraploide resultara menor que el rango de CV de la agrupación de a cuatro cromosomas (cuartetos), el citotipo tetraploide sería considerado de origen alopoliploide. Contrariamente, los resultados obtenidos indican que los tetraploides son el producto de la duplicación del genoma diploide. Otras técnicas citológicas son necesarias para individualizar a los grupos homólogos ancestrales de cromosomas con certeza y la obtención de híbridos sintéticos podría brindar evidencias de mayor soporte.
CONCLUSIÓN
La descripción del cariotipo es una herramienta útil para analizar la evolución cromosómica en numerosas especies de plantas, inclusive en complejos poliploides. En especies con poca variación en la morfología de los cromosomas, como es el caso de P. cromyorrhizon, el estudio del cariotipo en citotipos intraespecíficos disímiles permite obtener datos útiles para determinar el origen de los citotipos poliploides. Nuestros datos brindan soporte a la hipótesis de diferenciación intraespecífica a través de la formación de autopoliploides que conservan el cariotipo monoploide ancestral. Los caracteres cariológicos presentados aquí, brindan información sobre el germoplasma regional de especies forrajeras y serán de utilidad en estudios filogenéticos, evolutivos y en planes de conservación y domesticación.












 uBio
uBio