Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Medicina (Buenos Aires)
versión impresa ISSN 0025-7680versión On-line ISSN 1669-9106
Medicina (B. Aires) v.67 n.3 Buenos Aires mayo/jun. 2007
Falso diagnóstico de tuberculosis por cultivo
Valeria Alonso1, Roxana Paul2, Lucía Barrera2, Viviana Ritacco3
1 Agencia de Promoción Científica, SeCyT, beca PICT 9978,
2 Servicio de Micobacterias, Instituto Nacional de Enfermedades Infecciosas INEI ANLIS Carlos G. Malbrán,
3 Consejo Nacional de Investigaciones Científicas y Técnicas (CONICET)
Dirección postal: Dra. Viviana Ritacco, Servicio de Micobacterias, INEI ANLIS Malbrán, Av. Vélez Sarsfield 563, 1281 Buenos Aires, Argentina Fax: (54-11) 4302-7635 e-mail: vritacco@anlis.gov.ar
Resumen
Las herramientas de genotipificación intra-especie de Mycobacterium tuberculosis desarrolladas durante los años 90 no sólo dieron un impulso notable a la epidemiología de la tuberculosis, también pusieron de manifiesto un fenómeno hasta entonces soslayado en los laboratorios de tuberculosis: la contaminación cruzada de muestras. Este error consiste en la transferencia accidental de bacilos de una muestra con alta carga bacilar a la o las procesadas subsecuentemente. La consiguiente aparición de falsos cultivos positivos puede inducir al diagnóstico erróneo de tuberculosis y la instauración de tratamientos prolongados con drogas potencialmente tóxicas. Esa secuencia de errores conduce al mal manejo de los pacientes involucrados, la distracción de los recursos del sistema de salud y la distorsión de los resultados de análisis epidemiológicos. Se detectó contaminación cruzada en todos los laboratorios donde fue investigada sistemáticamente, con tasas de alrededor del 3% de los cultivos positivos. La confirmación requiere confrontar resultados bacteriológicos, clínicos, epidemiológicos y de genotipificación. Realizamos aquí una revisión de la información nacional e internacional sobre el tema y describimos las medidas recomendadas para minimizar el riesgo, vigilar la ocurrencia y evitar las consecuencias clínicas de este error de laboratorio que vulnera la certeza de un cultivo positivo.
Palabras clave: Contaminación cruzada de laboratorio; Mycobacterium tuberculosis; Genotipificación
Abstract
False diagnosis of tuberculosis by culture. A remarkable input to the epidemiology of tuberculosis was not the only benefit of the molecular tools developed in the early nineties for Mycobacterium tuberculosis intra-species differentiation. These genotyping methods served also to unveil specimen cross-contamination, which was until then overlooked in laboratories culturing mycobacteria. This error consists in the accidental carry-over of bacilli from a specimen with high bacterial load to that, or those, processed subsequently. The ensuing detection of falsely positive cultures can result in a wrong diagnosis of tuberculosis and the initiation of a long-lasting treatment with potentially toxic drugs. This series of errors implies the mismanagement of patients, the distraction of public health system resources, and the distortion of epidemiological data. M. tuberculosis laboratory cross-contamination was detected wherever investigated systematically, with a median rate of 3% of all positive cultures. The confirmation of this error requires a critical appraisal of bacteriological, clinical, epidemiological and genotyping results. We present here a review of national and international information on laboratory cross-contamination and describe measures recommended for minimizing the risk, surveying the occurrence, and avoiding clinical consequences of this laboratory error that raises a question on the reliability of a positive culture.
Key words: Laboratory cross-contamination; Mycobacterium tuberculosis; Strain typing
Ningún método de última generación ha desplazado todavía al cultivo como prueba gold standard para confirmar tanto el diagnóstico etiológico de tuberculosis como la curación del paciente. El diagnóstico bacteriológico incluye una serie de complejos pasos que van desde la obtención de la muestra clínica hasta la identificación de Mycobacterium tuberculosis. Es probable que en alguno de estos pasos se produzca una contaminación cruzada por transferencia accidental de bacilos de una muestra altamente positiva a la o las procesadas subsecuentemente. Este error, que genera uno o más resultados falsos positivos en los laboratorios que cultivan micobacterias, la mayoría de las veces pasa inadvertido en la rutina de trabajo1, 2.
La frecuencia con que ocurre este tipo de error es mayor de lo sospechado pero no resulta fácil comparar los porcentajes comunicados por los distintos laboratorios. Algunos autores refieren el número de falsos positivos al total de muestras procesadas en el laboratorio, otros al total de cultivos positivos, y otros al total de casos de tuberculosis diagnosticados. En ocasiones, la contaminación se confirma cuando se investiga un episodio sospechado, en otras es develada casualmente en el transcurso de un estudio epidemiológico. Según lo publicado, entre 0.33% y 16% de los cultivos positivos son falsos cultivos positivos debidos a este error, pero en la mayor parte de los casos el porcentaje ronda el 3%1, 3-14.
Dadas las serias consecuencias de un diagnóstico erróneo de tuberculosis, presentamos aquí un análisis del tema recopilando información actualizada sobre causas de contaminación cruzada de laboratorio, herramientas para la identificación, criterios para la confirmación, posibles consecuencias y recomendaciones para la vigilancia en el laboratorio de micobacteriología.
Causas
El error puede producirse en cualquiera de los pasos que conforman el procesamiento bacteriológico de una muestra clínica, desde la manipulación de la muestra fresca hasta las pruebas de identificación15, 16. No siempre es posible detectar la causa de una contaminación porque suelen pasar semanas desde el evento que la origina hasta el desarrollo de los cultivos contaminados, y aún más tiempo hasta la confirmación del error. En ese lapso, las circunstancias que favorecieron la contaminación pudieron haber cambiado y los instrumentos/reactivos involucrados haber sido reemplazados2, 4.
Uno de los principales factores que contribuyen a la contaminación cruzada es la notable capacidad de M. tuberculosis de sobrevivir en condiciones adversas. Los bacilos pueden permanecer viables en equipos o soluciones de trabajo contaminadas durante períodos prolongados y también en material biológico sometido a fijación por calor o formol12, 17, 18. La contaminación de la muestra se puede producir aun antes de su llegada al laboratorio por causa de un instrumento. Por ejemplo, los bacilos de un paciente sometido a broncoscopia pueden sobrevivir a la esterilización del equipo y desarrollar en la o las muestras de lavado broncoalveolar tomadas a continuación. Lo que es aún más grave, un broncoscopio contaminado puede transmitir la infección de un paciente a otro10, 18.
La complejidad técnica del cultivo de las micobacterias favorece sutiles errores de manipulación que suelen pasar inadvertidos. En este sentido, el paso más vulnerable es la decontaminación de la muestra. Todos los especímenes que provienen de sitios no estériles deben ser decontaminados con el objeto de evitar el crecimiento de microorganismos ambientales o de la flora normal de la muestra. Este procedimiento involucra la adición de una solución fuertemente alcalina, la neutralización con una solución de pH ácido o neutro, la concentración de los bacilos mediante centrifugación y finalmente la resuspensión del sedimento. Las soluciones usadas en estos pasos pueden contaminarse y albergar bacilos viables durante períodos prolongados10, 18. El roce inadvertido de bordes de recipientes de soluciones o pipetas con material contaminado, la formación de aerosoles al dispensar un reactivo sobre la muestra o al descartar suspensiones de bacilos en recipientes comunes pueden generar transferencia de esta bacteria de una muestra a otra.
La probabilidad de estos accidentes es mayor cuando existe sobrecarga de trabajo y se procesan lotes de muchas muestras por jornada5, 6, 11, 15, 18, 20. Por otro lado, cuando se procesa un exiguo número de muestras por año también existe riesgo de contaminación por falta de práctica. La incorporación de nuevo personal al laboratorio, más aún si no tiene entrenamiento adecuado, puede también considerarse un factor de riesgo.
Se atribuye un riesgo mayor de falsos cultivos positivos al uso de medios líquidos. Estos medios muy enriquecidos ofrecen óptimas condiciones de crecimiento a bacilos transferidos, aun a partir de un inóculo muy pequeño y/o después de sufrir estrés en el transporte, en el ambiente del laboratorio o durante el procesamiento de la muestra. La manipulación de líquidos favorece la formación de aerosoles, lo que acrecienta el riesgo de contaminación cruzada. Además, las pipetas y, en particular, las micropipetas empleadas para dispensarlos pueden contaminarse y transferir bacilos21, 22, 23. Recientemente se ha propuesto el cultivo en medio líquido en placas de microtitulación. Si bien es lógico pensar que estos micrométodos podrían favorecer la transferencia entre pocillos contiguos, esto no ha sido demostrado hasta el momento13.
El sistema de detección radiométrica BACTEC 460TB acrecienta el riesgo de contaminación cruzada. Para la lectura del crecimiento este sistema toma una muestra de la atmósfera del frasco de cultivo utilizando una aguja que perfora uno tras otro los tapones de goma. Una sutil falla en el funcionamiento del incinerador de la aguja trae como consecuencia el traslado de bacilos viables de un frasco de cultivo positivo a los siguientes2, 4.
La inoculación por aguja como factor de riesgo está siendo eliminado gradualmente. En efecto, en la actualidad se encuentran disponibles en el mercado sistemas automatizados de detección no radiométrica que detectan el crecimiento mediante indicadores fluorométricos o colorimétricos de lectura visual (MGIT Mycobacteria Growth Indicator Tube) o instrumental (BACTEC MGIT, MBBacT). La mayoría de los países, incluso el nuestro, está substituyendo el sistema de cultivo radiométrico por estos sistemas que, al obviar el uso de la aguja, reducen considerablemente el riesgo de contaminación respecto al del BACTEC46021.
Los cultivos de cepas de laboratorio usados asiduamente como testigos positivos pueden contaminar equipos, soluciones o material de trabajo y agregarse como factor de riesgo de contaminación cruzada de laboratorio19.
Herramientas para la identificación
Para confirmar un episodio de contaminación cruzada de laboratorio es necesario evaluar en forma conjunta la información provista por la historia clínica de los pacientes involucrados (tanto anterior a la toma de la muestra como su evolución posterior), la investigación de foco y la genotipificación. Si alguna de ellas no está disponible el proceso de identificación se complica y el evento puede quedar sin resolver. Aun cuando por sí sola es insuficiente, la genotipificación es la única herramienta disponible en el laboratorio para confirmar un episodio de contaminación y tiene el mérito agregado de haber puesto el tema en evidencia a nivel global.
Diversos métodos de exploración genotípica identifican segmentos repetitivos hipervariables del cromosoma, lo que permite la diferenciación entre distintos individuos de una misma especie. El genoma de M. tuberculosis es rico en este tipo de secuencias, entre las que se encuentran las Insertion Sequence (IS), las grandes familias de genes con secuencias polimórficas ricas en citosina y guanina, Polymorphic G C Rich Sequence (PGRS) y varias secuencias cromosomales cortas como los Direct Repeats (DR) y los Variable Number Tandem Repeats (VNTRs)23, 24. En particular, las ISs se duplican, se trasladan y se sitúan en diferentes posiciones dentro del cromosoma. Como consecuencia de este proceso, la posición y el número de repeticiones varían entre cepa y cepa, generando de este modo perfiles genéticos diferentes que sirven para la tipificación intra-especie. La IS más utilizada como marcador genotípico de M. tuberculosis es IS6110 ya que está presente sólo en el cromosoma de este microorganismo y generalmente en alto número de copias, lo que le otorga un alto grado de discriminación y especificidad. Sin embargo, cuando la cepa investigada posee una o pocas copias de IS6110, este elemento es poco o nada polimórfico y es necesario recurrir a otros tipos de marcadores genéticos que generen mayor discriminación25, 26.
Restriction Fragment Length Polymorphism (RFLP) con IS6110
Aún hoy, el RFLP IS611027 es considerado el método "gold standard" para la genotipificación de M. tuberculosis. Este método produce un patrón específico de cada cepa en base al número y la posición relativa de IS6110 en el cromosoma. Emplea grandes cantidades de ADN cromosómico, lo cual implica semanas y hasta meses, dado que M. tuberculosis es un microorganismo de crecimiento lento. Además, el RFLP es una técnica compleja, requiere reactivos importados muy costosos, personal altamente entrenado y un sofisticado programa de computación para comparar los perfiles de bandas obtenidos en distintos experimentos y/o laboratorios28.
Métodos que utilizan Polimerase Chain Reaction (PCR)
Para obviar estos inconvenientes se han desarrollado técnicas basadas en la amplificación de segmentos de ADN mediante reacción en cadena de la polimerasa (PCR). Estos métodos pueden ser aplicados sobre mínimas cantidades de ADN, aun de baja calidad o parcialmente degradado, y suelen arrojar resultados en 24-48 horas2, 28, 29. Los más conocidos se describen a continuación.
Mediante amplificación e hibridación reversa, el spoligotyping30 revela la presencia o ausencia de 43 segmentos espaciadores variables interpuestos entre los segmentos repetidos de la región DR. Esta técnica permite identificar taxones dentro del complejo M. tuberculosis y es particularmente útil para estudios filogenéticos. No es recomendable como único método para la investigación de contaminación cruzada porque tiene bajo poder de discriminación y puede sobreestimarla31. Patrones distintos descartan contaminación pero patrones idénticos deben ser investigados por otros métodos de genotipificación.
El Double Repetitive Element (DRE-PCR)32está basado en la amplificación mediante PCR de las regiones ubicadas entre dos elementos repetitivos hipervariables, la secuencia IS6110 y el segmento PGRS. Esta técnica tiene resultados poco reproducibles entre diferentes experimentos, aun realizados en el mismo laboratorio26. Sin embargo, resulta útil si sólo se aspira a comparar en un experimento perfiles de bandas relevantes entre aislamientos sincrónicos. Como es un método de baja complejidad accesible en nuestro medio y se completa en sólo 8 horas, resulta útil como método tamiz, pero sus resultados deben ser confirmados2, 28.
El Mixed-Linker PCR33 y, más aún, su reciente versión simplificada, el Fast Ligation Mediated PCR (FLiP)34, son ingeniosos métodos que revelan polimorfismos de IS6110. Son muy prometedores porque producen rápidamente resultados que, al parecer, son equivalentes a los de RFLP pero no están suficientemente reproducidos ni validados29.
El análisis de Mycobacterial Interspersed Repetitive Units (MIRU)35 revela polimorfismos generados por el número de repeticiones de un panel de 12 a 25 VNTRs36, 37. Cuanto mayor es el tamaño del panel, mayor es su poder de discriminación, que nunca supera el del RFLP IS6110, a menos que los aislamientos tengan bajo número de copias de IS6110 22, 26. Estos métodos son prácticos sólo si se los aplica en una versión automatizada.
Recientemente se compararon los métodos disponibles hasta el momento para la genotipificación del complejo M. tuberculosis. RFLP IS6110, FLiP y MIRUs-VNTR resultaron los métodos más reproducibles y con mayor poder de discriminación29.
Criterios para la confirmación
La sospecha de contaminación cruzada generalmente comienza en el laboratorio cuando aparecen cultivos positivos con escaso desarrollo a partir de una o varias muestras con baciloscopia negativa que fueron procesadas dentro de la misma semana que una muestra con alta carga bacilar, por el mismo operador, en la misma cabina de bioseguridad, y/o empleando los mismos lotes de reactivos1, 11. La obtención de una sola o muy pocas colonias ha sido identificada como señal de alarma de contaminación4, 8, 38. Este criterio es difícil de aplicar en el laboratorio de un hospital pediátrico porque la tuberculosis infantil es frecuentemente paucibacilar y el cultivo suele ser negativo o presentar muy pocas colonias2. Otras señales de alarma son el aumento repentino en la frecuencia de cultivos positivos en el laboratorio y la presunción clínica fuerte o confirmación diagnóstica de otra etiología en el momento del informe del cultivo positivo19, 39.
Estas señales son sólo el comienzo de una serie de pasos para la confirmación de la contaminación. El primer paso es la genotipificación de las cepas involucradas. Si los perfiles genéticos son idénticos, el proceso de confirmación sigue su curso. Si se dispone de otro aislamiento del paciente cuyo cultivo se presume contaminado, y su perfil genético es diferente al del episodio en cuestión, se sustenta la sospecha de contaminación. El perfil de resistencia a drogas anti-tuberculosis también puede contribuir a investigar el evento. La coincidencia tanto en el perfil genético como en el de resistencia a drogas de los aislamientos bajo sospecha refuerza aún más la presunción de contaminación26, 39.
Una vez obtenidos todos los perfiles genéticos disponibles para esclarecer el presunto episodio de contaminación, es necesario recurrir a la información clínica de los pacientes involucrados. Se refuerza la sospecha cuando el cuadro clínico de los pacientes cuyas muestras se presumen contaminadas era inespecífico en el momento de la investigación diagnóstica o, como se ha dicho, cuando la evolución posterior no resultó compatible con tuberculosis y, por supuesto, cuando en el ínterin se confirmó otra etiología4.
Por último, es importante evaluar los perfiles genéticos a la luz de la información epidemiológica. En áreas endémicas, la presencia de cultivos con idéntico perfil genético entre las muestras procesadas en un período breve de tiempo puede no ser suficiente para confirmar la contaminación. Esto se debe a que, en una población con alta incidencia y transmisión activa de la enfermedad, la diversidad genética de M. tuberculosis suele ser baja. Además, el estudio de foco es crítico pues genotipos idénticos pueden evidenciar transmisión real entre contactos40, 41.
Por otra parte, cabe considerar que distintos aislamientos de un mismo paciente pueden presentar genotipos diferentes en los casos de re-infección por otra cepa diferente o de infección simultánea por más de una cepa1, 42-45. Por lo tanto, cada caso presuntamente involucrado en un evento de contaminación cruzada deberá ser evaluado individualmente a la luz del conjunto de evidencias disponibles, clínicas, bacteriológicas, genotípicas y epidemiológicas8, 16.
Consecuencias
Debido al extenso período de tiempo que transcurre entre la sospecha de un evento de contaminación cruzada de muestras hasta su confirmación, la consecuencia más común es el inicio de un tratamiento antibiótico innecesario4, 10, 16. En Holanda se demostró que 31% de los pacientes con falso diagnóstico de tuberculosis recibieron tratamiento y 12% fueron hospitalizados11. El tratamiento de la tuberculosis no es inocuo y puede ocasionar efectos adversos. Además, el diagnóstico erróneo retrasa o impide el tratamiento de la verdadera etiología, a veces con consecuencias graves para el paciente8, 15.
La contaminación cruzada también tiene consecuencias socio-económicas. En primer lugar, un paciente sin tuberculosis puede sufrir discriminación laboral y marginación social en ambientes donde la enfermedad es un estigma. Por otra parte, este error genera un desvío de recursos tanto humanos como económicos, si se tiene en cuenta el costo laboral de personal de laboratorio y de atención médica, de materiales consumidos por el laboratorio, y de antibióticos, en caso de ser utilizados11.
Por último, si pasan inadvertidos, los eventos de contaminación falsean los resultados de estudios epidemiológicos tanto convencionales como moleculares, sobreestimando las tasas de incidencia y de transmisión de tuberculosis10,11. Se han descrito extensos episodios de contaminación sometidos a genotipificación que habían sido interpretados erróneamente como brotes de tuberculosis39, 46. Algunos de esos pseudobrotes fueron ocasionados por cepas multirresistentes y los pacientes con falso diagnóstico fueron gratuitamente sometidos a tratamiento con drogas de segunda línea, que son de mayor costo, más difícil acceso y más efectos adversos que las que integran el tratamiento estándar5, 41.
Contaminación cruzada en la Argentina
En nuestro país, los primeros estudios sistemáticos de contaminación cruzada se realizaron en el marco de investigaciones epidemiológicas. En esos estudios, el paso previo al análisis de transmisión consistió en excluir la contaminación cruzada como causa de clustering, es decir, de agrupamiento de aislamientos con genotipos idénticos. Así es como durante la documentación de un extenso brote de tuberculosis multirresistente ocurrido en el Hospital Muñiz, la contaminación cruzada se investigó en los diez pares de aislamientos que cayeron bajo sospecha. En base a los registros clínicos y bacteriológicos, la posibilidad de contaminación se descartó en todos los casos menos en uno47.
Durante el transcurso de un estudio de población realizado en un hospital suburbano con bajo volumen de trabajo que empleaba medio sólido a base de huevo, la bacterióloga sospechó tres episodios de contaminación cruzada que fueron confirmados rápidamente por el trabajo conjunto con el equipo médico y de laboratorio (Tabla 1, Hospital I). Este es el único estudio de Argentina en el que se investigó la tasa de contaminación cruzada del período: 2.7% (5/183) de los cultivos analizados por RFLP resultaron ser falsos positivos9.
TABLA 1.- Episodios de contaminación cruzada Mycobacterium tuberculosis de laboratorio investigados en hospitales de Argentina entre 1996 y 2003
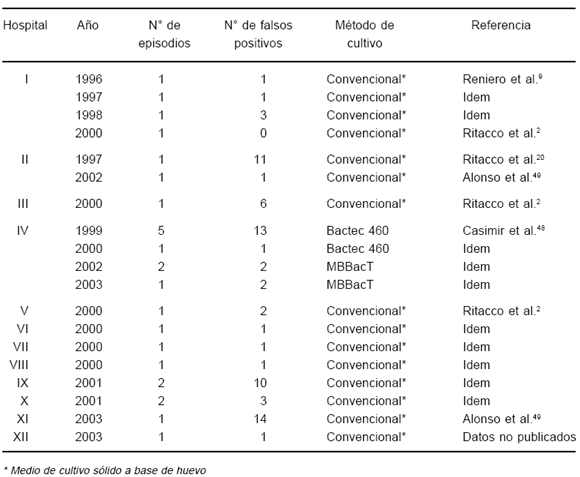
En el laboratorio de un hospital pediátrico que empleaba el sistema de cultivo BACTEC460 ocurrió el mayor número de episodios registrados en el país hasta el presente. La elevada frecuencia de contaminación cruzada se atribuyó al mal funcionamiento del incinerador de la aguja de ese sistema. El inconveniente desapareció transitoriamente al cambiar el sistema BACTEC 460 por el sistema MBBacT para el cultivo de micobacterias. Sin embargo, en cuanto se relajaron las rigurosas medidas que habían sido adoptadas para minimizar su ocurrencia se produjeron nuevos episodios, aunque en menor escala (Tabla 1, Hospital IV). A pesar del obstáculo agregado que representan los casos pediátricos paucibacilares para la identificación de contaminación cruzada, el alerta precoz de la bacterióloga permitió identificar precozmente todos los episodios sospechosos de contaminación y evitar así el tratamiento innecesario de los niños involucrados48.
En un laboratorio del Gran Buenos Aires con sobrecarga de trabajo se documentó un episodio de inusual duración (Tabla 1, Hospital II). En un lapso de 10 días consecutivos, todos los cultivos positivos eran falsos positivos y aparecieron intercalados entre otros cultivos que resultaron negativos. Como este episodio pasó inadvertido y fue un hallazgo retrospectivo en el transcurso de una investigación epidemiológica, no se pudo rastrear con certeza el origen de la contaminación. Se comprobó, sin embargo, que sólo las muestras que habían sido sometidas al procedimiento de Petroff produjeron falsos cultivos positivos. Por ese motivo, esos falsos positivos fueron probablemente consecuencia de la contaminación accidental con M. tuberculosis de alguna de las soluciones empleadas en dicho procedimiento durante el período en cuestión20.
En total, desde 1996 hasta la actualidad se han investigado en el laboratorio de referencia 26 episodios de contaminación cruzada producidos en 12 laboratorios de la Red Nacional de Tuberculosis. Con una sola excepción, los episodios ocurrieron en hospitales de la ciudad de Buenos Aires y sus alrededores, y de éstos, la vasta mayoría tenía una alta carga de trabajo. El único episodio confirmado en el interior del país se asoció a cambios de personal (Tabla 1, Hospital XII). En la mayoría de los casos, los grupos de aislamientos sospechosos de contaminación cruzada fueron enviados espontáneamente al laboratorio de referencia. En otros casos, la sospecha surgió en el mismo laboratorio de referencia ante la coincidencia entre perfiles de resistencia a drogas de aislamientos enviados simultáneamente por un laboratorio. Se solicitó información y/o cultivos adicionales y se llevó a cabo una investigación conjunta con el laboratorio en cuestión49. La mayoría de las muestras fueron analizadas empleando DRE-PCR32 como prueba tamiz interpretada con la flexibilidad descripta y RFLP IS611027 como método de confirmación. Ambas técnicas moleculares mostraron 100% de concordancia2.La sospecha de contaminación cruzada se confirmó en 24 de los 26 episodios investigados y en todos los laboratorios involucrados. En cada uno de los episodios con contaminación confirmada se identificó la muestra contaminante por ser la única muestra positiva al examen directo de esputo.
Recomendaciones
Medidas para minimizar el riesgo
A continuación se enumera una serie de medidas propuestas para prevenir este tipo de error de laboratorio; se encuentran entre las normas básicas para disminuir el riesgo y asegurar la calidad del trabajo en un laboratorio de bacteriología de la tuberculosis4, 7, 10, 23, 50.
Asegurar el correcto flujo de aire dentro de la cabina de seguridad biológica y en el laboratorio. Fraccionar los reactivos en alícuotas pequeñas, sobre todo los utilizados en el proceso de decontaminación. No procesar más de 12 muestras por sesión de trabajo, trabajando primero las muestras clínicas negativas a la baciloscopia y luego las positivas (algunos autores proponen asignar una cabina de bioseguridad a las muestras clínicas y otra a las operaciones con cultivos positivos, facilidad a la que, obviamente, muy pocos laboratorios pueden acceder). Establecer y respetar rigurosamente un protocolo de trabajo. Usar guardapolvos con puño. No abrir envases ni tubos conteniendo muestras antes de haber cerrado el anterior. Proteger el extremo superior de las pipetas con algodón. Utilizar pipetas individuales para cada muestra evitando mezclar las soluciones por pipeteo. No centrifugar en tubos de vidrio. Dejar reposar los tubos que han sido agitados o centrifugados antes de abrirlos. Descartar sobrenadantes con maniobras suaves en recipientes semi/cerrados, evitando la formación de aerosoles; una vez terminada la operación, decontaminar la superficie de trabajo y dejarla despejada para que la radiación UV alcance todos los sectores.
Medidas de vigilancia
Es importante adoptar mecanismos de vigilancia que permitan detectar estos casos tempranamente para evitar informar falsos resultados positivos. Algunas medidas simples para la vigilancia de la contaminación cruzada en el laboratorio de micobacterias consisten en4: procesar los cultivos y registrar los resultados en orden riguroso para poder rastrear retrospectivamente los procedimientos; vigilar la tendencia del número y la proporción de cultivos positivos; mantener una fluida comunicación con el equipo médico; ante una señal de alarma realizar un estudio retrospectivo de cada paciente teniendo en cuenta historia clínica, datos de laboratorio y datos epidemiológicos.
Señales de alarma
En el laboratorio, las señales son: la presencia de una o más muestras con examen directo de esputo negativo y cultivo positivo tardío con menos de 5 colonias y procesadas a continuación de otra con alta carga bacilar; el resultado positivo al cultivo de una sola de tres muestras de un paciente; el aumento súbito del número de cultivos positivos en el registro del laboratorio; la detección de idéntico perfil de resistencia en dos o más aislamientos de muestras procesadas el mismo día.
Las señales de alarma en la clínica son: la recepción de un informe positivo del laboratorio correspondiente a un paciente con presunción diagnóstica débil de tuberculosis, con evolución clínica no compatible con esta enfermedad y/o diagnóstico de otra etiología en el ínterin; la acumulación en un período de tiempo de informes de cultivo positivo de muestras tomadas con fibrobroncoscopio.
Se concluye que la contaminación cruzada no es un fenómeno inusitado, pues se la ha detectado en todos los laboratorios de micobacteriología en los que se realizó su vigilancia. Esto vulnera la certeza de un resultado positivo de cultivo. Un evento de contaminación cruzada provoca una secuencia de errores que implican un mal manejo clínico de los pacientes involucrados, una distracción de los recursos del sistema de salud y una distorsión en los resultados de los análisis epidemiológicos. Por ende, es muy importante aplicar medidas para minimizar su ocurrencia y, cuando aparece, identificarla a tiempo. El alerta y capacitación del personal del laboratorio son cruciales para sospechar precozmente este error. Su confirmación requiere una investigación integral llevada a cabo conjuntamente por el laboratorio y el equipo médico.
Agradecimientos: Trabajo realizado con apoyo de los proyectos PICT 9978 de SeCyT y PIP2373 de CONICET.
Bibliografía
1. Glynn JR, Yates MD, Crampin AC, et al. DNA fingerprint changes in tuberculosis: reinfection, evolution, or laboratory error? J Infect Dis 2004; 190: 1158-66.
2. Ritacco V, López B, Paul R, et al. Falsos cultivos positivos por contaminación cruzada en laboratorios de tuberculosis. Rev Argent Microbiol 2002; 34: 163-6.
3. Fischl MA, Uttamchanandi RB, Daikos GL, et al. An outbreak of tuberculosis caused by multiple-drug-resistant tubercle bacilli among patients with HIV infection. Ann Intern Med 1992; 117: 177-83.
4. Small PM, McClenny NB, Singh SP, Schoolnik GK, Tompkins LS, Mickelsen PA. Molecular strain typing of Mycobacterium tuberculosis to confirm cross-contamination in the mycobacteriology laboratory and modification of procedures to minimize occurrence of false-positive cultures. J Clin Microbiol 1993; 31: 1677-82.
5. Wurtz R, Demarais P, Trainor W, et al. Specimen contamination in mycobacteriology laboratory detected by pseudo-outbreak of multidrug-resistant tuberculosis: analysis by routine epidemiology and confirmation by molecular technique. J Clin Microbiol 1996; 34: 1017-9.
6. Burman WJ, Stone BL, Reves RR, et al. The incidence of false-positive cultures for Mycobacterium tuberculosis. Am J Respir Crit Care Med 1997; 155: 321-6.
7. Bhattacharya M, Dietrich S, Mosher L, et al. Crosscontamination of specimens with Mycobacterium tuberculosis: clinical significance, causes, and prevention. Am J Clin Pathol 1998; 109: 324-30.
8. Gutierrez MC, Vincent V, Aubert D, et al. Molecular fingerprinting of Mycobacterium tuberculosis and risk factors for tuberculosis transmission in Paris, France, and surrounding area. J Clin Microbiol 1998; 36: 486-92.
9. Reniero A, Moscatelli J, Kantor IN, Ritacco V. Early detection of Mycobacterium tuberculosis laboratory cross-contamination in a Buenos Aires hospital. Int J Tuberc Lung Dis 1999; 3 (Suppl. 1): 173.
10. Burman WJ, Reves RR. Review of false-positive cultures for Mycobacterium tuberculosis and recommendations for avoiding unnecessary treatment. Clin Infect Dis 2000; 31: 1390-5.
11. de Boer AS, Blommerde B, de Haas PE, et al. False-positive Mycobacterium tuberculosis cultures in 44 laboratories in The Netherlands (1993 to 2000): incidence, risk factors, and consequences. J Clin Microbiol 2002; 40: 4004-9.
12. Ruddy M, McHugh TD, Dale JW, et al. Estimation of the rate of unrecognized cross-contamination with Mycobacterium tuberculosis in London microbiology laboratories. J Clin Microbiol 2002; 40: 4100-4.
13. Moore DA, Caviedes L, Gilman RH, et al. Infrequent MODS TB culture cross-contamination in a high-burden resource-poor setting. Diagn Microbiol Infect Dis 2006; 56: 35-43.
14. Yan JJ, Jou R, Ko WC, Wu JJ, Yang ML, Chen HM. The use of variable-number tandem-repeat mycobacterial interspersed repetitive unit typing to identify laboratory cross-contamination with Mycobacterium tuberculosis. Diagn Microbiol Infect Dis 2005; 52: 21-8.
15. Bauer J, Thomsen VO, Poulsen S, Andersen AB. False-positive results from cultures of Mycobacterium tuberculosis due to laboratory cross-contamination confirmed by restriction fragment length polymorphism. J Clin Microbiol 1997; 35: 988-91.
16. van Duin JM, Pijnenburg JE, van Rijswoud CM, de Haas PE, Hendriks WD, van Soolingen. Investigation of cross contamination in a Mycobacterium tuberculosis laboratory using IS6110 DNA fingerprinting. Int J Tuberc Lung Dis 1998; 1: 425-9.
17. Kantor IN, Barcat JA., Ritacco V. Viability of mycobacterium in formalin fixed tissues. Int J Tuberc Lung Dis 1999; 3: 362.
18. Ramos MC, Soini H, Roscanni GC, et al. Extensive cross-contamination of specimens with Mycobacterium tuberculosis in a reference laboratory. J Clin Microbiol 1999; 37: 916-9.
19. Fitzpatrick L, Braden C, Cronin W, et al. Investigation of laboratory cross-contamination of Mycobacterium tuberculosis cultures. Clin Infect Dis 2004; 38: 52-54.
20. Ritacco V, Togneri A, López B, Moscatelli J, Reniero A, Barrera L. An unusually prolonged episode of mycobacterial cross-contamination in a diagnostic laboratory. Resumo, XX Congresso Brasileiro de Microbiologia. Salvador BA, Sociedade Brasileira de Microbiologia, Oct. 28-28, 1999, Salvador, Bahia, Brasil.
21. Tortoli E, Cichero P, Piersimoni C, Simonetti MT, Gesu G, Nista D. Use of BACTEC MGIT 960 for recovery of mycobacteria from clinical specimens: multicenter study. J Clin Microbiol 1999; 37: 3578-82.
22. Gascoyne-Binzi DM, Barlow RE, Frothingham R, et al. Rapid identification of laboratory contamination with Mycobacterium tuberculosis using variable number tandem repeat analysis. J Clin Microbiol 2001; 39: 69-74.
23. National TB Controllers Association. CDC Advisory Group on Tuberculosis Genotyping. Guide to the application of genotyping to tuberculosis prevention and control. Atlanta, GA: US Department of Health and Human Services, CDC; June 2004.
24. Cole ST, Brosch R, Parkhill J, et al. Deciphering the biology of Mycobacterium tuberculosis from the complete genome sequence. Nature 1998; 393: 537-44.
25. van Soolingen D, de Haas PEW, Hermans PWM, Groenen P, van Embden JDA. Comparison of various repetitive DNA elements as generic markers for strain differentiation and epidemiology of Mycobacterium tuberculosis. J Clin Microbiol 1993; 31: 1987-95.
26. Kremer K, van Soolingen D, Frothingham R, et al. Comparison of methods based on different molecular epidemiological markers for typing of Mycobacterium tuberculosis complex strains: interlaboratory study of discriminatory power and reproducibility. J Clin Microbiol 1999; 37: 2607-18.
27. van Embden JDA, Cave MD, Crawford JT, et al. Strain identification of Mycobacterium tuberculosis by DNA fingerprinting: recommendations for a standardized methodology. J Clin Microbiol 1993; 31: 406-9.
28. Varela G, Carvajales S, Gadea P, et al. Comparative molecular study of Mycobacterium tuberculosis strains, in times of antimicrobial drug resistance. Rev Argent Microbiol 2005; 37: 11-15.
29. Kremer K, Arnold C, Cataldi A, et al. Discriminatory power and reproducibility of novel DNA typing methods for Mycobacterium tuberculosis complex strains. J Clin Microbiol 2005; 43: 5628-38.
30. Kamerbeek J, Schouls L, Kolk A, et al. Simultaneous detection and strain differentiation of Mycobacterium tuberculosis for diagnosis and epidemiology. J Clin Microbiol 1997; 35: 907-14.
31. Gori A, Bandera A, Marchetti G, et al. Spoligotyping and Mycobacterium tuberculosis. Emerg Infect Dis 2005; 11: 1242-8.
32. Friedman CR, Stoekle MY, Jonhson Jr.WD, Riley LW. Double repetitive element PCR method for subtyping Mycobacterium tuberculosis clinical isolates. J Clin Microbiol 1995; 33: 1383-4.
33. Haas WH, Butler WR, Woodley CL, Crawford JT. Mixedlinker polymerase chain reaction: a new method for rapid fingerprinting of isolates of the Mycobacterium tuberculosis complex. J Clin Microbiol 1993; 31: 1293-8.
34. Reisig F, Kremer K, Amthor B, van Soolingen D, Haas WH. Fast ligation-mediated PCR, a fast and reliable method for IS6110-based typing of Mycobacterium tuberculosis complex. J Clin Microbiol 2005; 43: 5622-7.
35. Supply P, Lesjean S, Savine E, Kremer K, van Soolingen D, Locht C. Automated high-throughput genotyping for study of global epidemiology of Mycobacterium tuberculosis based on mycobacterial interspersed repetitive units. J Clin Microbiol 2001; 39: 3563-71.
36. Frothingham R, Meeker-O'Connell WA. Genetic diversity in the Mycobacterium tuberculosis complex based on variable numbers of tandem DNA repeats. Microbiology 1998; 144: 1189-96.
37. Gopaul KK, Brown TJ, Gibson AL, Yates MD, Drobniewski FA. Progression toward an improved DNA amplification-based typing technique in the study of Mycobacterium tuberculosis epidemiology. J Clin Microbiol 2006; 44: 2492-8.
38. Nivin B, Fujiwara PI, Hannifin J, Kreiswirth BN. Cross-contamination with Mycobacterium tuberculosis: an epidemiological and laboratory investigation. Infect Control Hosp Epidemiol 1998; 19: 500-3.
39. Cronin W, Rodriguez E, Valway S, et al. Pseudo-outbreak of tuberculosis in an acute-care general hospital: epidemiology and clinical implications. Infect Control Hosp Epidemiol 1998; 19: 345-7.
40. Carroll NM, Richardson M, van Helden PD. Criteria for identification of cross-contamination of cultures of Mycobacterium tuberculosis in routine microbiology laboratories. J Clin Microbiol 2003; 41: 2269.
41. Bearman G, Vaamonde C, Larone D, Drusin L, Zuccotti G. Pseudo-outbreak of multidrug-resistant Mycobacterium tuberculosis associated with presumed laboratory processing contamination. Infect Control Hosp Epidemiol 2002; 23: 620-2.
42. Braden CR, Morlock GP, Woodley CL, et al. Simultaneous infection with multiple strains of Mycobacterium tuberculosis. Clin Infect Dis 2001; 33: 42-7.
43. Das S, Narayanan S, Hari L, et al. Simultaneous infection with multiple strains of Mycobacterium tuberculosis identified by restriction fragment length polymorphism analysis. Int J Tuberc Lung Dis 2004; 8: 267-70.
44. van Rie A, Victor TC, Richardson M, et al. Reinfection and mixed infection cause changing Mycobacterium tuberculosis drug-resistance patterns. Am J Respir Crit Care Med 2005; 172: 636-42.
45. Warren RM, Victor TC, Streicher EM, et al. Patients with active tuberculosis often have different strains in the same sputum specimen. Am J Respir Crit Care Med 2004; 169: 610-4.
46. Segal-Maurer S, Kreiswirth BN, Burns JM, et al. Mycobacterium tuberculosis specimen contamination revisited: the role of laboratory environmental control in a pseudo-outbreak. Infect Control Hosp Epidemiol. 1998; 19: 101-5.
47. Ritacco V, Di Lonardo M, Reniero A, et al. Nosocomial spread of Human Immunodeficiency Virus-related multidrug-resistant tuberculosis in Buenos Aires. J Infect Dis 1997; 176: 637-42.
48. Casimir L, Lovadina L, Paul R, et al. Falsos diagnósticos de tuberculosis como resultado de contaminación cruzada de cultivos. Libro de Resúmenes, XVII Congreso Latinoamericano y X Argentino de Microbiología. Buenos Aires, Asociación Latinoamericana de Microbiología y Sociedad Argentina de Microbiología, Sept 25-27, 2004, Buenos Aires.
49. Alonso V, Paul R, López B, Barrera L, Ritacco V. Genotipificación de Mycobacterium tuberculosis para la vigilancia de contaminación cruzada en laboratorios de la Red Nacional de Tuberculosis. Libro de Resúmenes, II Congreso Argentino de Redes de Laboratorio, ANLIS Dr. C. G. Malbrán, sept 6-8, 2005, Buenos Aires.
50. Carroll NM, Richardson M, Engelke E, de Kock M, Lombard C, van Helden PD. Reduction of the rate of false-positive cultures of Mycobacterium tuberculosis in a laboratory with a high culture positivity rate. Clin Chem Lab Med 2002; 40: 888-92.
Recibido: 31-07-2006
Aceptado: 14-11-2006














