Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Medicina (Buenos Aires)
versión impresa ISSN 0025-7680
Medicina (B. Aires) vol.70 no.5 Ciudad Autónoma de Buenos Aires sep./oct. 2010
COMUNICACIÓN BREVE
Familias de la proteína de superficie PspA de Streptococcus pneumoniae. Relación con serotipos y localización
Clara Mayoral1, Mara Della Bianca1, María Rosa Baroni1, Rita Giani1, Mabel Regueira2, Fabián Zalazar1
1Laboratorio de Práctica Profesional, Facultad de Bioquímica y Ciencias Biológicas, Universidad Nacional del Litoral, Hospital Dr. J.M. Cullen, Santa Fe;
2Servicio Bacteriología Clínica, INEI-ANLIS. Dr. Carlos Malbrán, Buenos Aires, Argentina
Dirección postal: Dra. Clara Mayoral, Laboratorio de Práctica Profesional, Facultad de Bioquímica y Ciencias Biológicas, Universidad Nacional del Litoral, Hospital Dr. J.M. Cullen. Av. Freyre 2150, 3000 Santa Fe, Argentina Fax: (54-342) 4575221 e-mail: cmayoral@fbcb.unl.edu.ar
Resumen
PspA, proteína de superficie de Streptococcus pneumoniae es un factor de virulencia, fuertemente inmunogénica y común a todos los serotipos. Aunque el gen que codifica para esta proteína presenta una marcada heterogeneidad en la región correspondiente al N-terminal, la PspA contiene epitopes conservados de manera tal que la inmunización genera protección contra neumococos pertenecientes a diversos tipos capsulares y con distintas PspA. A pesar del marcado polimorfismo del gen pspA es posible agrupar las distintas variantes en 3 familias mayoritarias. Estas propiedades las convierten en candidatas ideales para elaborar vacunas. Debido a que la mayoría de los trabajos sobre identificación de familias fueron realizados sobre serotipos frecuentes en otros países, el objetivo fue identificar las familias de PspA de aislamientos de pacientes de nuestra región y relacionarlas con los serotipos prevalentes y patologías. Se estudiaron 70 aislamientos, provenientes de niños con infecciones invasoras. Se aplicó una PCR utilizando cebadores específicos de cada familia. El 60% fueron familia 1 y 34% familia 2. En un 6% no se identificó ninguna de las familias de PspA. Los serotipos 1 y 5 presentaron familia 1 únicamente; los serotipos 14, 6B, 19F y 18C mostraron genes de ambas familias. La familia 1 se observó en 60% de las neumonías y 50% de las meningitis. La familia 2 en 33% de neumonías y 50% de meningitis. Esta información podría ser un valioso aporte para la formulación de una vacuna regional efectiva utilizando PspA recombinante como inmunógeno.
Palabras clave: Streptococcus pneumoniae; Familias de PspA; Serotipos; Infecciones invasoras
Abstrac
Pneumococcal surface protein A (PspA) families. Relation with serotypes and clinical site of infection. PspA, a pneumococcal surface protein, is highly immunogenic and common to all serotypes. Although pspA gene shows a great heterogeneity at the N-terminal region, PspA protein has conserved epytopes which are able to elicit protective cross-reaction against various serotypes presenting different PspA. In spite of the high polimorfism of the PspA, three majority families can be identified. These properties convert PspA as ideal candidate for the formulation of a pneumococcal vaccine. Investigations of the PspA families were mostly carried out on prevalent serotypes in other countries. The aim of this study was to identify PspA families from Streptococcus pneumoniae isolates of our region as well as to associate them to prevalent serotypes or pathologies. We studied 70 isolates from pediatric patients with invasive infections. PCR was performed using specific primers for each family. In these studies we observed that 60% were PspA family 1, 34% were PspA family 2 and 6% remained unclassified. Serotypes 1 and 5 presented only family 1; serotypes 14, 6B, 19F y 18C showed genes from both families. Family 1 was observed respectively in 60 y 50% of pneumonias and meningitis. The family 2 was identified in 33 and 50% of pneumonias and meningitis. This information about the PspA family distribution could become a valuable contribution to develop an effective regional vaccine using recombinant PspA as immunogen.
Key words: Streptococcus pneumoniae; PspA families; Serotypes; Invasive infections
La alta morbi-mortalidad de las neumonías y meningitis causadas por Streptococcus pneumoniae (Spn) evidencian la necesidad del desarrollo de vacunas antineumocócicas eficaces para ser utilizadas como estrategia universal. Existen actualmente vacunas formuladas con polisacáridos unidos a proteínas no neumocócicas -las llamadas 7, 10 y 13 valente- que protegen contra infecciones invasoras. La primera1 contra aquéllas causadas por los serotipos 4, 6B, 9V, 14, 18C, 19F y 23F, con una eficacia del 56 al 93% según la región geográfica de la población en estudio2. Con la introducción de esta vacuna se observó, en numerosos países, una disminución significativa del número de infecciones invasoras neumocócicas causadas por los serotipos contenidos en la misma, entre niños menores de 2 años3. Sin embargo, el alto costo limita su uso, especialmente en los países donde el índice de morbi-mortalidad es muy alto.
En nuestro país los serotipos incluidos en la vacuna 7-valente representan el 61.2% de las cepas invasoras aisladas en menores de 2 años y 53.4% de las mismas en todos los niños, sin tener en cuenta el grupo etario4. Esta diferencia se fundamenta en la mayor frecuencia de los serotipos 1 y 5, no contenidos en esta vacuna, en los niños de 2 a 6 años.
Por otra parte, la vacuna 10-valente contiene, además, los serotipos 1, 5, y 7F; la 13-valente, además de todos los anteriores agrega, el 3, 6A y 19A, logrando una mayor protección. Actualmente la 10-valente está licenciada en la Argentina.
Además, como existen aproximadamente 30 serotipos patógenos y éstos varían sustancialmente con el lugar geográfico, las vacunas óptimas tendrían que tener diferentes formulaciones acordes a los serotipos prevalentes de cada región, de estar formuladas sobre la base de polisacáridos de la pared celular.
Desde hace varios años se están estudiando proteínas de superficie del Spn para la elaboración de vacunas1. Entre ellas, la llamada PspA tiene las ventajas de ser fuertemente inmunogénica en humanos5, inducir protección contra bacteriemia neumocócica y ser común a todos los serotipos6. Los anticuerpos anti-PspA mostraron protección cruzada para cepas de diferentes serotipos7, tanto en experiencias en ratones6 así como en ensayos clínicos en humanos.
Su estructura molecular presenta una región altamente variable6-8, lo que permite distinguir 3 familias de PspA (familias 1, 2 y 3). Hollingshead et al7 propusieron una clasificación para las PspA con base en las diferencias de las secuencias aminoacídicas localizadas inmediatamente antes de una región rica en prolina, designada clade-defining region.
La familia 1 está compuesta de clades 1 y 2. La familia 2, de los clades 3, 4, 5 y la familia 3 del clade 67. Estas familias son capaces de generar anticuerpos con protección cruzada entre distintos serotipos de la misma familia, lo que las convierte en candidatas ideales para una vacuna.
El desarrollo efectivo de una vacuna conteniendo antígenos de PspA requiere la información acerca de las familias presentes en los aislamientos neumocócicos de las regiones donde la vacuna será utilizada. Por otra parte, teniendo en cuenta la diversidad de PspA en algunas regiones de su estructura y a fin de minimizar el costo de una vacuna sería conveniente restringir el número de antígenos a los más relevantes.
En esta línea de trabajo, en un estudio anterior9 se determinaron los serotipos invasores prevalentes en nuestra zona mientras que en el presente trabajo intentamos identificar las familias de PspA contenidas en dichos aislamientos, con el fin de obtener información epidemiológica fehaciente para ser utilizada en el desarrollo de una vacuna efectiva para la región centro y litoral del país, conteniendo determinantes antigénicos de PspA.
Se analizaron 70 aislamientos de Spn, serotipificados con la tradicional técnica de Quellung, usando el esquema de Skov Sorensen, con antisueros producidos por Statens Seruminstitut (Copenhagen, Dinamarca); recolectados durante el período 2003 a 2006, provenientes de niños menores de 5 años de edad, internados en el Hospital Dr Orlando Alassia (Santa Fe, Argentina) por: a) neumonía (n = 57), b) meningitis (n = 10) y c) peritonitis espontánea (n = 3). Estos niños residían en la ciudad de Santa Fe y zona de influencia. Para el análisis estadístico de los resultados se calculó el riesgo mediante el Odds Ratio y la asociación entre las variables se estimó mediante Chi cuadrado.
La identificación de las familias de PspA se realizó inicialmente aplicando una técnica de reacción en cadena de la polimerasa (PCR) utilizando cebadores específicos de secuencia para familias 1 y 2, que son las más frecuentes según la literatura mundial. Con las muestras negativas para familias 1 y 2, se realizó una PCR con cebadores comunes a las 3 familias.
Para la extracción de ADN bacteriano fueron recolectadas entre 5 y 10 colonias a partir de cultivos de Spn de 18 h, resuspendidas en 100 μl de HCl -Tris buffer 0.1M, pH 8.0 y sometidas a ebullición durante 10 min. Luego de centrifugar 2 min a 10 000 g para eliminar restos celulares, se recuperó el sobrenadante conteniendo el ADN y se conservó a -20 °C hasta su análisis.
Las secuencias de oligonucleótidos utilizadas son las siguientes:
Familia 1: LSM12 (5´CCGGATCCAGCGTCGCTATCTTAGGGGCTGGTT3´)8 y SKH63 (5´TTTCTGGCTCATC/TAACTGCTTTC3´)14.
Familia 2: LSM12 y SKH52 (5´TGGGGGTGGAGTTTCTTCTTCATCT3´)14
Familias 1-3: LSM12 y SKH2 (5![]() -CCACATACCGTTTTCTTGTTTCCAGCC-3
-CCACATACCGTTTTCTTGTTTCCAGCC-3![]() )7
)7
La reacción de amplificación contuvo 10 mM Tris-HCl pH 9.0, 50 mM KCl, 0.1% Tritón X-100, MgCl2 2.5 mM, dNTP s 200 μM c/u, 50 pmol de cada primer, 0.8 U de Go Taq ADN polimerasa y 1μl de ADN en estudio en un volumen final de 25 μl.
En todas las reacciones se procesaron también las siguientes muestras controles:
Controles positivos: preparaciones de ADN provenientes de las cepas de Spn ATCC R36A y BG11703, correspondientes a las familias 1 y 2 respectivamente.
Control de reactivos: se utilizó agua en reemplazo de la muestra de ADN.
La amplificación fue realizada en un termociclador programable (Eppendorf Mastercycler Personal) en un total de 30 ciclos. Las condiciones de ciclado fueron: desnaturalización inicial a 94 °C durante 3 min seguida de 30 ciclos de 94 °C, 30 segundos (seg); 62 °C, 30 seg; 72 °C, 2 min, con una extensión final a 72 °C por 7 min.
Todo aislamiento que dio resultado negativo por PCR a 62 °C fue ensayado nuevamente a temperaturas de annealing menores (primero a 58 °C y luego a 55 °C) para compensar la/s posible/s divergencia/s en la secuencia de apareamiento de los oligonucleótidos utilizados.
Los amplicones fueron analizados mediante electroforesis en agarosa al 1.2 % buffer TAE/1X en presencia de 0.5 μg/ml bromuro de etidio a 5-10 V/cm durante 45 min. Finalizada la electroforesis el gel fue observado bajo iluminación ultravioleta (UV)
Los productos resultantes con los cebadores LSM12/SKH63 y LSM12/SKH52 presentaron tamaños de aproximadamente entre 1000 y 1200 pb, compatibles con los amplicones esperados para las familias PspA 1 y 2, respectivamente. De los aislamientos inicialmente negativos para familias 1 y 2, el ADN de sólo uno de ellos pudo ser amplificado con los cebadores LSM12/SKH2 (comunes a las 3 familias); el fragmento obtenido (de aproximadamente 1300 pb), fue secuenciado y, luego del análisis en Basic Local Alignment Search Tool (Blastn), incluido dentro de la familia 2.
Los resultados, que se presentan en la Tabla 1, mostraron que 42 de los 70 aislamientos fueron PspA familia 1 y 24 familia 2. En 4 no se identificó ninguna de las familias por el protocolo usado en este trabajo. Esta distribución de familias estuvo asociada a algunos serotipos: el 1 y 5 presentaron familia 1 únicamente.
TABLA 1.- Distribución de familias de PspA según serotipos y enfermedad

PBE: peritonitis bacteriana espontánea.
Se encontró asociación significativa entre el serotipo 1 y familia 1 (p = 0.000000). Esta observación no pudo ser confirmada para el serotipo 5 debido al bajo número de aislamientos encontrados. En los serotipos 14, 6B, 19F y 18C se detectaron genes correspondientes tanto a familia 1 como a la familia 2
Los aislamientos de nuestro estudio en los cuales no fue posible identificar alguna de las familias correspondieron a los serotipos 19F, 18C, 33F y 8.
La familia 1 se identificó en 34/57 neumonías y en 5/10 meningitis. También se observó en las 3 peritonitis espontáneas. La familia 2 se observó en 19/57 y 5/10 neumonías y meningitis, respectivamente. Los 4 aislamientos no tipificables fueron recuperados a partir de hemocultivos de pacientes con neumonía. No se detectó asociación entre las familias y la localización o extensión de la enfermedad (p = 0.538).
La Fig. 1 muestra una imagen típica de una electroforesis en agarosa al 1.2% de productos de PCR de los genes de PspA de algunos de los 70 aislamientos clínicos y de los controles positivos de Spn utilizados en cada PCR realizada.
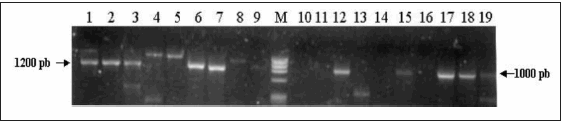
Fig. 1.- Imagen típica de una electroforesis en agarosa al 1.2% de productos de PCR de los genes de PspA de distintos aislamientos de Streptococcus pneumoniae. Calles: 1, Spn ATCC R36A (control positivo familia 1); 2, 3, 6, 7, 12, 15 y 17-19, PspA Familia 1; 4, Spn ATCC BG11703 (control positivo familia 2); 5 y 8, PspA Familia 2; 9, 10, 11 y 14, No tipificables; 16, control de reactivos; M, Marcador de tamaño molecular (![]() X174 DNA/Hae III, Promega Corp., USA).
X174 DNA/Hae III, Promega Corp., USA).
En los últimos años se han realizado varios trabajos sobre identificación de familias de PspA, en su mayoría sobre serotipos frecuentes en otros países. De los 40 aislamientos, a partir de infecciones invasoras en niños, estudiados en Colombia por Vela et al.10, 62.5% fueron PspA familia 1, y 35%, familia 2. En la Argentina, de los 149 aislamientos de infecciones invasoras en niños, caracterizados por Mollerach et al.11, 54.4% fueron PspA familia 1 y 41.6% familia 2. Por el contrario, Pimenta et al.12 en muestras nasofaríngeas de niños menores de 5 años y Hollinsghead et al.13 en muestras de adultos mayores de 50 años con enfermedad invasora, en Brasil y EE.UU. respectivamente, obtuvieron, en ambos estudios mayor prevalencia de la familia 2. En Alemania, Heeg et al.14 obtuvieron igual proporción de ambas familias en aislamientos a partir de niños con meningitis.
De manera similar a lo publicado por otros autores10-11, nuestros resultados evidenciaron una mayor frecuencia de familia 1 respecto a la 2.
En coincidencia con autores latinoamericanos10-11, en nuestro estudio los serotipos 1 y 5 presentaron únicamente familia 1. Ambos serotipos, no incluidos en la vacuna 7-valente, son altamente frecuentes en la población pediátrica de Sud América, en particular en países del Cono Sur, no así en Norte América. En el serotipo 6B de los aislamientos del presente trabajo se detectaron, en su mayoría, genes correspondientes a familia 2, a diferencia de Vela y col.10, que encontraron que todos los serotipos 6B fueron familia 1.
Pimenta et al.12, Brandileone et al.15 y Hollinsghead et al.13 no encontraron asociación significativa entre distribución de familia de PspA y tipo/extensión de la enfermedad. Mollerach et al.11, Vela et al.10 y Heeg et al.14, no hacen mención a este tipo de asociación.
En cuanto a los aislamientos en los cuales no se identificaron algunas de las familias de PspA, hubo diferencias entre nuestros resultados (6%) y los obtenidos por otros autores. Hollinsghead et al en EE.UU.13 obtuvieron un 0.2%, mientras que Pimenta et al., en Brasil, comunicaron un 20.2%12.
En conclusión, este estudio provee información epidemiológica acerca de la distribución de familias de PspA de los aislamientos de Spn recolectados de infecciones invasoras de nuestra zona, para ser utilizada en el desarrollo de vacunas recombinantes de PspA que tengan alta eficacia en nuestra región. Estos hallazgos son promisorios porque se podría lograr protección contra más serotipos que con las clásicas vacunas conjugadas a polisacáridos, lo que resultaría ser de menor costo e independiente del serotipo, factor importante para disminuir la tasa de morbi-mortalidad del Spn y su actual resistencia a los antimicrobianos.
Agradecimientos: La ejecución de este trabajo se llevó adelante con recursos económicos provenientes de la Universidad Nacional del Litoral, en el marco de Proyectos CAI+D.
Conflictos de interés: Los autores declaran que no existen conflictos de interés.
1. Bogaert D, Hermans PW, Adrian PV, Rümke HC, & de Groot R. Pneumococcal vaccines: an update on current strategies. Vaccine 2004; 22: 2209-22. [ Links ]
2. Pelton SI, Dagan R, Gaines BM, Klugman KP, Schmitt HJ. Pneumococcal conjugate vaccines: proceedings from an interactive symposium at the 41st Interscience Conference on Antimicrobial Agents. Vaccine 2003; 21: 1562-71. [ Links ]
3. Kaplan SL, Mason EO Jr, Wald ER, et al. Decrease of invasive pneumococcal infections in children among 8 children's hospitals in the United States after the introduction of the 7-valent pneumococcal conjugate vaccine. Pediatrics 2004; 113: 617-8. [ Links ]
4. Ruvinsky R. Epidemiology of invasive infections caused by Streptococcus pneumoniae in Argentina: A surveillance system, 1993-2004. Rev Hosp Niños BAires 2005; 47: 222-32. [ Links ]
5. Linder A, Hollingshead SL, Janulczyk R, Christensson B, Åkesson P. Human antibody response towards the pneumococcal surface proteins PspA and PspC during invasive pneumococcal infection. Vaccine 2007; 25: 341-5. [ Links ]
6. Crain MJ, Waltman II W D, Turner JS, et al. Pneumococcal surface protein A (PspA) is serologically highly variable and is expressed by all clinically important capsular serotypes of Streptococcus pneumoniae. Infect Immun 1990; 58: 3293-9. [ Links ]
7. Hollingshead SK, Becker R, Briles DE. Diversity of PspA: mosaic genes and evidence for past recombination in Streptococcus pneumoniae. Infect Immun 2000; 68: 5889-900. [ Links ]
8. Swiatlo E, Brooks-Walter A, Briles DE, McDaniel LS. Oligonucleotides identify conserved and variable regions of pspA and pspA-like sequences of Streptococcus pneumoniae. Gene 1997; 188: 279-84 . [ Links ]
9. Mayoral C, Baroni MR, Giani R, Virgolini S, Zurbriggen l, Regueira M. Distribución de serotipos de Streptococcus pneumoniae aislados de infecciones invasoras en el Hospital de Niños de Santa Fe. Rev Argent Microbiol 2008; 40: 13-6. [ Links ]
10. Vela Coral MC, Fonseca N, Castañeda E, Di Fabio JL, Hollingshead SK, Briles DE. Pneumococcal surface protein A of invasive isolates from Colombian children. Emerg Infect Dis 2001; 7: 832-6. [ Links ]
11. Mollerach M, Regueira M, Bonofiglio L, et al. Invasive Streptococcus pneumoniae isolates from Argentinean children: serotypes, families of pneumococcal surface protein A (PspA) and genetic diversity. Epidemiol Infect 2004; 132: 177-84. [ Links ]
12. Ribeiro-Dias F, Brandileone MC, Miyaji EN, Leite LC, Sgambatti de Andrade AL. Genetic diversity of PspA types among nasopharyngeal isolates collected during an ongoing surveillance study of children in Brazil. J Clin Microbiol 2006; 44: 2838-43. [ Links ]
13. Hollingshead SK, Baril L, Ferro S, King J, Coan P, Briles DE. Pneumococcal Proteins Epi Study Group. Pneumococcal surface protein A (PspA) family distribution among clinical isolates from adults over 50 years of age collected in seven countries. J Med Microbiol 2006; 55: 215-21. [ Links ]
14. Heeg C, Franken C, Van der Linden M, Al-Lahham A, Reinert, RR. Genetic diversity of pneumococcal surface protein A of Streptococcus pneumoniae meningitis in German children. Vaccine 2007; 25: 1030-5. [ Links ]
15. Brandileone MC, Andrade AL, Teles EM, et al. Typing of pneumococcal surface protein A (PspA) in Streptococcus pneumoniae isolated during epidemiological surveillance in Brazil: towards novel pneumococcal protein vaccines. Vaccine 2004; 22: 3890-6. [ Links ]
Recibido: 26-4-2010
Aceptado: 24-6-2010














