Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Medicina (Buenos Aires)
Print version ISSN 0025-7680
Medicina (B. Aires) vol.73 no.5 Ciudad Autónoma de Buenos Aires Oct. 2013
ARTÍCULO ORIGINAL
Caracteres clínico-patológicos y perfil genético en el carcinoma colorrectal
Florencia Perazzo1-2, Fernando Piaggio1, Hugo Krupitzki3, Alejandro García1, Alejandra Avagnina1, Boris Elsner1, Valeria Denninghoff1-3
1Servicio de Patología,
2Departamento de Oncología Clínica, Centro de Educación Médica e Investigaciones Clínicas (CEMIC),
3Dirección de Investigación CEMIC-Unidad Asociada a CONICET, Buenos Aires
Dirección postal: Dra. Valeria Denninghoff, Servicio de Patología, Centro de Educación Médica e Investigaciones Clínicas (CEMIC), Av. E. Galván 4102, 1431 Buenos Aires, Argentina
Fax: (54-11) 5299-0100 e-mail: vdenninghoff@cemic.edu.ar
Resumen
El cáncer colorrectal es el tercer cáncer más frecuente en hombres y el segundo más frecuente en mujeres, con una incidencia mundial aproximada de 1.2 millones de casos nuevos por año. Nuestro objetivo primario fue estudiar la relación existente entre las características clínico-histológicas en individuos con cáncer colorrectal y el estado mutacional de los codones 12 y 13 del gen KRAS (7 mutaciones validadas), con el fin de hallar un marcador histopatológico para los tumores mutados. El objetivo secundario fue determinar cuántos pacientes tenían mutaciones adicionales en los codones 15 y 61 del gen KRAS y 600 del gen BRAF que podrían modificar el fenotipo tumoral. Fueron seleccionados 60 individuos con cáncer colorrectal (30 wild-type y 30 con mutaciones validadas en los codones 12 y 13 del gen KRAS). Se amplificaron y secuenciaron del gen KRAS los exones 2 y 3, y del gen BRAF el exón 15. La información recolectada se examinó mediante un análisis descriptivo, análisis univariado y/o análisis multivariado, según correspondiese. En conclusión, no se encontró relación entre las características clínico-histológicas de los tumores de individuos con diagnóstico de cáncer colorrectal y el estado mutacional de los codones 12 y 13 del gen KRAS. No hallamos un marcador histopatológico para los tumores mutados. En pacientes con adenocarcinomas colorrectales avanzados y KRAS wild-type resulta de interés considerar el estudio del codón 600 del gen BRAF.
Palabras clave: KRAS; BRAF; Anti-EGFR; Cáncer colorrectal; Parámetros histopatológicos
Abstract
Clinical-pathological features and gene profile in colorectal cancer. Colorectal cancer is the third most frequent cancer in men and the second most frequent in women, with a worldwide incidence of approximately 1.2 million new cases per year. Our primary objective was to study the relationship between clinical and histological features of individuals with colorectal cancer and the mutational status of codons 12 and 13 of the KRAS gene (7 validated mutations), in order to find a histopathological marker to mutated tumors. The secondary objective was to determine how many patients had additional mutations in codons 15 and 61 of the KRAS gene, and codon 600 of the BRAF gene, which could modify the tumor phenotype. Sixty individuals with colorectal cancer (30 wild-type subjects and 30 with validated mutations in codons 12 and 13 of the KRAS gene) were selected. Exons 2 and 3 of the KRAS gene, and exon 15 of the BRAF gene were amplified and sequenced. The data collected were reviewed by a descriptive, univariate and/or multivariate analysis, as appropriate. In conclusion, no relation was found between clinical and histological features of individuals with colorectal cancer and their mutational status for codons 12 and 13 of the KRAS gene. This suggests that those easily available data do not allow predicting the response to anti-EGFR therapy. In patients with advanced colorectal adenocarcinomas and KRAS wild-type status, further study of codon 600 of the BRAF gene could be required.
Key words: KRAS; BRAF; Anti-EGFR; Colorectal cancer; Histopathological parameters
El cáncer colorrectal es el tercer cáncer más frecuente en hombres y el segundo más frecuente en mujeres, con una incidencia mundial aproximada de 1.2 millones de casos nuevos por año. Según la Organización Mundial de la Salud, aproximadamente 608 000 personas mueren anualmente en el mundo por esta causa, lo cual representa el 8% de las muertes por cáncer, siendo esta la cuarta causa más común1. En la enfermedad metastásica se recomienda utilizar como tratamiento complementario a la cirugía agentes quimioterápicos que han logrado en las últimas décadas aumentar la supervivencia global y el tiempo libre de enfermedad. El receptor de factor de crecimiento epidérmico (EGFR) representa un blanco importante en el tratamiento del cáncer, ya que su activación por parte de su ligando, el factor de crecimiento epidérmico (EGF), desencadena una cascada de reacciones intracelulares involucradas en el crecimiento y proliferación tumoral así como en los procesos de angioinvasión y metástasis2. En la última década se agregaron anticuerpos monoclonales, como por ejemplo el cetuximab o panitumumab (anti-EGFR) o bevacizumab (anti-VEGF) que aumentaron la supervivencia de estos pacientes. En pacientes con carcinomas colorrectales metastásicos (mCRC), al ser tratados con anticuerpos anti-EGFR, se ha observado una mejor supervivencia en el grupo que no posee la mutación del gen KRAS en el ADN del tumor cuando se los compara con aquellos pacientes portadores de la mutacion3. Por otro lado, la suma de estos agentes ha aumentado los efectos adversos y los costos de los tratamientos, por lo cual sería provechoso individualizar al grupo de pacientes que se beneficiaría con esta terapia. En los adenocarcinomas colorrectales se observa la mutación del oncogen KRAS en el 30-45% de los casos3-5.
Tanto la European Medical Agency (EMEA) como la Food and Drug Administration (FDA) de EE.UU., en el año 2008 aprobaron la utilización de anticuerpos monoclonales anti-EGFR en los pacientes con tumores que presentan KRAS no mutados (KRAS wild-type). La selección de pacientes para tratamiento anti-EGFR basada en el estado mutacional de los codones 12 y 13 del gen KRAS es altamente específica para los pacientes no respondedores: 95% de pacientes con CCR mutados para KRAS no se benefician con el tratamiento anti-EGFR. Pero no es suficientemente sensible debido a que solo la mitad de los pacientes con tumores KRAS wild-type responden al tratamiento6-7.
En el Centro de Educación Médica e Investigaciones Clínicas (CEMIC), a partir de junio de 2008, se realiza en forma rutinaria la detección de las mutaciones de los codones 12 y 13 del gen KRAS (G12V/D/S/A/C/R y en todos los pacientes con cáncer colorrectal con enfermedad metastásica o con estadio IV (luego de la aprobación del ANMAT). Las alteraciones que afectan al gen KRAS en adenocarcinomas, molecularmente bien caracterizadas y estables, son mayoritariamente sustituciones de aminoácidos en el codón 12 y 13 de la proteína. Existen 7 mutaciones/cambios aminoacídicos validados como responsables de la modificación en la respuesta al tratamiento anti-EGFR, y aprobados para la práctica clínica: G12V (GGT>GTT), G12D (GGT>GAT), G12S (GGT>AGT), G12A (GGT>GCT), G12C (GGT>TGT), G12R (GGT>CGT), y G13D (GGC>GAC). A su vez fueron descriptas mutaciones en los codones 15 y 61. Se ha descripto un grupo de pacientes de Taiwan que presentó una mayor frecuencia de mutaciones en el codón 15, con iguales características tumorigénicas6. Existe la necesidad de identificar marcadores predictivos adicionales a los codones 12 y 13 del gen KRAS (ya validadas), para poder seleccionar con mayor precisión al subgrupo de pacientes que responderían al tratamiento basado en anti-EGFR, como podrían ser los codones 15 y 61 de los exones 2 y 3 del gen KRAS respectivamente, o genes involucrados en el downstream de la cascada de señalización. Entre estos últimos se encuentra el gen BRAF (oncogén del sarcoma viral murino) involucrado en la regulación de la vía MAP/ERKs. Se han publicado trabajos retrospectivos los cuales demostraron que la mutación T1799A en el codón 600 del exón 15 del gen BRAF, que se traduce como un cambio aminoacídico valina por glutámico (V600E) afecta negativamente los tratamientos de pacientes con terapias anti-EGFR, que muestran menor supervivencia global y menor tiempo libre de enfermedad que aquellos pacientes con tumores BRAF wild-type7.
Nuestro objetivo primario fue estudiar la relación existente entre las características clínico-histológicas de individuos con cáncer colorrectal y el estado mutacional de los codones 12 y 13 del gen KRAS (7 mutaciones validadas), con el fin de hallar un marcador histopatológico para los tumores mutados. El objetivo secundario fue determinar cuantos pacientes tenían mutaciones adicionales en los codones 15 y 61 del gen KRAS y 600 del gen BRAF que podrían modificar el fenotipo tumoral.
Materiales y métodos
En el CEMIC, a partir de junio de 2008, se realiza en forma rutinaria la detección de las mutaciones de los codones 12 y 13 del gen KRAS en todos los pacientes con cáncer colorrectal con enfermedad metastásica o con estadio IV (G12V/D/S/A/C/R y (G13D). De estos pacientes, ya clasificados como wild-type o mutados para estas 7 mutaciones validadas, fueron seleccionados 60 individuos con cáncer colorrectal, entre septiembre de 2008 y septiembre de 2011, que fueron divididos en dos grupos según su estado predeterminado de las mutaciones validadas del gen KRAS: 30 wild-type (GrupoWT) y 30 con diferentes mutaciones (GrupoMT). Los criterios de inclusión fueron: pacientes adultos (mayores de 18 años), con una biopsia del tumor inicial preferentemente quirúrgica, con consentimiento informado firmado, y datos exigidos por el protocolo. Este estudio está adherido a la Declaración de Helsinki y fue aprobado por el Comité de Ética del CEMIC.
Se realizaron cortes histológicos de las biopsias incluidas en parafina, que se colorearon con hematoxilina-eosina para el posterior análisis de los parámetros morfológicos, que fueron volcados en una tabla.
Se purificó el ADN a partir de cortes de 7 μm de la biopsia incluida en parafina, se desparafinaron los cortes con xilol-etanol 100% (Carlo Erba, Italia) antes de la digestión enzimática con buffer-PK (100mM Tris ClH-ph = 8.25 mM EDTA, 0.5% SDS, 0.01% PK) a 42 °C. Las fracciones lipo-proteicas fueron extraídas con fenol-cloroformo-isoamílico (Carlo Erba, Italia). El ADN purificado fue precipitado con ClNa/isopropanol y resuspendido en T10E (Tris-EDTA). La pureza y el rendimiento del ADN obtenido fueron medidos por espectroscopia. Se amplificaron del gen KRAS2 los exones 2, antes llamado 1, (codones 1 al 37), y 3 (codones 38 al 81) que amplifican fragmentos de 111 y 133 pares de bases (pb) respectivamente, mediante los siguientes primers: K2-F (5'-ATGACTGAATATAAACTTGT-3'), K2-R (5'-CTCTATTGTTGGATCATATT-3') para el exón 2; y K3-F (5'-CAGGATTCCTACAGGAAGCAAGTAG-3'), K3-R (5'-CACAAAgAAAgCCCTCCCCA-3') para el exón 3 (chromosome 12 genomic contig-NCBI NT 009714.16). Las amplificaciones se llevaron a cabo en un volumen de reacción de 25 μl, utilizando entre 250 y 500 ng totales de ADN. A cada reacción se le incorporó 20 pmol de cada primer, 200 μM de cada dNTP, 10 mM Tris-HCl (pH 8.3), 50 mM KCl, 2 mM MgCl2, y 1 U de Taq ADN polimerasa (Promega Corporation, Madison, WI, USA). El ciclado térmico fue el siguiente: desnaturalización a 95 °C por 2 minutos, seguido de 35 ciclos (desnaturalización a 95 °C por 30 segundos, annealing a 49 °C por 30 segundos, y elongación a 72 °C por un minuto), con una elongación final a 72 °C por 5 minutos. Los primers diseñados resuelven la presencia de un pseudogen en el cromosoma 6. Por el otro lado, la presencia de las mutaciones del codón 600 del gen BRAF fue analizada por una tercera PCR con primers secuencia específicos que flanquean el exón 15 y amplifican un fragmento de 136 pb: BRAF600-F (5'-TGCTTGCTCTGATAGGAAAAT-3') y BRAF600-R (5'-AACTCAGCAGCATCTCAGGG-3'), (chromosome 7 genomic contig-NCBI NC_000007). La amplificación fue realizada como en los casos previos, a excepción de 1 mM MgCl2 y el annealing de 60 °C, por 30 segundos. Todas las PCR fueron evaluadas mediante electroforesis en gel de poliacrilamida al 9% en buffer TBE 1X (Tris-Bórico-EDTA) visualizadas con bromuro de etidio bajo luz ultravioleta. Se secuenciaron los fragmentos de los exones amplificados por PCR. La secuenciación se realizó con el BigDye® Terminator v3.1 Cycle Sequencing Kit (Applied Biosystem, Foster City, CA, USA). La secuencia fue separada por electroforesis capilar (ABI PRISM 310 Genetic Analyzer, Applied Biosystem, Foster City, CA, USA). La secuencia obtenida fue analizada con el Sequencing Analysis Software v5.2 (Applied Biosystem, Foster City, CA, USA).
Para realizar el análisis estadístico se confeccionó una base de datos con la información obtenida de la recolección de los datos epidemiológicos básicos, de los análisis de laboratorio y de los estudios histopatológicos y de biología molecular, para cada paciente. Se recolectó de cada paciente la siguiente información: sexo, edad, tabaquismo, antecedentes familiares de cáncer, antecedentes personales de otras neoplasias, localización del tumor, tamaño tumoral, presencia de obstrucción o perforación al diagnóstico, metástasis ganglionares, metástasis a distancia, estadio TNM, presencia de invasión vásculo-linfátia, y grado de diferenciación histológica. La información recolectada se examinó mediante un análisis descriptivo para clasificar, representar y resumir los resultados, lo cual nos permitió sintetizar la información contenida en los mismos. Asimismo, se aplicaron técnicas de análisis univariado y/o multivariado según correspondiese. El método estadístico usado es el chi cuadrado de Pearson con una confianza del 95% (la condición de una diferencia significativa está dada por un p menor a 0.05).
Resultados
De los 60 pacientes estudiados, 29/60 (48%) eran mujeres y 31/60 (52%) hombres, con una edad media de 61 años dentro de un rango etario entre 31 y 90 años. Tenían antecedentes de tabaquismo 31/60 (52%), mientras que 23/60 (38%) presentaban antecedentes familiares de cáncer y 11/60 (18%) tenían antecedentes personales de otras neoplasias. La localización del tumor primario fue 12/60 (20%) en colon derecho, 4/60 (7%) en colon transverso, 24/60 (40%) en colon izquierdo y 20/60 (33%) en recto. El diámetro tumoral máximo tuvo un rango entre 1.1 y 7.5 cm con un diámetro promedio de 3.8 cm. Fueron 6/60 (10%) T1, 6/60 (10%) T2, 21/60 (35%) T3, 22/60 (37%) T4a y 5/60 (8%) T4b. El estudio de los ganglios regionales determinó que 26/60 (43%) eran N0, 11/60 (18%) N1, 9/60 (15%) N2a y 11/60 (18%) N2b. No presentaban metástasis a distancia (M0) al momento del diagnóstico 41/60 (68%) casos y 18/60 (30%) sí los presentaban. Fueron estadificados de acuerdo a la clasificación TNM: 9/60 (15%) en estadio I, 16/60 (27%) en estadio II, 17/60 (28%) en estadio III y 18/60 (30%) en estadio IV. Presentaban evidencias de invasión vásculo-linfática 17/60 (28%) casos. Respecto a la diferenciación del tumor, 12/60 (20%) eran adenocarcinomas bien diferenciados, 37/60 (62%) moderadamente diferenciados, 4/60 (7%) indiferenciados y 7/60 (12%) tenían más del 50% de componente mucinoso.
No se encontraron diferencias significativas entre el estado mutacional del gen KRAS (7 mutaciones validadas) y el género (chi = 0.00 p =1.00), la edad (t = 0.70 p = 0.48), el tabaquismo (chi = 0.00 p = 1.00), los antecedentes familiares de cáncer (Fisher's exact = 0.03), los antecedentes personales de otras neoplasias (Fisher's exact = 1.00), la localización del tumor (chi = 6.52 p = 0.16), el tamaño tumoral (t = 1.01 p = 0.32), la invasión de la pared colónica (T) (Pearson chi2(5) = 2.00 Pr = 0.85), las metástasis ganglionares (N) (chi = 3.63 p = 0.46), las metástasis a distancia (M) (chi = 2.36 p = 0.50), el estadio TNM (Pearson chi2(5) = 5.70 Pr = 0.34), el estadio TNM con subestadios (chi= 9.37 p = 0.40), la presencia de invasión vásculo-linfática (chi = 2.08 p = 0.35), y el grado de diferenciación histológica (chi = 0.17 p = 0.98).
El análisis de los resultados de la secuenciación de los diferentes exones determinó que:
1. Codón 15 del exón 2 del gen KRAS: 60/60 (100%) fueron wild-type y 20/60 (33%) presentaban el polimorfismo G15G (GGC>GGT). De los 20 pacientes con este polimorfismo, 7/20 (35%) pertenecían al GrupoWT y 13/20 (65%) al GrupoMT. De los 13 pacientes del GrupoMT, 11 individuos tenían mutado el codón 12, y 2 individuos el 13 (Tabla 1).
TABLA 1.- Estado mutacional y polimorfismo del gen KRAS

2. Codón 61 del exón 3 del gen KRAS: 59/60 (98%) fueron wild-type y en 1/60 (2%) se encontraba mutado en E61R (CAA>CGA). Este único caso mutado es también G12V (GGT>GTT) y G15G (GGC>GGT), único tumor con 3 fenómenos genéticos en el mismo gen (Tablas 1 y 3).
TABLA 2.- Estado mutacional del codón 15 del gen KRAS en relación al estado mutacional del gen KRAS para los codones 12 y 13

(chi = 1.87, p = 0.18)
TABLA 3.- Estado mutacional del codón 600 del gen BRAF en relación al estado mutacional del gen KRAS
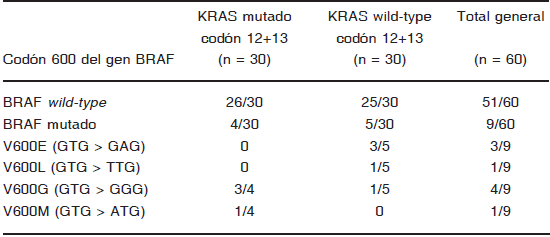
(chi=6.02, p = 0.20)
3. Codón 600 del exón 15 del gen BRAF: 51/60 (85%) fueron wild-type y 9/60 (15%) presentaban mutación. De los 9 casos mutados, 4/9 (44%) correspondían al GrupoMT, mientras que 5/9 (56%) correspondían al GrupoWT, aumentando así el número total de casos mutados (Tabla 3). En los 9 casos mutados se observaron 4 mutaciones: 4/9 (44%) para V600G (GTG>GGG), 3/9 (33%) V600E (GTG>GAG), 1/9 (11%) V600L (GTG>TTG) y 1/9 (11%) V600M (GTG>ATG). Las diferentes mutaciones presentaron una impronta distinta en ambos grupos, mientras que en el GrupoMT predominó la mutación V600G (3/4, 75%), en el GrupoWT predominó la mutación V600E (3/5, 60%). Además esta última mutación se presentó exclusivamente en el Grupo WT (Tabla 3).
El análisis total de las mutaciones se puede observar en la Tabla 4, solo 25/60 (42%) pacientes serían absolutamente wild-type si se realizara la sumatoria de los 5 codones. Si solo se tuviese en cuenta la mutación V600E del gen BRAF, que es la responsable comprobada de una activación constitutiva de la proteína, con su consiguiente oncogenicidad, el número de pacientes totalmente wild-type para los codones estudiados sería 28/60 (47%).
TABLA 4.- Estado mutacional de los codones 12, 13, 15 y 61 del gen KRAS y del codón 600 del gen BRAF

A) mutaciones individuales y B) sumatoria de casos wild-type para los 5 codones analizados. *No se resta el caso porque ya pertenecía al GrupoMT (E61R+G12V+G15G). #Se restan 5/9 casos porque los 4/9 casos restantes pertenecían al GrupoMT.
Discusión
El análisis de las características epidemiológicas de los individuos (género, edad, tabaquismo, antecedentes familiares de cáncer, y antecedentes personales de otras neoplasias), e histológicas del tumor (localización del tumor, tamaño tumoral, invasión de la pared colónica, metástasis ganglionares, metástasis a distancia, estadio TNM, presencia de invasión vásculo-linfática, y grado de diferenciación histológica) no arrojó diferencias significativas entre los distintos grupos de mutaciones estudiadas (GrupoWT vs. GrupoMT). No se encontraron marcadores histopatológicos para los tumores mutados (GrupoMT). Estos resultados son concordantes con los publicados por Weichert y col., donde no se hallaron diferencias significativas en los parámetros histológicos respecto de la determinación de KRAS por diferentes métodos8. Existen escasas publicaciones que aborden un análisis minucioso de las características histológicas de los cánceres colorrectales y su correlación con el estado mutacional de KRAS y BRAF. Las características histológicas de los tumores analizadas en nuestro trabajo, así como los datos epidemiológicos, no demostraron relación significativa con ninguno de los estudios moleculares realizados en este trabajo.
El estudio adicional de los codones 15 del exón 2 y 61 del exón 3 del oncogen KRAS permitió encontrar, dentro del GrupoMT, un paciente con tres fenómenos genéticos concomitantes (G12V, G15G y E61R). Estos resultados difieren con los obtenidos por Richman y col., quienes encontraron 23/711 (3% de mutaciones en el codón 61 en pacientes wild-type para los codones 12 y 13 del mismo gen10. El codón 15 presenta una tasa del 33% (20/60) de polimorfismo, debido a que no evaluamos supervivencia ni respuesta, no podemos determinar la importancia de este polimorfismo como posible factor predictivo/pronóstico.
Realizar, en todos los casos con KRAS wild-type (GrupoWT), el estudio complementario del codón 600 del exón 15 del gen BRAF permitió identificar un subgrupo de individuos que podrían tener un comportamiento biológico diferente y por lo tanto una respuesta diferente a la terapia. El estudio adicional del codón 600 agregó un número importante de casos mutados totales (5/30, 17%). De esta manera el genotipo de estos pacientes estaría determinando una resistencia al tratamiento monoclonal que no fue detectada en la primera etapa del trabajo. Este porcentaje es significativamente mayor que el publicado por Laurent-Puig y col, y Di Nicolantonio y col., quienes encontraron un 3% y un 10% respectivamente de casos mutados para BRAF dentro del subgrupo KRAS wild-type7,9. Por otra parte, encontramos 4/30 (13%) BRAF mutado dentro del GrupoMT, lo cual representa otra diferencia ya que los estudios antes mencionados sólo encontraron mutaciones en el GrupoWT y sostienen que ambas son excluyentes. La importancia de este hallazgo es que el agregado de una segunda mutación podría estar determinando un fenotipo tumoral diferente, aun no descripto ni evaluado. Ambas diferencias pueden ser explicadas por la metodología utilizada, dado que en nuestro trabajo usamos PCR-secuenciación por método Sanger y en los otros dos trabajos se usó discriminación alélica con sondas TaqMan. De esta forma si tuviésemos que hacer una comparación certera, solo 3/30 (10%) pacientes del GrupoWT tenía la mutación V600E, única mutación detectada por el método TaqMan. Por otro lado, dada esta limitación del método TaqMan para este tipo de análisis, en el GrupoWT, si solo pudiésemos ver la mutación V600E, no hubiésemos hallado las mutaciones V600G y V600M. Esto sugiere que sería necesario agregar el estudio del codón 600 del exón 15 del gen BRAF a todos los pacientes con KRAS wild-type antes de instaurar el tratamiento con anti-EGFR para identificar dentro de este subgrupo una mayor cantidad de casos que no responderán positivamente a la terapia. Cuando comenzamos a estudiar el KRAS se desconocía la respuesta diferencial al tratamiento según el tipo de mutación que el tumor del paciente poseía en los codones 12 y 13, por lo tanto seguimos creyendo conveniente el estudio de todas las posibles variaciones genéticas del codón 600 del gen BRAF, que posiblemente, al igual que como ocurrió con el KRAS, en un futuro estas alternativas genéticas puedan tener implicancia clínica.
La adición de defectos genéticos en una vía de señalización determinada podría modificar el fenotipo tumoral. En nuestro trabajo realizamos un estudio exhaustivo de la vía de señalización MAP/ERKs y de ese análisis se desprendieron cuatro datos interesantes: un paciente con tres fenómenos genéticos concomitantes en el gen KRAS (G12V, G15G y E61R), 3/30 (10%) pacientes del GrupoWT con la mutación V600E, dicha mutación solo se encontró en el GrupoWT, y encontramos, dentro del GrupoMT, 4/30 (13%) individuos con BRAF mutado (V600G y V600M). A pesar del bajo número de casos estudiados, este trabajo podría ser considerado un generador de hipótesis, porque de confirmarse estos resultados con un número mayor de casos, los métodos de análisis de las mutaciones tumorales, así como también la descripción precisa del tipo de mutación, podrían modificar el tratamiento.
En conclusión, no se encontró relación entre las características clínico-histológicas de los tumores de individuos con diagnóstico de cáncer colorrectal y el estado mutacional de los codones 12 y 13 del gen KRAS. No hallamos un marcador histopatológico para los tumores mutados. Esto sugiere, por el momento, la ausencia de factores predictivos más específicos factibles de ser usados en la práctica diaria que nos permitan predecir la respuesta con una mayor precisión. En pacientes con adenocarcinomas colorrectales avanzados y KRAS wild-type resulta interesante considerar el estudio del codón 600 del gen BRAF pues permitiría distinguir un subgrupo de pacientes que podría responder de una manera diferente al tratamiento basado en anticuerpos anti-EGFR.
Agradecimientos. Agradecemos a Mariana dos Santos por su colaboración técnica, a Myrthala Duguid (Directora de la Biblioteca del Instituto Universitario CEMIC), y a Valeria Melia por la revisión idiomática.
Conflictos de interés: Los autores declaran no tener conflicto de interés.
1. World Health Organization. WHO cancer mortality database (IARC) En: http://www-dep.iarc.fr/WHOdb/WHOdb.htm; consultado el 30/05/2011. [ Links ]
2. Chung KY, Shia J, Kemeny NE, et al. Cetuximab shows activity in colorectal cancer patients with turmos that do not express the epidermal growth factor receptor by immunohistochemistry. J Clin Oncol 2005; 23: 1803-10. [ Links ]
3. Lievre A, Bachet J-B, Le Corre D, et al. KRAS mutation status is predictive of response to cetuximab therapy in colorectal cancer. Cancer Res 2006; 66: 3992-5. [ Links ]
4. Moroni M, Veronese S, Benvenuti S, et al. Gene copy number for epidermal growth factor receptor (EGFR) and clinical response to antiEGFR treatment in colorectal cancer: a cohort study. Lancet Oncol 2005; 6: 279-86. [ Links ]
5. Siena S, Sartore-Bianchi A, Di Nicolantonio F, Balfour J, Bardelli A. Biomarkers predicting clinical outcome of epidermal growth factor receptor-targeted therapy in metastatic colorectal cancer. J Natl Cancer Inst 2009; 101: 1308-24. [ Links ]
6. Wang JY, Hsieh JS, Chen FM, et al. High frequency of activated K-ras codon 15 mutant in colorectal carcinomas from Taiwanese patients. Int J Cancer 2003; 107: 387-93. [ Links ]
7. Laurent-Puig P, Cayre A, Manceau G, et al. Analysis of PTEN, BRAF, and EGFR status in determining benefit from cetuximab therapy in wild-type KRAS metastatic colon cancer. J Clin Oncol 2009; 27: 5924-30. [ Links ]
8. Weichert W, Schewe C, Lehmann A, et al. KRAS genotyping of paraffin-embedded colorectal cancer tissue in routine diagnostics. Comparison of methods and impact of histology. J Mol Diagn 2010; 12: 35-42. [ Links ]
9. Di Nicolantonio F, Martini M, Molinari F, et al. Wild-type BRAF is required for response to panitumumab or cetuximab in metastatic colorectal cancer. J Clin Oncol 2008; 26: 5705-12. [ Links ]
10. Richman SD, Seymour MT, Chambers P, et al. KRAS and BRAF mutations in advanced colorectal cancer are associated with poor prognosis but do not preclude benefit from oxaliplatin or irinotecan: results from the MRC FOCUS trial. J Clin Oncol 2009; 27: 5931-7. [ Links ]
Recibido: 7-V-2012
Aceptado: 22-I-2013














