El coronavirus SARS-CoV-2, agente causal de la enfermedad pandémica COVID-19, ha causado efectos devastadores en todo el planeta, incluyendo la muerte de más de 3.9 millones de personas a nivel mundial, más de 105 000 en nuestro país con un dramático impacto socioeconómico1,2.
Si bien la mayor parte de los pacientes diagnosticados con COVID-19 desarrollan una enfermedad leve, el 5-10% de ellos cursa una enfermedad grave que requiere hospi talización, y un 2-5% debe ser ingresado en una unidad de cuidados intensivos3. En este último grupo de pacientes, la gravedad de la infección se asocia al desarrollo de un cuadro inflamatorio denominado Síndrome de Distrés Respiratorio Agudo (SDRA) y a fallas multiorgánicas4. La rápida propagación de esta pandemia, la falta de te rapias específicas, el advenimiento de nuevas variantes virales y la implementación de estrategias de vacunación diversas, exigen el compromiso urgente de la comunidad científica en la comprensión de la naturaleza, magnitud y persistencia de la respuesta inmunológica generada en pacientes recuperados de COVID-19 y en individuos vacunados con diferentes estrategias y dosis, a fines de tomar decisiones de salud pública basadas en evidencias locales e implementar nuevas políticas sanitarias.
Se ha reportado que los linfocitos T son capaces de controlar múltiples infecciones virales y brindar protección ante subsecuentes re-infecciones mediante la generación de memoria inmunológica (Fig. 1)5. En este sentido, es imprescindible comprender la naturaleza y magnitud de la respuesta de linfocitos T antígeno-específicos para el monitoreo de la efectividad de vacunas generadas a partir de diferentes plataformas y basadas en diferentes estra tegias [virus inactivados, vectores adenovirales, ácidos nucleicos, proteínas recombinantes y partículas similares a virus (VLP, del inglés virus-like particles)].
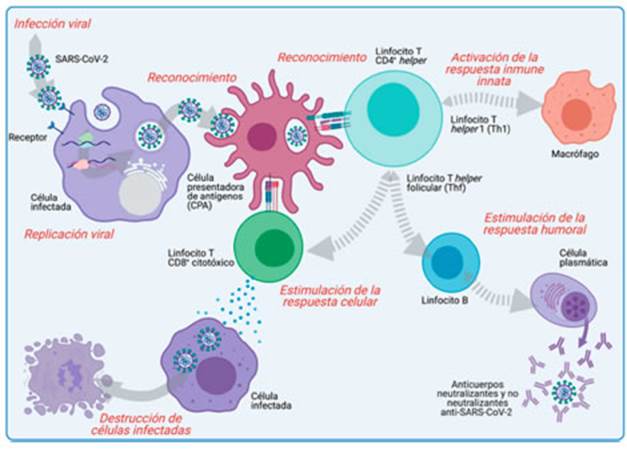
Fig. 1 Respuesta inmunológica humoral y celular frente a la infección por SARS-CoV-2. Las células presentadoras de antígeno (CPA; ej. células dendríticas) responsables de orquestar la respuesta inmunológica, reconocen el virus, lo procesan y presentan a linfocitos T CD8+ y CD4+ vírgenes (naïve) en ganglios linfáticos, en el contexto de moléculas del complejo mayor de histocompatibilidad (CMH) tipo 1 o 2, respectivamente. Los linfocitos T CD4+ activados (caracterizados por una alta expresión de citoquinas como IL-2 y moléculas de activación como CD40L/CD154), se diferencian en distintas subpoblaciones celulares, incluidos los linfocitos T helper 1 (Th1) productores de IFN-γ y TNF-α, capaces de activar macrófagos y los linfocitos T helper foliculares (Thf), productores de IL-21 responsables de cooperar con linfocitos B que, al diferenciarse a células plasmáticas, producen anticuerpos neutralizantes y no neutralizantes del virus, entre otros. Por otro lado, linfocitos T CD4+ activados facilitan la activación de linfocitos T CD8+ citotóxicos responsables de destruir células infectadas por SARS-CoV-2
En lo que respecta a otros coronavirus de la familia (SARS-CoV), se ha observado que los niveles de anti cuerpos caen por debajo del límite de detección entre 1 y 3 años6, mientras que los linfocitos T de memoria per manecen activos hasta incluso 11 años después7. A su vez, estudios recientes, en pacientes recuperados de CO VID-19, revelan el valor fundamental de la respuesta de linfocitos T (tanto CD4+ como CD8+) a fines de monitorear la memoria inmunológica desarrollada como consecuen cia de la infección. De hecho, un porcentaje sustancial de pacientes que no presentaron respuesta humoral tras la infección con SARS-CoV-2, sí evidenciaron respuestas específicas mediadas por linfocitos T8. En este sentido, Le Bert y colaboradores han propuesto que los pacien tes con COVID-19 tendrían la capacidad de desarrollar inmunidad a largo plazo mediada por linfocitos T CD4+ y CD8+ específicos de SARS-CoV-2, demostrando la im portancia clave de identificar estas poblaciones celulares con el objetivo de caracterizar la memoria inmunológica en sujetos recuperados9. A su vez, en función de la di versidad de estrategias de vacunación implementadas en nuestro país y en el mundo y el debate actual respecto a la inmunogenicidad de las mismas, y la duración de sus efectos, resulta de crucial importancia realizar estudios comparativos de la memoria inmunológica generada por las distintas vacunas en poblaciones locales con perfiles similares. Por otro lado, es un gran desafío científico el estudio de respuestas inmunológicas de pacientes recu perados e individuos vacunados frente a nuevas variantes emergentes del SARS-CoV-2. Finalmente, es relevante el estudio de reacciones cruzadas entre linfocitos T de individuos sanos frente a coronavirus estacionales y el SARS-CoV-29-12. En función de estas necesidades, deci dimos desarrollar e implementar la Plataforma COVID-T como herramienta para la determinación, caracterización y monitoreo de la respuesta linfocitaria T específica de SARS-CoV-2 en distintas poblaciones de pacientes recuperados e individuos vacunados con diferentes es trategias y esquemas.
En este contexto, se optimizó la metodología para el análisis, caracterización y monitoreo de la respuesta linfocitaria T a partir de muestras obtenidas de sangre de individuos vacunados y de pacientes recuperados de COVID-19 (Fig. 2). Los donantes son reclutados en base a su historia clínica de previa infección con SARS-CoV-2 (leve, moderada o grave) o su participación en los diferentes esquemas de vacunación determinados por los Ministerios de Salud nacionales, provinciales y de la ciudad de Buenos Aires, bajo protocolo aprobado por el Comité de Ética institucional, dependiente del Comité Central de Ética en Investigación del Ministerio de Salud de la Ciudad de Buenos Aires (Código de Registro: 4679). Las muestras requeridas para la optimización de la pla taforma fueron provistas inicialmente por el Biobanco del Instituto de Investigaciones Biomédicas en Retrovirus y SIDA (INBIRS, UBA-CONICET), el Hospital General de Agudos “Doctor Ignacio Pirovano” (Ciudad Autónoma de Buenos Aires) y el Ministerio de Salud de la Provincia de Buenos Aires.
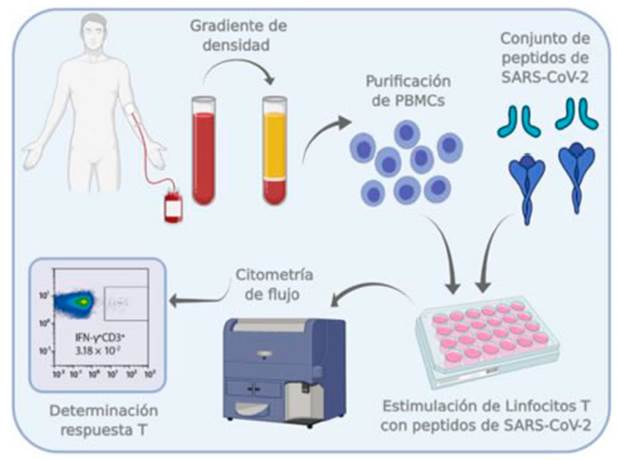
Fig. 2 Esquema de trabajo de la plataforma COVID-T. La sangre del paciente o individuo vacunado es centrifugada en gradiente de densidad a los fines de aislar y purificar las células mononucleares de sangre periférica (PBMCs). Estas células son cultivadas y estimuladas en presencia o ausencia de péptidos de SARS-CoV-2 in vitro. Finalmente, se analiza la producción de citoquinas y la expresión de moléculas de activación por parte de linfocitos T CD4+ y CD8+ activados, utilizando anticuerpos conjugados a moléculas fluorescentes, por citometría de flujo. La información obtenida se integra en coeficientes que indican la activación celular, de acuerdo a la medición de moléculas de superficie y citoquinas intracelulares.
La metodología central de la Plataforma COVID-T in volucra la utilización de muestras de sangre de pacientes recuperados, individuos vacunados o donantes sanos que no posean historia de contagio y cuyos resultados sero lógicos resulten negativos (ausencia de anticuerpos IgG e IgM contra SARS-CoV-2). Luego de la obtención de la muestra de sangre anticoagulada con EDTA, se procede a la separación de plasma, para el posterior análisis de anticuerpos específicos y neutralizantes de SARS-CoV-2, y al aislamiento de las células mononucleares de sangre periférica (PBMCs, por sus siglas en inglés, peripheral blood mononuclear cells).
La evaluación de la respuesta de linfocitos T especí ficos de SARS-CoV-2 se realiza utilizando un conjunto de péptidos seleccionados para activar linfocitos T específicos de SARS-CoV-2. Estos incluyen péptidos S (provenientes de la glicoproteína Spike), péptidos S1 (provenientes del dominio S1 de la glicoproteína Spike, el cual incluye el sitio de reconocimiento de la enzima convertidora de angiotensina 2; ACE2), péptidos N (provenientes de la proteína de la nucleocápside) y pép tidos M (provenientes de la proteína de membrana). Las PBMCs se cultivan en presencia (activación) o ausencia (control negativo sin activar) de los péptidos específicos de SARS-CoV-2. Los controles que se realizan implican: a) un control positivo con forbol-12-miristato-13-acetato (PMA) e ionomicina, y b) un control positivo de activa ción con péptidos de citomegalovirus (CMV), el cual se encuentra en alta frecuencia en la población, por lo que se pueden detectar respuestas positivas entre 50-100% de los individuos analizados13,14 (Fig. 2).
Finalmente, se determina la respuesta de linfocitos T específicos de SARS-CoV-2 mediante citometría de flujo al evaluar la expresión de marcadores de activación en linfocitos T CD4+ y CD8+. Entre ellos, se determina la ex presión de CD40L (CD154), molécula cuya expresión se induce tras la activación del receptor de células T (TCR por sus siglas en inglés, T cell receptor), cuyo papel es crucial en la generación de la respuesta humoral al inte ractuar con CD40 expresado en linfocitos B. Asimismo, se analiza la producción de citoquinas asociadas a una respuesta antiviral tales como interferón γ (IFN-γ), inter leuquina-2 (IL-2) y el factor de necrosis tumoral (TNF)-α. Para ello, tras la activación y estimulación con los péptidos anteriormente mencionados, se incuban las PBMCs con anticuerpos específicos para CD3, CD4, CD8, CD154, IFN-γ, TNF-α e IL-2. Para la discriminación de las células vivas se utiliza una sonda vital. Luego, las muestras se analizan en el citómetro de flujo BD FACSCanto II con el programa BD FACSDiva. Los datos obtenidos se pro cesan con el programa FlowJo 10.0 (BD) y se grafican con el programa GraphPad Prism 8.0. De esta manera, se pueden detectar las diferencias en la frecuencia y el fenotipo de las células T específicas de antígeno prove nientes de los distintos grupos de pacientes recuperados e individuos vacunados. Finalmente, mediante el análisis multivariado de los parámetros mencionados (citoquinas y marcadores de activación en diferentes subpoblaciones) se genera un coeficiente que permite determinar el grado de activación de la respuesta linfocitaria T. Como resulta do en un número de pacientes recuperados e individuos vacunados es posible verificar la presencia de linfocitos T CD4 y T CD8 productores de citoquinas, específicos de SARS-CoV-2, ya que fueron activados en presencia de péptidos específicos pertenecientes al virus.
Por otro lado, se ha observado que individuos que han sufrido infecciones previas con otros coronavirus estacio nales presentan linfocitos T de memoria que reaccionan en forma cruzada y podrían potencialmente proteger frente a una infección con SARS-CoV-29. Estos sujetos no vacunados ni infectados previamente con SARS-CoV-2 podrían ser identificados si sus linfocitos se activan al cultivarse con péptidos del virus.
En conclusión, la plataforma COVID-T recientemente implementada permitirá el análisis y monitoreo de la mag nitud, naturaleza y persistencia de la respuesta celular, particularmente linfocitos T específicos de SARS-CoV-2, en un amplio espectro de poblaciones que involucran: a) pacientes recuperados de COVID-19 (que cursaron la en fermedad leve, moderada o severa); b) individuos vacuna dos con diferentes estrategias y protocolos, y c) individuos vacunados de grupos poblacionales de riesgo. Asimismo, este desarrollo brindará la posibilidad de comparar en forma progresiva la respuesta inmunológica de individuos inmuni zados con diferentes esquemas de vacunación y pacientes recuperados de COVID-19. Finalmente, la plataforma COVID-T permitirá estudiar la respuesta inmunológica de individuos vacunados y recuperados frente a variantes virales emergentes. Es nuestro deseo que los resultados obtenidos a partir de esta plataforma brinden información relevante para la toma de decisiones y el diseño de nuevas estrategias preventivas y terapéuticas, no solo en esta pandemia sino en futuros desafíos de Salud Pública, ge nerando una nueva capacidad científico-tecnológica con impacto sanitario y social.














