PUNTOS CLAVE
Conocimiento actual
• La minería de texto aplicada a las publicaciones cien tíficas es una herramienta poderosa para analizar gran número de documentos y comportamientos, rastrear tendencias o hacer predicciones. Hasta 2020 (inclusive) aparecen 16 192 artículos que contienen los términos Síndrome Urémico Hemolítico (SUH) en la base de publicaciones ePMC.
Contribución del artículo al conocimiento actual
• Se analizó el texto de todas las publicaciones mencio nadas, obteniéndose resultados estadísticos de medios, autores, países participantes, colaboraciones científicas, evolución y frecuencia de términos significativos para el SUH. Se caracterizó el impacto de los fenómenos emer gentes -como la epidemia alemana- en la evolución de las publicaciones científicas.
Si bien existen alternativas para su definición1, el síndrome urémico hemolítico (SUH) se identifica como un síndrome clínico sistémico caracterizado por microan giopatía trombótica, anemia hemolítica, trombocitopenia e insuficiencia renal aguda. Es la causa más común de insuficiencia renal aguda en bebés y niños pequeños, aunque puede afectar a adolescentes y adultos. Puede causar graves secuelas permanentes y en ocasiones ser mortal. La causa generalizada del SUH es la infección por Escherichia coli productora de toxina Shiga (STEC), cuyo ingreso al organismo sucede por ingestión de alimentos o agua contaminados y por contacto tanto entre personas y animales infectados, como de persona a persona2-5. Las infecciones por STEC siguen siendo endémicas en América Latina6. La República Argentina tiene la mayor incidencia mundial de SUH en menores de 5 años7.
Por otro lado, y sin relación con infección por STEC, algunas enfermedades asociadas con mutaciones en ge nes que codifican factores de complemento y otras causas menos comunes, pueden dar lugar al mismo síndrome, en lo que se describe como “SUH atípico” (aSUH)8.
SUH fue descrito inicialmente en 19559. Desde ese primer trabajo, un gran número de publicaciones contri buyeron a la comprensión del síndrome, así como a paliar las afecciones de los pacientes.
Uno de los problemas para analizar la incidencia y evolución del SUH es el difícil acceso a registros clínicos, datos epidemiológicos y/o estadísticas, algunos existentes de manera dispersa y/o desactualizados. Además, y si bien el SUH es un cuadro de notificación obligatoria e inmediata, en algunos países -como la Argentina- no siempre se informan los casos. Por el contrario, el acceso a la información de repositorios científicos de consulta libre y permanentemente actualizados es sencillo. Sin embargo, debido al gran número de artículos biomédicos que se publican mundialmente, es cada vez más nece sario contar con sistemas automatizados para extraer información de la literatura especializada10,11.
La hipótesis de nuestro trabajo sostiene que la minería de texto (MT) en bases de datos científicos sobre SUH es una poderosa herramienta para extraer información no explícita, analizar comportamientos, realizar un se guimiento de tendencias, hacer predicciones y recabar información específica de interés médico.
Para contrastarla, realizamos un análisis detallado aplicando minería de texto en el corpus resultante de una búsqueda bibliográfica sobre SUH que rescató todas las publicaciones indexadas en la base de datos de Europe PMC (ePMC, https://europepmc.org/) entre 1955 y 2020 inclusive. ePMC es una plataforma científica abierta que proporciona acceso a una colección global de publicaciones en ciencias de la vida, de fuentes confiables. ePMC es desarrollado por el Instituto Europeo de Bioinformática (EMBL-EBI), una entidad asociada a PubMed Central, pero que la supera en más de 5 millones de resúmenes. Además, ePMC contiene patentes y otros registros.
Nuestro objetivo consistió en analizar el texto sub yacente a nivel de los descriptores utilizados en las búsquedas en ePMC, lo que aportó una visión diferente al de una revisión temática tradicional.
Este trabajo muestra los resultados del análisis con MT de un conjunto de datos contenidos en 16 192 artículos originales, un número difícil de abordar con las revisiones usuales. Identificamos así 89 850 autorías -de las cuales 52 203 son autores únicos-, extrajimos 54 250 lugares de trabajo de los autores y listamos los medios de publi cación de los artículos. Analizamos el texto de 13 008 resúmenes, extrayendo las palabras más utilizadas, realizando estadísticas, estudios temporales, pronósticos, correlaciones con brotes de SUH, detección de temas por medio de aprendizaje automático y cruce de información con otras fuentes de datos científicos. En resumen, en este trabajo presentamos e integramos información sobre el SUH, obtenida mediante el abordaje novedoso de la minería de texto.
Materiales y métodos
La sintaxis de la búsqueda en ePMC se construyó en inglés, teniendo en cuenta las tres formas en las que el SUH ha sido nombrado por diferentes autores: “Haemolytic uraemic syndrome”, “Hemolytic uremic syndrome” o “Hemolitic uremic syndrome”. El corpus de trabajo resultante está en inglés, y el análisis se realizó teniendo en cuenta las características de esta lengua.
Las herramientas informáticas utilizadas se detallan a continuación. KNIME Analytics Platform 4.1.0 (https://www.knime.com/), un software de acceso libre y gratuito, se usó para la construcción de la base de datos y su análisis. La plataforma KNIME permite crear visualmente flujos de trabajo de datos, utilizando nodos en pasos sucesivos, haciendo posible inspeccionar cada resultado parcial. VOSviewer 1.6.13 (https://www.vosviewer.com/) se usó para la creación de redes representativas de la relación entre autores. AntConc 3.5.7 (por Laurence Anthony, Universidad de Waseda, Japón) se usó para el análisis lingüístico del corpus conformado por todos los resúmenes. Microsoft Excel se utilizó para estadís ticas, pronósticos y trazados específicos.
Con AntConc se detectaron las unidades gramaticales, se contabilizó la suma de todas y se determinó también la frecuencia de uso de cada una de ellas. Se entiende por “unidad gramatical” a todo conjunto de caracteres o secuencia grafológica (por ejemplo, 2 palabras unidas entre sí por un guión) separados de otros por un espacio o por un signo de puntuación. En la minería de texto, esta unidad es denomina da token. Así, los tokens son todas las unidades gramaticales que hay en el texto (repetidas o no). Formas o types, en cambio, son un subconjunto dentro de los tokens: las unidades gramaticales distintas, sin tener en cuenta las repeticiones. AntConc se configuró para incluir letras, números y signos de puntuación en la definición de token.
En este trabajo utilizamos una lista de lema (lemma list) (https://lexically.net/downloads/BNC_lemmafile5.txt), corregida y ampliada para incluir los términos del tema SUH antes de ser utilizada en AntConc. La lista de lema es una lista que incluye una palabra y las variaciones de esa palabra. En KNIME, los términos fueron “lematizados” con la biblioteca PNL de Stanford Core.
Se utilizó además una lista de “palabras vacías” (stop words) para el análisis en KNIME y AntConc. La lista de “palabras vacías” es un conjunto de palabras que no tienen significado para el análisis, como preposiciones y determinan tes (por ejemplo, los términos the, a, in, to, from y otros) y que deben excluirse de los resultados antes de procesar un texto de lenguaje natural.
Mediante un flujo de trabajo digital armado en KNIME, se obtuvieron los resultados de la consulta a ePMC y se seleccionaron los campos: Id (identidad asignada a una publicación en el repositorio), source (fuente), pmid (identi ficador de PubMed de los trabajos indexados), pmcid (iden tificador de artículo con texto completo en PubMed Central), doi (identificador de objeto digital), title (título), authorString (autores de la publicación), pubYear (año de publicación), abstractText (texto del resumen), fullTextUrlList (link al texto digital completo del artículo), affiliation (lugar/es donde se realizó el trabajo; contiene en muchos casos los nombres de: institución, laboratorio, ciudad y país) y medlineAbbre viation (abreviatura de Medline referida al medio donde fue publicado el artículo). Se analizaron documentos publicados entre 1955 y 2020 inclusive, para extraer los siguientes descriptores: i) número de artículos por año; ii) número de artículos que incluyen resúmenes por año; iii) lista de todos los autores; iv) lista de primeros autores; v) número de ar tículos publicados por cada autor; vi) número de artículos publicados por cada primer autor; vii) número de artículos por sitio de publicación. Además, se obtuvo una “bolsa de palabras” de los resúmenes y la frecuencia de uso de pa labras. También se aplicó a los resúmenes un extractor de temas no supervisado (véase más adelante).
Para el análisis de filiación, debió tenerse en cuenta que los datos de filiación institucional (organización y dirección) no se encuentran estandarizados para los primeros años de búsqueda en la base de datos ePMC y que, en muchos ca sos, la filiación no figura siquiera en esta base de datos. Por lo cual, en ocasiones este campo no pudo ser minado para obtener el país de filiación.
En cuanto a la “bolsa de palabras”, es un conjunto que contiene los términos incluidos en un documento, sin tener en cuenta la gramática ni el orden de las palabras, aunque sí registrando el número de veces que aparecen en el texto. En KNIME, para etiquetar términos, se utilizó el nodo etique tador Abner Tagger, que reconoce entidades con nombre biomédico. Los términos fueron “lematizados” con la biblioteca PNL de Stanford Core. Se eliminaron todos los caracteres de puntuación, se aplicó un filtro con la lista de “palabras vacías” especificada y todos los términos contenidos en los documentos de entrada se convirtieron a letra mayúscula. Los valores resultantes en la “bolsa de palabras” se contaron y se ordenaron.
Debido a que antes de 1970, en la base ePMC, no hay un número representativo de resúmenes sobre SUH, solo se utilizaron los que aparecen a partir de ese año para construir la “bolsa de palabras”, con un resultado de 52 277 términos diferentes (types) de 1 486 645 tokens.
Para la detección de temas, se aplicó la Implementación simple en paralelo de la asignación latente de Dirichlet (LDA) al procesamiento del lenguaje natural. El nodo de KNIME utilizado se basa en Newman y col.12, con un esquema de muestreo LDA disperso y una estructura de datos según Yao y col.13. Utilizamos la biblioteca de modelado de temas de Machine Learning for Language Toolkit (MALLET). Las opciones elegidas fueron: semillas -1593552080; número de temas 5; número de palabras en un tema: 10; alfa: 0.1; beta: 0.01; iteraciones: 1000.
Las publicaciones comprendidas entre los años 2000 y 2020 inclusive se utilizaron para generar una predicción hasta 2025. Se empleó la versión AAA del algoritmo de suavizado exponencial, incluyendo un intervalo de confianza. El resultado de la previsión se validó comparando los datos previstos de 2000 hasta 2020 con los datos reales existentes.
Resultados
El objetivo de nuestro trabajo fue utilizar la minería de texto para analizar la vasta colección de publicaciones científicas referidas al SUH en el repositorio ePMC, trans formando el texto publicado -utilizado para representar el lenguaje y el conocimiento explícito- en datos que genera ran información adicional implícita. Las herramientas que facilitan la recuperación y articulación de la información digital son necesarias y útiles porque permiten integrar “fragmentos de conocimiento” en modelos que ayudan a gestionar la complejidad de los datos y, adicionalmente, aportan a la reducción de los costos de prevención y terapia14 de diferentes enfermedades.
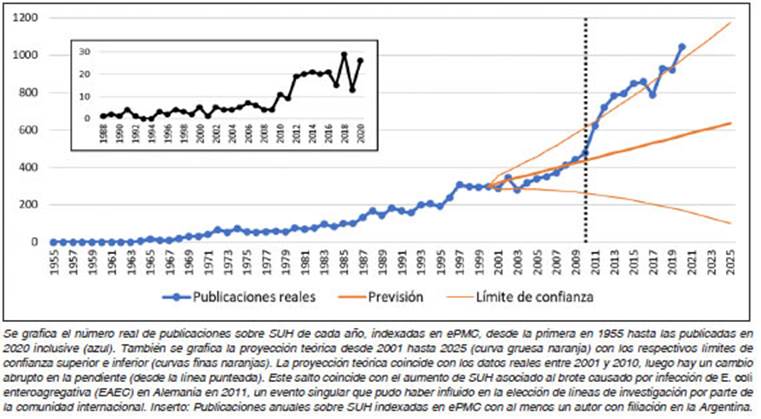
Analizamos 16 192 artículos en relación con su año de publicación, desde el primero aparecido en 1955 hasta los publicados en 2020 inclusive. No se encontraron registros de 1956 a 1960 ni de 1962 a 1963. Cuando fue necesario, la información se organizó en doce grupos de cinco años cada uno (de 1956-1960 a 2016-2020). El porcentaje de publicaciones de SUH conteniendo resúmenes fue incre mentándose con los años en ePMC (datos no mostrados). En nuestra búsqueda se detectaron y consideraron un total de 13 008 resúmenes. En nuestro conjunto de datos, el 78% de las publicaciones contiene resumen.
El estudio temporal de las publicaciones sobre SUH se muestra en la Figura 1, donde también se observa una proyección teórica a partir de 2001.
De mayo a julio de 2011, hubo en Alemania un brote significativo de una enfermedad transmitida por alimentos contaminados, caracterizada por diarrea sanguinolenta y con alta frecuencia de derivación a SUH. El brote se asoció inicialmente con infección por Escherichia coli enterohemorrágica (EHEC) del serotipo O104:H415. Más tarde se demostró que la causa del brote fue una cepa enteroagregativa de E. coli (EAEC) que había adquirido los genes productores de toxina Shiga tipo 2 (Stx2), un factor de virulencia ampliamente reconocido como el res ponsable más importante de SUH16. Como puede verse en la Figura 1, las curvas de publicaciones reales (azul) y esperadas (naranja) divergen claramente para los años posteriores a 2010. Probablemente esta divergencia está relacionada con el interés y la necesidad de la comunidad científica de aportar investigaciones para la resolución de una situación de emergencia puntual. En otra escala, pero en la misma dirección, un aumento inesperado de publicaciones científicas sobre coronavirus, el agente causal de la actual pandemia -otro evento singular y con fortísimas implicancias en la salud mundial- ocurre desde principios de 2020.
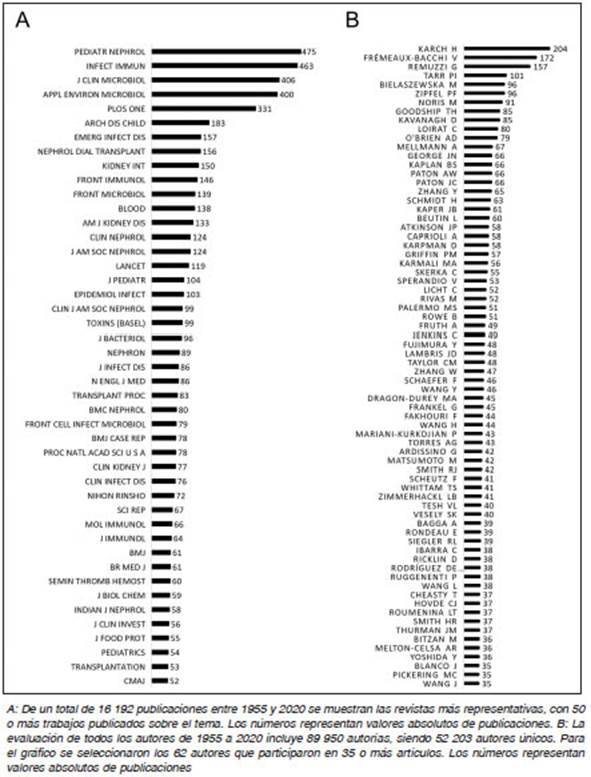
La Figura 2A muestra las revistas más representativas, con al menos 50 publicaciones sobre el SUH entre 1955 y 2020 inclusive. La revista Pediatric Nephrology enca beza el registro, luego Infection and Immunity, Journal of Clinical Microbiology y Apllied and Environmental Mi crobiology, todas estas con al menos 400 publicaciones sobre el tema.
Respecto de las autorías de las publicaciones, los cambios en las reglas en ePMC para especificar el nú mero de autores en cada artículo desde 1955 hasta 2020 presentaron un obstáculo para un análisis uniforme. Otro inconveniente para el análisis es la variedad de formas para mencionar al mismo autor. Por ejemplo, un mismo autor aparece citado a veces con un único apellido, otras con dos, o con la o las iniciales de sus nombres. Para nuestro análisis, se consideró cada variación como una entidad diferente, y los homónimos no se distinguieron. Esto indudablemente provoca un error en el recuento, aunque no significativo en términos del análisis total. Identificamos así 89 850 autorías -de las cuales 52 203 son autores únicos-. Los resultados se muestran en la Figura 2B. Helge Karch, Veronique Frémeaux-Bacchi, Giuseppe Remuzzi y Phillip I. Tarr son los únicos autores con más de 100 artículos publicados en SUH.
El análisis de autores se completó con las relaciones de colaboración científica entre ellos (Fig. 3). Se usó el software VOSviewer para construir un gráfico de redes de los 62 autores que publicaron 30 o más artículos sobre el tema (comparar con la Fig. 2B). En la visualización de la red, cada color está determinado por el grupo al que pertenecen los autores (que resultaron agrupados en 9 clústeres). El tamaño de letras y círculos es directamen te proporcional al peso de los elementos. Las líneas entre los autores representan 264 enlaces: cuanto mayor es el trabajo colaborativo, más cerca aparece un autor de otro en el mapa. Los vínculos más fuertes entre los elementos de cada clúster están representados por líneas más gruesas.
Aunque el lugar de trabajo de los autores suele incluir diferentes datos, nuestro interés se centró en el país donde se realizaron las investigaciones sobre SUH. Sin embargo, esta información no aparece en todas las pu blicaciones: a veces solo se detalla la región, la ciudad, o el nombre del laboratorio del autor; a veces ninguno de estos datos. Por lo tanto, la extracción de países se realizó automáticamente, seleccionando directamente el país cuando figuraba en la publicación, o indirectamente (fundamentalmente en el caso de EE.UU.), asociando el Estado mencionado con el país. De un conjunto de datos inicial con 16 192 títulos, se obtuvieron 54 250 filiaciones totales y 11 480 del primer autor.
Se reconocieron 139 países participantes. De ellos, solo 14 (EE.UU., Reino Unido, Alemania, Japón, Cana dá, Italia, Francia, China, India, Australia, Argentina, los Países Bajos, España y Brasil) contribuyeron con más de 150 publicaciones cada uno. De estos, solo tres (Ar gentina, Brasil y Australia) están en el hemisferio sur. La participación de estos 14 países se analizó agrupando los datos por períodos de 5 años (Fig. 4A). Como se observa, el número de publicaciones ha ido aumentando a lo largo de los años, y EE.UU. ocupa la primera posición entre los países con publicaciones sobre SUH, con un 32.5% de participación. Sin embargo, la Figura 4B muestra una disminución de la participación relativa de los EE.UU. (y también de Canadá), cuando se grafica la participación porcentual en relación con el total de publicaciones (con filiaciones explícitas) para cada lustro. Otros países han mantenido una participación porcentual constante a lo lar go del tiempo (Italia, Argentina, Países Bajos). Por último, se verificó un aumento de la participación de países como China, India, España y Brasil en estudios sobre SUH.
Ya fue mencionado el acontecimiento singular ocurrido en Alemania en 2011, una muestra dramática de la rapi dez con la que un agente infeccioso puede convertirse en una gran amenaza para la salud de un país17. Cabe señalar que el aumento de las publicaciones relacionadas con SUH en Alemania a partir del brote (Fig. 4A, flecha roja) puede atribuirse a la contribución de los equipos de investigación para comprender, superar y/o prevenir un fenómeno que afectaba la salud pública del país.
Si bien el objetivo de este trabajo es presentar la uti lización de las herramientas de minería de texto sobre el total de la bibliografía internacional sobre SUH indexada en ePMC, el análisis también puede focalizarse en las publicaciones de algún país en particular. Como ejemplo analizamos las publicaciones con autores que hayan especificado su filiación en la Argentina, en la misma base de datos en la que realizamos el análisis general y usando los mismos parámetros de búsqueda para hacer la minería de texto. Como ya se mencionó, no todas las publicaciones informan la filiación de los autores. Con estas consideraciones, esta parte del análisis se circuns cribió a las 272 publicaciones en las que se especifica el término “Argentina” en la filiación de alguno de sus auto res, dentro del período analizado. La primera publicación que cumple esas características es de 1988, habiendo un salto cuantitativo en el número de publicaciones a partir de 2010 y un pico en 2018 (inserto de Fig. 1 y Fig. 4). Las 272 publicaciones están firmadas por 1 883 autores, de los cuales no todos trabajan en instituciones argentinas, pero sí al menos uno de ellos. El número de autores únicos es de 968. Tres investigadoras argentinas firman 30 o más trabajos: Marta Rivas, Marina Palermo y Cristina Ibarra, quienes aparecen en la lista de autores que más han publicado mundialmente sobre el tema (Fig. 2B), además de formar parte de una red de colaboración entre ellas (Fig. 3). Respecto de las revistas en las que se publicaron estos 272 trabajos, Pediatric Nephrology (con 37), PLoS One (con 17), Frontiers in Cellular and Infection Microbiology (con 14) y Revista Argentina de Microbiología (con 13) son los medios más frecuentes.
Otro de los elementos que nos interesó estudiar fue la frecuencia del uso de las palabras y la evolución temporal en el uso de ciertos términos significativos en el SUH. Los resúmenes de 1955 a 2020 fueron seleccionados mediante un flujo de trabajo hecho en KNIME. Dado que las reglas del ingreso de datos en ePMC han cambiado a lo largo de los años, en algunos registros los resúmenes aparecen truncados y no completos. Se utilizó AntConc para contar la frecuencia de palabras, aplicando la lista de “palabras vacías” y una lista de lema incluyendo los términos propios del tema SUH. El corpus de resúmenes contenía 1 486 645 tokens y 52 277 types.
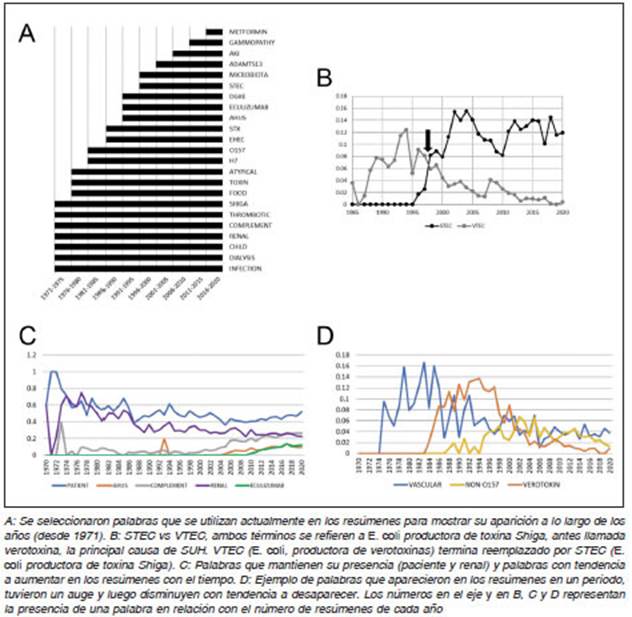
Se seleccionaron algunas palabras específicas que se emplean actualmente en la bibliografía, para detectar su año de aparición (Fig. 5A). A lo largo de los años apare cieron términos relacionados con la utilización de nuevos enfoques para el tratamiento del paciente, de nuevas técnicas experimentales y el uso de nuevos fármacos.
Un meticuloso análisis de los resúmenes muestra que el uso de algunas palabras sufrió cambios temporales abruptos al ser reemplazadas por un término alternativo, por acuerdo de la comunidad científica. Tal es el caso del par de términos VTEC (acrónimo de “verotoxigenic Escherichia coli”), que refiere a la citotoxicidad de STx sobre células Vero y STEC (acrónimo de “Shiga toxin-producing E.coli”); el segundo reemplazó al primero para nombrar la misma bacteria (Fig. 5B).
Relativizando el número de veces que una palabra aparece en los resúmenes con el número de resúmenes publicados en un período de tiempo, se pueden reconocer tres comportamientos temporales característicos: i) pala bras cuyo uso llega a una meseta en el tiempo; ii) palabras cuyo uso tiende a aumentar (Fig. 5C); iii) palabras que cada vez se utilizan menos, a pesar de que han tenido una presencia significativa en años anteriores (Fig. 5D). Los términos como paciente y renal son utilizados todo el tiempo por los autores; aSUH, complemento (elemento del sistema inmune innato relacionado con la causa de aSUH) y eculizumab (anticuerpo monoclonal humano quimérico contra la proteína C5 del complemento, utiliza do para tratar el aSUH), muestran curvas crecientes en el tiempo. Con diferentes puntos de aparición; vascular, no-O157 (en referencia a cepas que causan enfermedad en humanos y no son O157:H7, la cepa STEC más co múnmente identificada) y verotoxina (el primer nombre dado a la toxina Shiga), muestran decrecimiento en las curvas temporales.
Sobre el mismo corpus de resúmenes se llevó a cabo una detección no supervisada de temas, utilizando la Asignación Latente de Dirichlet (LDA), un modelo es tadístico generativo ampliamente aceptado, que puede aplicarse al procesamiento del lenguaje natural para el descubrimiento no supervisado de temas18,19. La detección o agrupamiento (clustering) automático y no supervisado de temas es una potente herramienta para analizar un corpus formado por todos los resúmenes de interés (que pueden contarse en cientos, miles o incluso millones de elementos según cada consulta), porque permite dividir la información discriminando en grupos (temas) elementos que se relacionan fuertemente entre sí (las palabras de un mismo tema). En el contexto de nuestra investigación, el nodo extractor de temas de KNIME hace uso del modelo para obtener tablas y nubes de palabras de los resúme nes, identificando temas y sus términos de mayor preva lencia. Esta discriminación temática puede verse tanto en la Tabla 1 como en la Figura 6. La Tabla 1 muestra los cinco temas con sus diez palabras correspondientes. La Figura 6 no solo muestra los distintos temas (uno por cada color), sino que da cuenta del peso de cada palabra dentro de un tema mediante la diferencia de tamaño de las letras. Como puede verse en la Tabla 1 y en la Figura 6, la detección no supervisada discrimina claramente entre los grupos SUH y aSUH y especifica los subgrupos temáticos dentro del grupo SUH (ver epígrafe de la Tabla 1). Des taca el predominio de la palabra paciente en algunos de los temas detectados, que se expresa con claridad en el tamaño de esta palabra respecto de las demás. Paciente es una de las palabras que, según se vio en el análisis mostrado en la Fig. 5C, sigue siendo muy usada en las publicaciones sobre SUH a lo largo de los años.

Tabla 1 Agrupamiento de palabras características detectado mediante aprendizaje automático no supervisado: se detallan las diez palabras correspondientes a cada uno de los cinco temas
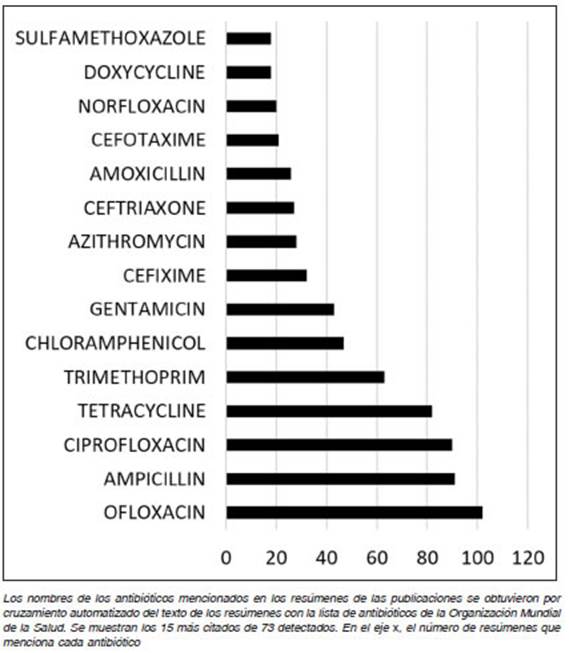
El cruzamiento automatizado de los resúmenes con otras bases de datos permite asimismo obtener infor mación específica de interés médico. Para mostrarlo cruzamos la información de nuestro corpus con la lista de antibióticos de la Organización Mundial de la Salud actua lizada a 202120. Los 15 antibióticos más mencionados en los resúmenes sobre SUH (de un total de 73 detectados) se muestran en la Figura 7. Este ejemplo muestra la po sibilidad de llevar a cabo un análisis cuantitativo de los intereses y elecciones de los autores de las publicaciones respecto de los diferentes tratamientos, y permite selec cionar con rapidez un conjunto de bibliografía determinada entre la gran cantidad de publicaciones existentes.
Discusión
El objetivo principal de este trabajo fue descubrir estruc turas y patrones de información no explícitos (a menudo ocultos) en los artículos científicos sobre SUH indexados en ePMC. Aunque en la literatura hay precedentes de minería de textos en temas relacionados con la salud21,22, nuestra indagación indica que esta es la primera vez que se presenta un estudio completo con esta herramienta aplicada al SUH.
Los datos analizados incluyeron todas las publica ciones indexadas sobre SUH, sin ningún sesgo pre establecido como factor de impacto, grupo de trabajo, países involucrados, lugar de publicación, u otros, dando objetividad al análisis llevado a cabo.
La minería de texto permitió relacionar datos previa mente dispersos y presentarlos de forma compacta y clara, lo que nos llevó a una comprensión más profunda de los descriptores y nos permitió detectar fluctuaciones y tendencias que pueden contribuir a mejorar la forma en que se previene, investiga y trata el síndrome. Esto se aplica tanto a los estudios a largo plazo, como a la toma de decisiones frente a eventos singulares (como el brote de 2011 en Alemania).
En resumen, este trabajo nos permite concluir que i) el número de publicaciones sobre SUH está aumentando, lo que indica que todavía hay muchos problemas por resolver y que aún no se ha llegado a una manera eficaz de erradicarlo, o de mejorar su detección temprana y su cura; ii) la comunidad científica internacional trabaja ar duamente tanto en el SUH como en el aSUH, proponiendo nuevos tratamientos y soluciones, de acuerdo con las características y el origen de cada uno; iii) más allá del número de publicaciones de cada autor, sin duda existe un importante trabajo colaborativo de la comunidad cien tífica, sumándose continuamente nuevos investigadores al estudio de SUH; iv) aunque EE.UU. lidera el número de publicaciones sobre el tema, se han interesado nue vos países y otros han aumentado su participación en el tema; v) la comunidad científica reacciona con prontitud a la aparición de eventos singulares que afectan a la salud de la población; vi) las palabras empleadas por los autores de las publicaciones explican los cambios en los enfoques adoptados, revelan éxitos e ideas abandonadas, y muestran colaboración y globalización del conocimiento, así como la aparición de tendencias temáticas.
En cuanto a la detección no supervisada de temas (Tabla 1 y Fig. 6), su utilidad es vasta. Entre otras cosas puede implementarse para un análisis imparcial de las decisiones que toman los autores, y un estudio de los posibles déficits de esas elecciones.
Respecto de las predicciones estadísticas, entende mos que deben tomarse con cuidado, porque no puede afirmarse la dirección futura de cualquier tema de in vestigación, especialmente debido a la posibilidad de la aparición de fenómenos emergentes que redireccionen las investigaciones científicas en uno o varios países. Basta el ejemplo de la enfermedad por Sars-CoV-2, CO VID-19, declarada pandemia en 2020 y que definió una crisis sanitaria global. Sin embargo, estas predicciones pueden ayudar a la toma de decisiones por parte de equipos científicos e instituciones de salud, aumentando la eficiencia en el uso de los recursos otorgados a los proyectos de investigación.
Aunque en el presente trabajo hicimos minería de texto sobre la base ePMC como fuente de datos, llegamos a la conclusión de que la metodología y las herramientas desarrolladas son fácilmente configurables para otros repositorios de literatura científica. Además, el flujo de trabajo propuesto para SUH puede aplicarse directamen te al estudio de diferentes enfermedades o cuestiones científicas.
Si bien se han publicado artículos que aplican la mi nería de textos no solo a los resúmenes sino a un corpus compuesto por el texto completo de las publicaciones23, y aun considerando que una mayor cantidad de datos en principio podría favorecer la riqueza del análisis, conside ramos que la información que los autores incluyen en los resúmenes de sus publicaciones contiene las principales ideas del trabajo, y resultan suficientes para hacer la minería con los objetivos que nos propusimos. Más aún, el cuerpo completo de la publicación suele contener in formación redundante, que distrae del centro del análisis, además de implicar un alto costo en términos de tiempo computacional, no siempre disponible para todos los equipos de investigación. El flujo de trabajo que presenta mos y la metodología que aplicamos permiten un análisis riguroso y fiable mediante computadoras accesibles.
Por último, la propuesta de utilizar la minería de textos en publicaciones científicas debe considerarse una contri bución para imaginar formas innovadoras de abordar los datos científicos, en un esfuerzo por mejorar los campos de la prevención, la investigación y el tratamiento de muchas enfermedades y otros aspectos relevantes en el campo de la salud humana mundial.