Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Archivos argentinos de pediatría
Print version ISSN 0325-0075On-line version ISSN 1668-3501
Arch. argent. pediatr. vol.115 no.4 Buenos Aires Aug. 2017
http://dx.doi.org/10.5546/aap.2017.e225
PRESENTACIÓN DE CASOS CLÍNICOS
http://dx.doi.org/10.5546/aap.2017.e225
Dos mutaciones novedosas en el gen de glicina descarboxilasa en un niño con hiperglicinemia no cetósica clásica: a propósito de un caso
Dr. Shu Liua, Dra. Zhiqing Wangb, Tec. Lab. Jinqun Lianga, Tec. Lab. Nuan Chena, Enf. Haimei OuYanga, Tec. Lab. Weihong Zenga, Tec. Lab. Liying Chena, Tec. Lab. Xunjie Xiea y Prof. Dr. Jianhui Jianga
a. Departamento de Trastornos Endocrinos y del Metabolismo en Niños, Hospital Materno Infantil de la Provincia de Cantón, Ciudad de Cantón, República Popular China.
b. Laboratorio Clave de Gastroenterología de la Provincia de Cantón, Departamento de Gastroenterología, Hospital de Nanfang, Universidad Médica del Sur, Ciudad de Cantón, República Popular China.
Correspondencia: Dr. Jianhui Jiang: cimedgd@163.com
Financiamiento: Ninguno.
Conflicto de intereses: Ninguno que declarar.
Recibido: 19-10-2016
Aceptado: 28-3-2017
RESUMEN
La hiperglicinemia no cetósica es una encefalopatía por glicina autosómica recesiva y hereditaria sumamente rara, causada por una deficiencia en el sistema enzimatico de división de la glicina mitocondrial, que provoca síntomas clínicos graves. La hiperglicinemia no cetósica se caracteriza por fenotipos diversos y complejos, por ejemplo, hipotonía, convulsiones, deterioro cognitivo, retrasos del desarrollo y espasmos mioclónicos que podrían causar apnea e incluso la muerte. En este artículo, presentamos el caso de un niño de 1 año con convulsiones mioclónicas, hipotonía y coma, con aumento de la concentración de glicina en el plasma y el líquido cefalorraquídeo y con un índice de glicina en líquido cefalorraquídeo/plasma de 0,24. Existen dos mutaciones heterocigotas novedosas que confirman el diagnóstico de hiperglicinemia no cetósica. Una es una mutación de aminoácido, c.2516A>G (p.Y839C), y la otra es una mutación en los sitios de corte y empalme, c.2457+2T>A, en el gen GLDC.
Palabras clave: Hiperglicinemia no cetósica; Gen GLDC; Mutaciones novedosas; Glicina descarboxilasa; Encefalopatía.
INTRODUCCIÓN
La hiperglicinemia no cetósica (HNC, OMIM 605899), también conocida como encefalopatía por glicina, es un trastorno autosómico recesivo devastador del metabolismo de la glicina.1,2 Puede provocar la acumulación de grandes cantidades de glicina en los líquidos corporales y también puede causar disfunción neurológica grave. Al igual que con muchas afecciones congénitas, este trastorno es resultado de una variedad de fenotipos. Sus variantes, denominadas neonatal, infantil, leve episódica, de inicio tardío y transitoria, se caracterizan por fenotipos heterogéneos, por ejemplo, convulsiones, trastornos de la conducta, déficits cognitivos y retrasos del desarrollo. En la mayoría de los pacientes se presenta la forma neonatal, que se manifiesta mediante encefalopatía por glicina en los primeros días de vida, junto con hipotonía, letargo y convulsiones resistentes al tratamiento. Algunos sobreviven y tendrán discapacidad intelectual en el futuro.3,4
La causa de la HNC es un defecto en el sistema enzimatico de división de la glicina mitocondrial,3,5 que se diagnostica bioquímica y molecularmente. Debe sospecharse HNC en los individuos con síntomas neurológicos acompañados de aumento de la concentración de glicina en la sangre y el líquido cefalorraquídeo (LCR). El aumento de la concentración de glicina en LCR (> 80 pmol/l) y un índice de glicina en LCR/plasma elevado (normal: < 0,02) indican inequívocamente hiperglicinemia no cetósica típica.5 El diagnóstico de HNC debe determinarse con la detección de la mutación en los genes del sistema enzimatico de división de la glicina. El sistema enzimatico de división de la glicina es un sistema mitocontrial multienzimatico formado por cuatro subinidades individuales, que incluyen tres componentes específicos: proteína P (glicina descarboxilasa), proteína T (aminometíl transferasa) y proteína H (proteína H del sistema enzimatico de división de la glicina), y una enzima constitutiva: la dihidrolipoamida deshidrogenasa, que son codificadas por los genes GLDC, AMT, GCSH y DLD, respectivamente.3,6,7 Las mutaciones en el gen GLDC son responsables por la mayoría de los casos de HNC.3 Por primera vez, se presenta el caso de un niño de 1 año con HNC neonatal heterocigoto para dos mutaciones novedosas en el gen GLDC.
Estudio de caso
Se trata de un paciente de 1 año de edad, con antecedentes de hipotonía y fasciculaciones frecuentes en las extremidades, nacido de padres no consanguíneos. El niño nació en otro hospital a las 40 semanas de gestación de una madre primípara de 25 años de edad tras un embarazo sin complicaciones y con movimientos fetales normales (peso al nacer: 3150 g). Se lo hospitalizó en la unidad de cuidados intensivos neonatales debido a alimentación deficiente y a disminución de la actividad al día de vida. Presentó letargo, hipotonía muscular generalizada, alimentación deficiente, ausencia de reflejos tendinosos profundos y retraso del desarrollo. A partir de los 3 meses de vida, se observaron convulsiones mioclónicas progresivas aproximadamente diez veces al día.
Los análisis de laboratorio habituales, entre otros, glucemia, amoníaco en suero, hemograma y coagulación, eran normales. En la resonancia magnética (RM) se observó displasia del cuerpo calloso. En el análisis del LCR se observó que el recuento de leucocitos y las concentraciones de proteínas y glucosa eran normales. El cultivo y el análisis viral del LCR fueron negativos. Las muestras de LCR se analizaron con el kit NeoBase Non-derivatized MS/MS kitT (3040-001Z) de Perkin Elmer, conforme al protocolo estándar. Se usó el sistema AB Sciex API 3200 LC/MS/MS para el análisis, y los datos se gestionaron con el programa ChemoviewT de Perkin Elmer. En el análisis metabólico se observó un aumento de la concentración de glicina plasmática a 947,8 μM/l (valor de referencia: 115-600 μM/l) y la concentración de glicina en el LCR alcanzó 226,4 μM/l (valor de referencia: 3-20 μM/l). El índice de glicina en LCR/plasma era 0,24 (normal: < 0,02). Por lo tanto, se probó el diagnóstico de hiperglicinemia no cetósica con el análisis de aminoácidos en LCR y plasma, junto con las manifestaciones clínicas típicas.
Se confirmó el diagnóstico mediante la secuenciación directa de todos los exones y los límites intrón-exón de los genes GLDC, GCSH y AMT, con ADN obtenido a partir de linfocitos periféricos. El niño es portador de dos mutaciones heterocigotas novedosas, c.2457+2T>A y c.2516A>G(Y839C), en los exones 20 y 21 del gen GLDC, heredadas de su padre y su madre, respectivamente (Figura 1 B1-B4). Hasta el momento, según las investigaciones realizadas, no se habían notificado previamente estas dos mutaciones.
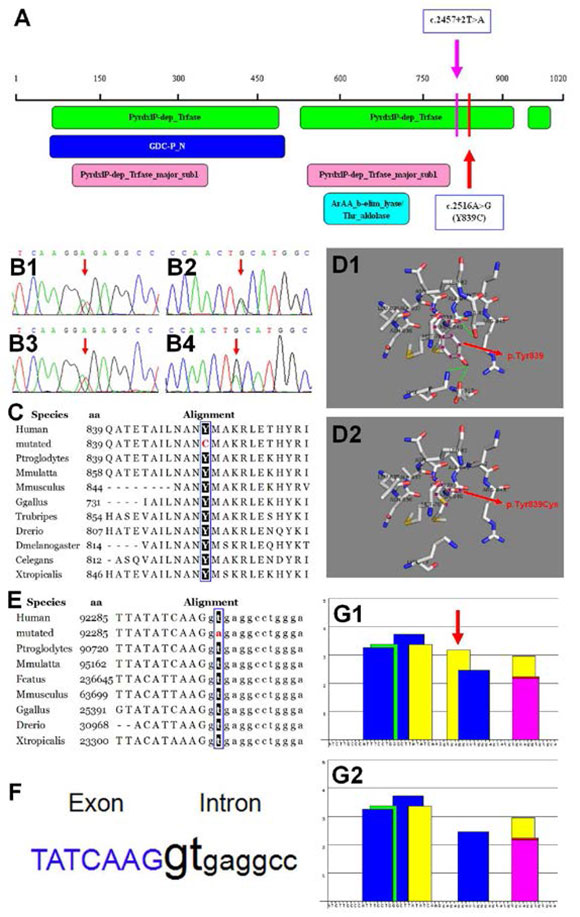
Figura 1. Identificación de las mutaciones c.2457+2T>A y c.2516A>G (Y839C) en los exones 20 y 21 del gen GLDC
(A) Representación gráfica del dominio proteico y la estructura del gen GLDC. Los recuadros de color verde, azul, rosado y turquesa, respectivamente, representan los dominios de la transferasa dependiente de piridoxal fosfato (IPR015424), la proteína P, terminal N del sistema enzimático de división de la glicina (IPR020580), la transferasa dependiente de piridoxal fosfato, región mayor, subdominio 1 (IPR015421) y la liasa de eliminación de beta-aminoácidos aromáticos/treonina aldolasa (IPR001597). Las mutaciones puntuales de c.2457+2T>A y c.2516A>G (Y839C) en el gen GLDC se muestran con líneas violetas y rojas, respectivamente.
(B) Cromatogramas de secuencia de las mutaciones en el gen GLDC. B1 y B2 representan, respectivamente, las secuencias mutadas de c.2457+2T>A y c.2516A>G en el niño, heredadas de su padre (B3) y su madre (B4). El sitio de la mutación se indica con flechas rojas.
(C) Comparación BLAST de la secuencia alrededor de los aminoácidos en la posición 839 en el gen ortólogo GLDC entre las diferentes especies. El recuadro y los caracteres indican el aminoácido altamente conservado entre las especies en las que se identificó p.Tyr839Cys.
(D1 y D2) Simulación computarizada de los aminoácidos y las estructuras alrededor de la posición 839 en el gen GLDC. Las estructuras animadas del tipo salvaje (D1) y el tipo mutante (D2) del gen GLDC muestran la mutagénesis que reemplaza la tirosina con cisteína en la posición 839 en el dominio transferasa dependiente de piridoxal fosfato de la glicina-eshidrogenasa. En D1, las líneas punteadas de color verde indican las interacciones estéricas alrededor de la tirosina en la posición 839, mientras que D2 indica la pérdida de las interacciones estéricas y la conformación del cambio en el dominio como resultado de la sustitución.
(E) Comparación BLAST de la secuencia alrededor del nucleótido c.2457+2 en el gen ortólogo GLDC entre las diferentes especies. El recuadro y los caracteres indican el aminoácido altamente conservado entre las especies en las que se identificó c.2457+2T>A.
(F) Diagrama de secuenciación de c.2457+2 que indica que la mutación se encontraba cercana al límite intrón-exón.
(G1 y G2) Predicción computarizada del efecto del cambio c.2457+2T>A en la composición de los potenciadores exónicos de corte y empalme. G1: La T en la posición +2 del intrón 20 del gen GLDC incluye un potenciador exónico de corte y empalme. G2: Cabe observar que la sustitución de T por A pierde el potenciador exónico de corte y empalme en el intrón 20 reconocido por la proteína SRSF6. La flecha roja indica la posición de la mutación.
El análisis bioinformático de estas dos mutaciones se hizo mediante SIFT, Mutation Taster, PolyPhen2, BDGP y ESE finder (Figura 1 A, C-G). Los resultados se indican a continuación: (1) c.2457+2T>A: Mutation Taster: causante de la enfermedad, prob: 1; ESE finder: la mutación conlleva a la pérdida del sitio emblemático; BDGP: las predicciones del sitio de corte y empalme indicaron que este sitio natural formó un sitio donante de corte y empalme alternativo, con un puntaje de 0,99. (2) c.2516A>G(Y839C): SIFT: perjudicial, con un puntaje de 0; Mutation Taster: causante de la enfermedad, prob: 0,999999999819036; PolyPhen2: probablemente perjudicial con un puntaje de 1,000 (valor máximo). Según los resultados del análisis bioinformático, se consideró inequívocamente que eran las mutaciones causantes de la enfermedad.
Hasta el momento, no existe un tratamiento eficaz para la HNC. El tratamiento actual consiste en reducir la concentración de glicina con benzoato y bloquear los efectos del exceso de glicina sobre los receptores de N-metil-D-aspartato (NMDA) con dextrometorfano.8 En este paciente, parte del tratamiento experimental incluye la administración por vía oral de dosis de 500 mg/kg/día de benzoato de sodio, 300 mg/día de piridoxina y 6 mg/kg/día de dextrometorfano. Entretanto, se inició la restricción alimentaria de proteínas naturales como método terapéutico suplementario, que era de 3 g/kg/día al momento del diagnóstico. Para lograr el efecto terapéutico deseado, se realizaron ajustes adicionales de la dosis sobre la base de la concentración real de glicina en la sangre y la respuesta del paciente. Después de un mes de tratamiento combinado, en la primera visita de seguimiento, la hipotonía muscular había mejorado levemente y la frecuencia de las convulsiones había disminuido a una o dos al día; además, la concentración de glicina en plasma cayó a 752 μM/l (valor de referencia: 115600 μM/l). Sobre la base de estos resultados, se inició el ajuste de la dosis de benzoato de sodio a 600 mg/kg/día y de dextrometorfano a 12 mg/kg/día. La concentración de glicina en plasma se normalizó (297 pmol/l, valor de referencia: 158302 pmol/l). La ingesta de proteínas naturales bajó a 2 g/kg/día después de aumentar la dosis de benzoato de sodio. Antes de que el niño dejara de asistir al seguimiento, después de seis meses de tratamiento, la concentración de glicina en plasma variaba entre 546 y 1132 μM/l (valor de referencia: 115-600 μM/l) debido al bajo cumplimiento del tratamiento, como interrumpir los medicamentos y llevar un control deficiente de la ingesta de proteínas. La última evaluación de seguimiento fue seis meses después de completar el tratamiento inicial (cuando el niño tenía 1 año y 7 meses); la dieta con restricción protéica y el tratamiento farmacológico mejoraron los reflejos tendinosos profundos activos y la hipotonía muscular, pero no había tenido efectos evidentes sobre la alimentación deficiente y el retraso mental.
DISCUSIÓN
La patogenia de la HNC es relevante para la acumulación de glicina en el sistema nervioso central como resultado de la deficiencia del sistema enzimatico de división de la glicina.9 Los síntomas se explican por la función neurotransmisora de la glicina. La glicina cumple una función inhibidora sobre receptores específicos en el tronco encefálico. Esta función inhibidora se realza cuando la concentración de glicina aumenta, lo que conduce a insuficiencia respiratoria y letargo. La glicina es también un coagonista obligatorio, junto con el glutamato, del receptor del NMDA excitador del glutamato en la corteza cerebral y el cerebelo, por lo que el exceso de glicina explicaría las convulsiones. La mutaciones en el gen GLDC son responsables por el 70% al 75% de los casos de HNC, mientras que las mutaciones en los genes AMT y GCSH representan solo el 20% y < 1% de los casos de HNC, respectivamente.3 Según las investigaciones recientes, en unos pocos pacientes con deficiencia enzimática no se identificó una mutación en el gen GLDC, AMT o GSH. Estos casos se deben a la deficiencia en la síntesis del ácido lipoico cofactor10,11 o a la disrupción del sistema de transporte de glicina, que está mediado por dos portadores dependientes de sodio, principalmente GLYT1 y GLYT2.12 En relación con el principal gen causante de la enfermedad, existen hasta el momento 214 variantes en el gen GLDC aunque no se hallaron puntos específicos donde se produzca la mutación. Las mutaciones de aminoácido representan más del 70% de las variantes; el resto son deleciones de mayor escala, inserciones/deleciones de menor escala y mutaciones que podrían afectar el sitio de corte y empalme (The Human Gene Mutation Database 2016).13
Se prevé que la mutación c.2457+2T>A en el exón 20 interrumpa el sitio de corte y empalme, y afecte el corte y empalme alternativo del ARNm. La mutación de aminoácido c.2516A>G en el exón 21 provoca el reemplazo de tirosina con cisteína en la posición 839 de la proteína P. No se han notificado estas dos mutaciones en la base de datos de mutaciones genéticas humanas (HGMD), la base de datos dbSNP ni PubMed. Según varios programas de bioinformática, es sumamente probable que estas dos mutaciones sean patogénicas. Dado que no se realizó un análisis funcional, se requieren más estudios y pacientes.
La identificación de dos mutaciones novedosas en el gen GLDC en este niño indica que la hiperglicinemia no cetósica es la causa más probable de las manifestaciones clínicas observadas en el paciente. Las dos mutaciones novedosas en el gen GLDC, asociadas con ausencia de actividad enzimática y aumento de la concentración de glicina, también se correlacionaban con el fenotipo grave presente en este niño. Asimismo, la verificación de estas dos mutaciones novedosas en los padres confirma su estado de portadores. La disponibilidad de un informe sobre estas mutaciones permitiría ofrecer un diagnóstico prenatal ante embarazos futuros.
1. Madu AE, Oliver L. Non-ketotic hyperglycinaemia: case report and review of medical literature. JMatern Fetal Neonatal Med 2013;26(5):537-9. [ Links ]
2. Beyoğlu D, Idle JR. The glycine deportation system and its pharmacological consequences. Pharmacol Ther 2012;135(2):151-67.
3. Van Hove J, Coughlin C II, Scharer G. Glycine Encephalopathy. En: Pagon RA, Adam MP, Ardinger HH, et al. GeneReviewsR [Internet]. Seattle (WA): University of Washington, Seattle; 1993-2017.
4. Hennermann JB, Berger JM, Grieben U, et al. Predicition of long-term outcome in glycine encephalopathy: a clinical survey. J Inherit Metab Dis 2012;35:253-61. [ Links ]
5. Wang W, Wu Z, Dai Z, et al. Glycine metabolism in animals and humans: implications for nutrition and health. Amino Acids 2013;45(3):463-77. [ Links ]
6. Coughlin CR 2nd, Swanson MA, Kronquist K, et al. The genetic basis of classic nonketotic hyperglycinemia due to mutations in GLDC and AMT. Genet Med 2017;19(1): 104-11. [ Links ]
7. Narisawa A, Komatsuzaki S, Kikuchi A, et al. Mutations in genes encoding the glycine cleavage system predispose to neural tube defects in mice and humans. Hum Mol Genet 20121;21(7):1496-503. [ Links ]
8. Bjoraker KJ, Swanson MA, Coughlin CR 2nd, et al. Neurodevelopmental Outcome and Treatment Efficacy of Benzoate and Dextromethorphan in Siblings with Attenuated Nonketotic Hyperglycinemia. J Pediatr 2016;170:234-9. [ Links ]
9. Love JM, Prosser D, Love DR, et al. A novel glycine decarboxylase gene mutation in an Indian family with nonketotic hyperglycinemia. J Child Neurol 2014;29(1):122-7. [ Links ]
10. Baker PR 2nd, Friederich MW, Swanson MA, et al. Variant non ketotic hyperglycinemia is caused by mutations in LIAS, BOLA3 and the novel gene GLRX5. Brain 2014;137 (Pt 2):366-79. [ Links ]
11. Mayr JA, Zimmermann FA, Fauth C, et al. Lipoic acid synthetase deficiency causes neonatal-onset epilepsy, defective mitochondrial energy metabolism, and glycine elevation. Am J Hum Genet 2011;89(6):792-7. [ Links ]
12. Alfadhel M, Nashabat M, Qahtani HA, et al. Mutation in SLC6A9 encoding a glycine transporter causes a novel form of non-ketotic hyperglycinemia in humans. Hum Genet 2016;135(11):1263-8. [ Links ]
13. The Human Gene Mutation Database at the Institute of Medical Genetics is Cardiff. 2016. Professional 2016.4. [Acceso: 29 de marzo de 2017] Disponible en: http://www.hgmd.cf.ac.uk/ac/index.php.














