Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Acta bioquímica clínica latinoamericana
versión impresa ISSN 0325-2957versión On-line ISSN 1851-6114
Acta bioquím. clín. latinoam. v.40 n.1 La Plata ene./mar. 2006
SECCIÓN PERMANENTE LATINOAMERICANA
Optimização da detecção do polimorfismo Ala 677 – Val do gene que codifica a metilenotetrahidrofolato redutase (MTHFR)*
Darlene Camati Persuhn1, Sandra Soares Melo2, Carlos Leonardo Maccagnan Pinheiro Besen3, Fabiano Sansão4, Iriane Eger Mangrich5
1. Professora dos Cursos de Farmácia e Medicina – UNIVALI.
2. Professora do Curso de Nutrição – UNIVALI.
3. Bolsista do ProBIC/UNIVALI – Aluno do Curso de Farmácia – UNIVALI.
4. Aluno do Curso de Farmácia – UNIVALI.
5. Professora do curso de Medicina – UNIVALI.
* Projeto executado no laboratório de Biologia Molecular do CCS/UNIVALI-Itajaí/SC.
Resumen
A mutação (C677T) no gene que codifica a enzima metilenotetrahidrofolato redutase (MTHFR) tem sido associada a hiperhomocisteinemia e possivelmente ao risco elevado para doenças vasculares. O objetivo deste trabalho foi otimizar sua detecção por PCR para faciliar estudos populacionais. Foram testadas metodologias alternativas de purificação do DNA genômico humano e os resultados obtidos permitiram padronizar uma técnica com redução de reagentes para extração de DNA por precipitação com NaCl. Foram realizados experimentos variando a concentração dos componentes da reação de PCR que permitiram definir que reações contendo metade do volume inicial de deoxinucleotídeos e iniciadores (primers), assim como 0,5U de Taq DNA polimerase produzem essencialmente a mesma quantidade de amplificado que a reação anteriormente utilizada. Testou-se ainda, a redução do volume da reação de PCR com objetivo de utilizar o produto de amplificação diretamente para digestão com a enzima HinfI, evitando assim, etapas de pipetagem que acarretam em risco de contaminação. Obteve-se sucesso com 15 mL de volume final; que permitiu a utilização do sistema de PCR completo para digestão do produto amplificado. A importância deste estudo pode ser justificada pela significância que as doenças vasculares apresentam em termos populacionais e por este polimorfismo estar potencialmente relacionado com o risco de DAC (doença artério-coronariana) precoce.
Palavras-chave: metilenotetrahidrofolato redutase * C677T * PCR
Summary
Optimization of Methylenetetrahydrofolate Reductase C677T polimorphism detection
A common mutation C677T in methylenetetrahydrofolate reductase (MTHFR) gene, involved in the metabolism of homocysteine, has been suggested to play a role in increasing cardiovascular disease risk. In order to facilitate the obtainment of population data, the objective of this work was improve its PCR detection. Leucocytes DNA purification alternative protocols were tested and a new low cost method of NaCl precipitation was developed. The composition of PCR mixture was also tested and it defined that the concentration of deoxynucleotides, primers and Taq DNA polimerase could be reduced in a half. The final volume of PCR mixture was reduced to 15 µL, enabling the utilization of the same tube to amplification and HinfI digestion processes, avoiding extra steps. The significance of cardiovascular disease in populational data and the possibility of involvement of this mutation as risk factor of precocious ACD (arterial cardiovascular disease) justify this work.
Key-words: methylenetetrahydrofolate reductase * C677T * PCR
INTRODUÇÃO
A hiperhomocisteinemia (elevação de homocisteína no plasma), tem sido reconhecida como um fator de risco para o desenvolvimento de doenças macrovasculares - doença cerebral, coronariana e arterial periférica (Munshi et al., 1996). As manisfestações clínicas da hiperhomocisteinemia grave incluem anormalidades neurológicas, retardo mental, episódios recorrentes de tromboembolismo e doenças vasculares na infância (Kang, 1996).
Existem vários fatores que influem nas concentrações de homocisteína. Suas concentrações aumentam gradativamente em indivíduos idosos quando comparados a indivíduos adultos de meia-idade, fato este que pode ser explicado pela diminuição da produção ou da atividade enzimática para o metabolismo da homocisteína, além da diminuição da biodisponibilidade de vitaminas como a B6 e a B12 ou folato, ou também mulheres com diminuição hormonal pós-menopausa podem explicar este fenômeno (FOLSON et al., 1998).
Várias formas de hiperhomocisteinemia são causadas por defeito(s) genéticos ou não genéticos, ou pela combinação de ambos fatores (Tabela 1).
O polimorfismo C677T no gene da MTHFR foi descrito em 1995 por Frosst e colaboradores. Estes mesmos autores demonstraram que a mutação reduziu a atividade enzimática da MTHFR e estava associada com concentrações plasmáticas elevadas de homocisteína. O papel desta mutação na predisposição à aterosclerose foi estudado posteriormente e o relato mostrou que houve uma maior freqüência do polimorfismo C677T dentre os 60 pacientes com aterosclerose prematura em relação ao grupo controle (KLUIJTMANS et al., 1997). Vários outros estudos envolvendo a avaliação da prevalência deste polimorfismo e seu envolvimento com predisposição a doenças foram realizados (Arai et al., 1997, Kluijtmans et al. 1997, RADY et al., 2002) e os resultados mostraram que existe grande variabilidade de ocorrência em diferentes grupos étnicos assim como exibiram discussões controversas relativas ao seu real envolvimento como fator de risco genético para doenças vasculares.
A metodologia para detecção do polimorfismo Ala 677 – Val do gene que codifica a enzima metilenotetrahidrofolato redutase já foi descrita (ARRUDA et al., 1997), no entanto, um dos principais fatores da restrição de sua utilização é o elevado custo dos reagentes e conseqüentemente do exame. Assim, padronização de uma técnica rápida e barata de extração do DNA e de amplificação do fragmento gene que codifica a enzima metilenotetrahidrofolato redutase é de grande interesse para a realização de triagens populacionais.
Tabela 1. Etiologia da hiperhomocisteinemia genética e não genética.

CASUÍSTICA E MÉTODOS
Seleção de Pacientes
Para a caracterização genotípica, participaram do estudo pacientes com Diabetes mellitus Tipo 2, assistidos pelo Programa de Diabetes e Hipertensão da Unidade de Saúde e Saneamento – Balneário Camboriú – SC. Amostras de DNA de pacientes com genótipos heterozigoto foram selecionados e utilizados no estudo de otimização da detecção do polimorfismo C677T.
Anteriormente a coleta de dados, todos os pacientes foram informados sobre a finalidade do estudo e sobre os procedimentos experimentais que participaram, sendo solicitado as suas assinaturas em um termo de consentimento pós-informação.
O presente trabalho foi aprovado pela Comissão de Ética para Pesquisas em Humanos da Universidade do Vale do Itajaí – SC.
Purificação de DNA de leucócitos humanos
Após centrifugação de 1,5 mL de sangue periférico coletado em presença de EDTA (3 minutos, 5.000 rpm), o tapete de leucócitos resultante foi recolhido em tubo plástico limpo de 1,5 mL. Às células foi adicionado 1,0 mL de tampão de lise I. A mistura resultante foi centrifugada por 5 minutos, 5.000 rpm, ressuspensa em 250 µL de tampão de liseII e 5 µL de SDS 20%. Após homogeinização em vortex, adicionou-se 100 µL de NaCl saturado. A mistura foi novamente homogeneizada em vortex e centrifugada por 3 minutos, 5.000 rpm. O sobrenadante resultante foi recolhido em tubo limpo. Adicionou-se 300 µL de isopropanol absoluto. O DNA foi centrifugado por 5 minutos, 5.000 rpm e lavado duas vezes com solução de etanol 75%. Após 12 h em repouso a temperatura ambiente, o DNA foi hidratado com 50 µL de água ultra pura autoclavada. Utilizamos 5 µL de amostra nas reações de amplificação (metodologia modificada a partir de Miller et al., 1988).
Tampão de Lise I
0,3 M de Sacarose (PM = 342,3)
10 mM Tris-HCl (pH = 7,5)
5 mM MgCl
Triton x-100 1%
Estocado em recipiente âmbar, sob refrigeração (4 ºC).
Tampão de Lise II
0,075 M NaCl
0,024 M Na-EDTA
pH = 8,0, ajustado com NaOH.
Estocado à temperatura ambiente.
Amplificação por PCR de um fragmento do gene que codifica a enzima MTHFR e que compreende o local da mutação C677T
Modificou-se a composição do sistema de PCR, tanto na concentração de reagentes quanto no volume final de reação.
A reação original utilizava 2 µL de solução de DNTPs (solução estoque 5 mmol/L), 1µL de cada primer (solução estoque 10 pmol/mL), 1,5 µL de solução de MgCl2 (solução estoque 50 mmol/L), 2,5 µL de tampão de Taq DNA Polimerase (fornecido pelo fabricante da enzima), 1U de Taq DNA Polimerase e 2,5 µL de DNA purificado conforme descrito por Miller e colaboradores (1988) num volume final de 25 µL. O sistema era incubado por 3 minutos a 94 ºC, seguido de 30 ciclos de incubação a 94 ºC (um minuto), 55 ºC (um minuto) e 72 ºC (dois minutos), além de uma etapa extra de extensão de 5 minutos. Para diagnosticar o polimorfismo C677T, o fragmento de 198 pb obtido necessita ser digerido com enzima HinfI. Oito microlitros do amplificado foi recolhido em novo tubo plástico ao qual foi adicionado tampão e enzima HinfI (1U) além de água suficiente para completar 15 µL. O sistema foi incubado a 37 °C por 12 horas e o produto analisado em gel de eletroforese em poliacrilamida 7% ou agarose 1,5%.
A fim de reduzir custos e etapas de pipetagem, testamos diversos sistemas alternativos até chegarmos ao que produziu resultados satisfatórios: 1 µL de solução de DNTPs (Gibco-BRL), 0,5 µL de cada primer (Gibco-BRL), 1,5 µL de solução de MgCl2, 1,5 µL de tampão de Taq DNA Polimerase (fornecido pelo fabricante da enzima), 0,5 U de Taq DNA Polimerase (Life Technologies) e 1,5 µL de DNA purificado conforme descrito num volume final de 15 µL. O sistema de incubação foi idêntico ao citado acima. Para a digestão com a enzima HinfI (Gibco-BRL), bastou adicionar ao tubo onde foi realizada a amplificação uma mistura contendo enzima, tampão e água totalizando um volume final de 20 µL.
RESULTADOS
A Figura 1 apresenta o resultado da amplificação do fragmento de 198 pb do gene da MTHFR utilizando o sistema com concentrações reduzidas de reagentes e volume final de 15 microlitros na presença de 1,0 ou 0,5 unidade de Taq DNA polimerase, mostrando que houve sucesso em ambas as reações de amplificação. Para testar a eficiência da digestão com a enzima HinfI, submetemos o material amplificado em volume final de 15 µL à digestão no mesmo tubo da amplificação conforme descrito na metodologia. Utilizamos somente DNA de pacientes previamente diagnosticados como heterozigotos para evidenciar o resultado, uma vez que no homozigoto normal, não ocorre digestão do fragmento de 198 pb, e no DNA portador de alelo alterado, o fragmento é digerido e gera duas bandas: 175 e 23 pares de base, conforme aparecem na Figura 2, mostrando o sucesso da reação e da digestão.
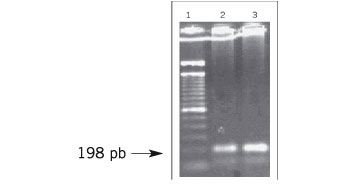
Figura 1. Teste de redução da concentração de Taq DNA polimerase As reações de amplificação do fragmento de 198 pb correspondente á região do gene da MTHFR que compreende o polimorfismo C677T foram conduzidas conforme descrito em Materiais e Métodos. O volume final das reações foi 15 µL, contendo 1,5 µL de DNA; 1,0 µL de DNTPs; 1,5 µL de MgCl2; 1,5 µL de tampão de Taq DNA Polimerase; 0,5 µL de primers (1 e 2); 0,5 U ou 1,0 U de Taq DNA polimerase. Linha 1: marcador de peso molecular (100 pb) Linha 2: 1,0 U de Taq DNA polimerase Linha 3: 0,5 U de Taq DNA polimerase
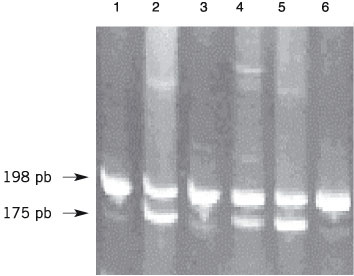
Figura 2. Teste de redução de volume e digestão com HinfI As reações de amplificação do fragmento de 198 pb correspondente á região do gene da MTHFR que compreende o polimorfismo C677T foram conduzidas conforme descrito em Materiais e Métodos. O volume final das reações foi 15 µL, contendo 1,5 µL de DNA; 1,0 µL de DNTPs; 1,5 µL de MgCl2; 1,5 µL de tamp
DISCUSSÃO
A técnica de purificação de DNA adaptada de Miller et al. (1988) mostrou-se efetiva. A vantagem desta em relação a original é a utilização de um volume reduzido de amostra e de reagentes.
A otimização da composição do sistema de PCR mostrou que é possível reduzir a quantidade de reagentes de alto custo da reação, inclusive da enzima Taq DNA polimerase mantendo a capacidade de amplificação e reduzindo os custos da análise. Além disso, os experimentos com volume final reduzido da reação de PCR permitiram economizar uma etapa de pipetagem e utilização de tubos plásticos para a digestão com a enzima HinfI. A visualização de duas bandas após digestão com a enzima na Figura 2, demonstra que o processo de identificação do polimorfismo não foi prejudicado pelas modificações efetuadas.
CONCLUSÃO
Através do protocolo estabelecido neste trabalho é possível realizar o dobro de análises de identificação do polimorfismo C677T da MTHFR com a mesma quantidade de reagentes, sem prejuízo na qualidade de interpretação.
Agradecimientos
Agradecemos ao programa de financiamento PROBIC/UNIVALI pela concessão de recursos para execução deste projeto.
Correspondencia
DARLENE CAMATI PERSUHN
Rua Uruguai, 458 Bloco 27, Sala 313
Laboratório de Biologia Molecular
CEP:88302-202 Itajaí -SC
E-mail: darlene@ccs.univali.br
Referências
1. Arai K, Yamasaki Y, Kajimoto Y, Watada H, Umayahara Y, Kodama M, et al. Association of methylenetetrahidrofolate reductase gene polymorphism with carotid arterial wall thickening and myocardial infarction in NIDDM. Diabetes 1997; 46: 2102-4.
2. Arruda VR, Zuben PM, Chiaparini LC, Annichino-Bizzacchi JM, Costa FF. The mutation Ala 677ÆVal in the methylene tetrahydrofolate reductase gene: a risk factor for arterial disease and venous thrombosis. Thromb Haemost 1997; 77(s): 818-21.
3. Folson AR, Nieto J, McGovern PG, Tsai MY, Malinow MR, Eckfeldt JH. Prospective study of coronary heart disease incidence in relation to fasting total homocysteine related genetic polymorphims and B vitamins. The atherosclerosis risk in communities (ARIC) study. Circulation 1998; 98: 204-10.
4. Frosst P, Blom HJ, Milos R, Goyette P, Sheppard CA, Matthews RG, et al. A candidate genetic risk factor for vascular disease: a commom mutation in methylenetetrahydrofolate reductase. Nat Genet 1995; 10: 111-3.
5. Kang S S. Treatment of hyperhomocyst(e)inemia: physiological basis. J Nutr 1996; 125: 1273S-5S.
6. Kluijtmans LAJ, kastelein JJP, Lindemans J, Boers GHJ, Heil SG, Bruschke AVG, Jukema JW, Heuvel LPWJ, Trijbels FJM, Boerma GJM, Verheugt FWA, Willwms F, Blom HJ. Thermolabile methylenetetrahydrofolate reductase in coronary artery disease. Circulation 1997; 90: 2573-7.
7. Miller SA, Dykes DD, Polesky HF. A simple salting out procedure for extracting DNA from nucleated cell. Nuc A Res 1988; 16: 1215.
8. Munshi MN, Stone A, Fink L, Fonseca V. Hyperhomocysteinemia following a methionine load in patients with non-insulin-dependent diabetes mellitus and macrovascular disease. Metabolism 1996; 45(1): 133-5.
9. Rady PL, Szucs S, Grady J, Hudnall SD, Kellner LH, Nitowsky H, Tyring SK, Refsum H, Ueland PM, nygard O & Vollset SE. Annu Rev Med 2002; 49: 31-62. [ Links ] [ Links ] [ Links ] [ Links ] [ Links ] [ Links ] [ Links ] [ Links ] [ Links ]














