Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Acta bioquímica clínica latinoamericana
versión impresa ISSN 0325-2957versión On-line ISSN 1851-6114
Acta bioquím. clín. latinoam. v.42 n.1 La Plata ene./mar. 2008
MICROBIOLOGÍA
Identificación y sensibilidad de bacilos gram-negativos
Evaluación del uso de un sistema automatizado directamente de los frascos de hemocultivo*
Identification and antimicrobial susceptibility of gram-negative bacilli
Evaluation of the use of automatic system directly from blood culture bottles
María Alejandra Blanco1, Lidia Casimir2, Horacio Angel Lopardo3
1. Bioquímica.
2. Médica.
3. Dr. en Ciencias Bioquímicas.
* Servicio de Microbiología. Hospital de Pediatría "Prof. Dr. Juan P. Grarrahan", Combate de los Pozos 1881, (1245) Ciudad Autónoma de Buenos Aires, Argentina.
Premio Federación Bioquímica 2007
Resumen
La rapidez en el informe de los hemocultivos es esencial en el manejo clínico-infectológico de pacientes con bacteriemia. En el presente estudio se evaluó la utilidad del sistema Vitek para la identificación y sensibilidad antimicrobiana partiendo de las botellas de hemocultivo. También se evaluó el método de sensibilidad antibiótica de difusión con discos partiendo directamente de las botellas de hemocultivo. En un período de un año se procesaron 101 botellas de hemocultivo que resultaron positivas con bacilos gram-negativos (71 aislamientos de enterobacterias y 26 de bacilos gram-negativos no fermentadores). Los resultados de identificación obtenidos partiendo de las botellas de hemocultivo se compararon con las pruebas manuales convencionales realizadas a partir de colonia aislada y los resultados de sensibilidad antibiótica, con el método de dilución en agar. El 86% de las enterobacterias fueron identificadas correctamente a nivel de especie y el 14% no fueron identificadas. El 92,3% de los bacilos gram-negativos no fermentadores fue identificado correctamente a nivel de especie, 3,8% fueron mal identificados y otros 3,8% no fueron identificados. La concordancia global en la sensibilidad fue del 92,7% para el sistema Vitek y del 89,9% para el método de difusión con discos. Se encontraron errores very major (0,1% y 0,4%), major (2,4% y 1,3%) y minor (7,6% y 5,5%) para difusión con discos y sistema Vitek respectivamente. La inoculación de las tarjetas Vitek a partir de las botellas de hemocultivo que contenían bacilos gram-negativos, otorgó resultados satisfactorios tanto de identificación como de sensibilidad antibiótica. El método de difusión con discos realizado a partir de las botellas de hemoculivos también resultó confiable, pero el sistema Vitek tiene la ventaja adicional de proveer resultados similares en tiempos menores.
Palabras clave: Identificación; Sensibilidad antimicrobiana; Hemocultivo; Vitek
Summary
Reducing the turnaround time of laboratory diagnosis of bacteremia by means of blood cultures is of paramount importance to perform an appropriate clinical management of septic patients. In the present study, the performance of Vitek system for identification and antimicrobial susceptibility and disk diffusion antibiotic susceptibility method, directly from positive blood culture bottles, were evaluated. During one year, 101 positive bottles with gram-negative bacilli (71 Enterobacteriaceae and 26 non-fermenting gram-negative rods) were selected at random, and identification and antimicrobial susceptibility were studied. Identification obtained using Vitek system directly from the bottles has been compared to conventional biochemical tests with previously isolated bacteria. In a similar way, results of antimicrobial susceptibility have been compared to those obtained by agar dilution method. A good identification at species level was obtained for 86% of Enterobacteriaceae while 14% could not be identified. Almost ninety three percent of non-fermenting gram-negative rods were correctly identified with 3.8% badly identified, and 3.8% not identified. Global concordance with the reference method in susceptibility was 92.7% for Vitek system and 89.9 for disk diffusion method. very major (0.1% and 0.4%), major (2.4% and 1.3%) and minor (7.6% and 5.5%) errors were found for the disk diffusion method and Vitek system respectively. Vitek results obtained from direct inoculation of blood culture bottles containing gram-negative bacilli were satisfactory for identification as well as for antimicrobial susceptibility. The disk diffusion method directly performed from the bottles also showed acceptable results. However the advantage of Vitek system was its ability to significantly reduce the turnaround time.
Key words: Identification; Antimicrobial susceptibility; Blood cultures; Vitek
INTRODUCCIÓN
La rapidez en el diagnóstico de bacteriemia disminuye significativamente la morbi-mortalidad especialmente en pacientes inmunodeficientes, también reduce los días de internación, racionaliza la administración empírica de antibióticos y por consiguiente los costos (1-3). Los sistemas automatizados permiten el monitoreo continuo y automático de las botellas de hemocultivo. Una vez que se detecta la positividad del hemocultivo, se realiza un subcultivo a medio sólido que debe incubarse entre 18 y 24 horas. A partir de las colonias aisladas que crecen en los medios sólidos, recién pueden efectuarse las pruebas de identificación y de sensibilidad antimicrobiana. La reducción de esos tiempos podría lograrse con el uso acoplado de otro sistema automatizado, que realice la identificación y las pruebas de sensibilidad directamente del desarrollo en medio líquido, sin necesidad de esperar el desarrollo en el subcultivo.
Los objetivos de este trabajo fueron:
1. Comparar la identificación de bacilos gram-negativos por un sistema automatizado (Vitek) directamente de las botellas de hemocultivo con la identificación determinada con el método convencional realizado a partir de colonias aisladas.
2. Comparar el método de difusión con discos con el método automatizado (Vitek), realizados directamente de botellas de hemocultivo en las que desarrollaron bacilos gram-negativos, en forma controlada con un estándar apropiado (dilución en agar) para determinar la sensibilidad a los antimicrobianos.
MATERIALES Y MÉTODOS
Este estudio fue realizado en el hospital en el período comprendido entre agosto de 2005 y julio de 2006. Todos los aislamientos fueron procesados por ambos métodos, en forma ciega y por distintos operadores (M.A.B y M.A.M).
Como método de referencia para la identificación se utilizaron las pruebas manuales convencionales realizadas a partir de colonias aisladas. Como método de referencia para la sensibilidad se realizó el método de dilución en agar a partir de colonias aisladas, según las recomendaciones del CLSI (4) (5). El medio de cultivo utilizado fue agar Mueller-Hinton y se emplearon las siguientes cepas de referencia: Escherichia coli ATCC 25922, Escherichia coli ATCC 35218 y Pseudomonas aeruginosa ATCC 27853. Básicamente se utilizaron concentraciones crecientes de los siguiente antibióticos: ampicilina (1-128 µg/mL), ampicilina/sulbactam (1-128 µg/mL), piperacilina (0,5-128 µg/mL), piperacilina/tazobactam (0,25-128 µg/mL), ceftacidima (0,5-128 µg/mL), cefotaxima (4-128 µg/mL), cefepime (0,5-128 µg/mL), imipenem (0,5-32 µg/mL), meropenem (0,125-32 µg/mL), gentamicina (0,25-32 µg/mL), amicacina (0,5-128 µg/mL) y ciprofloxacina (0,125-8 µg/mL).
Muestras ensayadas
Se incluyeron 101 botellas de hemocultivo, 59 aeróbicas y 42 anaeróbicas, en las que se observaron bacilos gram-negativos, pertenecientes a 77 pacientes. Para la identificación se excluyeron 4 botellas que resultaron polimicrobianas al analizar los subcultivos realizados en medios sólidos. Para las pruebas de sensibilidad se excluyeron 14 botellas: las 4 que resultaron polimicrobianas, 3 que fueron identificadas como Stenotrophomonas maltophilia, dado que el sistema Vitek no provee de resultados de sensibilidad para este germen, 3 que resultaron no identificadas por lo que el sistema no refiere resultados de sensibilidad antibiótica, una que resultó mal identificada como Sphingomonas paucimobilis, una de la que no se obtuvo resultados por los métodos directos de los frascos por escaso inóculo y 2 aislamientos que no resultaron viables al momento de realizar el método estándar (dilución en agar). Cada vez que el sistema automatizado para hemocultivos (Bact/Alert 3D) detectó la positividad de algún frasco se efectuó el correspondiente subcultivo y a través de la tinción de Gram se identificó el desarrollo de bacilos gram-negativos. Esas botellas se procesaron de la siguiente forma:
A) Para la identificación y sensibilidad antimicrobiana se utilizó el sistema Vitek, software versión 9.01 (VTK- R 9.01) (Laboratorio bioMérieux, Marcy l´Etoile, Francia). Se efectuó la inoculación directa de las tarjetas GNI y GNS-651. Se colocaron 5 mL de caldo de las botellas en tubos separadores (SST gel and clot Activator, Vacutainer BD), se centrifugaron a 3.000 rpm durante 10 min, se descartó el sobrenadante y posteriormente con el sedimento se inocularon las tarjetas GNI y GNS para identificación y pruebas de sensibilidad respectivamente siguiendo las instrucciones del fabricante.
B) Para la difusión con discos, directamente de las botellas de hemocultivos, se dispensaron una o dos gotas del caldo de los frascos sobre placas de agar Mueller-Hinton y se hisoparon en tres direcciones. Luego se colocaron los discos correspondientes y se incubaron a 35 ºC durante 18 a 24 horas. La interpretación se efectuó de acuerdo con las recomendaciones del CLSI (5).
C) Para la identificación manual convencional se utilizaron para enterobacterias las siguientes pruebas: agar hierro triple azúcar (TSI), agar citrato de Simmons, descarboxilación de lisina y ornitina, producción de ureasa y movilidad e indol en medio SIM. Para bacilos gram-negativos no fermentadores se utilizaron las pruebas de oxidasa, oxidación/fermentación de glucosa, movilidad en caldo, DNasa, desarrollo en agar Mac Conkey y/o API 20NE (bioMérieux).
Se compararon los tiempos de demora en la emisión de los resultados de identificación y sensibilidad por ambos métodos.
Análisis estadístico
Los resultados obtenidos por los métodos alternativos cuyas interpretaciones difirieron levemente con las del método de referencia [sensible (S) vs. intermedio (I), resistente (R) vs. I, I vs. R, ó I vs. S] fueron considerados errores minor. Los resultados que fueron sensibles por los métodos alternativos y resistentes por dilución en agar fueron interpretados como errores very major, y los resistentes por los métodos alternativos pero sensibles por dilución en agar, como errores major (6).
RESULTADOS
Identificación por inoculación directa de las tarjetas GNI
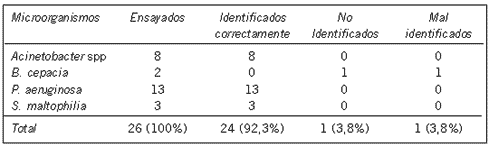
De las 97 botellas monomicrobianas que se analizaron, 71 aislamientos fueron enterobacterias y los 26 restantes fueron bacilos gram-negativos no fermentadores. Las enterobacterias halladas fueron: 2 Citrobacter freundii, 5 Enterobacter cloacae, 13 Escherichia coli, 40 Kebsiella spp, 1 Proteus spp, 2 Salmonella spp, 7 Serratia marcescens y 1 Shigella spp. Los bacilos gram-negativos no fermentadores hallados fueron: 8 Acinetobacter spp, 2 Burkolderia cepacia, 13 Pseudomonas aeruginosa y 3 Stenotrophomonas maltophilia. Sesenta y una de las enterobacterias (86%) fueron correctamente identificadas y 10 (14%) no fueron identificadas (Tabla I). De los 26 bacilos gram-negativos no fermentadores 24 (92,3%) fueron correctamente identificados, uno (3,8%) fue mal identificado como Sphingomonas paucimobilis y otro (3,8%) no fue identificado (Tabla II).
El análisis de las pruebas impresas positivas permitió identificar correctamente 8 de los 10 aislamientos de enterobacterias que habían resultado previamente no identificadas (2 Citrobacter freundii, 2 Salmonella spp y 4 Serratia spp). Las pruebas positivas de fermentación de glucosa, producción de sulfuro de hidrógeno, sorbitol y ONPG permitieron la identificación de Citrobacter freundii. Las pruebas positivas de fermentación de glucosa, producción de sulfuro de hidrógeno, sorbitol y ornitina permitieron la identificación de Salmonella spp. La resistencia a colistín además de las pruebas positivas de fermentación de glucosa, adonitol, ornitina y maltosa permitió la identificación de Serratia spp.
Tabla I. Comparación entre los resultados de identificación obtenidos por Vitek a partir de botellas de hemocultivo y la identificación bioquímica manual convencional de 71 aislamientos de enterobacterias.
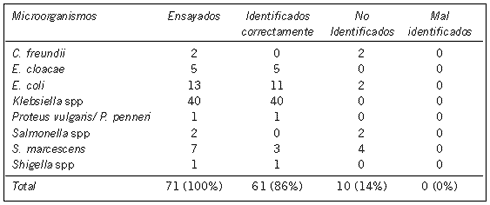
Tabla II. Comparación entre los resultados de identificación obtenidos por Vitek a partir de botellas de hemocultivo y la identificación bioquímica manual convencional y/o API 20 NE de 26 aislamientos de bacilos gram-negativos no fermentadores de glucosa.
Sensibilidad antimicrobiana realizada directamente de las botellas de hemocultivo
La concordancia global para los métodos de difusión con discos y Vitek comparados con el método de referencia fue 89% y 92,7% respectivamente.
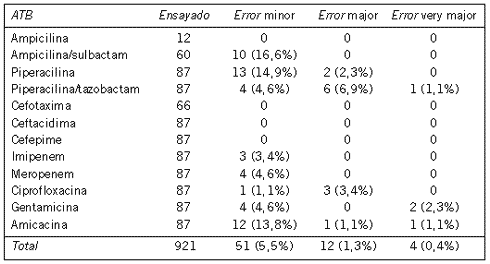
El porcentaje de error para cada droga ensayada por los métodos evaluados comparados con el método patrón (dilución en agar) se muestra en las Tablas III y IV. En la comparación de 12 antibióticos, se encontraron errores very major (0,1% y 0,4%), major (2,4% y 1,3%) y minor (7,6% y 5,5%) para difusión con discos y Vitek respectivamente. Con piperacilina, piperacilina/tazobactam, ampicilina/sulbactam y amicacina se observaron la mayor parte de los errores para ambos métodos y para difusión con discos se observó además un importante porcentaje de errores minor con ciprofloxacina.
Ambos métodos detectaron el total de las beta-lactamasas de espectro extendido presentes en enterobacterias (22 aislamientos de Klebsiella spp y 3 de Serratia marcescens). Hubo un caso de Klebsiella pneumoniae con probable carbapenemasa (Masuda positivo) (7) que fue intuida por ambos métodos por la resistencia a meropenem.
Si bien el sistema Vitek no identifica la producción de beta-lactamasas de espectro extendido en bacilos gram-negativos no fermentadores, dio pautas para alertar sobre su presencia por punto de corte.
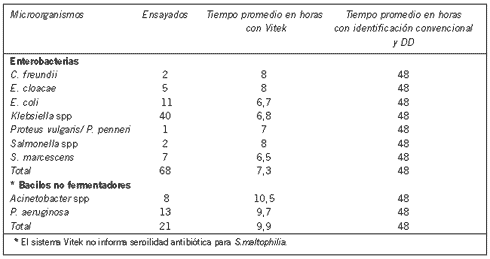
El tiempo promedio de demora en los informes de identificación y sensibilidad antimicrobiana por el sistema Vitek realizado directamente del caldo de las botellas y el método convencional para enterobacterias fue de 7,3 h y 48 h respectivamente y para los bacilos gram-negativos no fermentadores fue de 9,9 h y 48 h respectivamente (Tabla V).
Tabla III. Correlación de los ensayos de sensibilidad antimicrobiana realizados directamente de las botellas de hemocultivo con el método de difusión con discos y el método estándar (dilución en agar).
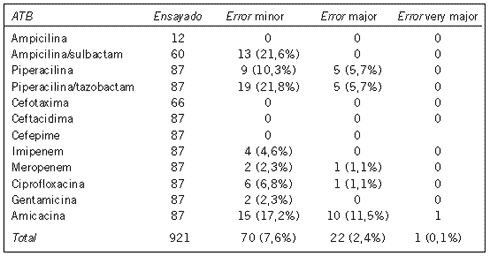
Tabla IV. Correlación de los ensayos de sensibilidad antimicrobiana realizados directamente de las botellas de hemocultivo con el Vitek y el método estándar (dilución en agar).
DISCUSIÓN Y CONCLUSIONES
Distintos estudios han evaluado el rendimiento del sistema Vitek 2 en la identificación y sensibilidad antimicrobiana partiendo directamente de las botellas de hemocultivo.
De Cueto et al. (8) estudiaron 50 bacilos gram-negativos y obtuvieron una correcta identificación en el 62% de los aislamientos, 28% no fueron identificados y 10% fueron mal identificados. Con respecto al análisis de sensibilidad antimicrobiana obtuvieron 2,4% de errores very major, 0,6% de errores major y 3,6% de errores minor.
Bruins et al. (9) estudiaron 344 bacilos gram-negativos (311 enterobacterias y 33 Pseudomonas aeruginosa), 93% fueron correctamente identificados, 2,6% fueron reportados por el sistema Vitek 2 como "de bajo poder discriminatorio" entre 2 especies distintas, 2,3% no fueron identificados y 2% fueron mal identificados. Con respecto a la sensibilidad antimicrobiana obtuvieron 0,1% de errores very major, 0,02% de errores major y 2,1% de errores minor.
Los resultados de este trabajo mostraron que el método Vitek directamente de las botellas de hemocultivo resulta confiable en la identificación de enterobacterias, Stenotrophomonas maltophilia, Acinetobacter spp y Pseudomonas aeruginosa. Los resultados de identificación correcta fueron mejores que los reportados por de Cueto et al. (8), 86% para las enterobacterias y 92,3% para los bacilos gram-negativos no fermentadores. Realizando un posterior análisis de las pruebas positivas impresas en enterobacterias, el porcentaje de identificación global correcta se acercó al 96%.
Al comparar los resultados de sensibilidad antimicrobiana obtenidos con el método de difusión con discos y Vitek directamente de las botellas, controlados con el método patrón (dilución en agar), se puede observar que ambos métodos resultan confiables para los bacilos gram-negativos ya que su concordancia con el método de referencia fue de aproximadamente 90% y los errores very major fueron 0,1% y 0,4% respectivamente, muy por debajo del límite de 3% considerado por Jorgensen como adecuado (10) e incluso inferiores a los mencionados por otros autores (1 y 1,5%) (11). No obstante, la suma de errores major y minor dieron cifras aceptables (menores al 7%) sólo para Vitek (6,8%) (10). Dicha suma para el método de difusión fue del 10%. Es de destacar que, sin embargo, este último método es potencialmente más apto que los de dilución para la detección de ciertos mecanismos de resistencia. La mayor parte de los errores se observaron con piperacilina, piperacilina/tazobactam, ampicilina/sulbactam y amicacina. No obstante, dada la aparición de nuevos mecanismos de resistencia (carbapenemasas, beta-lactamasas de espectro extendido de diferentes afinidades por los antibióticos, sensibilidad disminuida a fluoroquinolonas, etc.) (12)(13), estos resultados deberán seguir siendo validados en el tiempo y en las distintas zonas geográficas.
Según conocimiento de los autores este sería el primer trabajo que utilizó exitosamente un sistema Vitek, modelo anterior al Vitek 2 usado por los investigadores arriba mencionados, acoplado al estudio automatizado de hemocultivos.
Tabla V. Tiempos de demora en el informe de bacilos gram-negativos con Vitek realizado directamente de las botellas de hemocultivo y el método de identificación convencional acoplado a la difusión con discos (DD).
Agradecimientos
Los autores agradecen el excelente trabajo técnico realizado por Alejandra Mastroianni y el aporte de Química Erovne, sin el cual no se hubiera podido concretar este trabajo.
Correspondencia
DRA. MARÍA ALEJANDRA BLANCO
Gallo 1098, 14º Piso "A"
(1172) Ciudad Autónoma de Buenos Aires, Argentina
Tel: (+54)(011) 4963-1404/15-54722-2752
Referencias bibliográficas
1. Barenfanger J, Drake C, Kacich G. Clinical and financial benefits of rapid bacterial identification and antimicrobial susceptibility testing. J Clin Microbiol 1999; 37: 1415-8. [ Links ]
2. Doern GV, Vautor R, Gaudet M, Levy B. Clinical impact of rapid in vitro susceptibility testing and bacterial identification. J Clin Microbiol 1994; 32: 1757-62. [ Links ]
3. Jordá Vargas L, Vila A, Lanza A, Bonvehi P, Nazar J, Mikietuk A, et al. Utilidad del sistema Vitek en la identificación bacteriana y estudios de sensibilidad antimicrobiana. Acta Bioquím Clin Latinoam 2005; 39: 19-25. [ Links ]
4. Clinical and Laboratory Standards Institute. Methods for dilution antimicrobial susceptibility tests for bacteria that grow aerobically. Approved standard M7-A7, Clinical and Laboratory Standards Institute, Wayne PA, 2006. [ Links ]
5. Clinical and Laboratory Standards Institute. Performance standards for antimicrobial susceptibility testing, sixteenth informational supplement. Document M100-S16, Clinical and Laboratory Standards Institute, Wayne PA, 2006. [ Links ]
6. Barry AL. Surrogate disk for predicting cefotaxime and ceftriaxone susceptibilities of Streptococcus pneumoniae. J Clin Microbiol 1996; 34: 2609-12. [ Links ]
7. Masuda G, Tomioka S, Hasegawa M. Detection of b-lactamase production by gram negative bacteria. J Antibiot 1976; 29: 662-4. [ Links ]
8. De Cueto M, Ceballos E, Martinez L, Perea EJ, Pascual A. Use of positive blood cultures of direct identification and susceptibility testing with the Vitek 2 system. J Clin Microbiol 2004; 42: 3734-8. [ Links ]
9. Bruins MJ, Bloembergen P, Ruijs G, Wolfhagen M. Identification and susceptibility testing of Enterobacteriaceae and Pseudomonas aeruginosa by direct inoculation from positive BACTEC blood culture bottles into Vitek 2. J Clin Microbiol 2004; 42: 7-11. [ Links ]
10. Jorgensen JH. Selection criteria for an antimicrobial susceptibility testing system. J Clin Microbiol 1993; 31: 2841-4. [ Links ]
11. Metzler CM, Dehaan RM. Susceptibility test of anaerobic bacteria: statistical and clinical considerations. J Infect Dis 1974; 130: 588-94. [ Links ]
12. Robin F, Delmas J, Schweitzer C, Tournilhac O, Lesens O, Chanal C, et al. Evolution of TEM-type enzymes: biochemical and genetic characterization of two new complex mutant TEM enzymes, TEM-151 and TEM-152, from a single patient. Antimicrob Agents Chemother. 2007; 51: 1304-9. [ Links ]
13. Castanheira M, Pereira A, Nicoletti GA, Pignatari ACC, Barth AL, Gales AC. First report of plasmid-mediated qnrA1 in a ciprofloxacin-resistant Escherichia coli strain in Latin America. Antimicrob Agents Chemother 2007; 51: 1527-9. [ Links ]
Aceptado para su publicación el 12 de diciembre de 2007














