Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO  uBio
uBio
Share
Acta bioquímica clínica latinoamericana
Print version ISSN 0325-2957
Acta bioquím. clín. latinoam. vol.46 no.2 La Plata Apr./June 2012
MICROBIOLOGÍA
Caracterización genotípica de Staphylococcus aureus resistente a la meticilina prevalente en hospitales de Pakistán
Genotypic characterization of prevalent methicillin resistant Staphylococcus aureus in Pakistani community hospitals
Caracterização de Staphylococcus aureus resistente à meticilina prevalecente em hospitais da comunidade no Paquistão
Zahid Majeed1, Yasrab Arafat2, Zainab Ajab1, Salman Akbar Malik2, Waseem Shehzad Abbasi2, Huma Ajab1
1 M. Phil
2 PhD
* Department of Biochemistry, Faculty of Biological Sciences, Quaid-i-Azam University, Islamabad, Pakistan.
Resumen
El gen de la proteína A se usó como marcador genético para la caracterización de aislados de Staphylococcus aureus resistentes a la meticilina (SAMR). De un total de 130 aislados de Staphylococcus aureus, 90 fueron identificados como SAMR y 81 de éstos se pudieron caracterizar por tipificación spa. Todos estos aislados fueron obtenidos de cinco Hospitales Nacionales de la Comunidad. Se utilizaron dos juegos diferentes de cebadores para amplificar la región-X del gen de la proteína A en las cepas de SAMR. Un conjunto de cebadores, spa-F/spa-R ha identificado tres tipos de repeticiones diferentes, a saber, 7 repeticiones (spa 2), 8 repeticiones (spa 3) y 10 repeticiones (spa 4) y otro conjunto de cebadores, spa-1113F/spa-1514R ha identificado 4 tipos de repeticiones diferentes, a saber, 6 repeticiones (spa 1), 15 repeticiones (spa 6), y 17 repeticiones (spa 7) y 19 repeticiones (spa 8). Se identificó la repetición 11 (spa 5) con ambos conjuntos de cebadores. Los tipos de SAMR esporádicos que portaban las repeticiones 6, 7, 10, 17 y 19 fueron poco prevalentes mientras que los SAMR epidémicos con 8, 11, y 15 repeticiones fueron más prevalentes y se los consideró involucrados en la transmisión entre los pacientes dentro de los diferentes hospitales. Este trabajo concluye que la técnica spa es lo suficientemente eficiente como para diferenciar las cepas epidémicas, esporádicas y aquéllas que se transforman lentamente de esporádicas a epidémicas.
Palabras clave: Genotipo; Staphylococcus aureus; Proteína A; Tipificación spa; Polimorfismo
Summary
Protein A gene was used as a genetic marker for the characterization of Pakistani methicillin resistant Staphylococcus aureus (MRSA) isolates. Out of a total of 130 Staphylococcus aureus isolates, 90 were identified as MRSA and of these 90 MRSA, 81 MRSA isolates were characterized by spa typing. All of these isolates were collected from five National Community Hospitals. Two different sets of primers were used to amplify the X-region of Protein A gene in MRSA strains. One set of primers i.e, spa-F/spa-R identified three types of different repeats viz., 7 repeats (spa 2), 8 repeats (spa 3) and 10 repeats (spa 4) and another set of primers i.e. spa-1113F/spa-1514R identified 4 types of different repeats viz., 6 repeats (spa 1), 15 repeats (spa 6), and 17 repeats (spa 7) and 19 repeats (spa 8). Repeat 11 (spa 5) was identified with both sets of primers. Sporadic MRSA types carrying 6, 7, 10, 17 and 19 repeats were less prevalent, while the epidemic MRSA with 8, 11 and 15 repeats were more prevalent and considered to be involved in transmission among the patients within different hospitals. Research work concludes that spa technique is efficient enough to differentiate spa strains carrying variations in general and those slowly transforming from sporadic to epidemic outbreak in particular.
Keywords: Genotyping; Staphylococcus aureus; Protein A; Spa typing; Polymorphism
Resumo
O gene da proteína A foi usado como marcador genético para a caracterização de isolados de Staphylococcus aureus resistentes à meticilina (SAMR). De um total de 130 isolados de Staphylococcus aureus, 90 foram identificados como SAMR e 81 destes puderam se caracterizar por tipificação spa. Todos estes isolados foram obtidos de cinco Hospitais Nacionais da Comunidade. Utilizaram-se dois jogos diferentes de cevadores para amplificar a região-X do gene da proteína A nas cepas de SAMRUn conjunto de cevadores, spa-F/spa-R tem identificado três tipos de repetições diferentes, a saber, 7 repetições (spa 2), 8 repetições (spa 3) e 10 repetições (spa 4) e outro conjunto de cevadores, spa-1113F/spa-1514R tem identificado 4 tipos de repetições diferentes, a saber, 6 repetições (spa 1), 15 repetições (spa 6), e 17 repetições (spa 7) e 19 repetições (spa 8). Foi identificada a repetição 11 (spa 5) com ambos os conjuntos de cevadores. Os tipos de SAMR esporádicos que tinham as repetições 6, 7, 10, 17 e 19 foram pouco prevalecentes enquanto que os SAMR epidêmicos com 8, 11, e 15 repetições foram mais prevalecentes e são considerados envolvidos na transmissão entre os pacientes dentro dos diferentes hospitais. Este trabalho conclui que a técnica spa é o suficientemente eficiente como para diferenciar as cepas epidêmicas, esporádicas e aquelas que se transformam lentamente de esporádicas em epidêmicas.
Palavras chave: Genótipo; Staphylococcus aureus; Proteína A; Tipificação spa; Polimorfismo
Introducción
Staphylococcus aureus es uno de los agentes causales de enfermedades infecciosas humanas de distinta gravedad y frecuencia. A través de los años esta especie ha desarrollado resistencia a la meticilina lo que constituye un riesgo significativo para los pacientes y suma una importante carga financiera a los recursos destinados al cuidado de la salud (1), particularmente en los países subdesarrollados, entre ellos Pakistán. Un gran número de infecciones clínicas como la foliculitis, la forunculosis, la celulitis, la mastitis, el impétigo, la osteomielitis, la endocarditis aguda y la meningitis están ligados a S. aureus (2). La patogénesis de S. aureus ha sido atribuida a su capacidad de producir una variedad de proteínas. La proteína de superficie del estafilococo conocida como proteína A, de 40-60 kD, está codificada por el gen spa y está presente tanto en las cepas sensibles a la meticilina como en las resistentes (SAMR). Esta proteína protege a la bacteria de mecanismos de defensa del huésped (3).
El gen spa está compuesto por aproximadamente 2.150 bp y aloja un número de regiones con funciones diferentes (Figura 1) que incluyen la región ligando Fc-de la inmunoglobulina G (IgG), la región X y la C-terminal (4). La región ligando Fc IgG está compuesta por cinco repeticiones 160 bp. La región X contiene un número variable de repeticiones de 24 bp y está ubicada inmediatamente arriba de la región que codifica la secuencia de adhesión a la pared celular de C-terminal (5) (6).
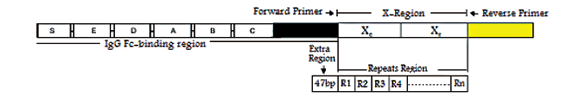
Figura 1. Mapa genético de la proteína A (23) (25). Reproducido con algunas modificaciones.
La tipificación es un prerrequisito importante de las medidas de control para los microorganismos clínicamente importantes. Hay muchas técnicas de genotipificación estandarizadas para identificar las cepas de S. aureus geográficamente aisladas y bien definidas. Éstas son: electroforesis en gel de campos pulsados (PFGE), electroforesis de enzimas multilocus (MLEE), y tipificación de secuencia multilocus (MLST), tipificación del gen coagulasa A (coa) y técnicas de microarreglos. En comparación con estas técnicas, la tipificación spa es un marcador de locus único para la identificación rápida de las cepas de S. aureus en general. La región X de la proteína A es un marcador de locus único que se utiliza en la tipificación spa. Esta tiene la ventaja de que sus datos pueden ser fácilmente comparados en todo el mundo por medio del software en línea Ridom Staph Type (7).
En Pakistán es muy escasa la literatura publicada sobre las cepas locales de SAMR. Los trabajos previos (8-13) son incompletos y no efectuaron técnicas de epidemiología molecular de SAMR que los haga clínicamente importantes. La no disponibilidad de las cepas pretipificadas, su depósito y acceso son los problemas más importantes que exhiben.
La aplicación de varias nuevas técnicas de tipificación molecular proporcionó una comprensión más minuciosa de la epidemiología y de la estructura poblacional de S. aureus en regiones del mundo geográficamente aisladas. En el entorno clínico, la terapia con drogas de rutina requiere la identificación exacta, rápida y a bajo costo, de los principales patógenos bacterianos. En el presente estudio se ha realizado una caracterización genotípica de SAMR en los hospitales de la comunidad pakistaníes y se presenta un análisis de los tipos spa.
Materiales y Métodos
MUESTRAS MRSA
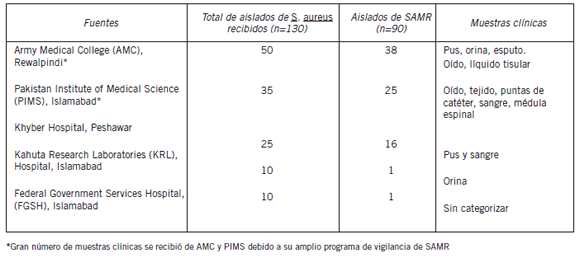
De un total de 130 cepas de Staphylococcus aureus, 90 fueron identificadas como SAMR y de éstas se han podido caracterizar 81 por tipificación spa. Se recolectaron muestras de S. aureus de cinco hospitales comunitarios diferentes durante el período 2007-2009. En la Tabla I se muestran los detalles del número de aislados de S. aureus disponibles de cada hospital. La cepa control NCTC 11561 provenía del Army Medical College (AMC), de Rawalpindi (13). Estas cepas se reconfirmaron por medio de pruebas bioquímicas y de sensibilidad antibiótica recomendadas. Se determinó la resistencia de S. aureus a la meticilina por medio del método de difusión con discos (14). Se reconocieron los aislados resistentes y sensibles en base a criterios estándares (14).
Tabla I. Aislados de SAMR obtenidos en diferentes hospitales.
En la Tabla III se presenta la visión detallada de un número de SAMR identificados con el origen de la infección.
Tabla III. Diferentes tipos spa de SAMR identificados a partir de distintas muestras clínicas

TINCIÓN DE GRAM Y PRUEBAS BIOQUÍMICAS
Se llevaron a cabo tinción de Gram y pruebas bioquímicas de acuerdo al National Standard Method desarrollado por la Health Protection Agency, UK (http://www.hpa-standardmethods.org.uk/).
EXTRACCIÓN DE ADN
Con algunas modificaciones, se adoptó el método de Japoni et al, para la extracción de ADN (15). Un cultivo bacteriano de 18 a 24 horas de incubación en caldo infusión cerebro corazón (BHI) (Oxoid, Inglaterra), se llevó a un tubo Eppendorf de 1,5 mL. El cultivo fue centrifugado a 14.000 rpm durante 10 minutos a 4 ºC. El sobrenadante se descartó y el sedimento se resuspendió en 467 µL de buffer Tris-EDTA. Se agregaron 30 µL de dodecil sulfato de sodio (SDS) al 10% y 3 µL de una solución de 20 µg/mL de proteinasa K (MB, Biochemicals Inc.). Se mezcló esta solución suavemente y se la incubó durante 1 hora a 37 ºC. Se agregó un volumen igual de fenol y cloroformo (1:1) y se lo mezcló muy suavemente para evitar el cizallamiento del ADN invirtiendo el tubo hasta que las dos fases se mezclaran completamente. Luego se lo hizo girar a 12.000 rpm durante 10 minutos. La fase acuosa superior se transfirió a un tubo Eppendorf nuevo. Una vez más, la fase acuosa se mezcló bien con un volumen igual de fenol y cloroformo (1:1), y se centrifugó a 14.000 rpm durante 10 minutos a 4 ºC. Nuevamente se pasó la fase acuosa a un tubo nuevo y se le agregaron 30 µL de acetato de sodio 3M. El ADN se precipitó con 99% de etanol frío seguido por 3-4 min de centrifugado a 10.000 rpm. El sedimento de ADN se suspendió en 50 µL de buffer Tris-EDTA (pH 8) y se almacenó a -20 ºC.
CUANTIFICACIÓN DE ADN
Se determinó la concentración de ADN (ng/µL) midiendo la absorbancia de la muestra a 260 nm. Cada muestra de ADN se diluyó 300 veces y se la cuantificó por medio de un espectrofotómetro UV (Serie 8453, Agilant, EE.UU.). El rango de longitud de onda de la absorbancia fue de 200 nm - 340 nm y se aplicó la siguiente fórmula para calcular la cantidad final de ADN en la muestra.
ADN (ng/µL) = [DO260 x factor de dilución × 50 × (260/280)]/180
CARACTERIZACIÓN DE LA PROTEÍNA A
En los SAMR, la región X del gen spa fue amplificada con dos conjuntos de cebadores específicos para spa, en primer lugar, spa 1113F 5'-TAAAGACGATCCTTCGGTGAGC-3', spa 1514R 5'-CAGCAGTAGTGCCGTTTG CTT-3' y en segundo lugar spa F 5'- AGCACCAAAAGAGGAAGACAA-3', spa R 5'-GTTTAACGACATGTACTCCGT-3' (16) (17). En la alícuota de 20 µL de la reacción de PCR simple se agregaron 2 µL de buffer para PCR de 10X, 2 µL de MgCl2 25 mM, 4 µL dNTPs 2 mM, 0,5 U de Taq polimerasa (5 U/µL) (MBI, Fermentas, EE.UU.), 2 µM de cebadores de 0,5 mM cada uno, 2 µL de la plantilla de ADN y 6,8 µL de ddH2O. La amplificación por PCR de la región X en el gen spa se llevó a cabo con Tpersonal Thermocycler (Biometra, Alemania). El protocolo incluyó una desnaturalización inicial a 94 ºC durante 5 min, seguida por 35 ciclos de desnaturalización a 94 ºC durante 30 segundos, anillado a 55 ºC durante 30 s, y extensión a 72 ºC durante 1 min, y extensión final a 72 ºC durante 10 min. Se utilizó la cepa NCTC-11561 como control positivo para S. aureus y la cepa EMRSA-15 como positiva para spa en la PCR. Como control negativo se usó una mezcla maestra sin plantilla de ADN.
ELECTROFORESIS EN GEL DE AGAROSA
Los productos de PCR se corrieron por electroforesis en 2% agarosa en presencia de bromuro de etidio (EtBr) en tampón 1X TBE (Trisborato-EDTA) durante 80 min y 120 voltios. Se incluyeron ADN ladder de 50 bp (Fermentas, EE.UU.) y 100 bp (Pomega) en cada corrida. Se visualizaron las bandas de ADN en un transiluminador UV y se las fotografió. Se evaluaron visualmente los patrones de bandas y se los analizó por medio del software Quantity One (versión 4,2, Bio Rad, Alemania). Las cepas de SAMR que mostraban una banda diferenciada entre 200 bp y 600 bp se consideraron como un tipo diferente de spa. El número de repeticiones se determinó tal como se lo describe en la próxima sección.
CÁLCULO DE NÚMERO VARIABLE DE REPETICIONES EN TÁNDEM
Los amplicones analizados en el gel se usaron para calcular el número de repeticiones variables en tándem (VNTRs) en la región X del gen spa. El amplicón de 263 bp contiene 9 repeticiones en la región X del gen spa en S. aureus (18). Se usó como amplicón de referencia para el cálculo de repeticiones en este estudio. En todas las muestras el número de repeticiones se calculó de acuerdo a los criterios dados más abajo.
. Tamaño de repeticiones simples en la región X = 24 bp
. Tamaño de nueve repeticiones en la región X = 24 bp x 9 repeticiones = 216 bp
. Tamaño de nueve repeticiones en la región X (18) = 263 bp
. Región extra 263 - 216 = 47 bp
. Tamaño del amplicón en este estudio = X
. Tamaño del amplicón usado para el cálculo de las repeticiones = X-47 = Y
. Número de repeticiones determinadas en las cepas de SAMR = Y/24 = R repeticiones
La Figura 1 muestra la región extra (47bp) y la región de repeticiones usada para el cálculo de número variable de repeticiones en tándem en la región-X amplificada del gen A de la proteína.
CRITERIOS DE INCLUSIÓN Y EXCLUSIÓN PARA LA TIPIFICACIÓN spa
Se establecieron criterios de inclusión y exclusión para la tipificación spa. Los cultivos puros de SAMR en los que se identificó el gen spa, fueron posteriormente subcultivados al menos tres veces para reconfirmarlo en términos del número de repeticiones, siguiendo los pasos mencionados anteriormente en materiales y métodos.
CRITERIOS DE DIFERENCIACION DE STAPHYLOCOCCUS AUREUS RESISTENTE A LA METICILINA EPIDÉMICO (SAMR-E) Y ESPORÁDICO (SAMR-S)
En este estudio, sumado al valor de corte propuesto anteriormente de 7 repeticiones (23), se han caracterizado los SAMR-E y el SAMR-S siguiendo estos criterios:
a. Si el tipo spa específico fuera epidémico, debería estar presente en más de un hospital a partir de una importante cantidad de muestras clínicas. La recolección de más de un hospital es válida para decir que el tipo de spa es epidémico.
b. Si la expansión del tipo spa estuviera limitada al interior de un solo hospital y en un número escaso de muestras de pacientes, se lo denominaría esporádico.
c. Si la cepa esporádica tuviera una importante dise
minación sólo en un hospital pero muy poca en cualquier otro, se podría pensar que se encontraría en un período de transformación de diseminación esporádica a epidémica.
ANÁLISIS ESTADÍSTICO
Prueba de chi cuadrado
Se aplicó la prueba de chi cuadrado a un nivel de significación del 95% y se evaluó la diferencia entre los tipos de spa (n=81). Se usó la siguiente fórmula y los valores se calcularon manualmente.
?2 = (o-e)2/e , donde o = valores observados y e = valor esperado
Agrupamiento jerárquico
Se analizaron las repeticiones numéricas usando el agrupamiento jerárquico UPGMA (unweighted pair group with arithmetic mean), versión 15 del Statistical Package for Social Sciences (SPSS). Se analizaron los agrupamientos a una distancia similar apropiada, representando los tipos spa. El tamaño del amplicón y sus respectivas repeticiones se usaron en el agrupamiento jerárquico para definir los diferentes tipos de grupos de SAMR.
Resultados
TIPIFICACIÓN
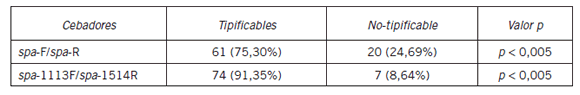
Se usaron dos grupos diferentes de cebadores para tipificar las cepas de SAMR Se halló que el cebador "spa-1113F/spa-1514R" tipificó 74 aislados de SAMR (91,35%) mientras que el cebador "spa-F y spa-R" tipificó 61 SAMR (75,30%) (Tabla II). Se observó una diferencia altamente significativa (prueba chi cuadrado, p <0,005) entre el número de SAMR tipificados y no tipificados. En este estudio, el gen spa mostró una alta variabilidad ya que se obtuvieron ocho spa diferentes sobre la base de su polimorfismo de región X. Se hallaron ocho productos diferentes de PCR que oscilaban entre 200 bp y 500 bp. El producto de PCR del cebador "spa-F/spa-R" mostró tres tipos claramente diferenciados de amplicones de 225 bp, 250 bp y 275 bp. Los productos de PCR del cebador "spa-1113/spa-1514R" mostraron cuatro tipos claramente diferenciados de amplicones de 200 bp, 400 bp, 450 bp y 500 bp. El producto de PCR de 300 bp se obtuvo con ambos cebadores. Se halló que la primera selección podría cambiar los criterios para la caracterización de la cepa.
Tabla II. Comparación de tipificación y no tipificación de SAMR por cebadores específicos spa.
En este estudio se observó que el cebador spa-F/spa-R podría usarse donde haya interés en identificar los tipos de SAMR con amplicones de 200, 225 bp y 275 bp, mientras que el cebador "spa-1113F/spa-1514R" es un marcador epidemiológicamente más conveniente para encontrar distintos tipos de SAMR con amplicones de 400 bp, 450 bp y 500 bp. Es evidente que los tipos de SAMR con 300 bp pueden ser tipificados por un cebador "spa-R/spa-F" o "spa-1113F/spa-1514R" (Figuras 2 y 3).
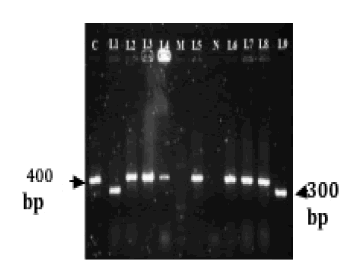
Figura 2. Electroforesis en gel de agarosa (3%) con dos tipos spa hallados con el Cebador "spa-1113F/spa-1514R" (M = 50 bp). El cebador "spa-1113F/spa-1514" mostró amplicones de 300 bp y 400 bp. Los SAMR que se muestran en la franja número 1 y 9 fueron llevadas al tamaño de 300 bp. Los SAMR en las franjas número 2 a 8 fueron llevados al tamaño 400 bp. La franja C tiene el control positivo de EMRSA -15 que se llevó al tamaño 400 bp. La franja N tiene control negativo.
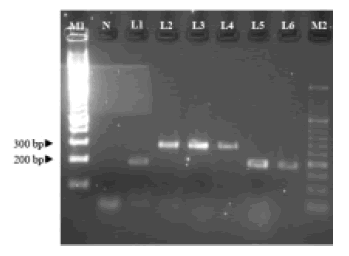
Figura 3. Electroforesis en gel de agarosa (3%) con dos tipos spa hallados con el cebador "spa-R/spa-F" (M1=100 bp, M2= 50 bp). El cebador "spa-R/spa-F" mostró los tipos SAMR con 200 bp y 300 bp. El SAMR en las franjas número 1, 5 y 6 fueron llevados al tamaño 200 bp. Las cepas SAMR en franjas 2, 3 y 4 fueron llevadas al tamaño 300 bp.
POLIMORFISMO ALÉLICO
En un tipo de SAMR en particular, el número de repeticiones se evaluó por la región X. Las repeticiones calculadas para ocho productos diferentes de PCR correspondieron a ocho tipos de polimorfismo alélico en el gen spa. El alelo con 6 repeticiones, llamado tipo spa 1 (n=2, 2,46%) y el alelo con 7 repeticiones llamado spa 2 (n=1, 1,23%). El tipo spa 1 estuvo asociado con muestras clínicas de orina y pus mientras que el tipo spa 2 estuvo relacionado solamente con las muestras clínicas de fractura ósea. Los tipos spa más importantes fueron spa 3, spa 5 y spa 6, ya que estos tipos se recuperaron de distintas muestras (Tabla III). El alelo con 8 repeticiones, llamado tipo spa 3 (n=25, 30, 86%), estuvo asociado con muestras clínicas de lavado broncoalveolar no dirigido (BAL), punta de catéter (CT), pus, líquido tisular, orina y esputo. El alelo con 11 repeticiones, llamado tipo spa 5 (n=36, 44,44%) estuvo relacionado con las muestras clínicas de orina, el sistema nervioso central (SNC), el oído y pus. El alelo con 15 repeticiones, llamado tipo spa 6 es el único tipo que se ha recogido de un número máximo de muestras clínicas como pus, SNC, fractura ósea, médula espinal, lavado bronqueo-alveolar, líquido tisular, esputo y orina. El alelo con 17 repeticiones, llamado tipo spa 7 (n = 2, 2,46%) estuvo asociado solamente con muestras clínicas de pus. El alelo con 19 repeticiones, llamado tipo spa 8 (n=4, 4,93%) se halló en muestras clínicas de pus, esputo y punta de catéter.
SAMR-E y SAMR-S
Hubo una diferencia altamente significativa (prueba chi cuadrado p <0,005) en el número de tipos spa; spa 3, spa 5 y spa 6 (Tabla III). Estos tipos spa fueron muy prevalentes. Hubo una diferencia no significativa (prueba chi cuadrado p <0,05) en el número de tipos spa; spa 1, spa 2, spa 4, spa 7 y spa 8. Se halló que estos últimos fueron menos prevalentes (Figura 4). La mayor prevalencia se observó para el tipo spa 6 (71,60%) mientras que la menor prevalencia se observó para el tipo spa 2 (1,23%). De acuerdo a los criterios epidemiológicos de los autores, se halló que los tipos spa; spa 3 (8 repeticiones), spa 5 (11 repeticiones) y spa 6 (15 repeticiones) se diseminaron como mínimo en 3 a 5 hospitales (AMC, PIMS, Khyber, KRL, FGSH) y el número de aislados para cada tipo spa fue también elevado. Estos tipos spa también poseían más de siete repeticiones. Por lo tanto, por su número de repeticiones junto con la dispersión temporal y espacial de los aislados en distintos hospitales se los consideró como SAMR-E. Los tipos spa SAMR-S: spa 1 (6 repeticiones), spa 2 (7 repeticiones) se diseminaron sólo dentro de su hospital de su origen (AMC). Estos tipos spa tenían también 7 o menos repeticiones en la región X. Los aislados que tenían más de 7 repeticiones fueron muy pocos: spa 4 (10 repeticiones) de PIMS, spa 7 (17 repeticiones) de AMC y spa 8 (19 repeticiones) de los hospitales AMC y PIMS. Su diseminación espacial-temporal estuvo limitada a un mínimo de uno hasta un máximo de dos hospitales con un número menor de aislados. Además, tuvieron una ocurrencia infrecuente (Tabla III). Estos últimos tres tipos spa fueron la excepción a la regla de = 7 para discriminar entre SAMR-S y SAMR-E.

Figura 4. Prevalencia (%) de los diferentes tipos spa de SAMR
Las muestras clínicas de pus incluyeron una mayor variedad de tipos spa comparados con otras muestras. Hubo siete tipos spa de SAMR que estuvieron particularmente asociados con las muestras clínicas de pus excepto spa 2. Los spa 1, 2, 4, 7 y 8, portadores de 6, 7, 10, 17 y 19 repeticiones respectivamente, se observaron en muy pocas ocasiones. Los tipos spa 3, 5 y 6 con 8, 11 y 15 repeticiones respectivamente, fueron más prevalentes en las diferentes muestras clínicas.
PREVALENCIA INTER E INTRA HOSPITALARIA
El cebador "spa-F/spa-R" mostró que los tipos spa 3 y spa 5 se hallaban en porcentajes de prevalencia de 62,50% y 37,50% respectivamente en el Hospital AMC de Rawalpindi. Estas dos cepas se hallaron también en el Hospital de Khyber en Peshawar pero el porcentaje de la prevalencia fue significativamente diferente. Además, el tipo spa 3 fue menos prevalente (26,66%) y el spa 5 fue altamente prevalente (73,33%). El spa 2 y el spa 4 fueron solamente detectados en el Hospital PIMS. El spa 2 fue solamente identificado en una muestra (5,00%) (Tabla IV).
Tabla IV. Tipos spa de SAMR (n = 61) tipificados con el cebador "spa-F/spa-R"

El SAMR tipificado con el cebador "spa-1113F/spa-1514R" mostró que el tipo spa 6 estuvo presente en todos los hospitales excepto en el FGSH, pero fue el tipo más prevalente tanto en el hospital AMC como en el PIMS. El spa 5 se observó en muy pocas muestras. Este tipo fue un poco más prevalente en el hospital PIMS (8,0%) que en el hospital AMC (3,22%). El spa 6 fue el tipo más prevalente detectado en los hospitales AMC, PIMS y Khyber con prevalencia de 80,64%, 88,00% y 68,75% respectivamente (Tabla V). El spa 1 estuvo sólo disponible en el hospital Khyber de Peshawar. Los tipos spa 2, spa 3, spa 4 pudieron identificarse con el cebador "spa-F/spa-R" mientras que los tipos spa 1, spa 6, spa 7 y spa 8 pudieron identificarse con el cebador "spa-1113F/spa-1514R". El spa 5 pudo identificarse por medio de cualquiera de los dos cebadores (Tabla IV y V).
Tabla V. Tipos spa de SAMR (n = 74) tipificados con el cebador "spa-1113F/spa-1514R".

El cebador "spa-F/spa-R" agrupó 61 aislados de SAMR tipificados en cuatro agrupamientos (Figura 5) utilizando el agrupamiento jerárquico en el valor de similitud de 14%. Del mismo modo, "spa-1113F/spa-1514" agrupó 74 MRSA tipificados en cinco agrupamientos distintivos representados por tipos spa utilizando el agrupamiento jerárquico a distancias similares de 10% (Figura 6).
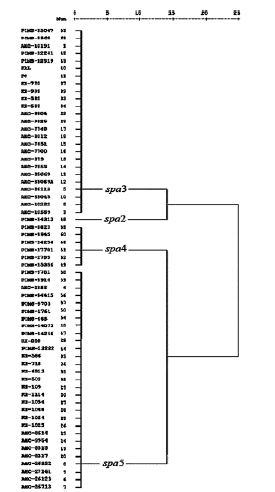
Figura 5. El dendrograma muestra los tipos spa basados en el número de repeticiones en la región X del gen spa con el cebador spa-F/spa-R.

Figura 6. El dendrograma muestra los tipos spa sobre la base del número de repeticiones en la región X del gen spa con el cebador "spa-1113F/spa-1514R".
Discusión y Conclusiones
TIPIFICACIÓN
El porcentaje de tipificación mostró que el cebador "spa-1113F/spa-1514R" tendía a ser más específico y poseía mayor fidelidad para la región X del gen spa que el otro cebador "spa-F/spa-R". Se halló que la probabilidad de un número mayor de tipos de SAMR podría lograrse cuando se usaba el primer cebador. La capacidad de tipificación del cebador "spa-1113F/spa-1514R" fue alta 91,35% (n =74) a diferencia de spa-F/spa-R" que fue 75,30% (n = 61). Estos resultados son similares a los de otro estudio en el que spa tipificó 29 (80,55%) de 36 cepas SAMR (21). Se observaron porcentajes de no tipificación de 8,64% y 24,69% para los cebadores "spa-1113F/spa-1514R" y "spa-F/spa-R" respectivamente. Se concibe que en aislados no tipificables, la región X de la proteína A podría estar ausente (20). La mutación del gen de la proteína A podría también anular la tipificación spa (22).
Un solo conjunto de cebadores ha sido frecuentemente usado para estudiar la tipificación spa (21) (23) (24) lo que hace que la comparación y discriminación del tipo spa a través de PCR sean ambiguas y subjetivas. Tal como se observó en estos estudios, diferentes cebadores amplificaron diferentes productos de PCR de distintos tamaños para la misma "región X". No obstante, podría ser beneficioso predecir de modo más confiable los diferentes tipos spa. Además, también es importante considerar que a veces un solo cebador puede no ser compatible debido a cambios en la conformación en la región del ADN específica donde realmente se va a ligar el cebador. Estos resultados podrían afectar los resultados de la tipificación.
Para poder comparar este trabajo con el resto de trabajos publicados, han sido calculados los tipos spa en base al tamaño del amplicón y se utilizó como referencia 263 bp, que consiste en 9 repeticiones tal como lo sugirieron otros autores (18). Se hallaron productos de PCR que oscilaban entre 200 bp y 500 bp. La amplificación por PCR de la región X del gen de la proteína A arrojó tamaños de amplicón que oscilaron entre 90±10 y 350±10 bp en aislados de S. aureus (25). En la tipificación spa (21) se observó un amplicón simple de 200 a 600 bp. Los productos de PCR oscilaron entre 250 bp y 637 bp en longitud para las distintas cantidades de repeticiones en la región X del gen spa (24). En este estudio, la diferencia en el tamaño del amplicón estuvo relacionada con la diferencia en las repeticiones, las que podían deberse a deleciones, mutaciones puntuales, duplicación o inserción de las repeticiones en la región X (16) (26).
POLIMORFISMO ALÉLICO
La tipificación spa mostró ocho tipos de polimorfismo alélico en la región X del gen spa. En un estudio, 12 tipos spa fueron clasificados sobre la base del tamaño del amplicón y se describieron de 2 a 13 repeticiones (25). En este estudio, los tipos que transportaban las repeticiones 6, 7, 8 y 11 concuerdan con los observados en estudios previos. Sin embargo, los tipos con 15, 17 y 19 repeticiones son únicos. Distintos estudios han descrito diferente número de repeticiones para diferentes tipos de cepas de SAMR. Un estudio informó 4 a 13 repeticiones en 142 aislados (27). Estas cepas de SAMR, tenían 4, 5, 7, 8, 9, 10 y 11 repeticiones en la región X y el tipo spa más común tuvo 10 repeticiones correspondientes a 556 bp (24). También observaron tipos con 2, 7, 17 y 21 repeticiones en algunas muestras. En este estudio, la cantidad de repeticiones calculadas para diferentes tipos spa por medio del análisis de los productos de PCR también está en concordancia con estos estudios.
En otro trabajo (18) se caracterizaron los tipos spa sobre la base del tamaño del amplicón y se los categorizó en cuatro tipos spa. Su estudio mostró que el tipo spa 1 (11 repeticiones, n = 92,72%) era el más común y estaba implicado en la mayoría de los brotes epidémicos. El tipo spa 2 (10 repeticiones, 20,16%) estuvo relacionado con casos raros de brotes en hospitales. Los tipos spa 3 (9 repeticiones, 14,11%) y spa 4 (5 repeticiones, 2,2%) se observaron sólo en casos esporádicos. En este estudio, el tipo más común fue el spa 6 (15 repeticiones, 71,60%), que fue altamente prevalente. Los tipos spa 3, spa 5 y spa 6 resultaron significativamente más prevalentes que spa 1, spa 2, spa 4, spa 7 y spa 8 (prueba de chi cuadrado p < 0,005). En este estudio, se identificaron siete tipos spa de muestras clínicas de pus. La alta prevalencia de los tipos spa asociados con el pus puede deberse al gran número de muestras de pus disponibles en los hospitales. Otra razón puede ser la preferencia de los médicos clínicos de tomar muestras de pus para el screening de SAMR.
ANÁLISIS DE SAMR-E. Y SAMR-S
La tipificación spa surgió como un marcador de locus simple para la tipificación de SAMR. Los tipos spa 3 (8 repeticiones), spa 5 (11 repeticiones) y spa 6 (15 repeticiones) y spa 1 (6 repeticiones), spa 2 (7 repeticiones) y spa 3 (8 repeticiones), spa 4 (11 repeticiones), spa 7 (17 repeticiones) y spa 8 (19 repeticiones) fueron SAMR-S. Se ha sugerido una regla para la discriminación entre SAMR-E y SAMR-S sobre la base del gen del polimorfismo de la proteína A (23). Hallaron que la presencia de siete o menos repeticiones en la región X tendía a ser esporádica mientras que más de siete repeticiones eran indicativas del carácter epidémico de una cepa de SAMR. Los aislados SAMR-E en Pakistán cumplieron con los criterios definidos en la sección de los materiales y métodos y estuvieron en concordancia con los estudios previos (19) (23). Todos los SAMR-E tenían más de siete repeticiones pero unos pocos SAMR-S no siguieron completamente los criterios de =7 repeticiones. Estos SAMR-S spa 4 (10 repeticiones), spa 7 (17 repeticiones) y spa 8 (15 repeticiones) fueron la excepción a la regla. Parecería que estos tipos son inherentemente SAMR-S y de algún modo debido a la duplicación o inserción en la región X han adquirido una cantidad mayor de repeticiones. Los aislados de SAMR con estos tipos spa mostraron una prevalencia epidemiológica menor entre las muestras clínicas, lo que indica que la adquisición de una región X más larga probablemente no esté correlacionada positivamente con la diseminación de la infección. Una región X más larga permite una exposición más favorable de las regiones ligando Fc a nivel de la superficie celular, facilita la infección y esto parece ser importante para la diseminación epidémica (23). Esto no se da para el SAMR-S transitorio, que tiene una región X más larga pero una diseminación restringida. Tener una región X más larga podría ser una ventaja para que SAMR-S se transforme en SAMR-E. Sin embargo este aspecto necesita ser más profundamente investigado. Estas consideraciones demandan atención y vigilancia del spa en el largo plazo.
La diferencia entre estos estudios y los anteriores (23) puede deberse al hecho de que no existen criterios exactos para clasificar SAMR en SAMR-E y SAMR-S de manera precisa. Aquí es importante considerar que el carácter epidémico y esporádico puede no sólo depender del número de repeticiones pero sí también de la fuente de transmisión inter/intra hospitalaria que puede originarse en el personal paramédico y en los pacientes que se trasladan de hospital a hospital. La forma con la que se puede restringir una diseminación epidémica de SAMR-S está ligada a condiciones higiénicas rigurosas.
Los loci múltiples en el cromosoma pueden contribuir al carácter epidémico y la región X del gen spa puede estar asociada con uno de estos loci. En un estudio anterior se halló que todos los brotes epidémicos correspondían a cepas con tipos spa que portaban 10 y 11 repeticiones mientras que los aislados spa con 5 ó 9 repeticiones eran sólo detectados en casos esporádicos (19). Un número mayor de repeticiones puede constituir una ventaja/selección natural para las cepas y ayudar a la diseminación epidémica de las mismas. Una región X más larga es importante para el carácter epidémico de la cepa (23).
PREVALENCIA DE LOS TIPOS CARACTERIZADOS EN LOS HOSPITALES PAKISTANÍES
La prevalencia inter e intrahospitalaria de diferentes tipos spa mostró que tanto spa 3 como spa 5 fueron cepas prevalentes en el Hospital AMC en Rawalpindi y el Hospital Khyber en Peshawar. El tipo spa 5 fue más prevalente en el hospital de Khyber vs. el hospital AMC (73,33% vs. 62,50%). Sin embargo, spa 3 fue más prevalente en el hospital AMC vs. el hospital Khyber (62,50% vs. 26,66%). Se mostró que la diseminación de spa 3 fue alta en el hospital AMC mientras que la diseminación de spa 5 fue alta en el hospital Khyber. Los tipos spa 2 y spa 4 solamente fueron detectados en el hospital PIMS, lo que sugiere que estos tipos podrían haber emergido recientemente en respuesta a los diferentes tratamientos con antibióticos que tuvieron los pacientes. PIMS es un hospital importante, que recibió pacientes de los cuatro distritos del país. En estos tipos de spa (spa 2 y spa 4), no debe descartarse la posibilidad de ocurrencia debida a nuevas internaciones de pacientes externos.
Spa tipo 5 se observó en unas pocas muestras pero fue más prevalente en el hospital PIMS (8,0%) que en el hospital AMC (3,22%) Spa tipo 6 transportaba 15 repeticiones en su región X y fue significativamente el tipo más prevalente detectado en los hospitales AMC, PIMS y Khyber. Este tipo spa fue aislado de la mayoría de las distintas muestras clínicas. Se podría considerar que spa 6 tiene más diversidad, lo que da lugar a más diseminación en los hospitales que se encuentran ubicados a cierta distancia uno del otro (hospital Khyber de Peshawar vs. otros hospitales). El spa tipo 1 sólo se encontró en el hospital Khyber de Peshawar. Este tipo mostró ser raro o un emergente reciente debido a algún motivo desconocido que merecería mayor investigación. Se ha concluido que el presente estudio ha aportado información confiable y básica acerca de la prevalencia epidemiológica de distintos tipos spa SAMR implicados en los hospitales de la comunidad. Además, la tipificación spa puede proporcionarle conocimientos adicionales al epidemiólogo del hospital, ya que estas técnicas son lo suficientemente eficientes para diferenciar las nuevas cepas spa que transportan variaciones en general y aquellas que lentamente se transforman de esporádicas a epidémicas.
CORRESPONDENCIA
ZAHID MAJEED C/O PROF. DR. SALMAN AKBAR MALIK, CHAIRMAN
Department of Biochemistry
Faculty of Biological Sciences
Quaid-i-Azam University, ISLAMABAD, Pakistan.
P.O Box No. 44000 Tel.: +92 51 90613012, +92 51 2601164
1. Kim T, Oh PI, Simor AE. The economic impact of methicillin-resistant Staphylococcus aureus in Canadian hospitals. Infect Control Hosp Epidemiol 2001; 22: 99-104. [ Links ]
2. Waldvogel FA. Staphylococcus aureus (including staphylococcal toxic shock). En: Mandell G, Bennett J, Dolin R, editors. In Principles and practice of infectious diseases, Mandell G, Bennett J, Dolin R, (ed) Philadelphia: Churchill Living Stone; 2000, p.p 2069-92. [ Links ]
3. National Standard Method. Health Protection Agency, UK (http://www.hpa-standardmethods.org.uk/). [ Links ]
4. Schneewind O, Model P, Fischetti VA. Sorting of protein A to the staphylococcal cell wall. Cell 1992; 70: 267-81. [ Links ]
5. Guss B, Uhlen M, Nilsson B, Lindberg M, Sjoquist J, Sjodahl J. Region X, et al. tThe cell-wall-attachment part of staphylococcal protein A. Eur J Biochem 1984; 138: 413-20. [ Links ]
6. Uhlen M, Guss B, Nilsson B, Gatenbeck S, Philipson L, Lindberg M, et al. Complete sequence of the staphylococcal gene encoding protein A. A gene evolved through multiple duplications. J Biol Chem 1984; 259: 1695-702. [ Links ]
7. Harmsen D, Claus H, Witte W, Rothganger J, Claus H, Turnwald D, et al. Typing of methicillin resistant Staphylococcus aureus in a university hospital setting by using novel software for spa repeat determination and database management. J Clin Microbiol 2003; 41: 5442-8. [ Links ]
8. Ashiq B, Tareen AK. Methicillin Resistant Staphylococcus aureus in a teaching hospital of Karachi: A Laboratory study. J Pak Med Assoc 1989; 39: 6-9. [ Links ]
9. Siddiqui GM, Karamat KA, Hanan A. Prevalence of Methicillin Resistant Staphylococcus aureus: A study at PAFHospital Sargodha. Pak J Pathol 1999; 10: 26-8. [ Links ]
10. Forsgren A, Nordstrom K, Philipson L, Sjoquist J. Protein A mutants of Staphylococcus aureus. J Bacteriol 1983; 107: 245-50. [ Links ]
11. Khatoon N, Bukhari MH, Riaz JR, Sheikh AS, Iqbal A, Naeem S, et al. Prevalence of Methicillin Resistant Staphylococcus aureus (MRSA) infection laboratory study at Mayo Hospital Lahore. Biomedica 2002; 18: 49-52. [ Links ]
12. Quereshi AH, Hanan A. The prevalence and susceptibility pattern of Methicillin Resistant Staphylococcus aureus. Pak Armed Forces Med J 2003; 53: 198-201. [ Links ]
13. Bukhari M, Iqbal A, Khatoon N, Iqbal N, Naeem S, Qureshi GR et al. A laboratory study of susceptibility of Methicillin resistant Staphylococcus aureus (MRSA). Pak J Med Sci 2004; 20: 229-33. [ Links ]
14. Ali AM, Abbasi SA, Ari S, Mirza IA.: Nosocomial infections due to methicillin resistant Staphylococcus aureus in hospitalized patients. Pak J Med Sci 2007; 23: 593-6. [ Links ]
15. Andrews JM. BSAC standardized disc susceptibility testing method (version 3). J Antimicrob Chemother 2004; 53: 713-28. [ Links ]
16. Japoni A, Alborzi A, Rasouli M, Pourabbas B. Modified DNA Extraction for rapid PCR detection of Methicillin resistant staphylococci. Iranian Biomedical J 2003; 8 (3): 161-5. [ Links ]
17. Strommenger BC, Kettlitz T, Weniger D, Harmsen A, Friedrich W, Witte W, et al. Assignment of Staphylococcus isolates to groups by spa typing, SmaImacrorestriction analysis, and multilocus sequence typing. J Clin Microbiol 2006; 44: 2533-40. [ Links ]
18. Sabat A, Krzyszton-Russjan J, Strzalka W, Filipek R, Kosowska K, Hryniewicz W, et al. New method for typing Staphylococcus aureus strains: multiple-locus variable-number tandem repeat analysis of polymorphism and genetic relationships of clinical isolates. J Clin Microbiol 2003; 41: 1801-4. [ Links ]
19. Montesinos I, Salido E, Delgado T, Cuervo M, Sierra1. Epidemiologic genotyping of methicillin-resistant Staphylococcus aureus by pulsed-field gel electrophoresis at a University Hospital and comparison with antibiotyping and potein A and coagulase gene polymorphism. J Clin Microbiol 2002; 40: 2119-25. [ Links ]
20. Frenay HM, Bunschoten AE, Schouls LM, van Leeuwen WJ, Vandenbroucke-Grauls CM, Verhoef J, et al. Molecular typing of methicillin-resistant Staphylococcus aureus on the basis of protein A gene polymorphism. Eur J Clin Microbiol 1996; 15: 60-4. [ Links ]
21. Hoefnagels-Schuermans A, Peetermans WE, Struelens MJ, Van Lierde S, Van Eldere J. Clonal analysis and identification of epidemic strains of methicillin-resistant Staphylococcus aureus by antibiotyping and determination of protein A gene and coagulase gene polymorphisms. J Clin Microbiol 1997; 35: 2514-20. [ Links ]
22. Koreen L, Ramaswamy SV, Graviss EA, Naidich S, Musser JM, Kreiswirth BN:. spa Typing method for Discrimination among Staphylococcus aureus isolates: Implications for use of a single marker to detect genetic micro-and macrovariation. J Clin Microbiol 2004; 42: 792-9. [ Links ]
23. Frenay HM, Theelen JP, Schouls LM, Vandenbroucke-Grauls CM, Verhoef J, van Leeuwen WJ, et al. Discrimination of epidemic and nonepidemic methicillin-resistant Staphylococcus aureus strains on the basis of protein A gene polymorphism. J Clin Microbiol 1994; 32: 846-7. [ Links ]
24. Frenay HM, Theelen JP, Schouls LM, Vandenbroucke-Grauls CM, Verhoef J, van Leeuwen WJ, et al. Discrimination of epidemic and nonepidemic methicillin-resistant Staphylococcus aureus strains on the basis of protein A gene polymorphism. J Clin Microbiol 1994; 32: 846-7. [ Links ]
25. Shopsin B, Gomez M, Montgomery SO, Smith DH, Waddington M, Dodge DE, et al. Evaluation of protein A gene polymorphic region DNA sequencing for typing of Staphylococcus aureus strains. J Clin Microbiol 1999; 39: 3556-63. [ Links ]
26. Vimercati C, Cremonesi P, Castiglioni B, Pisonil G, Boettcher PJ, Stella A, et al. Molecular typing of Staphylococcus aureus isolated from Cows, Goats and Sheep with intramammary infections on the basis of gene polymorphisms and toxins genes. J Vet Med 2006; 53: 423-8. [ Links ]
27. Brigido MDM, Barardi CR, Bonjardin CA, Santos CL, Junqueira ML, Brentani RR, et al. Nucleotide sequence of a variant protein A of Staphylococcus aureus suggests molecular heterogeneity among strains. J. Basic Microbiol 1991; 31: 337-45. [ Links ]
28. Kahl BC, Mellmann A, Deiwick S, Peters G, Dag Harmsen.: Variation of the polymorphic region X of the protein A gene during persistent airway infection of cystic fibrosis patients reflects two independent mechanisms of genetic change in Staphylococcus aureus. J Clin Microbiol 2005; 43: 502-5. [ Links ]
Aceptado para su publicación el 20 de marzo de 2012














