Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO  uBio
uBio
Share
Acta bioquímica clínica latinoamericana
Print version ISSN 0325-2957On-line version ISSN 1851-6114
Acta bioquím. clín. latinoam. vol.50 no.4 La Plata Dec. 2016
QUÍMICA BIOLÓGICA
Aislamiento y caracterización celular de exosomas de plasma para su uso como biomarcadores de diagnóstico
Isolation and cellular characterization of exosomes from plasma for their use as diagnostic biomarkers
Purificação e caracterização celular de exossomos para seu uso como biomarcadores de diagnóstico
María de Jesús Orozco-Romero1, Raúl Borja-Urby2, Martha Verónica Ponce-Castañeda3, Noé Valentín Durán-Figueroa4
1 Maestra en Ciencias en Bioprocesos.
2 Doctor en Ciencias en Óptica.
3 Doctora en Ciencias.
4 Doctor en Ciencias en Biotecnología.
a Instituto Politécnico Nacional, Unidad Profesional Interdisciplinaria de Biotecnología.
b Centro de Nanociencias y Micro y Nanotecnologías, Instituto Politécnico Nacional.
d Centro Médico Nacional Siglo XXI, Instituto Mexicano del Seguro Social.
CORRESPONDENCIA NOÉ VALENTÍN DURÁN-FIGUEROA Av. Acueducto de Guadalupe S/N, Gustavo A Madero, Barrio La Laguna Ticoman, 07340 CIUDAD DE MÉXICO, México D.F. E-mail: nduranf@ipn.mx
Resumen
Los exosomas son vesículas membranosas extracelulares esenciales en la comunicación intercelular a larga distancia, viajan en los fluidos corporales y entregan mensajes moleculares dirigidos a la mayoría de las células de todo el organismo. La liberación de mensajes vía exosomas ocurre en forma de ADN, ARN o proteínas; dicha liberación se ha asociado a diferentes condiciones fisiológicas normales y patológicas, como el cáncer. Por lo anterior, el aislamiento eficiente y caracterización celular de exosomas de plasma es clave para su uso como biomarcadores no invasivos de diversas enfermedades. En el presente estudio se purificaron exosomas a partir de muestras clínicas de plasmas de pacientes previamente diagnosticados con retinoblastoma y de individuos sanos como control. Los exosomas recuperados fueron caracterizados a nivel celular por microscopia electrónica de transmisión empleando una técnica de criogenia. Para demostrar la correcta purificación de exosomas se confirmó la presencia de las proteínas transmembranales CD63 y CD81 mediante immunoblot. Adicionalmente de los exosomas purificados, se identificaron ARNs pequeños no codificantes llamados microARNs. En general, se describe la purificación y caracterización celular de exosomas obtenidos de plasma humano para su potencial uso como biomarcadores.
Palabras clave: Biomarcadores; Exosomas; Retinoblastoma.
Summary
Exosomes are small extracellular vesicles essential in intercellular communication; they act as vehicles of broad scope. They are travelling in body fluids and delivering molecular messages to cells in the organism. Messages released by exosomes like DNA, RNA and proteins are associated with different pathological conditions including cancer. Therefore, the efficient isolation and cellular characterization of exosomes from plasma is essential to use them as biomarkers in many diseases. Here, exosomes were purified from patients diagnosed with pediatric cancer and healthy individuals as control. The exosomes recovered were characterized using cryogenic transmission electron microscopy. Moreover, the presence of CD63 and CD81 transmembrane proteins was confirmed using Western blot. Besides, miRNAs presence was identified from exosomes. This work describes a complete technique to isolate and characterize exosomes from human plasma, recognizing their potential as biomarkers.
Key words: Biomarker; Exosome; Retinoblastoma.
Resumo
Os exossomos são vesículas membranosas extracelulares essenciais na comunicação intercelular de longa distância; eles viajam em fluidos corporais e entregam mensagens moleculares dirigidas à maioria das células de todo o organismo. A liberação de mensagens através dos exossomos ocorre em forma de DNA, RNA ou proteínas; essa liberação foi associada a diferentes condições fisiológicas normais e patológicas, tais como o câncer. Por tudo isso, o eficiente isolamento e caracterização celular de exossomos de plasma é chave para sua utilização como biomarcadores não invasivos de várias doenças. No presente estudo, exossomos foram purificados a partir de amostras clínicas de plasmas de pacientes que tinham sido diagnosticados previamente com retinoblastoma e de indivíduos saudáveis como controle. Os exossomos recuperados foram caracterizados a nível celular por microscopia eletrônica de transmissão usando uma técnica de criogenia. Para demonstrar a correta purificação dos exossomos, foi confirmada a presença de proteínas transmembranares CD63 e CD81 usando inmunoblot. Além dos exossomos purificados foram identificados ARNs não codificantes pequenos chamados microARNs. Em geral os métodos de purificação e caracterização celular de exossomos obtidos de plasma humano são descritos por seu potencial utilização como biomarcadores.
Palavras-chave: Biomarcadores; Exossomos; Retinoblastoma.
Introducción
El cáncer se caracteriza por un crecimiento celular anormal que puede invadir tejidos adyacentes e incluso diseminarse en otros órganos. El origen del cáncer es multifactorial, intervienen elementos como la alimentación, infecciones virales, factores hereditarios, ambientales y geográficos. Pese a la recurrencia sintomatológica, hay algunos casos que se desarrollan de forma silenciosa y asintomática (1).
El cáncer engloba más de un ciento de enfermedades que afectan a todas las poblaciones, edades y estratos sociales; por lo tanto, se le considera un problema de salud pública. En 2012 se le atribuyeron 8,2 millones de muertes y se prevé que en 2020 se registren 15 millones de nuevos casos (2). Anualmente más de 250 mil niños son diagnosticados con cáncer. Entre los ocho tipos más comunes se encuentra el retinoblastoma, un tipo de cáncer pediátrico que afecta a la retina (3); se estima que en el mundo 9 mil casos nuevos son diagnosticados cada año (4).
EL RETINOBLASTOMA EN MÉXICO
El retinoblastoma es un problema oncológico que afecta al 4,3% de la población infantil en México. En menores de un año es la segunda neoplasia más común y constituye la segunda causa de muerte infantil en menores de dos años. La primera manifestación visible es un brillo blanco en la pupila, llamada leucocoria. Entre los síntomas secundarios se presenta la inflamación, posteriormente se genera el glaucoma que ocurre cuando el tumor empuja el diafragma del cristalino (5).
Puede presentarse de forma unilateral o bilateral, puede tener un patrón congénito y/o esporádico (no hereditario). Se estima que la sobrevida de los pacientes afectados es mayor al 80%. Sin embargo, las metástasis aparecen durante el primer año de diagnóstico. Para los casos metastásicos la sobrevida no supera los seis meses posteriores al diagnóstico (6). Aunque el retinoblastoma no afecta a una gran población en México al ser comparado, por ejemplo, con leucemia sí es un excelente modelo de estudio para elucidar los mecanismos celulares, genéticos y bioquímicos que provocan el cáncer(7).
El diagnóstico del retinoblastoma ocurre en estadios intermedios-avanzados de la enfermedad. Reportes previos sugieren que el retraso del diagnóstico predice el grado de invasión extraocular del tumor y por consiguiente, los casos metastáticos (8). El tiempo perdido entre la etapa asintomática y el diagnóstico hace que el tratamiento clínico sea la cirugía, con la consecuente remoción de uno o los dos ojos. Para contrarrestar esto, se requiere el desarrollo e implementación de métodos de diagnóstico tempranos no invasivos.
EL USO DE BIOMARCADORES EN ONCOLOGÍA
Los biomarcadores son indicadores de estados o condiciones fisiológicas. Pueden ser útiles para determinar la propensión a alguna enfermedad, evaluar su progresión, o bien, comprobar la eficacia de algún tratamiento. El potencial de los biomarcadores ofrece ventajas al área oncológica, entre las que se destaca una detección temprana en enfermedades asintomáticas. Otro de los beneficios es la disminución de dolor al aplicar métodos no invasivos durante la toma de muestras. Es por ello que identificar biomarcadores resulta una herramienta invaluable para la evaluación clínica (9).
Hoy en día, entre los biomarcadores más novedosos y trascendentales están las microvesículas; este tipo de biomarcadores son llamados "biopsias líquidas" (10). Las vesículas más ampliamente estudiadas son los exosomas, cuyos diámetros oscilan entre 30-140 nm (11). Los exosomas viajan a través de distintos fluidos biológicos como la leche materna, orina, saliva y plasma para llevar un mensaje a las células receptoras (12) y por mecanismos no completamente descritos, se fusionan en su membrana plasmática para liberar su contenido, entregando así mensajes íntegros y funcionales a la célula blanco (13).
EL ROL DE LOS EXOSOMAS EN EL CÁNCER
La relevancia de los exosomas en el cáncer aumentó al demostrarse que llevaban en su interior diferentes ARN y proteínas involucrados en el desarrollo y angiogénesis tumoral (14)(15). Recientemente se descubrió que los exosomas participan de forma activa durante la metástasis provocando la formación de un microambiente celular que inicia la colonización de órganos específicos como pulmón, es decir, son la señal de detonación de metástasis (16).
Otro elemento clave sobre el estudio de exosomas fue la identificación de "oncomirs", es decir, ARNs no codificantes llamados microARN que funcionan como genes oncológicos. Actualmente se usan a los microARNs de exosomas como biomarcadores (15). En cáncer de ovario, por ejemplo, a partir del suero de cuatro pacientes con diferentes estadios patológicos se identificó un grupo de ocho microARNs con niveles de expresión similares en todos los estadios (15). En melanoma se identificó la presencia de tres tipos distintos de microARNs a partir de exosomas aislados de dos líneas celulares tumorales (17).
Por lo anterior, el presente trabajo se enfocó en el aislamiento y purificación de exosomas en plasma de pacientes con retinoblastoma y de individuos sanos como control. Se implementó y estandarizó la caracterización celular mediante microscopia electrónica y se demostró mediante immunoblot la presencia de proteínas transmembranales características de exosomas. En este trabajo se establece un protocolo eficiente para la purificación de exosomas, para su potencial uso como biomarcadores de diagnóstico no invasivo.
Pese al potencial que los biomarcadores ofrecen, las limitaciones que se ven reflejadas en un gran número de publicaciones son la ausencia de validación de éstos en la práctica clínica, la ausencia de mediciones en cada fase de la condición fisiológica, la deficiencia o ausencia en las estrategias de validación y caracterización, junto con la deficiencia de análisis y ensayos clínicos (18)
Materiales y Métodos
CONSIDERACIONES ÉTICAS DEL ESTUDIO
Las muestras de sangre periférica fueron colectadas de cinco pacientes sanos y cinco pacientes con retinoblastoma en el Centro Médico Nacional Siglo XXI, siguiendo las reglas de bioética y bioseguridad del Comité de Investigación Científica Nacional (México). El proyecto fue autorizado por el Comité Local de Investigación y de Bioética con el número 2002/718/0034 adscrito al instituto Mexicano del Seguro Social. Los padres dieron su consentimiento para participar en el presente estudio luego de ser informados de los objetivos del estudio. Se incluyeron a individuos entre 18 a 24 meses; se consideró a los pacientes con retinoblastoma en estadio II de acuerdo con la clasificación intraocular internacional de retinoblastoma.
AISLAMIENTO DE EXOSOMAS DEL PLASMA
Muestras de plasma de pacientes con retinoblastoma se descongelaron y el volumen de trabajo fue de 500 μL. Las muestras se centrifugaron a 2000 g durante 20 min para eliminar cualquier célula. Luego, el sobrenadante se transfirió a un tubo nuevo para centrifugarse a 10000 g durante 20 min para remover cualquier residuo celular. En seguida, el sobrenadante se recuperó y se mezcló con PBS en una proporción 1:1 y se homogenizó con vortex. A continuación, se añadió el reactivo Total Exosome Isolation (Invitrogen, Thermo Fisher) en una proporción 1:5 y se mezcló hasta formar una solución homogénea. Las muestras se incubaron en hielo durante 10 min, después se centrifugaron a temperatura ambiente a 10000 g durante 10 min. El sobrenadante se desechó, mientras que el botón que contenía los exosomas fue resuspendido en PBS. La suspensión fue almacenada a -20 °C. La concentración de exosomas se relacionó indirectamente con la concentración de proteínas de la muestra por ensayo de Bradford.
MICROSCOPÍA ELECTRÓNICA DE TRANSMISIÓN POR CRIOGENIA
Los exosomas se observaron a través de microscopía electrónica de transmisión implementando una técnica criogénica (Cryo-TEM) con un sistema dosis mínima (MDS). Usando un microscopio electrónico de transmisión JEOL JEM2100 (Pleasanton, CA, EE.UU.) Para la caracterización morfológica de las poblaciones de exoso
mas, se vitrificaron las muestras en una rejilla de carbono Lacey dentro del dispositivo Gatan CryoplungeTM3 (EE.UU.) Después de la transferencia, la muestra se congeló por inmersión en etano líquido; para su crio-preservación se mantuvo en nitrógeno líquido hasta su observación en el microscopio. Con un soporte criogénico especial (Mo. 914) se transfirió al Gatan para mantener la temperatura de la muestra hasta -170 °C durante la observación. Las micrografías fueron adquiridas a 80 kV, de excitación débil, la dosis de radiación menor de 4 pA/cm² bajo condiciones altamente desenfocadas para obtener una mejor imagen de las poblaciones. Todas las imágenes fueron grabadas por una cámara CCD acoplada (Ultrascan XP, Gatan; EE.UU.) y procesadas en Micrografía Digital.
ANÁLISIS DE POBLACIONES DE EXOSOMAS CON ASISTENCIA BIOINFORMÁTICA
Las fotomicrografías capturadas durante la observación de exosomas por Cryo-TEM fueron analizadas usando el software ImageJ (19). Las micrografías fueron duplicadas al inicio del análisis para evitar la pérdida de las originales. Las imágenes se convirtieron a formato 8-bits, posteriormente se ajustó el umbral de detección. Por el contraste de las imágenes fue necesario realizarlo independiente para cada toma. En seguida, las imágenes se convirtieron a un formato binario (blanco y negro) con el objetivo de llenar espacios y marcar líneas divisorias donde hubiese superposiciones de las muestras. Se analizaron las partículas con tamaños mayores a 30 nm² y una circularidad entre 0,3 y 1,0, solicitando que se mostraran las líneas de contorno para cada partícula. Como parámetros de medición se consideraron el área, la media, el diámetro mínimo y máximo en los ejes X y Y, el perímetro, la circularidad y la solidez de la partícula. La escala se calibró con la regla original de las micrografías, encontrándose una relación 1:1 (pixel-nm).
INMUNOTRANSFERENCIA
Los exosomas aislados de plasma se mezclaron con amortiguador Laemmli, se calentaron a baño María por 5 min fueron cargados en geles SDS-PAGE. Se utilizó el marcador de peso molecular Protein Precision Plus (Bio- Rad) y se corrió a 100 V durante 1,5 h. Se transfirieron en una membrana Immun-Blot PVDF (Bio-Rad) durante 50 min a 18 V utilizando Trans-Blot Cell transferencia semi húmeda (Bio-Rad). En seguida se bloquearon las membranas con PBS, 0,1% de Tween 20 y 5% de Blotting-Grade Blocker (Bio-Rad) toda la noche. Después, las membranas se lavaron 4 veces durante 5 min usando amortiguador de lavado (PBST 0,1%). Las membranas se incubaron durante 1 hora con su respectivo anticuerpo primario recién preparado: CD63 con dilución 1:5000 (ab8219, Abcam), CD9 con dilución 1:10000 (ab2215, Abcam), CD81 con dilución 1:10000 (ab35026). Posteriormente, las membranas se lavaron para continuar con la incubación del anticuerpo secundario acoplado a peroxidasa de rábano HRP con dilución 1:15000 (ab97420, Abcam). Inmediatamente, las membranas se lavaron y se revelaron con Clarity Western Blotting Substrato (Bio-Rad) y se visualizaron mediante el Sistema de ChemiDoc (Bio-Rad).
PURIFICACIÓN DE ARN A PARTIR DE EXOSOMAS
Los exosomas purificados fueron transferidos a tubos libres de ARNasas. El volumen se ajustó a 200 μL con PBS. Para la extracción del ARN se usó el equipo Total Exosome RNA Isolation kit (Invitrogen, LifeTechnologies). Para lisar los exosomas se añadieron 200 μL de solución desnaturalizante y se agitó por 5 min en vortex. Luego, las muestras se incubaron en hielo durante 5 min. Inmediatamente se añadió a las muestras 400 μL ácido-fenol: cloroformo, se mezclaron por agitación durante 60 segundos. A continuación, las muestras se centrifugaron durante 5 min a 13000 g a temperatura ambiente. Se recuperó la fase acuosa y se transfirieron a un tubo libre de ARNasas. A la fase acuosa se le añadió 1,25 volúmenes de etanol absoluto. Estas soluciones fueron filtradas y recuperadas en los tubos colectores. Finalmente, el ARN se eluyó tres veces con 20 μL de agua DEPC. La concentración se midió por espectrofotometría con Nanodrop 2000 (Thermo Scientifics).
INICIADORES DE microARN TIPO STEM-LOOP
Los iniciadores se diseñaron considerando una estructura sintética conservada Stem-Loop que forma una horquilla. La especificidad de los iniciadores tipo Stem-Loop RT para cada microARN es conferida por seis nucleótidos (nt) en la orientación final 3’, que son complementarios al microARN maduro.
Los iniciadores utilizados en la PCR fueron diseñados considerando su orientación. Para el iniciador "adelante", se tomaron 8 nucleótidos complementarios a la secuencia del microARN maduro en dirección 5’. Para el iniciador "reverso" se consideró una secuencia complementaria de 18 nucleótidos conservados del iniciador sintético Stem-Loop en dirección 3’(20). La secuencia de los iniciadores se muestra en la Tabla I.
Tabla I. Secuencias de los iniciadores de microARN tipo Stem-Loop.

IDENTIFICACIÓN DE microARN PROVENIENTES DE EXOSOMAS
A partir del ARN purificado de los exosomas se realizó una Steem-Loop RT-PCR (20). En tubos libres de nucleasas se añadieron 0,5 μL del mix 10 mMdNTP (R1121, Thermo Scientific Ficher), 11,5 μL de agua libre de nucleasas y 1 μL de ARN molde (50 ng/μL). Las muestras se calentaron a 65 °C durante 5 min y se incubaron en hielo durante 2 min. En seguida, a cada muestra se añadió 1 μL del iniciador específico para los microARNs (miR-21, miR-155, miR-142 y miR-125b), 4 μL del amortiguador enzimático M-MuLV (New England Biolabs), 2 μL de 0,1 M DTT (Affymetrix), 0,1 μL de ARNasa out (40 unidades/μL) (Invitrogen) y 0,2 μL de la transcriptasa inversa M-MuLV (New England Biolabs). La reacción de retrotranscripción (RT) se realizó en un volumen total de 20 μL a 16 °C durante 30 min, seguido por 60 ciclos (30 °C por 30 s, 42 °C por 30 s y 50 °C durante 1 s), finalmente se inactivó la enzima a 85 °C por 5 min. El cDNA producto de la RT se conservó a 4 °C.
La PCR se realizó con 14 μL de agua estéril, 2 μL de producto de RT, 1 μL del iniciador derecho (10 mM), 1 μL del iniciador reverso universal (10 mM), 21 μL de la mezcla maestra Taq 5X (New England Biolabs). Las reacciones se amplificaron durante 30 ciclos (95 °C por 15 s, la temperatura media (Tm) por 15 s y 68 °C por 30 s). Los productos de PCR se conservaron a 4 °C. Se analizó la presencia de los microARNs con una electroforesis en geles de agarosa al 2,5%, y se visualizaron mediante el Sistema de ChemiDoc (Bio-Rad).
Resultados
Se trabajó con un volumen mínimo de plasma de 500 μL, el cual fue suficiente para evaluar la presencia de exosomas en muestras clínicas.
Para confirmar la presencia, forma y distribución de tamaños de los exosomas provenientes del plasma de pacientes con retinoblastoma y de individuos sanos, las muestras fueron observadas por Cryo-TEM. En la Figura 1 se muestra la forma redonda y cóncava característica de los exosomas. La distribución de tamaños de los exosomas fue analizada con el software ImageJ que permitió examinar y evaluar las imágenes generadas por Cryo-TEM. Derivadas de dicho análisis se encontraron dos poblaciones principales, una con diámetros que oscilaban entre 30 y 110 nm (Fig. 1A) y otra entre 100 y 195 nm diámetro (Fig. 1B). En la micrografía se observan como lunares suspendidos entre el espacio de la rejilla de carbón; además, se distingue con facilidad la doble membrana que envuelve y da estructura a los exosomas. Como resultado de la digitalización de las fotomicrografías (Fig. 1C) se determinó la frecuencia y tamaño de los exosomas purificados a partir de plasma, encontrándose mayoritariamente diámetros entre 55 y 95 nm (Fig. 1D).
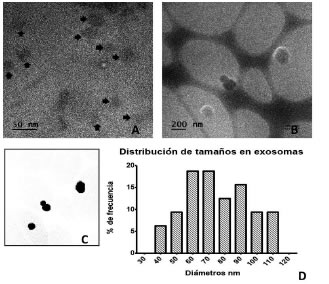
Figura 1. Micrografías electrónicas de transmisión. Imágenes representativas que muestran la estructura y tamaños de los exosomas purificados a partir de plasma.
A: Conjuntos de exosomas con tamaños de entre 30 y 60 nm. B: Exosomas con diámetros superiores a 100 nm. C: Digitalización de fotomicrografías durante el análisis con ImageJ. D: Clasificación de exosomas por tamaño y frecuencia relativa.
Los exosomas tienen una combinación única de proteínas y lípidos que permiten su reconocimiento por parte de las células receptoras. Aprovechando la presencia de proteínas transmembranales de la familia de tetranspaninas específicas de los exosomas, es posible su identificación por medio de Western blot.
Para corroborar la identidad bioquímica de los exosomas se muestra en las Figuras 2A y 2B el perfil proteico de los exosomas purificados de plasma. Para comprobar la identidad celular de los exosomas se detectaron por inmunotransferencia los marcadores CD63, CD81, Figuras 2C y 2D, respectivamente. La presencia de estas proteínas confirma la integridad de los exosomas recuperados a partir de plasma de pacientes y controles.
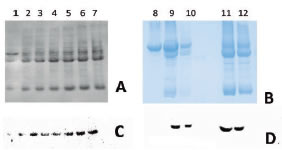
Figura 2. Inmunotransferencia.
A: Perfil proteico de exosomas aislados del plasma de individuos sanos (líneas 1 al 7). B: Perfil proteico de exosomas aislados de pacientes (líneas 11, 12), y de controles sanos (líneas 9,10), BSA como control negativo (línea 8). C revela la presencia de CD63 en exosomas. D indica la presencia de CD81 en exosomas humanos.
Uno de los métodos más simples y rápidos para evidenciar que los exosomas llevan un mensaje, es mediante la detección de microARNs, ARNs y proteínas. De acuerdo con literatura reciente sobre identificación de miARNs en exosomas (15), se confirmó la presencia de algunos de ellos mediante la técnica de steem-loop RT-PCR. Se evaluó la presencia de los microARNs 21, 155, 142 y 125b. En las Figuras 3A y 3B se demuestra la presencia de los miR-21 y miR-142 que en exosomas de pacientes y controles. El miR-155 sólo está presente en exosomas de pacientes (Fig. 3C) y el miR-125b está ausente en los exosomas de pacientes y controles (Fig. 3D), con lo cual se evidencia la diversidad del contenido de los exosomas provenientes de plasma de individuos con condiciones fisiológicas distintas.
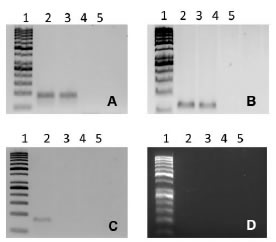
Figura 3. Gel de agarosa. Los productos de PCR de microARNs presentes en exosomas de pacientes y controles.
A: miR-21, B: miR-142, C: miR-155 y D: miR-125b. Línea 1 corresponde al marcador de peso molecular, líneas 2 y 3 al producto amplificado de pacientes y controles respectivamente. Líneas 4 y 5 a los controles negativos de la reacción.
Discusión y Conclusiones
Se han encontrado exosomas en una variedad de fluidos biológicos que incluyen orina, saliva, leche materna y plasma (21). Sin embargo, no se había precisado el volumen mínimo de muestra necesaria para hacer la caracterización celular y molecular de exosomas. Se demostró que 500 μL son suficientes para aislar una cantidad suficiente de exosomas y poder hacer su caracterización.
Por otra parte, en años recientes se han empleado otras técnicas de microscopia de alta resolución e incluso la implementación de costosos equipos que permiten la cuantificación de exosomas (22). Se propone la técnica de Cryo-TEM como una alternativa viable en la caracterización de los exosomas. Asimismo, se sugiere el uso del software ImageJ para el análisis cuantitativo de la distribución de tamaños. Si bien ImageJ es usado para el análisis de fotomicrografías fluorescentes y de campo claro, en el estudio de tejidos por inmunohistoquímica (18) resulta sumamente útil en el análisis de imágenes digitales. Esta combinación de Cryo-TEM e ImageJ permitió identificar poblaciones, observar estructuras y morfología. Además, se confirmó por inmunotransferencia la identidad de los exosomas recuperados con los marcadores específicos CD63 y CD81 previamente reportados (23). Estos marcadores CD63 y CD81 juegan un rol fundamental durante el proceso de transporte y distribución de los exosomas, además de coadyuvar en el reconocimiento por parte de la célula receptora (23).
Se verificó con experimentos sencillos y rápidos el contenido de los exosomas provenientes de plasma de pacientes y controles, encontrando diferencias cualitativas en el material confinado y resguardado por exosomas de pacientes con retinoblastoma. Estas evidencias sugieren que conocer el perfil completo de las moléculas transportadas por exosomas de pacientes puede dar información veraz y útil en el diagnóstico temprano de retinoblastoma. Su implementación como biomarcadores de la enfermedad agilizará el diagnóstico de los infantes.
AGRADECIMIENTOS
Este trabajo fue realizado con el apoyo de CONACyT-Salud 201388 y SIP-IPN-20152018. MJOR agradece a CONACyT-México y BEIFI-IPN por la beca proporcionada. Los autores agradecen al Instituto Politécnico Nacional y el Centro de Nanociencias y Micro y Nanotecnologías (CNMN) del IPN.
1. DeSantis CE, Lin CC, Mariotto AB, Siegel RL, Stein KD, Kramer JL, et al. Cancer treatment and survivorship statistics. CA Cancer J Clin 2014; 64 (4): 252–71.
2. Siegel RL, Miller KD, Jemal A. Cancer statistics 2015. CA Cancer J Clin 2015; 65 (1): 21254. [ Links ]
3. American Cancer Society. Cancer Facts & Figures 2014. Cancer Facts and Figures. Available from: URL: http://www.cancer.org/acs/groups/content/@research/documents/webcontent/acspc-042151.pdf (Fecha de acceso 5 de octubre de 2015). [ Links ]
4. Thériault BL, Dimaras H, Gallie BL, Corson TW. The genomic landscape of retinoblastoma: A review. Clin Exp Ophthalmol 2014; 42 (1): 33–52.
5. Alvarado-Castillo B, Campos-Campos LE, Villavicencio-Torres A. Características clínicas y metastásicas en retinoblastoma. Rev Med Inst Mex Seguro Soc 2009; 47 (2): 151–6.
6. Alvarado-Castillo B, Campos-Campos LE, Villavicencio- Torres A. Prevalencia de retinoblastoma del 2002 al 2006 en una unidad médica de alta especialidad. Rev Mexicana Oftalmol 2007; 81(6): 336–9.
7. Conkrite K, Sundby M, Mukai S, Thomson JM, Mu D, Hammond SM, et al. miR-17~92 cooperates with RB pathway mutations to promote retinoblastoma. Genes Dev 2011 Aug 15; 25 (16): 1734–45.
8. Ramírez-Ortiz MA, Ponce-Castañeda MV, Cabrera-Muñoz ML, Medina-Sansón A, Liu X, Orjuela MA. Diagnostic delay and socio-demographic predictors of stage at diagnosis and mortality in unilateral and bilateral retinoblastoma. Cancer Epidemiol Biomarkers Prev 2014 May; 23 (5): 784-92. [ Links ]
9. Ajit SK. Circulating microRNAs as biomarkers, therapeutic targets, and signaling molecules. Sensors (Basel) 2012; 12 (3): 3359–69.
10. Best MG, Sol N, Kooi I, Tannous J, Westerman BA, Rustenburg F, et al. (2015). Rna-Seq of tumor-educated platelets enables blood-based pan-cancer, multiclass, and molecular pathway cancer diagnostics. Cancer Cell 2015 Nov 9; 28 (5): 666–76.
11. Skog J, Würdinger T, van Rijn S, Meijer DH, Gainche L, Curry WT, et al. Glioblastoma microvesicles transport RNA and proteins that promote tumour growth and provide diagnostic biomarkers. Nat Cell Biol 2008 Dec; 10 (12): 1470–6.
12. Wiklander OPB, Nordin JZ, O’Loughlin A, Gustafsson Y, Corso G, Mäger I, et al. Extracellular vesicle in vivo biodistribution is determined by cell source, route of administration and targeting. J Extracell Vesicles 2015 Apr 20; 4: 26316.
13. Valadi H, Ekström K, Bossios A, Sjöstrand M, Lee JJ, Lötvall JO. Exosome-mediated transfer of mRNAs and microRNAs is a novel mechanism of genetic exchange between cells. Nat Cell Biol 2007 Jun; 9 (6): 654–9.
14. Melo SA, Luecke LB, Kahlert C, Fernandez AF, Gammon ST, Kaye J, et al. Glypican-1 identifies cancer exosomes and detects early pancreatic cancer. Nature 2015; 523 (7559): 177–82.
15. Taylor DD, Gercel-Taylor C, inventors; University of Louisville Research Foundation, assignee. Exososme-Associated microRNAs as diagnostic marker. US patent 8, 637, 254. 2014 Jan 28. [ Links ]
16. Hoshino A, Costa-Silva B, Shen TL, Rodrigues G, Hashimoto A, Tesic M, et al. Tumour exosome integrins determine organotropic metastasis. Nature 2015; 527 (7578): 329-35. [ Links ]
17. Xiao D, Ohlendorf J, Chen Y, Taylor DD, Rai SN, Waigel S, et al. Identifying mRNA, microRNA and protein profiles of melanoma exosomes. PloS One 2012; 7 (10): 1-15. [ Links ]
18. Drucker E, Krapfenbauer K. Pitfalls and limitations in translation from biomarker discovery to clinical utility in predictive and personalized medicine. EPMA J 2013; 4 (7): 1-10. [ Links ]
19. Hartig SM. Basic Image Analysis and Manipulation in ImageJ. En Current Protocols in Molecular Biology 1st. ed. Houston, Texas, EE.UU. John Wiley Sons Inc; 2013; p. 1-12. [ Links ]
20. Varkonyi-Gasic E, Wu R, Wood M, Walton E F, Hellens RP. A highly sensitive RT-PCR method for detection and quantification of microRNAs. Plant Methods 2007; 3 (1): 12-6. [ Links ]
21. An T, Qin S, Xu Y, Tang Y, Huang Y, Situ B, et al. Exosomes serve as tumour markers for personalized diagnostics owing to their important role in cancer metastasis. J Extracell Vesicles 2015; 4 (4): 27522-37. [ Links ]
22. Théry C, Amigorena S, Raposo G, Clayton A. Isolation and characterization of exosomes from cell culture supernatants and biological fluids. En: Current protocols in cell biology. 1st. ed. París: John Wiley Sons Inc; 2006. p. 221-29. [ Links ]
23. Pols MS, Klumperman J. Trafficking and function of the tetraspanin CD63. Exp Cell Res 2009; 315 (9): 1584–92.
Recibido: 23 de noviembre de 2015
Aceptado: 28 de junio de 2016














