Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Acta bioquímica clínica latinoamericana
versión impresa ISSN 0325-2957versión On-line ISSN 1851-6114
Acta bioquím. clín. latinoam. vol.51 no.4 La Plata dic. 2017
BIOLOGÍA MOLECULAR
Mutaciones en JAK2, MPL y CALR en neoplasias mieloproliferativas: análisis de disociación de alta resolución
Mutations in JAK2, MPL and CALR in myeloproliferative neoplasms: High Resolution Melting analysis
Mutações em JAK2, MPL e CALR em neoplasias mieloproliferativas: análise de dissociação em alta resolução
Yanina Paola Videla1a,b, Silvina Quintana2a,c, Josefina Pérez Maturo1a,b, Vanesa Di Gerónimo3a, Nazarena Martín4d, Fernando Pagani4d
1 Licenciada en Ciencias Biológicas.
2 Doctora en Farmacia y Bioquímica.
3 Técnica.
4 Médico.
a Laboratorio de Biología Molecular, Fares Taie Instituto de Análisis. Rivadavia 3343, CP7600 Mar del Plata. Argentina.
b FCEyN. Universidad Nacional de Mar del Plata. Deán Funes 3350, CP7600 Mar del Plata. Argentina.
c Consejo Nacional de Investigaciones Científicas y Técnicas (CONICET), Rivadavia 1917, C1033AAJ, Buenos Aires, Argentina.
d Servicio de Hematología. Clínica Colón. Av. Colón 3629, CP7600 Mar del Plata. Argentina.
CORRESPONDENCIA Dra. SILVINA QUINTANA Laboratorio de Biología Molecular, Fares Taie Instituto de Análisis. Rivadavia N° 3343, MAR DEL PLATA E-mail: biologiamolecular@farestaie.com.ar
Resumen
La policitemia vera (PV), la trombocitemia esencial (TE) y la mielofibrosis idiopática (MI) constituyen las Neoplasias Mieloproliferativas cromosoma Filadelfia negativas (NMP Ph-neg). La mutación V617F en el exón 14 del gen JAK2 ha sido descripta en un 90% de los casos de PV y en un 50% de TE y MI. Recientemente, se identificaron mutaciones en el exón 10 del gen MPL y en el exón 9 del gen CALR, presentes en un 5 y 73% de pacientes con TE y MI sin mutaciones en JAK2, respectivamente. En el presente trabajo se estudió la detección de dichas mutaciones en 52 pacientes con NMP, mediante amplificaciones por PCR en Tiempo Real con posterior análisis por High Resolution Melting (HRM) y secuenciación. La mutación V617F en JAK2 fue registrada en un 83,3% de pacientes con PV y 42,8% con TE y MI. Un 6,25% y 56,25% de pacientes con TE y MI JAK2 negativos resultaron positivos para mutaciones en el exón 10 de gen del receptor de la trombopoyetina (MPL) y el exón 9 de gen de la calreticulina (CALR). El análisis por HRM puede ser considerado como herramienta diagnóstica eficaz para las NMP debido a su alta sensibilidad, bajo costo y tiempo de procesado, teniendo en cuenta el impacto clínico que podría tener en los pacientes la detección temprana de dichas mutaciones.
Palabras clave: Neoplasias mieloproliferativas; JAK2; Gen de la calreticulina; Gen del receptor de la trombopoyetina; Análisis de disociación de alta resolución; Diagnóstico.
Abstract
Polycythemia vera (PV), essential thrombocythemia (TE) and idiopathic myelofibrosis (MI) are Philadelphia chromosome–negative myeloproliferative neoplasms (MPN-Ph. Neg). The presence of the V617F mutation in exon 14 of the JAK2 gene has been described in 90% of cases of PV and 50% of MI and TE. Recently, mutations in exon 10 of the MPL gene and exon 9 of CALR gene have been identified, which are present in 5 to 73% of patients with TE and MI without mutations in JAK2, respectively. In this work, the detection of these mutations was studied in 52 patients with NMP, using real time PCR amplifications with subsequent High Resolution Melting (HRM) analysis and sequencing. A total of 83.3% of patients with PV and 42.8% with MI and TE were recorded as positive for the V617F mutation in JAK2. A total of 6.25% and 56.25% of the patients with MI and TE with non-mutated JAK2 were positive for mutations in MPL exon 10 and CALR exon 9. HRM analysis could be considered an effective diagnostic tool for NMP due to its high sensitivity, low cost and processing time, taking into account the clinical impact that early detection of such mutations could have on patients.
Key words: Myeloproliferative neoplasms; Mutation; JAK2; CALR; MPL; High resolution melting; Diagnosis.
Resumo
Policitemia vera (PV), trombocitemia essencial (TE) e mielofibrose idiopática (MI) constituem as Neoplasias Mieloproliferativas cromossomo Filadélfia negativas (NMP Ph-neg). A mutação V617F no exon 14 do gene JAK2 foi descrita em 90% dos casos de PV e em 50% de TE e MI. Recentemente, foram identificadas mutações no exon 10 do gene MPL e no exon 9 do gene CALR, presentes em 5 a 73% de pacientes com TE e MI sem mutações em JAK2, respectivamente. Neste trabalho foi estudada a detecção de tais mutações em 52 pacientes com NMP, usando amplificações por PCR em Tempo Real, com posterior análise por High Resolution Melting (HRM) e sequenciamento. 83,3% dos pacientes com PV e 42,8% com TE e MI foram positivos para a mutação V617F em JAK2. 6,25% e 56,25% de pacientes com TE e MI JAK2 negativo foram positivos para mutações no exon 10 de gene do receptor da trombopoietina (MPL) e o exon 9 de gene da calreticulina (CALR). A análise por HRM pode ser considerada como ferramenta de diagnóstico eficaz para as NMP, devido à sua alta sensibilidade, baixo custo e tempo de processamento, tendo em conta o impacto clínico que poderia ter a detecção precoce de tais mutações nos pacientes.
Palavras-chave: Neoplasias mieloproliferativas; JAK2; Gene da calreticulina; Gene do receptor da trombopoietina; High Resolution Melting; Diagnóstico.
Introducción
Las neoplasias mieloproliferativas crónicas (NMP) comprenden desórdenes clonales hematológicos originados por la transformación de células madre hematopoyéticas y la proliferación de una o más de las líneas mieloides. Según criterios clínicos, moleculares y anatomopatológicos de la médula ósea determinados por la Organización Mundial de la Salud (OMS) (1), la policitemia vera (PV), la trombocitemia esencial (TE) y la mielofibrosis idiopática (MI) constituyen las actualmente clasificadas NMP crónicas cromosoma Filadelfia negativas clásicas (NMP Ph-neg). Es decir, no presentan la aberración cromosómica BCR-ABL (2). Estos trastornos se caracterizan por presentar fibrosis progresiva de la médula ósea (MO) y el desarrollo de hematopoyesis extramedular en la MI, la presencia de eritrocitosis en PV y el compromiso en forma primaria de la línea de megacariocitos de MO en TE. Entre los síntomas y manifestaciones clínicas comunes a las tres entidades se detallan esplenomegalia, prurito y fatiga, acompañado de una clara reducción en la calidad de vida del paciente.
En los últimos años, el rol de la vía JAK-STAT en el desarrollo de distintos tipos de cáncer ha sido el foco de diferentes investigaciones. Tal es así, que se han detectado mutaciones que generan la activación constitutiva o hiperactividad de este mecanismo en diversos desórdenes hematológicos, especialmente en neoplasias mieloproliferativas. En 2005 se reportó una mutación puntual en el gen JAK2, el cual codifica para la quinasa JAK2 y cuyo rol es fundamental en la vía JAK-STAT. Esta mutación consiste en la sustitución de una guanina (G) por una timina (T) en el nucleótido 1849 del exón 14, resultando en el reemplazo de un aminoácido valina (V) por fenilalanina (F) en la posición 617 de la proteína (3) generando activación constitutiva de la misma. Kralovics et al han demostrado que esta mutación participa en la patogenia de las NMPs, debido a que contribuye a la expansión clonal de estos desórdenes (4). La presencia de JAK2 V617F ha sido reportada en la mayoría de los pacientes con NMPs, detectándose en el 95% de los casos de PV y en la mitad de los casos de TE o MI (3).
En los años subsiguientes se han reportado mutaciones somáticas en el gen MPL (5)(6). Este gen codifica para el receptor de la trombopoyetina (TpoR), hormona estimulante de la diferenciación de plaquetas (7). Las mutaciones descriptas hasta el momento se sitúan en el exón 10 e involucran 5 tipos (W515L, W515Ki, W515Kii, W515R y W515A) que afectan el residuo de triptófano en la posición 515 (5-8). Este residuo (W515)
forma parte del dominio RWQFP, clave en el mantenimiento del receptor dentro de su estado inactivo en ausencia de ligando (9-10). Hasta el momento, estas mutaciones han sido reportadas sólo en un 5% de pacientes con TE (5) y MI (8), que no poseen mutaciones en el gen JAK2, y en ningún paciente con PV.
Continuando con la investigación de la base molecular de las NMP, a fines de 2013 dos grupos en simultáneo reportaron mutaciones somáticas en el gen CALR (11)(12). Este gen codifica para una proteína sumamente importante en la proliferación celular, la calreticulina. Hasta el momento se han descripto 36 tipos de mutaciones en el gen CALR las cuales incluyen deleciones e inserciones que resultan en un nuevo C-terminal en la proteína (11)(12). Está reportado que dos de estas mutaciones son las más frecuentes en pacientes con NMP: una deleción de 52 pares de bases (pb) (tipo 1) y una inserción de 5 pb (tipo 2). Hasta la fecha, han sido halladas en el 73% de pacientes con TE y MI sin mutaciones en JAK2 y MPL, y no han sido descriptas en pacientes con PV (11)(12).
Es importante destacar que existen estudios que indican que las NMP CALR positivas tendrían un curso clínico más benigno que las asociadas a mutaciones en JAK2 o MPL (11-13), por lo que la implementación de una técnica diagnóstica eficaz resultaría sumamente útil tanto para los médicos como para los pacientes. Dado que los tres trastornos estudiados poseen características clínicas en común, que pueden incluso solaparse con patologías secundarias o reactivas a determinados desórdenes fisiológicos, el desarrollo de técnicas moleculares de detección de las mutaciones en JAK2, MPL y CALR ha proporcionado una herramienta por demás importante para la diferenciación de una entidad neoplásica de una reactiva.
Debido a que las mutaciones reportadas son de origen clonal, es necesario que la técnica utilizada para su análisis sea precisa y con alta sensibilidad. Si bien las técnicas PCR Alelo específica (del inglés Polymerase Chain Reaction), RFLP (del inglés Restriction Fragment Length Polymorphism) y secuenciación están ampliamente difundidas, poseen cierta limitación a la hora de un diagnóstico preciso en aquellos casos donde la carga alélica de la mutación es baja. El análisis de disociación de alta resolución (HRM; del inglés High Resolution Melting) es considerado una herramienta emergente para diagnóstico molecular. Es una metodología post-PCR que permite la identificación de variaciones en la secuencia de ADN, basada en el análisis de curvas de fusión de fragmentos de ADN amplificados mediante PCR en tiempo real. Entre las mutaciones que se pueden detectar con este análisis se encuentran: SNPs (del inglés Single Nucleotide Polymorphism), pequeñas deleciones e inserciones, mutaciones nuevas, o incluso alguna variante de ADN mutado minoritario en una muestra con predominio de la secuencia Wild Type (WT) (14)(15). En el análisis de HRM, la región de interés se amplifica mediante PCR en presencia de un intercalante fluorescente. Tras la PCR, la temperatura es elevada gradualmente de manera que la doble hebra de ADN se desnaturaliza. La fluorescencia emitida se mide en un instrumento especializado para generar una curva característica. A medida que la temperatura aumenta, la fluorescencia va disminuyendo, al principio lentamente, hasta que a una determinada temperatura, decae de manera abrupta, reflejando la temperatura de melting (Tm). Aspectos como el contenido de guanina (G) y citocina (C), la longitud, la secuencia y la heterocigosidad determinarán la curva de fusión específica para cada amplicón (15). El software utilizado permite la normalización de las curvas, para poder identificar claramente las diferencias entre muestras.
El objetivo del presente trabajo fue el desarrollo de protocolos de HRM para la detección de mutaciones en el exón 10 del gen del receptor de la trombopoyetina (MPL) y en el exón 9 del gen de calreticulina (CALR) en pacientes con neoplasias mieloproliferativas (NMP Ph-neg).
Materiales y Métodos
MUESTRAS
Se utilizaron muestras de sangre obtenidas de 52 pacientes diagnosticados por el Servicio de Hematología de la Clínica Colón de Mar del Plata, Argentina. Todos los pacientes dieron su consentimiento escrito voluntario y se respetaron las recomendaciones de Helsinki durante el mismo.
EXTRACCIÓN DE ADN
La extracción de ADN se llevó a cabo a partir de sangre periférica, obtenida por punción venosa (antebrazo) y recolectada en un tubo de plástico estéril con anticoagulante EDTA. Se realizó a partir de 200 μL de sangre entera, utilizando el kit comercial “ADN High Pure PCR Template preparation Kit” (Roche, Indianapolis, EE.UU.), según instrucciones del fabricante. Se cuantificó el ADN presente en las muestras mediante el kit Quant-iT™ PicoGreen® dsDNA Assay (Invitrogen, Carlsbad, EE.UU.), utilizando el termociclador en tiempo real Rotor Gene 6000 (Qiagen, Hilden, Alemania) según indicaciones del fabricante. Todas las muestras utilizadas en este estudio, presentaron una concentración de ADN entre 10 y 50 ng/μL.
DETECCIÓN DE MUTACIONES
Con el fin de detectar las mutaciones descriptas se llevaron a cabo amplificaciones por PCR en Tiempo Real en un termociclador Rotor Gene 6000, utilizando una mezcla fluorescente EvaGreen preformada y optimizada con los componentes de la reacción (Master Mix 2X KAPA HRM, KAPA Biosystems, Londres, Inglaterra), exceptuando cebadores y agua, en un volumen final de 20 μL, y por duplicado. Para cada gen se validaron los sets de cebadores elegidos de la bibliografía y se pusieron a punto los parámetros en la PCR, tomando en cuenta el tamaño del amplicón a generarse y la Tm provista por el proveedor de los mismos, mediante un gradiente de temperaturas de annealing (TA) de manera empírica, en un termociclador VERITI (Applied Biosystems). Los productos de la reacción fueron verificados en un gel de agarosa al 1,5% y visualizados bajo luz UV, con el fin de corroborar la amplificación de los productos del peso molecular esperado. Luego de realizar pruebas de validación con los pares de cebadores se seleccionó para el estudio de HRM el par más adecuado y se fijó el programa de ciclado determinado.
Para la correcta validación de las metodologías de HRM, aquellos productos de amplificación con patrones de melting diferentes fueron separados por electroforesis en geles de agarosa, purificados y secuenciados. Luego se compararon las secuencias obtenidas con las secuencias disponibles en la base de datos del Centro Nacional para la Información Biotecnológica de EEUU (NCBI).
La búsqueda de mutaciones comenzó siguiendo el algoritmo diagnóstico recomendado (16), utilizando pacientes con patologías confirmadas, dado que no se contaban con controles positivos para las mutaciones en estudio.
PROTOCOLOS PARA LA DETECCIÓN DE MUTACIONES EN EL GEN JAK2
Para la detección de la mutación JAK2 V617F y mutaciones en el exón 12 en el gen JAK2 se utilizaron los protocolos previamente validados por Quintana et al (17) y Schoenfeld et al (18), respectivamente.
PROTOCOLO PARA DETECCIÓN DE MUTACIONES EN EL EXÓN 9 DEL GEN CALR
Se realizaron amplificaciones por PCR en Tiempo Real de un fragmento de 298 pb, correspondiente al hot-spot de mutaciones halladas en el exón 9 de dicho gen, con los cebadores CALRHRM fw 5´-TAACAAAGGTGAGGCCTGGT-3´ y CALRHRM rv 5´- GCCTCTCTACAGCTCGTCCTT- 3´ (19). La decisión de amplificar sólo el hot-spot y no el exón completo se tomó teniendo en cuenta el tamaño del amplicón adecuado para un análisis de HRM, el cual debe ser menor de 300 pb. El exón entero consta de 777 pb, por lo que al amplificarlo completamente se pierde sensibilidad para dicho análisis. El programa de ciclado consistió en una desnaturalización inicial de 2 min, seguido de 45 ciclos de 15 s a 95 ºC, 15 s a 61 ºC y 20 s a 72 ºC. El análisis por HRM se llevó a cabo en un rango de temperaturas de 80 a 95 ºC con un delta de temperatura de 0,1 ºC.
PROTOCOLO PARA LA DETECCIÓN DE MUTACIONES EN EL EXÓN 10 DEL GEN MPL
Se llevaron a cabo amplificaciones por PCR en Tiempo Real utilizando los cebadores MPL HRM fw 5´-GCCGAAGTCTGACCCTTTTT-3´ y MPLHRM rv 5´-ACAGAGCGAACCAAGAATGCCTGTTTACA-3´ (6), los cuales amplifican un fragmento de 209 pb del exón 10 del gen MPL. El programa de ciclado consistió en una desnaturalización inicial de 2 min a 95 ºC, y 45 ciclos de 15 s a 95 ºC, 20 s a 60°C y 30 s a 72 ºC. El análisis por HRM se llevó a cabo en un rango de temperaturas de 85 a 95 ºC.
CÁLCULO DE PREVALENCIA DE LAS MUTACIONES
Una vez validados los protocolos, se llevó a cabo la búsqueda de las mutaciones en la totalidad de pacientes. Siguiendo el algoritmo diagnóstico (16), se analizaron un total de 52 pacientes que ingresaron al servicio de Hematología de la Clínica Colón de la ciudad de Mar del Plata. En base a los datos aportados por los médicos a cargo del servicio (parámetros clínicos y hematológicos), se subdividieron los pacientes de acuerdo con su patología, a fin de calcular la ocurrencia de las mutaciones descriptas en cada una de ellas.
Resultados
DETECCIÓN DE MUTACIONES EN EL EXÓN 9 DEL GEN CALR
Al momento de la validación de la técnica no se contaba con controles positivos y negativos para mutaciones en el exón 9 del gen CALR, de modo que para su correcta validación se realizó en primer lugar con pacientes con TE, negativos para JAK2 V617F. Se llevaron a cabo amplificaciones por PCR en Tiempo Real, seguidas del análisis de melting de alta resolución. Luego de cada PCR, se normalizaron las lecturas de fluorescencia. Para ello se utilizó el software Rotor Gene 6000, permitiendo identificar áreas de pre- y post-melting en la zona estable de fluorescencia. Se colocaron las barras de normalización, lo cual permitió eliminar las diferencias en el background de fluorescencia de modo de incrementar la habilidad del software de detectar las diferencias sutiles que pueden existir en el perfil de melting. Aquellos productos de amplificación que mostraban patrones diferentes fueron separados por electroforesis en geles de agarosa, purificados y secuenciados (Figura 1). Dado que se desconocía a qué mutación correspondía cada perfil de melting, la búsqueda de las mismas se llevó a cabo in sílico, analizando las secuencias obtenidas tanto por observación de los electroferogramas como por alineamientos en BLAST (del inglés Basic Local Alignment Search Tool, www.ncbi.nlm.nih.gov/BLAST/). En base a esto, se asignaron en el software de HRM los genotipos correspondientes a los patrones obtenidos, tanto al WT como a aquellos que presentaban mutación.
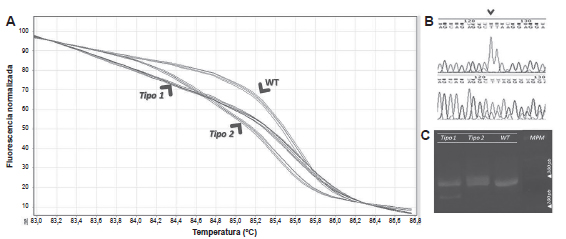
Figura 1. Validación del análisis por HRM para detección de mutaciones en el hot-spot del exón 9 de CALR. Se amplificó por PCR en tiempo real un fragmento de 298 pb, con el posterior análisis de melting de alta resolución. La asignación de genotipos se realizó luego de secuenciar los productos con patrones de melting diferentes. A. Curvas de melting normalizadas. Se observan las curvas correspondientes a los genotipos homocigota normal (rojo) y heterocigota para las mutaciones tipo 1 (violeta) y tipo 2 (celeste). B. Electroferogramas de secuencias parciales del hot-spot de mutaciones del exón 9 de CALR. Arriba: Secuencia WT (homocigota normal); Abajo: Mutación Tipo 1 (heterocigota mutado). C. Electroforesis en gel de agarosa. Se observan los patrones de bandas correspondientes a muestras con la mutación tipo 1 (del 52 pb), tipo 2 (ins. 5 pb) y WT. MPM: Marcador de peso molecular.
En la Figura 1 A se visualizan los tres patrones de melting obtenidos en el análisis, a los cuales se asignaron los genotipos correspondientes luego del análisis de las secuencias obtenidas. El patrón con mayor Tm corresponde al genotipo homocigota normal, mientras que los dos restantes, con menor Tm, corresponden a dos genotipos heterocigotas mutados distintos. Se pudo comprobar que estos coincidían con los dos tipos de mutaciones más frecuentes descriptos hasta el momento: la mutación tipo 1 (deleción de 52 pb) y la tipo 2 (inserción de 5 pb). Asimismo, se pudo constatar la presencia de amplicones con diferentes pesos para cada muestra secuenciada, a partir de la separación de las mismas por electroforesis en gel (Figura 1 B), confirmando que las mutaciones halladas se presentan en heterocigosis y que la presencia de las mismas puede ser corroborada en geles de agarosa.
DETECCIÓN DE MUTACIONES EN EL EXÓN 10 DEL GEN MPL
Se llevaron a cabo amplificaciones por PCR en Tiempo Real, con posterior análisis por HRM (Figura 2 A). Aquellos productos que mostraban patrones de melting diferentes fueron separados por electroforesis en geles de agarosa, purificados y secuenciados (Figura 2 B). Cabe destacar que se inició la búsqueda de mutaciones sin controles positivos y negativos para mutaciones en el exón 10 del gen. Por lo tanto, la misma se llevó a cabo in sílico, analizando las secuencias obtenidas, tanto por observación de los electroferogramas como por alineamientos en BLAST (Basic Local Alignment Search Tool, programa informático de alineamiento de secuencias de tipo local, ya sea de ADN, ARN o de proteínas).

Figura 2. Validación del análisis por HRM para detección de mutaciones en el exón 10 de MPL. Se amplificó por PCR en tiempo real un fragmento de 209 pb, con el posterior análisis de melting de alta resolución. La asignación de genotipos se realizó luego de secuenciar los productos con patrones de melting diferentes. A. Curvas de melting normalizadas. Se observan las curvas correspondientes a los genotipos homocigota normal y heterocigota para la mutación W515K. B. Electroferogramas de secuencias parciales del exón 10 de MPL. Arriba: Secuencia WT (Homocigota normal). Abajo: Mutación W515K, cambio TGG>AAG (heterocigota mutado). C. Electroforesis en gel de agarosa. Se observan los patrones de bandas correspondientes a muestras WT y con la mutación W515K. MPM: Marcador de peso molecular
Como consecuencia del análisis de las secuencias de los productos de PCR, se pudo determinar la presencia de la mutación W515K en aquella muestra que presentaba un patrón de melting diferencial, con una Tm menor (Figura 2 A). La curva con mayor Tm fue coincidente con el genotipo homocigota normal. Una réplica de la muestra secuenciada fue analizada mediante electroforesis en gel de agarosa, sin observarse diferencias en el patrón de bandas obtenidas (Figura 2 C).
PREVALENCIA DE MUTACIONES EN PACIENTES CON NMP
Luego de la validación de los protocolos, se procedió a realizar el screening de las mutaciones en la población en estudio (Figura 3). En el caso de los pacientes con PV (n=24), un 83,3% presentó genotipo heterocigota para la mutación JAK2 V617F, mientras que en los restantes no fue detectada (Figura 3 A). En dichos pacientes, tampoco se hallaron mutaciones en el exón 12 del gen JAK2 por lo que se asume poseen genotipo homocigota normal. Como se ilustra en la Figura 3 B, la subpoblación con TE (n=22) presentó mutaciones en los tres genes analizados. Un 50% resultaron heterocigotas para la mutación JAK2 V617F, un 31,8% para mutaciones tipo 1 y 2 en CALR y 4,6% la mutación W515K en MPL. Sólo un 13,6% de los pacientes no presentaron ninguna mutación de las analizadas, clasificándose a los mismos como Triples Negativos (TN). Por último, en los pacientes con MI (n=6) sólo pudieron hallarse las mutaciones JAK2 V617F (16,7%) y las más frecuentes en CALR (33,3%). Un 50% de los pacientes fueron clasificados como TN (Figura 3 C).

Figura 3. Prevalencia de mutaciones en JAK2, exón 10 de MPL y exón 9 de CALR. Resultados obtenidos en pacientes
diagnosticados con PV (A), TE (B) y MI (C) y para TE y MI homocigotas normales para JAK2 (D).
Además, se analizó la frecuencia de las mutaciones en el exón 9 de CALR y en el exón 10 de MPL, en pacientes con TE y MI negativos para JAK2 V617F. Tal como se observa en la Figura 3 D, en el 56,3% de los mismos se detectó la presencia de mutaciones tipo 1 y 2 en CALR, y en un 6,3% en el exón 10 de MPL. Un 37,5% de los individuos analizados no presentaron ninguna de las mutaciones buscadas. Asimismo, se corroboró que las mutaciones analizadas son mutuamente excluyentes.
Discusión y Conclusiones
Con el advenimiento de la medicina personalizada, existe una gran necesidad de métodos rápidos y precisos para la detección de mutaciones asociadas con las diferentes patologías. Teniendo en cuenta las características clínicas de estos desórdenes y el origen de las mutaciones que las generan, una tecnología ideal debería ser lo suficientemente sensible como para detectar las mutaciones aun existiendo cantidades significativas de contaminación con células WT. La técnica de HRM es una metodología emergente para la detección de mutaciones en muestras clínicas. Es un método no destructivo que permite un análisis posterior de los productos de PCR mediante electroforesis en gel o secuenciación. Provee un sistema cerrado que reduce el riesgo de contaminación, el tiempo de análisis y no requiere de procesamiento o separación después de la PCR. Basado en su facilidad de uso, bajo costo, rapidez y sensibilidad, la HRM es una herramienta de gran utilidad en diagnóstico clínico para distintas aplicaciones: screening y detección de mutaciones, metilación, genotipado, búsqueda de SNPs, entre otras (14)(15-20).
La validación de los protocolos de detección por HRM se llevó a cabo de manera exitosa, corroborando los resultados obtenidos en el análisis de perfiles de melting con los aportados por la secuenciación de los productos. La elección de los cebadores que amplificaban fragmentos de no más de 300 pb permitió la detección de SNPs, inserciones y deleciones de interés. Debido a la alta sensibilidad que presenta la HRM, el diseño y elección de los mismos debió ser cuidadoso, verificando que la variación en el perfil de melting se debía a la presencia de la mutación buscada y no a un polimorfismo sin valor diagnóstico. Para esto se analizaron las secuencias de los productos de amplificación, en busca de las mutaciones específicas, ya que no se contaba con controles positivos en ninguno de los genes analizados. Una vez hallada la mutación, se procedió exitosamente a la asignación del genotipo correspondiente a cada curva de melting obtenida, utilizando el software específico para HRM. Debido a la baja frecuencia de mutaciones en MPL, y a un número relativamente bajo de pacientes analizados, sólo pudo hallarse un tipo de mutación (W515K) en el exón 10 del gen, de los cuatro indicados por Beer en 2008 (5). Por lo tanto, la técnica pudo ser correctamente validada sólo para dicha variación, debiéndose corroborar por secuenciación los productos que en un futuro estudio obtengan patrones de melting diferentes a éste o al WT. Como parte del protocolo de validación, y para complementar el análisis, se estudiaron los productos de amplificación por electroforesis en geles de agarosa. Gracias a esto, se pudo constatar además la utilidad de la técnica de HRM para la detección de variaciones de pocos nucleótidos. En el caso de la mutación W515K (TGG>AAG) en el exón 10 de MPL, no fue posible detectarla mediante electroforesis en gel de agarosa debido a la baja resolución que presenta esta técnica para diferenciar productos que difieren en pocas bases, mientras que sí lo fue mediante HRM y secuenciación. En el caso de las mutaciones en el exón 9 de CALR, sólo se detectaron dos de las 36 reportadas (11)(12) siendo éstas las más frecuentes en pacientes con TE y MI. Al igual que para MPL, las muestras que presenten un patrón de melting diferente deberán ser analizadas por secuenciación. Si bien las mutaciones encontradas en este trabajo pudieron ser detectadas también por secuenciación, el tiempo de procesado de la muestra es mayor que el necesario para llevar a cabo el análisis de HRM.
Habiendo validado la técnica, se realizó el estudio de las mutaciones en la totalidad de pacientes, siguiendo el algoritmo diagnóstico recomendado (16). En cuanto a la frecuencia de las mutaciones en cada patología, en general los resultados fueron coincidentes con los reportados por la bibliografía (3)(12)(13)(21). En pacientes con PV, para la prevalencia de la mutación JAK2 V617F se obtuvo un porcentaje en concordancia con lo esperado. También se buscaron mutaciones en el exón 12 del gen JAK2, no registrándose resultados positivos. Los porcentajes obtenidos para los pacientes con TE fueron similares a los esperados. Por el contrario, para la subpoblación correspondiente a pacientes con MI no se hallaron mutaciones en el exón 10 de MPL, sino sólo la mutación JAK2 V617F y las más frecuentes en CALR. Este resultado podría estar sesgado al bajo número de pacientes incluidos en el estudio (n=6) y a la baja frecuencia de la mutación.
En cuanto a los pacientes TN, que no presentaron ninguna de las mutaciones estudiadas para ambas patologías, resta analizar por secuenciación el exón 9 del gen CALR completo para poder confirmar dicha clasificación, ya que hasta el momento sólo se analizó el hot-spot para las mutaciones reportadas.
La no detección de las mutaciones analizadas en el presente estudio no descarta la existencia de una entidad neoplásica por completo, dado que existen otros genes candidatos que podrían influir en la fisiopatogenia de las NMP. Sin embargo, en aquellos pacientes que requieren la toma de decisiones de manera urgente, la confirmación de la misma puede reducir el tiempo de espera para un tratamiento eficaz, previniendo al paciente de secuelas de gravedad.
La validación de protocolos basados en la técnica de HRM es un gran avance para la comunidad médica de la ciudad de Mar del Plata y la zona, dado que se trata de una herramienta para diagnóstico de vanguardia que aporta resultados precisos en pocas horas. Además, es importante destacar que mediante esta técnica las mutaciones pueden detectarse aún cuando se encuentran en baja carga alélica, y es posible hallar nuevas variantes no reportadas. En patologías que poseen síntomas clínicos que revisten gravedad es importante otorgar resultados certeros en poco tiempo, teniendo en cuenta las secuelas que se pueden generar en el paciente, como es el caso de las NMP, que presentan riesgo de trombosis y/o hemorragias.
AGRADECIMIENTOS
Los autores agradecen en la Agencia Nacional de Promoción Científica y Tecnológica de la República Argentina por la financiación de proyecto mediante su instrumento FONTAR ANR 600 BIO Bioingeniería 2010 Nº 018/10.
1. Swerdllow SH, Campo E, Harris NL. WHO classification of tumours of haematopoietic and lymphoid tissues. France: IARC Press; 2008. [ Links ]
2. Campbell PJ, Green AR. The myeloproliferative disorders. NEJM 2006; 355 (23): 2452-66. [ Links ]
3. Baxter EJ, Scott LM, Campbell PJ, East C, Fourouclas N, Swanton S, et al. Acquired mutation of the tyrosine kinase JAK2 in human myeloproliferative disorders. The Lancet 2005; 365 (9464): 1054-61. [ Links ]
4. Kralovics R, Passamonti F, Buser AS, Teo SS, Tiedt R, Passweg JR, et al. A gain-of-function mutation of JAK2 in myeloproliferative disorders. NEJM 2005; 352 (17): 1779-90. [ Links ]
5. Beer PA, Campbell PJ, Scott LM, Bench AJ, Erber WN, Bareford, D, et al. MPL mutations in myeloproliferative disorders: analysis of the PT-1 cohort. Blood 2008; 112 (1): 141-9. [ Links ]
6. Pietra D, Brisci A, Rumi E, Boggi S, Elena C, Pietrelli, A, et al. Deep sequencing reveals double mutations in cis of MPL exon 10 in myeloproliferative neoplasms. Haematologica 2011; 96 (4): 607-11. [ Links ]
7. Kaushansky K. Lineage-specific hematopoietic growth factors. NEJM 2006; 354 (19): 2034-45. [ Links ]
8. Pardanani A, Lasho TL, Finke C, Mesa RA, Hogan WJ, Ketterling RP, et al. Extending JAK2 V617F and MplW515 mutation analysis to single hematopoietic colonies and B and T lymphocytes. Stem Cells 2007; 25 (9); 2358-62. [ Links ]
9. Staerk J, Lacout C, Sato T, Smith SO, Vainchenker W, Constantinescu SN. An amphipathic motif at the transmembrane-cytoplasmic junction prevents autonomous activation of the thrombopoietin receptor. Blood 2006; 107 (5): 1864-71. [ Links ]
10. Vainchenker W, Constantinescu SN. JAK/STAT signaling in hematological malignancies. Oncogene 2013; 32 (21): 2601-13. [ Links ]
11. Klampfl T, Gisslinger H, Harutyunyan AS, Nivarthi H, Rumi E, Milosevic, J et al. Somatic mutations of calreticulin in myeloproliferative neoplasms. NEJM 2013; 369 (25): 2379-90. [ Links ]
12. Nangalia J, Massie CE, Baxter EJ, Nice FL, Gundem G, Wedge DC et al. Somatic CALR mutations in myeloproliferative neoplasms with nonmutated JAK2. NEJM 2013; 369 (25): 2391-405. [ Links ]
13. Rumi E, Pietra D, Ferretti V, Klampfl T, Harutyunyan AS, Milosevic JD et al. JAK2 or CALR mutation status defines subtypes of essential thrombocythemia with substantially different clinical course and outcomes. Blood 2014; 123 (10): 1544-51. [ Links ]
14. Liew M, Pryor R, Palais R, Meadows C, Erali M, Lyon E, et al. Genotyping of single-nucleotide polymorphisms by high-resolution melting of small amplicons. Clin Chem 2004; 50 (7): 1156-64. [ Links ]
15. Reed GH, Kent JO, Wittwer CT. High-resolution DNA melting analysis for simple and efficient molecular diagnostics. Pharmacogenomics 2007; 8 (6): 597-608. [ Links ]
16. Grupo Español de Neoplasias Mieloproliferativas Filadelfia Negativas (GEMFIN). Manual de recomendaciones en Neoplasias Mieloproliferativas Crónicas Filadelfia Negativas. Barcelona, España: Marketing Farmacéutico & Investigación Clínica, S.L.; 2014. [ Links ]
17. Quintana S, Schoenfeld E, Di Gerónimo V, Martín N, Pagani, F. Detección de la mutación V617F del gen JAK2 mediante análisis de disociación de alta resolución. Acta Bioquím Clín Latinoam 2014; 48 (4): 447-55. [ Links ]
18. Schoenfeld EA. Aplicación de la técnica de High Resolution Melting (HRM) para el estudio de mutaciones en los exones 14 y 12 del gen JAK2 en Síndromes Mieloproliferativos. FCEyN, UNMDP. Argentina, 2013. [ Links ]
19. Chi J, Nicolaou KA, Nicolaidou V, Koumas L, Mitsidou A, Pierides C, et al. Calreticulin gene exon 9 frameshift mutations in patients with thrombocytosis. Leukemia 2014; 28 (5): 1152-4. [ Links ]
20. Vossen RH, Aten E, Roos A, den Dunnen JT. High‐Resolution Melting Analysis (HRMA). More than just sequence variant screening. Human Mutation 2009; 30 (6): 860-6. [ Links ]
21. Tefferi A. The history of myeloproliferative disorders: before and after Dameshek. Leukemia 2008; 22 (1): 3-13. [ Links ]
Recibido: 31 de octubre de 2016
Aceptado: 7 de julio de 2017














