Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Acta bioquímica clínica latinoamericana
versión impresa ISSN 0325-2957versión On-line ISSN 1851-6114
Acta bioquím. clín. latinoam. vol.54 no.3 La Plata set. 2020
CARTA AL EDITOR
Conociendo al coronavirus SARS-CoV-2, protagonista de una pandemia
Knowing the SARS-CoV-2 coronavirus, protagonist of a pandemic
Conhecendo o coronavírus SARS-CoV-2, protagonista de uma pandemia
Manuel E. Cortés1a
1 Biólogo, Profesor de Estado en Química y Biología, Magíster y Doctor en Ciencias.
a Departamento de Ciencias Pedagógicas, Facultad de Educación; Programa de Doctorado en Educación, Universidad Bernardo O’Higgins, Avenida Viel # 1497, Santiago, Chile.
Correspondencia Dr. MANUEL E. CORTÉS Decano Interino Facultad de Educación Vicerrectoría Académica Universidad Bernardo O’Higgins, CHILE Tel.: +562 2477 2244 Correo electrónico: manuel.cortes@ubo.cl
Señor Editor:
La grave situación que enfrenta el mundo debido a la pandemia de enfermedad por coronavirus 2019 (COVID-19) requiere que todos los profesionales de la salud estén adecuadamente informados acerca de las características de esta enfermedad. Esta Carta al Editor resume de forma clara, pero rigurosa, las principales características del coronavirus 2 del síndrome respiratorio agudo severo (SARS-CoV-2), agente etiológico causante de COVID-19.
SARS-CoV-2 se clasifica como coronavirus (familia Coronaviridae) por su apariencia de “corona” en las microfotografías electrónicas y pertenece específicamente al género Betacoronavirus, subgénero Sarbecovirus (1). Sus viriones tienen un diámetro entre 60–140 nm, su genoma es ARN monocatenario de sentido positivo y posee envoltura (1). La secuencia de ARN de SARS-CoV-2 consiste en unos 29.870 nucleótidos de longitud (unos 9.744 aminoácidos) (1). El análisis de las secuencias genómicas completas de pacientes con COVID-19 demostró que la secuencia de SARS-CoV-2 comparte 79,6% de identidad con la secuencia del SARS-CoV, coronavirus causante del brote de síndrome respiratorio agudo severo (SARS) en 2003 (2). SARS-CoV-2 posee cuatro proteínas estructurales: la proteína S, homotrímero que forma las espículas en la superficie viral y es responsable de la unión a los receptores del hospedador (3); la proteína M, que tiene tres dominios transmembrana y da forma a los viriones, promueve la curvatura de la membrana y se une a la nucleocápside (3); la proteína E, importante para el ensamblaje y la liberación del virus, que participa en la patogénesis viral (3); y la proteína N, que contiene dos dominios, los cuales pueden unirse al ARN viral a través de diferentes mecanismos (3) (Fig. 1). Para infectar a las células, el dominio de unión al receptor de la proteína S de SARS-CoV-2 es reconocido por el dominio de la peptidasa extracelular de la enzima convertidora de angiotensina 2 (ACE2), que es su receptor (2) (4). Este reconocimiento principalmente se da a través de residuos polares (4).
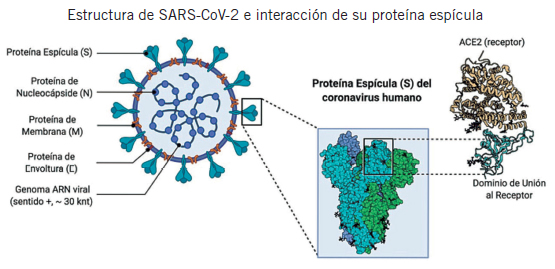
Figura 1. Estructura del coronavirus SARS-CoV-2 e interacción de la proteína espícula (S) con su receptor.
ACE2: enzima convertidora de angiotensina 2.
Figura creada con el software BioRender.com en base a lo reportado por Cascella et al. (10).
Se considera a SARS-CoV-2 como agente zoonótico. SARS-CoV-2 tiene un ancestro común con virus de murciélagos y es probable que su reservorio sean quirópteros, según lo reportado por estudios filogenéticos de secuencias de virus aislados de murciélagos (2) (5). También se ha propuesto a pangolines como hospederos intermediarios en base a análisis filogenéticos (6). Los resultados no han sido concluyentes al respecto, por lo cual las investigaciones continúan sobre este tema.
El contagio por SARS-CoV-2 produce la enfermedad conocida como COVID-19, para la cual algunas personas son asintomáticas. Para quienes presentan síntomas, los más habituales son fiebre, tos seca y cansancio (2). Éstos suelen ser leves y comienzan gradualmente. En algunos pacientes se han observado también síntomas gastrointestinales (7) y neurológicos (8). Cuando el paciente con COVID-19 se agrava, se observa un cuadro severo de síndrome respiratorio, caracterizado por gran dificultad para respirar, fiebre alta, dolor torácico, pulso irregular, entre otros síntomas y puede llegar a evolucionar a una falla multisistémica.
Finalmente, para retrasar la propagación de COVID-19, los profesionales sanitarios así como los organismos de salud públicos y privados deben poner especial énfasis en implementar y difundir las medidas que eviten más contagios. Según lo recomendado por la Organización Panamericana de la Salud, entre las precauciones estándar se destacan: la higiene de manos, el uso de equipo de protección personal adecuado (gorro, guantes de nitrilo y cubrebocas), prácticas de seguridad de inyección, una gestión segura de residuos, limpieza ambiental y esterilización de equipos de atención al paciente (9).
Fuentes de financiación
El presente trabajo fue realizado sin recibir una financiación específica.
Conflictos de intereses
El autor declara no tener conflictos de intereses respecto del presente trabajo.
1. Wang C, Liu Z, Chen Z, Huang X, Xu M, He T, et al. The establishment of reference sequence for SARS‐CoV‐2 and variation analysis. J Med Virol 2020; 92 (6): 667–74.
2. Zhou P, Yang X-L, Wang X-G, Hu B, Zhang L, Zhang W, et al. A pneumonia outbreak associated with a new coronavirus of probable bat origin. Nature 2020; 579 (7798): 270–3.
3. Chen Y, Liu Q, Guo D. Emerging coronaviruses: genome structure, replication, and pathogenesis. J Med Virol 2020; 92 (4): 418–23.
4. Yan R, Zhang Y, Li Y, Xia L, Guo Y, Zhou Q. Structural basis for the recognition of SARS-CoV-2 by fulllength human ACE2. Science 2020; 367 (6485): 1444-8. [ Links ]
5. Zhou H, Chen X, Hu T, Li J, Song H, Liu Y, et al. A novel bat coronavirus closely related to SARS-CoV-2 contains natural insertions at the S1/S2 cleavage site of the spike protein. Curr Biol 2020; 30 (11): 2196‐203.e3. [ Links ]
6. Lam TT-Y, Shum MH-H, Zhu H-C, Tong Y-G, Ni X-B, Liao Y, et al. Identifying SARS-CoV-2 related coronaviruses in Malayan pangolins. Nature 2020. In Press. DOI: https://doi.org/10.1038/s41586-020-2169-0. [ Links ]
7. Cortés ME. Síntomas digestivos en la enfermedad por coronavirus 2019 (COVID-19). Rev Gastroenterol Perú 2020; 40 (1): 100-1. [ Links ]
8. Baig AM, Khaleeq A, Ali U, Syeda H. Evidence of the COVID-19 virus targeting the CNS: tissue distribution, host–virus interaction, and proposed neurotropic mechanisms. ACS Chem Neurosci 2020; 11 (7): 995-8.
9. OPS. Instructivo - Lista de verificación de alistamiento para la respuesta al COVID-19 en Hospitales. Acciones de alistamiento en hospitales para la respuesta al COVID-19. Washington (DC): Organización Panamericana de la Salud (OPS/OMS); 2020. [ Links ]
10. Cascella M, Rajnik M, Cuomo A, Dulebohn SC, Di Napoli R. Features, evaluation and treatment coronavirus (COVID-19). En: StatPearls [internet]. Treasure Island (FL): StatPearls Publishing; 2020 Jan. 2020 May 18. [Fecha de acceso: 13 de junio de 2020]. Disponible en: https://www.ncbi.nlm.nih.gov/books/NBK554776/figure/article-52171.image.f3/ [ Links ]
Recibido: 19 de abril de 2020
Aceptado: 15 de junio de 2020














