Introducción
El control epidemiológico de las enfermedades metabólicas, inflamatorias y entéricas constituye uno de los principios más importantes en la prevención, dentro de los sistemas de salud y de las organizaciones sanitarias internacionales. La Organización Mundial de la Salud (OMS) y la Organización Panamericana de la Salud (OPS) se han referido al efecto que causan en la salud humana los desastres naturales, el propio hombre y el hacinamiento humano en campos de refugiados, identificados como algunos de los factores que contribuyen a la propagación de las epidemias (1).
Actualmente, el patrón bacteriano de un órgano o tejido se tiene en cuenta para analizar la etiología de las enfermedades, gracias al esclarecimiento del papel de las bacterias en las alteraciones moleculares en el sistema inmune y su aporte como probióticos en el reestablecimiento de los géneros bacterianos y levaduras, beneficiosos en el tracto gastrointestinal, como: Lactobacillus rhamnosus GG, Lactobacillus reuteri, Lactobacillus casei, Lactobacillus paracasei, Bacillus coagulans, Bacillus clausii, Bifidobacterium infantis, Bifidobacterium longum, Streptococcus thermophilus, Escherichia coli, Saccharomyces boulardii y Saccharomyces cerevisiae (2).
Después de más de dos décadas desde los primeros estudios genómicos de las comunidades bacterianas presentes en el intestino (como los descriptos por Zoetendal et al. (3), se han explorado los principales géneros y los mecanismos complejos en los que están implicados estos géneros, la posibilidad de su regulación a través del control de la expresión del llamado núcleo (core) de bacterias de mayor representación en el intestino y el papel del consumo de alimentos (4). Todo ello ha permitido el desarrollo de lo que por entonces se anticipaba con el término de la “era de la metagenómica”.
El objetivo de este trabajo de actualización fue agrupar y analizar algunas referencias que aportan elementos acerca del porqué de la estabilidad en el patrón de la microbiota intestinal individual y de la conservación de la tendencia en las poblaciones. Éste es un elemento a tener en cuenta en los estudios de vigilancia preventiva de las epidemias relacionadas con los cambios en este patrón.
Desarrollo
El tema de la microbiota intestinal tiene cada vez mayor importancia en la dilucidación de los complejos mecanismos etiológicos de las enfermedades y su connotación en el desarrollo de las epidemias. En el año 2011 comenzó uno de los primeros estudios de caracterización realizados en España, en el Hospital Comarcal de Inca (Islas Baleares), en colaboración con la Universidad de las Islas Baleares, acerca del patrón bacteriano presente en heces de sujetos sanos (grupo control), pacientes con diabetes mellitus tipo 2 y pacientes obesos (no diabéticos), a través de tecnologías metagenómicas para la identificación de los géneros bacterianos más frecuentes en el intestino humano. El grupo control incluyó sujetos en los que no se detectaron variables clínicas alteradas ni enfermedades de tipo digestivas inflamatorias y a los que no se les aplicaron tratamientos con antibióticos o medicamentos antiinflamatorios (5).
En general, los resultados de este trabajo constituyeron una evidencia más de que existe una estrecha relación entre la variedad y cantidad de bacterias intestinales con las variables clínicas características del estado sano y el estado patológico. Se identificaron algunos de los géneros bacterianos como posibles marcadores de diabetes y obesidad (Clostridium coccoides, Eubacterium rectale, Bacteroides spp. y Bifidobacterium spp.) y posibles marcadores en individuos sanos (Prevotella spp. y Lactobacillus spp.), así como su estrecha relación con el patrón de consumo de alimentos del llamado “estilo de consumo mediterráneo”. Este estudio multicomponente demostró un efecto dominante en cuanto a la variabilidad bacteriana (16,9% de varianza), representado por Clostridium coccoides, Eubacterium rectale, Bacteroides spp., Lactobacillus spp. y Bifidobacterium spp. Un segundo componente en orden de importancia, estuvo relacionado con las fuentes de prebióticos: (a) de fructo-oligosacáridos (FOS), los cuales pueden encontrarse en cebolla, ajo, cebolleta, endivias, puerros, arvejas, tirabeques, acelga, alcauciles, espárragos, tomates, leguminosas, plátanos, maíz, papas, soja en grano, pipas de girasol, avellanas, almendras, nueces, castañas de cajú y maníes (b) de manano-oligosacáridos (MOS), presentes en productos fermentados como cerveza, aceituna, vino y salsa de soja y (c) con fuentes de grasas poliinsaturadas y monoinsaturadas (5).
El análisis mostró un tercer componente en orden de importancia que incluyó la fuente animal (carne roja, carne blanca y la grasa de origen animal) y el consumo de isomalto-oligosacáridos (IMOS; harina de trigo, de centeno, de avena y de maíz) (5).
El desarrollo posterior de las herramientas metagenómicas (metodologías desarrolladas por la plataforma Illumina: 16S Amplicon y XT Nextera Metagenomic), a través de la preparación de bibliotecas genómicas permitió ampliar la identificación de 451 géneros en una sola secuenciación masiva, lo cual fue aprovechado por este grupo de investigación para ampliar el estudio hacia las enfermedades inflamatorias intestinales. Martínez-Cabrera et al. (6) caracterizaron la abundancia bacteriana en la enfermedad de Crohn, comparada con colitis ulcerosa y presencia de pólipos colorrectales. Se tomaron muestras de diferentes zonas transversales del intestino (lumen y mucosa en la misma área sana; o lumen y mucosa en la misma área afectada) y longitudinales (muestras tomadas a lo largo del intestino donde se habían visualizado afectaciones y se seleccionaron muestras de zonas sanas en el lado contrario, anteriores o posteriores a las afectadas), a partir de los estudios realizados en colitis ulcerosa por Lavelle et al. (7).
Los resultados obtenidos por Martínez-Cabrera et al. (6) no permitieron hablar de un núcleo o marcadores bacterianos (por el número reducido de pacientes), pero proporcionaron algunas recomendaciones acerca del uso de estos métodos para el seguimiento complementario a los estudios de colonoscopía.
Actualmente, se refiere a la microbiota como uno de los ecosistemas más importantes en la regulación, extensión de las epidemias y protección frente a las mismas. No sólo las epidemias producidas por patógenos son el resultado del desequilibrio en este ecosistema; también deben considerarse las producidas por trastornos metabólicos o inflamatorios. La obesidad es una enfermedad metabólica con implicaciones etiológicas complejas que ha adquirido connotaciones epidémicas, sobre todo en los países de estilo de vida y alimentación de tipo “occidental”. Esta patología y su relación con determinados patrones de microbiota intestinal presentes en estas poblaciones dependen de sus orígenes étnicos, edad o estatus socioeconómico. Existen evidencias de que algunos genes humanos están asociados con la abundancia de determinados géneros de bacterias intestinales, lo cual provee a la mucosa de una mayor susceptibilidad para tolerar determinadas bacterias favorables o no al desarrollo de estas patologías (8).
Las bases genéticas de las enfermedades de tipo inflamatorio pulmonar y entérico se relacionan con la capacidad del organismo humano a tolerar y/o protegerse de determinados desbalances de la microbiota a través de los mecanismos de homeostasis (9) (10) (11). Tomando como eje fundamental a la homeostasis-microbiota, podrían explicarse las causas por las que determinadas enfermedades pueden convertirse en epidemias, e incluso, en pandemias.
Control homeostático inmunitario de la estabilidad de un determinado patrón de microbioma intestinal
Se conoce como microbiota intestinal al conjunto de comunidades de bacterias, hongos, arqueas y virus que forman parte del intestino humano y animal, tanto en la mucosa como en el lumen intestinal. La determinación de los genomas colectivos presentes se conoce como microbioma intestinal (9).
En el organismo humano (hospedador) se presenta un sistema de regulación que garantiza una determinada estabilidad y convivencia simbiótica, en cuanto al tipo de microbio comensal (especies, géneros, familias, filos) y cantidad de estos, conocido como mecanismo de “homeostasis inmunitaria”. Este sistema está destinado a mantener la estabilidad funcional en el hospedador a través del balance entre vigilancia y tolerancia en presencia de microbios comensales, agentes infecciosos y patógenos oportunistas presentes en los tejidos y órganos. Este sistema vincula a los factores genéticos, inmunológicos, metabólicos, hormonas, señalizadores, receptores y macroestructuras de las membranas, entre otros (9).
Este sistema está sujeto, además, a variaciones externas al hospedador como factores socio-demográficos, edad y estilo en el consumo de alimentos, lo cual determina el origen y estabilidad de un determinado patrón de microbiota intestinal, beneficioso para el hospedador y para los microorganismos (9).
Existen dos ejes que relacionan los principales sistemas de órganos importantes para el control de la microbiota a través de un mecanismo homeostático eficiente coordinado en el hospedador: el sistema gastrointestinal y el respiratorio y el gastrointestinal y el sistema nervioso central que conforman los llamados “eje intestinopulmón” y “eje intestino-cerebro” (9) (10) (12).
Para conocer cómo opera este mecanismo de regulación es necesario tener en cuenta los orígenes de un determinado perfil de microbiota. Los inicios se atribuyen al nacimiento del niño porque se ha descripto que, en estado fetal, los pulmones están llenos de líquido amniótico estéril libre de microbios y que una vez nacido el niño, las bacterias colonizan las mucosas de una manera homogénea, hasta que con el crecimiento, se diferencian por comunidades de órganos (10). Esta adquisición de la microbiota por el niño, en una primera instancia, se produce a través del canal del parto de la madre en el nacimiento natural o por la vía de la cesárea (10), lo cual será analizado en esta revisión.
En condiciones normales, la mucosa intestinal adquiere una determinada carga microbiana, de manera que la proximidad física entre estas poblaciones presentes en el tejido del hospedador, favorece el intercambio metabólico y brinda oportunidades inmunológicas para beneficio de éste; sin embargo, al mismo tiempo, puede constituir una amenaza constante a la salud humana (9). Esta carga microbiana favorable, en términos de cantidad y diversidad, se distribuye desde el lumen hacia la mucosa (11). La distribución morfológica de la multicapa intestinal permite establecer una barrera para la convivencia de las especies microbianas, sin atravesar el epitelio intestinal, lo cual evita la invasión microbiana a tejidos profundos del hospedador (11) (13).
Una gran franja de moco (compuesto por mucina) separa la zona luminal (lumen) del epitelio en los sujetos sanos, hacia la que se secretan diariamente las IgA (lgAs), en la primera línea de defensa inmune. Esta IgAs actúa como centinela y agente neutralizante (11) (13).
Los microbios beneficiosos producen factores y metabolitos solubles secretados hacia la mucosa intestinal (como ácidos grasos de cadena corta y vitaminas), utilizando sustratos de la dieta. Estos compuestos bioactivos afectan la función del epitelio intestinal y las células inmunitarias de la mucosa (células dendríticas y macrófagos) que se activan y el resultado será la producción de citoquinas y factores relacionados (el ligando inductor de la proliferación y el factor de activación de células B) (11) (13). Todos estos mediadores e inmunoglobulinas son importantes para mantener sano al hospedador o en estado de portador, sin desarrollar una determinada enfermedad.
La lgAs es parte del mecanismo homeostasis-simbiosis o mecanismo de exclusión inmune que evita la interacción de los antígenos neutralizados (toxinas, virus y bacterias patógenas) con el epitelio, lo cual contribuye a aliviar la carga funcional del sistema inflamatorio y evita daños epiteliales. A la lgAs se le atribuye, además, una función inmunopotenciadora, inductora de respuestas inmunes efectoras en un contexto no inflamatorio favorable para preservar la homeostasis local (11) (13).
Otros de los guardianes presentes en el moco son las α-defensinas (expresadas en las células de Paneth del intestino), a las que se les atribuyen múltiples funciones bactericidas o bacteriostáticas. Algunos tipos, como HD5, proceden de forma similar a los péptidos de neutrófilos ya que interactúan con las membranas celulares bacterianas y las alteran, pero también se les atribuyen efectos en el citoplasma de la célula blanco. En cambio, el tipo HD6 se autoensambla espontáneamente en nanorredes de péptidos múltiples, al entrar en contacto con estructuras bacterianas, como flagelos y fimbrias. Esta función le confiere a la defensina HD6 un papel clave en la agregación y el secuestro de bacterias que ingresan a las criptas del intestino delgado y así previene la muerte celular directa, como si HD6 detuviese a las bacterias (14).
Pero la vigilancia inmunitaria del hospedador hacia la comunidad comensal intestinal no se reduce a esta primera línea de defensa. Implica, además, el reconocimiento de una diversidad de patrones moleculares asociados a patógenos (PAMP) como el lipopolisacárido (LPS) y otros componentes de la pared celular bacteriana a través de receptores tipo Toll (TLR), que median la tolerancia a la microbiota, y de proteínas de dominio de oligomerización de unión a nucleótidos (NOD) que son mediadoras en la activación de las α-defensinas (9).
Estos receptores actúan en distintos compartimentos celulares y se sitúan en la superficie epitelial expuesta hacia el lumen para promover la tolerancia y la respuesta inflamatoria saludable. Sin embargo, su activación en la superficie basolateral de los colonocitos conduce a fuertes respuestas proinflamatorias. La interacción de algunos ligandos microbianos presentes, como por ejemplo, en especies de Salmonella no patógenas, Bacteroides thetaiotaomicron, Lactobacillus casei, Clostridium spp. y Bacteroides fragilis, entre otros, actúa como moduladora favorable de la homeostasis inmunitaria (9).
En la Figura 1 se resume solo una parte del complejo mecanismo, referido por varios autores, destinado a mantener el control homeostático recíproco durante la convivencia entre el hospedador y la microbiota, así como el alcance de este mecanismo en otros sistemas de órganos que componen los ejes con el intestino (15) (16) (17).
Skelly et al. (15) explicaron ampliamente los mecanismos que garantizan el equilibrio entre las respuestas antiinflamatorias y proinflamatorias, inducidas por algunos géneros de la microbiota en el intestino, lo cual permite interpretar el importante papel de algunas poblaciones microbianas que pueden autorregular su representación e inhibir o activar estas vías de respuesta del hospedador, manteniendo el balance entre estas dos respuestas inmunoinflamatorias.
Ejemplos de la inmunomodulación antiinflamatoria
La presencia de especies bacterianas como B. infantis promueven la expresión de células dendríticas (CD) productoras de la enzima retinaldehído deshidrogenasa 2 (Aldh1a2), responsable de la producción de altos niveles de ácido retinoico (AR), principal precursor de la vitamina A. El AR, junto con el factor de crecimiento transformante β (TGF-β), promueve la diferenciación de células T vírgenes en células que expresan la proteína forkhead P3 (FOXP3) y en células T reguladoras (Treg). FOXP3 es el principal factor de transcripción que controla la función de las células T reguladoras (células Treg o CD4+) y puede inhibir la transcripción de genes clave tras la estimulación de los receptores de las células T. Por su parte, AR y TGF-β también regulan positivamente la expresión de los receptores de alojamiento integrina α4β7 y el receptor 9 de quimioquinas (CCR9), cuando las células T efectoras (CD4+ y CD8+) y de memoria, migran a la mucosa intestinal, favoreciendo el tropismo intestinal. Esto se traduce en la inhibición de una fuerte respuesta inmunitaria del hospedador frente a inmunógenos dietéticos y microbios comensales (15).
La disponibilidad epitelial de los precursores vitamínicos o de las vitaminas proporcionados al hospedador a través de la dieta o disponibles a partir de suplementos, cuya absorción está regulada por algunos géneros bacterianos ha sido uno de los objetivos de análisis para la dilucidación de las causas de algunas enfermedades, tratamiento y prevención (18).
En una revisión realizada por Yamamoto y Jørgensen (18), se sostiene que la disponibilidad de vitamina D se ha relacionado clásicamente con la regulación de la transcripción genómica involucrada en los mecanismos homeostáticos del calcio y el crecimiento óseo, a través de la vía del receptor de unión a la vitamina D (VDR), lo cual se asocia con la inhibición de las respuestas Th17 y Th1 (respuesta proinflamatoria) y con la estimulación de las células Treg con dominio FOXP3+ (respuesta antiinflamatoria). Por otra parte, algunos de los trabajos analizados en esta revisión, se refirieron al incremento de Prevotella presente en heces de sujetos sanos con una mayor ingesta de vitamina D, mientras que la representación fue menor en géneros como: Haemophilus (Proteobacteria) y Veillonella (Firmicutes). Sin embargo, los revisores hacen énfasis en que el tipo de muestra analizada puede ser importante en la dilucidación de estas asociaciones, poniendo mayor interés en muestras de biopsias de endoscopías y colonoscopías que en muestras de heces.
Se conoce poco acerca de los efectos directos de la vitamina D sobre las bacterias. Yamamoto y Jørgensen (18) identificaron un único estudio que demostró que esta vitamina inhibía el crecimiento in vitro de especies de micobacterias específicas. Según estos autores, si se corrobora este hallazgo, los efectos antimicrobianos de la vitamina D serían compatibles con las propiedades inmunorreguladoras conocidas y, si no, sería probable que la microbiota estuviese regulada, indirectamente, por las propiedades inmunológicas de la vitamina D. No obstante, los autores argumentaron que las bacterias influyen también en el metabolismo de la vitamina D, de manera que algunas bacterias expresan enzimas involucradas en la hidroxilación de esteroides y, por lo tanto, son capaces de procesar y activar la vitamina D de forma similar a como ocurre en los seres humanos (como por ejemplo, Streptomyces griseolus) (18).
Otro caso de inducción de este tipo de la respuesta antiinflamatoria del hospedador lo promueve el polisacárido A (PSA) de la cápsula de B. fragilis. Esta molécula puede ser transportada formando parte de vesículas de membrana externa hacia la lámina propia intestinal, a través de una proteína relacionada con la autofagia (ATG16L1) y una proteína que contiene el dominio de oligomerización de unión a nucleótidos (NOD2). El PSA interactúa con el receptor tipo Toll 2 (TLR2) presente en las células Treg con dominio FOXP3+, para inducir su proliferación y la producción de IL-10, promoviendo así un estado antiinflamatorio, favorable en la protección contra colitis y enfermedad de Crohn (15).
Estos efectos antiinflamatorios se pueden lograr, además, mediante ácidos grasos de cadena corta (AGCC), como butirato y propionato, productos del metabolismo bacteriano en el intestino, los cuales son aportados a la célula epitelial del hospedador por consorcios de cepas de Clostridium. El butirato inhibe las histonas desacetilasas, lo que aumenta la transcripción en el promotor FOXP3. Las señales de propionato producen este efecto a través de GPR43, cuyo resultado final es la producción de IL-10 antiinflamatoria (15).
El butirato interviene en otro mecanismo de respuesta antiinflamatoria, mediante el cual promueve la polarización de macrófagos de tipo M2, regulando el incremento de la expresión de la enzima arginasa 1 (ARG1), lo que finalmente promueve la disminución en la producción del factor de necrosis tumoral (TNF) y la expresión de NOS2, IL-6 e IL-12b (15).
Es precisamente éste uno de los puntos de regulación importantes en el dominio de una u otra respuesta inflamatoria: la activación alternativa de un tipo u otro de macrófago. Los macrófagos tienen una alta adaptabilidad funcional que les permite manifestar propiedades proinflamatorias o antiinflamatorias, en respuesta a varias citoquinas y productos microbianos. Los macrófagos se dividen en dos grupos: los de fenotipo M1 activados clásicamente (estimulados por IFN-γ y LPS) que ejercen efectos proinflamatorios y los de fenotipo M2 activados alternativamente (que son estimulados por IL-4 o IL-13), los cuales realizan funciones antiinflamatorias (19).
Estos mecanismos de alternancia están relacionados con el origen y evolución de muchas enfermedades inflamatorias. Se ha demostrado que en la obesidad los macrófagos del tejido adiposo cambian del macrófago polarizado fenotipo de M1 al macrófago polarizado fenotipo de M2, reduciendo así las señales inflamatorias derivadas del tejido adiposo. Algunas enfermedades como la artritis reumatoidea, están estrechamente relacionadas con el desequilibrio de los macrófagos M1 y M2 y podrían atenuarse restableciendo el equilibrio M1/M2. De la misma manera, se ha demostrado que en modelos de ratones, la enfermedad del intestino inflamado podría mejorarse, cambiando del fenotipo proinflamatorio (M1) al antiinflamatorio (M2) (19).
El mecanismo homeostático en el cual está involucrado el aminoácido esencial triptófano (Trp) procedente del consumo de alimentos, es otro de los ejemplos de regulación de la respuesta antiinflamatoria. Su metabolismo en el intestino incluye la transformación directa de Trp por bacterias intestinales (L. reuteri y Lactobacillus murinus) en varias moléculas, como el indol y sus derivados. Estos derivados del indol envían señales, a través del receptor de hidrocarburos arilo (AhR) presentes en los linfocitos intraepiteliales CD4+ para regular la reducción en la expresión del factor T helper inductor de POZ/Kruppel-like (ThPOK) y así inducir la expresión de RUNX3, lo que facilita la diferenciación en CD4+ y en CD8αα+ (15).
La señalización de AhR se considera un componente clave de la respuesta inmune en los puntos cuya función es de barrera, crucial para la homeostasis intestinal al actuar sobre la renovación epitelial, la integridad de la barrera y numerosos tipos de células inmunes (linfocitos intraepiteliales, células Th17, células linfoides innatas, macrófagos, células dendríticas y neutrófilos) (20).
Se han identificado algunas vías metabólicas de Trp en otros miembros de la microbiota intestinal humana, como Clostridium sporogenes, capaz de descarboxilar al Trp y conducir a la producción del neurotransmisor triptamina. En esta especie también se han descriptovías oxidativas y reductoras que producen ácido indolacético y ácido indolpropiónico, dos metabolitos de Trp que afectan la permeabilidad intestinal y la inmunidad del huésped. Contribuyen a este tipo de regulación los transportadores activos de Trp e indol identificados en E. coli, así como la triptofanasa (expresada en E. coli y Lactobacillus spp.) que convierte Trp en indol (20).
En las últimas décadas, se ha demostrado la relación entre la microbiota intestinal y la etiología de enfermedades asociadas con los estilos de vida occidentales, con los productos finales del metabolismo de Trp y con deficiencias de Trp, como: enfermedades inflamatorias del intestino (enfermedad de Crohn y colitis ulcerosa), síndrome del intestino irritable (SII), síndrome de las enfermedades metabólicas y complicaciones asociadas (diabetes, obesidad, hígado graso no alcohólico, resistencia a la insulina y aterosclerosis) y rasgos neuropsiquiátricos (en particular, ansiedad, depresión y autismo) (20).
Skelly et al. (15) se refirieron a otros copartícipes de las respuestas antiinflamatorias, como: Bifidobacterium bifidum. Esta bacteria produce polisacáridos de β-glucano/ galactano (CSGG) hacia la superficie epitelial, lo cual estimula señales gracias a su interacción con TLR2 presente en las células dendríticas (CD) y las acondicionan para inducir células Treg FOXP3+. Estas últimas son capaces de suprimir la producción de IL-2, IL-4 e IFN-γ (15).
Con algunas similitudes a este mecanismo descripto, pero con mayor potencia, Clostridium ramosum y algunas especies de Helicobacter inducen la diferenciación de células Treg FOXP3+ para expresar el receptor huérfano de retinoides (RORαt). El resultado de esta última inducción es la inhibición de las respuestas Th1, Th2 y Th17. Esta ruta podría estar relacionada con el componente inflamatorio en algunas enfermedades intestinales (15).
Ejemplos de inmunomodulación proinflamatoria
Existen consorcios bacterianos (bacterias filamentosas segmentadas) que se adhieren al epitelio intestinal para inducir al factor de transcripción C/EBPδ (que interviene en procesos inflamatorios, de inducción de la proliferación celular, diferenciación y estabilidad genómica), responsable de que aumente la expresión de la proteína amiloide A sérica (sintetizada fundamentalmente en el hígado, en respuesta a las citoquinas liberadas por los macrófagos en fase aguda de infección). La proteína amiloide A sérica induce las células T helper 17 (Th17) y produce un incremento en la producción de IL-17. A su vez, la proteína amiloide A sérica estimula a las CD para producir IL-1β, mientras que IL-1β establece un bucle de amplificación de la proteína amiloide A sérica. Este fragmento de regulación está relacionado con la señalización que generan ambos tipos de bacterias (filamentosas segmentadas y no filamentosas segmentadas), a través de la producción y señalización de IL-23 hacia las células linfoides innatas del grupo 3 (ILC3), para que produzcan IL-22. Esta última IL-22 envía señales por la vía del activador de la transcripción 3 (STAT3) presente en las células epiteliales para inducir, aún más, la expresión de amiloide A sérica y Th17 proinflamatoria. El resultado de este último mecanismo podría ser, por una parte desfavorable si se produjese la exacerbación de la artritis autoinmune o, por otro lado favorable, relacionado con la protección ante las infecciones enteropatogénicas (15).
Una gran variedad de géneros de bacterias se han asociado con una intensa respuesta proinflamatoria, entre las que se destaca Bifidobacterium adolescenti. En un caso descripto de un paciente en el que se aisló esta bacteria, se relacionó la adhesión epitelial bacteriana con la inducción de células Th17 y la citoquina IL-17 (15).
A través de un mecanismo similar, algunas especies de Helicobacter promueven este efecto con la consiguiente exacerbación de enfermedades autoinmunes. Sin embargo, Prevotella copri lo hace a través de la producción de succinato, destinado a la inducción de señales por mediación del receptor GPR91 presente en las CD, con el objetivo de mejorar las respuestas inmunitarias específicas de antígeno y las respuestas dirigidas por TLR (15).
A través de otros mecanismos muy diferentes a los referidos, Bilophila wadsworthia reduce la taurina desconjugada a sulfito, que utiliza para el metabolismo y la inducción de células Th1 productoras de IFN-γ, responsables de la exacerbación de la colitis. Con la presencia de Klebsiella pneumoniae en el intestino, el resultado es el mismo, pero de una manera dependiente de los receptores CD11b-CD103 representados en las CD dependientes de TLR y de IL-18 (15).
Como se puede constatar, las bacterias utilizan diferentes mecanismos de regulación de la respuesta inflamatoria en el hospedador de manera que exista un equilibrio entre ambos tipos de inmunomodulación (Fig. 1). Sin embargo, no son menos importantes los mecanismos involucrados en el reconocimiento, tolerancia y protección frente a los hongos que forman parte de la microbiota (Fig. 1).
Si bien los principales brotes epidémicos registrados en el mundo han sido por causas bacterianas o víricas (https://www.who.int/csr/don/archive/year/es/) una de las infecciones más frecuentes en los pacientes hospitalizados con enfermedades de alta complejidad, ha sido a causa del hongo Candida albicans. Esta infección fúngica en el sistema nervioso central (SNC) constituye una complicación potencialmente mortal que agrava el pronóstico de los pacientes (21).
En revisiones realizadas por algunos autores (21) (22) se analizaron y propusieron las principales vías homeostáticas de control de las poblaciones fúngicas comensales e invasoras, relacionadas con el eje intestino- cerebro (Fig. 1). En primer lugar, los receptores de patrones de reconocimiento (PRR) y los PAMPs son importantes en el mecanismo de señalización en infecciones fúngicas sistémicas y en las mucosas. Algunos factores de riesgo intrínsecos están determinados por defectos genéticos innatos en las células efectoras antifúngicas del huésped o en moléculas de señalización, que resultan relevantes en la defensa de diferentes órganos frente a hongos oportunistas. Estas alteraciones involucran mutaciones en los genes que codifican la síntesis de enzimas que se encuentran en los lisosomas de los neutrófilos y que se asocian con la candidiasis diseminada, las cuales conducen a fallas en la fagocitosis, a trastornos en la señalización de citoquinas (como IL- 17), o a defectos en la vía Dectina-1/CARD9 (21) (22).
Los receptores más conocidos en la respuesta frente a patógenos fúngicos son los de lectina tipo C (conocidos como PRR) que reconocen a hidratos de carbono como mananos, glucanos y quitina, presentes en la pared celular de los hongos. Estos receptores son necesarios para el reconocimiento y la fagocitosis, la inducción de mecanismos efectores antimicrobianos y de mediadores inflamatorios que dirigen y modulan la inmunidad adaptativa, incluidas las respuestas Th1 y Th17. Los principales miembros de este grupo de receptores que participan en la actividad antifúngica son: las familias de Dectina, el receptor Mincle, el CR3, el receptor de manosa y el DC-SIGN, que se expresan principalmente en células mieloides. Algunos trastornos en la expresión del receptor Dectina-1 han contribuido a deficiencias en el reconocimiento de los glucanos de la pared de C. albicans, lo que provocaba una disminución en la secreción de citoquinas, principalmente IL-17, TNF e IL-6 (21).
Muchas de las respuestas antifúngicas dependen exclusivamente de la activación de la proteína tirosinaquinasa no receptora (SYK) presente en células mieloides del intestino y, además, de la proteína adaptadora CARD9. La molécula adaptadora CARD9 interviene en la respuesta antifúngica frente a C. albicans, al ubicarse en la cascada de señalización por debajo de la quinasa SYK. La molécula CARD9 constituye una vía común a los receptores de lectinas de tipo C y se vincula con la vía de activación del factor nuclear kappa B (NF-κB) que permite el equilibrio entre respuestas proinflamatorias y antiinflamatorias. Algunas fallas en este mecanismo están relacionadas con el desarrollo de enfermedades digestivas inflamatorias como la colitis ulcerosa y la enfermedad de Crohn (21).
El micobioma intestinal es otro miembro esencial del eje intestino-cerebro, junto al bacterioma. El SNC puede influir en la composición de este micobioma, a través del eje hipotalámico-pituitario-suprarrenal (HPA), estrechamente asociado con la dinámica gastrointestinal y la hipersensibilidad colónica. A su vez, el micobioma puede favorecer la liberación de neuromediadores para reaccionar sobre el SNC e intervenir en procesos cognitivos, en respuestas de estrés, excitabilidad neuronal y en las vías de activación inmuno-neuroendocrina (21).
Gracias a todos estos mediadores de la respuesta inmune que son transportados hacia los sistemas de órganos componentes de estos ejes a través de los sistemas linfático y circulatorio o generados localmente por mediación de la microbiota intestinal, es posible lograr la respuesta sistémica en el órgano, ante los cambios o presencia de antígenos (21) (Fig. 1).
Si uno o varios de estos mecanismos homeostáticos estabilizadores fallasen por causas endógenas (como la edad del individuo) o exógenas (como radiaciones, quimioterapias o antibióticos, variaciones en la ingesta de alimentos, estrés, infecciones respiratorias e intestinales, entre otras) el resultado sería la alteración de las poblaciones de la microbiota intestinal (disbiosis), lo cual facilitaría la translocación de patógenos hacia el interior del hospedador y produciría un incremento en la respuesta proinflamatoria asociada con un gran número de enfermedades y un alto grado de riesgo frente a la virulencia en las infecciones (9).
Se ha identificado un amplio espectro de microbios oportunistas que se aprovechan de las posibles fallas en los mecanismos homeostáticos. En este amplio espectro de acción, se ha demostrado que la alteración en el contenido de hongos comensales intestinales puede ser una de las causas o consecuencias de la infección por parásitos platelmintos. Una gran diversidad de poblaciones de hongos intestinales ayudan directa o indirectamente a mantener una homeostasis intestinal saludable, de tal manera que su disbiosis tiene consecuencias inmunológicas. Con relación a este tema, Osakunor et al. (23) realizaron su contribución acerca de la posible asociación de géneros específicos de bacterias y hongos con la infección por el parásito Schistosoma haematobium en niños con edad preescolar (de 5 años o menores), en zonas de Zimbabue (África).
Estos autores refirieron que el aumento de Aspergillus se ha asociado con el incremento en los niveles de eosinófilos y una respuesta Th2 exagerada, una de las características también similares a la infección causada por Schistosoma (gusanos platelmintos que causan un gran número de muertes en África subsahariana). El incremento de los niveles de los hongos Aspergillus, Tricholoma y Periglandula estuvo acompañado del incremento de bacterias Pseudomonas, Stenotrophomonas, Derxia y Thalassospira y la disminución de Azospirillum, en los niños afectados. En todos los casos, la variación de estas poblaciones microbianas fue consistente con la intensidad de la infección provocada por el gusano. Las especies de Schistosoma más comunes son la forma urogenital (S. haematobium) e intestinal (Schistosoma mansoni). La sintomatología de la enfermedad se debe, principalmente, a reacciones inmunológicas frente a los huevos del gusano que intenta migrar a la vejiga o al lumen intestinal, según la especie involucrada. La infección causa efectos inmunomoduladores que ayudan a promover la supervivencia del parásito en el huésped y las consecuencias pueden provocar la desnutrición, deficiencias en el crecimiento y la cognición, la reducción en el efecto protector de las vacunas y el pronóstico alterado de las coinfecciones (23).
Acerca del control homeostático frente a los virus presentes de manera habitual en la microbiota intestinal, quedan aspectos en los que se continúa investigando.
¿Existen también virus comensales, identificados como componentes de un núcleo o viroma? Puede existir un llamado viroma al que se refieren Shkoporov et al., en estrecha relación con un determinado bacterioma (24). Estos autores realizaron un estudio metagenómico de la población de bacteriófagos fecales presentes en seres humanos encontrados en 9 adultos sanos. La colección de las muestras se realizó durante 12 meses en todos los sujetos y durante 26 meses en uno de ellos. El estudio demostró que la composición del viroma intestinal humano (en términos de diversidad-α y recuentos virales) fue significativamente estable a lo largo del tiempo y muy variable entre los individuos. A esta variabilidad la denominaron viromas personales persistentes. En este trabajo se identificó un núcleo filogenético de 22 grupos virales, compuesto por fagos similares a los crAss (fagos presentes en aguas contaminadas por heces humanas), por bacteriófagos de ADN bicatenario del orden Caudovirales y por fagos virulentos de ADN monocatenario de la familia Microviridae (bacteriófagos virulentos). Como resultado de este estudio, se postuló la posibilidad de que los fagos similares a crAss podrían ser un elemento importante en el viroma intestinal, dada la probabilidad de ser compartidos entre individuos y dominar el pequeño núcleo filogenético de los virus (24).
Las correlaciones estrechas observadas en cuanto a la composición microbiana entre microbiomas bacterianos y viromas, se evidenciaron de manera inversa entre los altos niveles relativos de Prevotella y los bajos niveles virales totales con una mayor diversidad-α del viroma. A partir del análisis de las coincidencias de la secuencia espaciadora CRISPR (secuencias de ADN de bacteriófagos presentes en el genoma y utilizado por las bacterias como uno de sus mecanismos de defensa desarrollados contra bacteriófagos) se demostró que los fagos infectaban a las bacterias abundantes y persistentes de los géneros Bacteroides, Faecalibacterium, Eubacterium, Prevotella y Parabacteroides (24).
Los mecanismos homeostáticos respondedores ante una posible disbiosis relacionada con algunos fagos Caudovirales se resumen en dos posibles vías principales de estimulación del sistema inmune (25): una primera vía en la que el bacteriófago lisa a las bacterias en el lumen intestinal y son liberados productos proinflamatorios; mientras que en la segunda posibilidad, los fagos transitan a través del epitelio intestinal y estimulan directamente el sistema inmunológico.
Los resultados obtenidos por Gogokhia et al. (26) permitieron proponer, además, un modelo en el que se sugirió que los fagos del orden Caudovirales (con la cápside intacta) inducen la producción de IFN-γ en células dendríticas y en células T CD4 +; por otra parte, una amplia gama de fagos Caudovirales purificados de E. coli, Lactobacillus plantarum o B. thetaiotaomicron estimularon la producción de citoquinas a partir de las células inmunes in vitro. La segunda alternativa de estimulación en este modelo sugirió que el ADN del fago era capaz de estimular la producción de IFN-γ por las células inmunes, pero no las cápsides vacías del fago sin ADN. También se observó la presencia de ADN de fagos marcados dentro de los endosomas de las células inmunes lo que permitió sugerir que el receptor tipo Toll 9 (TLR9), situado dentro de los endosomas, era esencial para la producción de citoquinas mediadas por fagos, por la vía de la proteína adaptadora MyD88. Esta proteína se sitúa por debajo de la mayoría de los TLR. En este modelo, el ADN del fago desencadena la activación de células inmunitarias dependientes de TLR9/MyD88 (25) (26).
Por todas estas razones y para comprender la posible relación entre las variaciones en el perfil microbiano intestinal, el origen, evolución, erradicación y prevención de las epidemias con bases etiológicas metabólicas, inmunológicas e infecciosas se referirán algunos de los factores relacionados con estos cambios.
Conformación de un patrón de microbioma intestinal en seres humanos
Hasta hace poco tiempo se hablaba del estado saludable del individuo y no se hacía referencia al estado saludable del microbioma para formar un sistema mutualista con el ser humano. Cuando las condiciones fisiológicas y morfológicas del individuo son favorables para que la microbiota esté en equilibrio mutualista con él, se establece una relación beneficiosa para ambos. Se requiere una microbiota intestinal estructurada y equilibrada en los consorcios fermentativos, para garantizar un estado de salud óptimo, mantener el equilibrio en el sistema inmune y evitar la presencia de patógenos (27) (28) (29).
La microbiota proporciona la posibilidad de transformar y absorber moléculas que no son producidas por el organismo o que no pueden ser transportadas naturalmente a través de la mucosa del intestino del ser humano. Estas habilidades proporcionan un metaboloma saludable al aumentar las concentraciones de ácidos grasos de cadena corta (ácidos pirúvico, cítrico, fumárico y málico), marcadores del incremento en el metabolismo energético en el intestino y protectores de los colonocitos contra eventos carcinogénicos (30) (31) (32).
La microbiota intestinal se conforma a partir del nacimiento del niño, crecimiento y envejecimiento en el adulto, gracias a variables internas y externas en el individuo.
Contribución de la madre al patrón microbiano del niño, durante el embarazo y el posparto
Un niño será saludable si desde el embarazo las condiciones prenatales de la madre son adecuadas. El conocimiento acerca de las variaciones en el patrón bacteriano durante la gestación, nacimiento, lactancia y primeros años de vida del niño, es importante para la comprensión de la etiología de algunas de las enfermedades que se manifiestan desde la niñez así como el surgimiento de epidemias en infantes y jóvenes (33) (34).
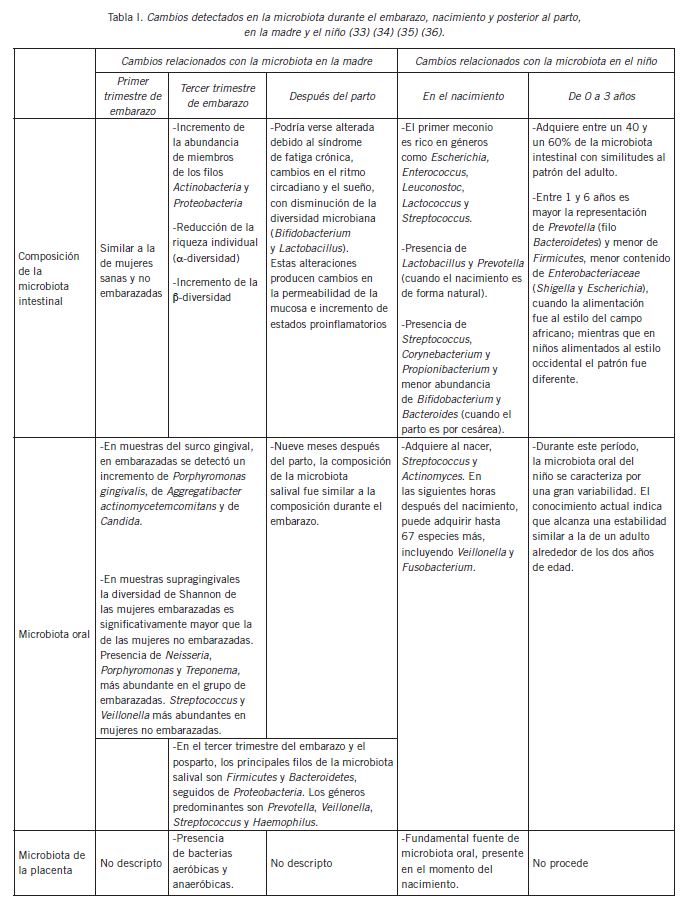
En la revisión realizada se han encontrado, fundamentalmente, referencias relacionadas con la evolución del patrón bacteriano durante la gestación y nacimiento del niño. En la Tabla I se muestra un resumen de algunos cambios en el patrón bacteriano, descriptos durante la gestación, posparto y primeras etapas de crecimiento del niño.
Durante la gestación, las condiciones fisiológicas maternas le proporcionan el entorno favorable para que el feto adquiera un determinado patrón bacteriano (35) (36). Al inicio del embarazo, la diversidad bacteriana en la vagina es estable; sin embargo, con la edad de gestación, se produce una variación del contenido y tipos de bacterias, de modo que el enriquecimiento de Lactobacillus en la vagina de la madre (Tabla I), permite la producción de ácido láctico para el mantenimiento de un pH vaginal bajo (protector para el feto), frente a la infección de patógenos (33).
Este mecanismo protector se ha demostrado en algunas especies de Lactobacillus (Lactobacillus crispatus, Lactobacillus iners, Lactobacillus gasseri y Lactobacillus jensenii), las cuales se adaptan muy bien a este ambiente vaginal; sin embargo, pueden manifestarse variaciones en la estabilidad de las mismas, con relación a la edad de gestación. La etiología de estas diferencias aún no está bien establecida, pero se ha relacionado con los niveles de hormonas sexuales. El incremento de estrógenos aumenta el grosor de la mucosa vaginal, lo cual, a su vez, aumenta la deposición de glucógeno. Esta deposición actúa como un agente quimiotáctico para los microbios, ya que el glucógeno es el sustrato principal utilizado por estas bacterias para disponer de la glucosa que fermentan llevándola a ácido láctico. Este último es el responsable de la reducción del pH vaginal (37).
El contenido de algunos géneros de Bacteroides . Staphylococcus en el intestino materno puede variar en correspondencia con las variables antropométricas en la madre, como el índice de masa corporal (IMC). En este sentido, algunos estudios realizados en mujeres embarazadas con sobrepeso demostraron diferencias en el contenido de estas bacterias intestinales, con relación a embarazadas que presentaban un peso corporal en el rango normal. Ambos grupos fueron analizados en el mismo período de gestación, pero no se compararon con mujeres no embarazadas como grupo control (35) (38).
Se detectó un número reducido de Bifidobacterium y Bacteroides, con un mayor número de Staphylococcus, Enterobacteriaceae y E. coli en mujeres con sobrepeso en comparación con mujeres embarazadas de peso normal. El contenido de E. coli fue mayor en mujeres con aumento excesivo de peso que en mujeres con aumento normal de peso, durante el embarazo. En cambio, los niveles de Bifidobacterium y Akkermansia muciniphila mostraron una tendencia opuesta. Por otra parte, el aumento del número de bacterias totales y de Staphylococcus se relacionó con un aumento de los niveles de colesterol plasmático. El mayor contenido de Bacteroides se relacionó con un mayor nivel de colesterol HDL y ácido fólico y una reducción de los niveles de triacilglicéridos. Un contenido mayor de Bifidobacterium estuvo relacionado con un aumento de los niveles de ácido fólico. Sin embargo, el incremento de Enterobacteriaceae y E. coli se relacionó con mayor presencia de ferritina y con la reducción de transferrina, mientras que los niveles de Bifidobacterium mostraron la tendencia opuesta (35) (38).
Algunas evidencias apuntan a que el intestino infantil es colonizado desde su permanencia en el útero por bacterias procedentes de la madre y que el primer meconio es rico en géneros de los filos Firmicutes . Proteobacteria (40) (41). La presencia de uno u otro género depende de la forma en que nace el niño, de modo que si el niño nace de forma natural se favorece la presencia de Firmicutes (Lactobacillus) y Bacteroidetes (Prevotella) (41). Mientras que en niños nacidos por cesárea, dominan bacterias características de la piel como Streptococcus, Corynebacterium y Propionibacterium (39) (40) (41) (Tabla I).
Es evidente que el paradigma acerca del entorno estéril del útero durante el embarazo, ha ido cambiando en los últimos tiempos. Prueba de ello es que se han encontrado algunos hongos en muestras intestinales de niños con edad entre 0 y 2 años, con una representación de los géneros Penicillium, Aspergillus y Candida y se ha demostrado que algunas especies de C. albicans y Malassezia spp. se transfieren de la madre a su descendencia a través del canal del parto. La prevalencia de Candida spp. puede manifestarse en un 23% en recién nacidos, lo cual podría duplicarse hasta el 50% a los 4 meses de su nacimiento (42).
En la fase posparto, el recién nacido adquiere un patrón microbiano enriquecido a partir de la leche materna. La presencia de géneros de los tres principales filos (Firmicutes, Bacteroidetes y Actinobacteria) (Tabla I) en el intestino del niño, procedente de esta fuente materna, es un factor esencial para la degradación de determinados hidratos de carbono que se van incorporando a partir de la dieta y facilitan la posterior evolución en su crecimiento corporal (30)(43)(44).
Gracias a la dieta que va cambiando en la medida que se le incorporan alimentos de origen vegetal (frutas, verduras como dieta blanda, cereales, derivados proteicos, etc.), con el tiempo (aproximadamente 3 años), el niño adquiere la microbiota intestinal con similitudes al patrón del adulto (43) (Tabla I). Estos cambios dependen en gran medida, de si la leche es de origen materno o fórmula, del estilo de vida (condiciones socio-demográficas), eventos, factores ambientales y frecuencia en el consumo de antibióticos (30).
El mecanismo de aceptación de determinados géneros depende del naciente sistema homeostático del niño. Al nacer, el intestino delgado del niño es colonizado por una microbiota de baja diversidad, la cual expone el antígeno microbiano y los patrones moleculares asociados a patógenos (PAMPs). Al mismo tiempo, se producen estímulos inmunes innatos endógenos; que consiguen que la estimulación del receptor tipo Toll (TLR) perinatal induzca una hiporrespuesta innata y una reprogramación en el epitelio intestinal y de las células mieloides (45).
En fechas cercanas al nacimiento, las células T y B salen del timo y de la médula ósea, respectivamente. Posteriormente, se dirigen a los tejidos linfoides secundarios, incluidos los ganglios linfáticos mesentéricos y los tejidos linfoides asociados al intestino (placas de Peyer y tejidos linfoides intestinales solitarios). Es probable que la microbiota (inicialmente transferida de la madre al nacer) se una a la IgAs materna cedida a través de la leche materna, la cual protege al antígeno microbiano del sistema inmunitario adaptativo. La IgAs materna y las células T reguladoras derivadas del timo neonatal (Treg) contribuyen al estado inicial del sistema inmunitario adaptativo, durante la fase posnatal (45).
Adquisición de un patrón bacteriano durante el crecimiento del niño
En la etapa de destete se incrementa la variedad de microbios en el hospedador, a través de la ingestión de alimentos sólidos que contienen hidratos de carbono complejos. Paralelamente, se produce la iniciación de la respuesta inmune adaptativa frente a los microbios y antígenos procedentes de la dieta. De este modo, en esta etapa, se pueden encontrar criptas con células de Paneth productoras de péptidos antimicrobianos y se favorece la producción de moco regulada en las células caliciformes, las cuales protegen al epitelio de la microbiota (45).
Al mismo tiempo, las células caliciformes comienzan a transportar los antígenos del lumen a las células dendríticas subyacentes (CD), en la lámina propia. Las CD, a su vez, presentan los antígenos a las células T vírgenes y se induce una activación inmunitaria transitoria (adaptativa). La etapa de destete está caracterizada por citoquinas proinflamatorias, factor de necrosis tumoral-α (TNF-α) e interferón-γ (IFN-γ). En esta fase se inducen células Treg que promueven la tolerancia y ajustan el sistema inmunológico de la mucosa para una respuesta adecuada a los estímulos inmunitarios en la vida posterior, protegiendo al niño de enfermedades inmunomediadas (45).
Después del destete, la composición de la microbiota se estabiliza y es menos sensible a perturbaciones, como la entrada de patógenos o el tratamiento con antibióticos. Los antígenos del lumen se facilitan a las células presentadoras de antígenos de una manera altamente controlada y la respuesta inmune homeostática está dominada por células Treg promotoras de tolerancia que, a su vez, ya son capaces de inducir la producción de IgA endógena por las células plasmáticas (45).
El uso de antibióticos en el niño, las continuas hospitalizaciones y los alimentos contaminados incrementan la susceptibilidad a enfermedades digestivas ocasionadas por Clostridioides difficile (46) y por C. albicans (infecciones fúngicas más frecuentes en sangre, pulmones y vía gastrointestinal), en niños menores de 12 meses (47). Estas enfermedades se consideran una de las causas de muerte más frecuentes por alteración de la microbiota intestinal (46) (47) (48).
Estos factores se han relacionado con la susceptibilidad encontrada en los niños menores de 3 años a padecer asma, alergias (eczemas, rinitis alérgicas y sensibilización atópica), a consecuencia de un desequilibrio en la relación en el contenido de los filos Bacteroides y Firmicutes, en el intestino (49). A esto podrían sumarse las condiciones carenciales en el consumo de alimentos, la resistencia de los patógenos a los antibióticos y la poca efectividad de las vacunas para erradicar completamente las enfermedades microbianas como causas de la alta incidencia y prevalencia de epidemias históricas en todos los rangos de edad (50).
De Filippo et al. (51) realizaron uno de los trabajos de mayor significación en cuanto al efecto positivo de la dieta de carácter primitivo en los niños, procedentes de zonas rurales de África. Estos autores analizaron la composición bacteriana de las heces de estos niños que se alimentaban acorde con el patrón de subsistencia de los campesinos del Neolítico y de otro grupo de niños sanos alimentados al estilo de los países europeos desarrollados (estilo occidental). Los resultados demostraron el incremento del filo Bacteroidetes y menor representación de Firmicutes en los niños africanos, con un menor contenido de Enterobacteriaceae que el encontrado en los niños europeos (Tabla I). El género Prevotella estuvo relacionado con el estado saludable de estas comunidades africanas (51).
Géneros bacterianos presentes en el adulto sano
Aunque la microbiota intestinal está integrada por bacterias, virus, hongos y arqueas, las bacterias han sido las más estudiadas. En las bases de datos de Pub- Med (National Library of Medicine, NIH), mientras 30 de los resultados de búsquedas están relacionados con los virus presentes en la microbiota intestinal, poco más de 500 referencias se relacionan con hongos y más de 25 000 referencias han contribuido al conocimiento de los complejos mecanismos que vinculan al hospedador con las bacterias comensales y patógenas (pubmed. ncbi.nlm.nih.gov, consultado: 10/01/2021).
Cada individuo tiene un núcleo estable de bacterias en una escala temporal de meses que se retroalimenta de otros factores ambientales, modulando al conjunto de microbios residentes (autóctonos) y a los llamados microbios viajeros (alóctonos). La dieta es uno de los determinantes de la variedad e incremento en el número de bacterias para garantizar un microbioma intestinal del sujeto adulto con un contenido aproximado de 1014 microbios (52). Las alteraciones en la dieta ocasionan el 57% de las variaciones, mientras que los factores genéticos son responsables del 12% (53).
Los principales filos bacterianos Firmicutes, Bacteroidetes, Proteobacteria, Actinobacteria y Verrucomicrobia colonizan normalmente el intestino del sujeto sano adulto (54). No es posible establecer los valores de rangos normales en este contenido porque existen diferencias entre individuos sanos de una misma zona geográfica, entre diferentes zonas geográficas, estilos de vida y entre zonas de muestreo en el sistema gastrointestinal de un mismo individuo (55).
Se ha demostrado que en el núcleo bacteriano intestinal de la población europea, más del 80% de los individuos estudiados contenían 32 especies de los géneros: Faecalibacterium, Roseburia, Bacteroides, Dorea, Clostridium, Eubacterium, Coprococcus, Alistipes, Collinsella, Parabacteroides y Bifidobacterium (31). En este estudio se encontró que Faecalibacterium, Eubacterium, Clostridium, Blautia y Roseburia son géneros representativos, tanto en las poblaciones rurales como urbanas del mundo. De ellos, Faecalibacterium, Eubacterium, Clostridium, Ruminococcus y Roseburia son importantes para la fermentación de hidratos de carbono precursores de butirato que intervienen en el mantenimiento saludable de las células colónicas (31).
En el colon del sujeto adulto también coexisten agentes patógenos primarios tales como: Campylobacter jejuni, Salmonella enterica, Vibrio cholerae, E. coli y B. fragilis pero con una abundancia baja (57) (58).
Por otra parte, se refiere que la abundancia del filo Proteobacteria es notablemente baja, en contraste con la alta abundancia de los géneros Bacteroides, Prevotella y Ruminococcus en el microbioma intestinal saludable (58).
La distribución de determinados géneros es diferencial teniendo en cuenta la localización longitudinal bacteriana (a lo largo del tracto gastrointestinal) y la localización axial (desde el lumen a la superficie de la mucosa del intestino) (6) (59). Según estudios realizados por Swidsinski et al. (59), predominan en el lumen (que pueden ser identificados en las heces): Bacteroides, Bifidobacterium, Streptococcus, Enterobacteriaceae, Enterococcus, Clostridium, Lactobacillus . Ruminococcus, mientras que Clostridium, Lactobacillus, Enterococcus y Akkermansia son predominantes en la mucosa y están asociados con el moco (detectados en la capa de moco y criptas epiteliales del intestino delgado) (59).
Que se favorezca la presencia de un género de bacterias u otro depende de algunos sistemas de regulación de la expresión génica en la población microbiana local. La presencia de algunos metabolitos inductores del crecimiento bacteriano y moléculas de señalización que son moduladoras de dicha expresión, garantizan la comunicación entre las bacterias gram negativas y gram positivas a través del llamado quorum sensing (sistema de señalización QS). En este mecanismo participan las moléculas QS, responsables de la autoinducción en la densidad celular circundante y la activación de la regulación compensatoria de la función celular apropiada (60). El adecuado funcionamiento de estos sistemas puede ser una de las razones por las que en sujetos sanos existe un equilibrio relativo entre los filos bacterianos.
Estos sistemas podrían proporcionarle a la bacteria la capacidad de burlar el mecanismo de defensa inmunitaria del hospedador o evadir la acción de los compuestos antimicrobianos y medicamentos, en los eventos patogénicos a través de la formación de biofilms. En este marco, se establece una cooperación entre grupos de bacterias para construir una barrera, la cual no consiguen alcanzar de manera individual (61).
Sin embargo, este tipo de regulación podría significar un mecanismo favorable para el hospedador si el resultado fuera el crecimiento de bacterias beneficiosas que depende de los compuestos que adquiere durante su consumo de alimentos. En algunos estudios se ha demostrado que el sistema de activación celular QS (AI-2) en especies de Prevotella (filo Bacteroidetes), es inducido por la glucosa (60), mientras que el crecimiento de algunas especies de Roseburia .Firmicutes) es inducido por un sistema QS, en presencia de L-fucosa disponible en algunos polisacáridos de la dieta (62).
Estos sistemas de regulación interfilos son parte de los complejos mecanismos regulatorios en el intestino (62) y resulta interesante que no existe un solo tipo de modelo de regulación QS, lo cual podría explicar el hecho de que la diversidad en la alimentación proporciona una mayor variedad de moléculas activadoras de estos mecanismos y, por lo tanto, un incremento en la diversidad bacteriana.
Uno de los sistemas QS en el que se centra la atención actual es en el que interviene el aminoácido triptófano, procedente de la dieta, mediante el cual se regula la secreción de péptidos antimicrobianos por parte de las células de Paneth del epitelio intestinal a través de la vía de activación de mTOR. En esta vía, el triptófano disponible es internalizado en la célula por el transportador B.AT1 que a su vez, necesita de ACER2 para su expresión. En ausencia de ACER2, el triptófano no puede ser absorbido eficientemente por las células epiteliales y se produce una alteración en la secreción de péptidos antimicrobianos que podría ocasionar la modificación del patrón de la microbiota intestinal y el incremento de la susceptibilidad del intestino a la inflamación (17).
Perlot y Penninger sugirieron desde el año 2013 (17) que éste podría ser uno de los mecanismos mediante el cual los coronavirus SARS-CoV podrían actuar negativamente sobre la diversidad bacteriana comensal, bloqueando la disponibilidad del receptor ACR2 para la cooperación en la internalización de triptófano (17).
En los continentes europeo y asiático se han realizado estudios referidos en la presente revisión. Sin embargo, existen pocos estudios desarrollados en América Latina relacionados con la microbiota intestinal y su variabilidad de acuerdo con la zona geográfica y sus diferencias con respecto a América del Norte. Esta caracterización sería de gran interés para poder comprender la influencia de los factores socio-demográficos en el patrón microbiano (63).
En este sentido, es importante destacar un estudio realizado en los EE.UU. que contribuyó significativamente al conocimiento del posible efecto de la emigración en la conformación de un microbioma y su estabilidad generacional. El estudio partió de la premisa de que los emigrantes procedentes de América Latina y el Caribe constituyen la mayoría de la población de origen extranjero que vive en los EE.UU. El análisis metagenómico de muestras de heces de adultos hispanos y de nacidos en los EE.UU. mostró mayor abundancia relativa de Bacteroides que el resto de los géneros bacterianos. Sin embargo, se encontró un mayor contenido de Prevotella en personas nacidas en América Latina que en hispanos nacidos en los EE.UU. La heterogeneidad presente en el patrón bacteriano de la población de latinos se explicó por las diferencias detectadas en primera generación (emigrantes nacidos en América Latina) y segunda generación (descendientes de los latinos emigrantes y que nacieron en EE.UU.). Cada uno de estos grupos tenía su propio patrón de microbioma, de manera que algunos taxones (Ruminococcaceae, Clostridiales, Bifidobacterium, Blautia, Enterobacteraceae y Sutterella) fueron abundantes según el lugar de nacimiento del sujeto (64).
Como bien plantearon Carbonetto et al. (65), todos los estudios que formaron parte del Proyecto del Microbioma Humano, se realizaron en poblaciones de EE.UU. y Europa y en ningún caso, en América Latina. Los resultados del análisis metagenómico realizado por estos autores a partir de diferentes muestras del sistema gastrointestinal de 20 sujetos sanos, durante el desarrollo de un estudio piloto acerca de la microbiota en la Argentina, mostraron diferencias en el microbioma de los sujetos, con relación a poblaciones de EE.UU. En la población argentina procedente de una zona metropolitana prevaleció Ruminococcaceae, Lachnospiraceae y Prevotellaceae, mientras que la familia Bacteroidaceae fue menos abundante (65).
De esta manera, sería una gran contribución establecer qué patrones de microbioma caracterizan las poblaciones con mayor resistencia ante el contagio y propagación de una enfermedad de tipo bacteriana, viral o fúngica.
Alteración de la microbiota en el sujeto enfermo
Una de las revisiones más completas acerca de la implicación de la microbiota en la etiología de gran número de enfermedades y las posibilidades de su caracterización como herramienta para el diagnóstico y la aplicación terapéutica, es la realizada por Kho y Lal (66). Estos autores abordaron cómo el desequilibrio en el patrón bacteriano conduce a la disfunción de la maquinaria del hospedador, contribuyendo a la patogénesis y/o a la progresión hacia un amplio espectro de enfermedades inflamatorias intestinales (enfermedades innmunomediadas), enfermedad celíaca (trastorno autoinmune multisistémico), trastornos neuropsiquiátricos, metabólicos (obesidad), diabetes tipo 1 y tipo 2, enfermedades cardiovasculares e hipertensión, entre otras (66).
La obesidad y la diabetes mellitus tipo 2 alcanzan magnitudes epidémicas, a consecuencia del incremento alarmante de la prevalencia de enfermedades crónicas no transmisibles (66) (67). Este incremento se ve favorecido por cambios evidentes en los estilos de vida, en niveles económicos y sociales que conducen a la ingestión de alimentos de bajo costo y bebidas envasadas (67). Los altos costos de los tratamientos de la diabetes mellitus tipo 2 y las enfermedades relacionadas, así como la mortalidad prematura de aproximadamente el 80% de los afectados, son las mayores preocupaciones de los sistemas de salud, por lo que la prevención es uno de los objetivos de la Organización Mundial de la Salud (67).
La diabetes tipo 2 es el resultado de complejas interacciones entre los genes del individuo y algunos factores de riesgo como la edad, antecedentes familiares, dieta, el estilo de vida sedentario y la obesidad. Uno de los factores externos con mayor relevancia es la variación en el patrón de la microbiota intestinal, como causa y/o consecuencia de estas enfermedades, e incluso, de enfermedades cardiovasculares (68).
Karlsson et al. realizaron un estudio de caracterización metagenómica bacteriana en las heces de 145 mujeres europeas en un grupo control y un grupo de pacientes con diabetes tipo 2. Los resultados mostraron una correlación positiva entre el incremento de los niveles de glucosa sérica y la abundancia de especies de Lactobacillus. Estas especies no se comportaron de la misma forma en mujeres chinas con esta enfermedad, analizadas en un estudio anterior. Como resultado, se encontró en las mujeres diabéticas, un incremento en Clostridium (mezcla de tres patógenos oportunistas: C. bolteae, C. hathewayi . C. clodtridioforme asociados con bacteriemia e infecciones en seres humanos) y la disminución de Roseburia (68).
La etiología de las enfermedades inflamatorias digestivas sigue siendo desconocida; sin embargo, tanto en colitis ulcerosa (CU) y enfermedad de Crohn (EC) las causas pueden ser la presencia de un agente infeccioso o de un antígeno procedente de la alimentación que provoque una respuesta inflamatoria alterada que termina atacando al propio intestino. Estas enfermedades aparecen en individuos genéticamente predispuestos, en los que la conjunción de factores ambientales exteriores y las bacterias intestinales producen una respuesta inflamatoria intestinal anómala que se perpetúa en el tiempo, produciendo la enfermedad (69).
En estas enfermedades inflamatorias se describe una significativa disminución en la diversidad microbiana, en particular, de la abundancia de las bacterias gram positivas anaerobias (por ejemplo, Ruminococcaceae y Lachnospiraceae) y un concomitante incremento en las bacterias anaerobias facultativas, tales como enterococos y estreptococos (70) (71) y de gram negativas, tales como Proteobacterias (en particular los miembros de la familia Enterobacteriaceae) (72) (73) (74).
En algunos estudios se ha observado la disminución de Bacteroidetes (72) (73), mientras que en otros, se describió el incremento en la cantidad de estas bacterias (74) (75); sin embargo, no se registraron cambios en la abundancia de Lactobacillus y de Actinobacteria (Bifidobacterium) (75) (76).
En cualquier caso, la mayoría de los estudios coincide en términos de un incremento significativo de Enterobacteriaceae, especialmente E. coli, asociada a la mucosa y en muestras fecales de los pacientes. En los pacientes con enfermedad de Crohn se ha podido identificar una variante de E. coli específica de esta enfermedad, denominada enteroadherente (77) (78).
Algunas enfermedades dérmicas pueden estar vinculadas a los trastornos en el perfil bacteriano intestinal. En un estudio realizado en Buenos Aires (Argentina) se analizaron muestras de heces de pacientes afectados por psoriasis y sujetos no afectados (grupo control) para profundizar en el conocimiento de aquellos géneros bacterianos como marcadores en distintas etapas de esta enfermedad. En estos pacientes se detectó un incremento en el contenido de Firmicutes y una reducción de Bacteroidetes. Los géneros con mayor representación en pacientes, fueron Faecalobacterium y Blautia, mientras que Bacteroides y Paraprevotella fueron mayoritarios en los controles. El incremento de severidad de la enfermedad en los pacientes se vio caracterizado por la disminución en la biodiversidad bacteriana (79).
Una aproximación en el establecimiento de biomarcadores diferenciales de tipo bacteriano en las heces, podría representar una herramienta complementaria en el seguimiento de los pacientes y su aplicación en determinadas terapias de restauración de la mucosa intestinal.
Contribución del agua en el patrón bacteriano de la microbiota intestinal
Otro de los factores externos a tener en cuenta por su posible aporte de microbios al patrón intestinal, es el consumo de agua y sus fuentes de disponibilidad.
Las fuentes de agua envasada contienen, generalmente, una microbiota muy variada que incluye las siguientes especies bacterianas: Achromobacter spp., Aeromonas spp., Flavobacterium spp., Alcaligens spp., Acinetobacter spp., Cytophaga spp., Moraxella spp., y Pseudomonas spp. (80).
Las cuencas de suministro de agua mineral pueden afectarse a causa de inundaciones provocadas por los cambios climáticos y por contaminaciones de las aguas subterráneas (81).
Andrade et al. (81) refirieron que sólo existen 14 estudios relevantes publicados entre 1980 y 2017 que profundizaron en cuanto a la posibilidad del agua subterránea como vector importante de infecciones.
La abundancia de bacterias en el agua, como E. coli y Pseudomonas aeruginosa, entre otras, puede constituir un riesgo para la salud humana. Algunos casos de gran connotación relacionados con la contaminación de agua envasada, se han producido a causa de la presencia de Vibrio cholerae en 82 casos confirmados en Portugal en 1974. También se detectó este tipo de contaminación por Campylobacter jejuni en el personal militar norteamericano que se encontraba en prácticas en Grecia en el año 1997. Posteriormente, en el año 2006, se demostró la asociación estadística entre la contaminación por Salmonella enterica en niños inmunodeprimidos en Gran Canaria (España) y el consumo de agua envasada, con la presencia de esta bacteria en el agua y en la fábrica donde se envasaba (82).
La Food and Drug Administration (FDA) de los EE.UU. ha incrementado las exigencias en el control microbiológico del agua, procedente de fuentes públicas y de agua envasada destinada al consumo. Estas regulaciones exigen la ausencia de bacterias coliformes y E. coli, para garantizar la salud (83).
Patrón de consumo de alimentos y salud
La composición de la dieta que consume el individuo es uno de los factores fundamentales en el equilibrio mutualista de los microbios beneficiosos y el hospedador. Las bacterias intestinales están involucradas en la descomposición de diversos polifenoles y productos secundarios polifenólicos presentes en una gran variedad de plantas, frutas y derivados de plantas como el té, el cacao y el vino, beneficiosos para ese estado saludable, en seres humanos. Estos polifenoles se manifiestan como derivados glicosilados de glucosa, galactosa y ramnosa, entre otros (30).
Los alimentos que contienen estos polifenoles forman parte del patrón de consumo al “estilo mediterráneo” (CAEM), beneficioso por la disponibilidad de productos ricos en estos derivados (como las aceitunas y el aceite de oliva). Los metabolitos resultantes de la transformación bacteriana de estos polifenoles presentan múltiples efectos protectores asociados con la inhibición del estrés oxidativo, la reducción de los niveles de lipoproteínas de baja densidad oxidadas y de factores de riesgo cardiovascular, así como también la inhibición de la inflamación (32).
El CAEM se caracteriza por un consumo frecuente de verduras, frutas, cereales (preferentemente granos enteros), legumbres, nueces, de un consumo reducido de pescado o mariscos, carne blanca y huevos, cantidades moderadas de productos avícolas y lácteos y baja ingesta de alcohol (vino) (83). Este estilo de alimentación constituye un paradigma en el mantenimiento de las mejores condiciones (dieta, ejercitación, ausencia de estrés) para garantizar la salud. La alta adherencia a este patrón, reduce la mortalidad y la incidencia de enfermedades crónicas (cáncer, síndrome metabólico y cardiovascular, enfermedades neurodegenerativas, diabetes mellitus tipo 2, enfermedades del hígado graso y alergia (84).
En una revisión realizada por Ostan et al. (85) se sugiere continuar profundizando en el conocimiento de las vías moleculares y celulares del efecto individual de los componentes de este estilo de alimentación.
En España, se han abordado estudios que demuestran la asociación entre el grado de ingesta y la presencia de determinados géneros bacterianos, en sujetos con una alta adherencia al CAEM. En un estudio realizado en la Comunidad Valenciana (86) se demostró una menor proporción de hombres con ingesta de tipo CAEM (45,5%) que de mujeres (62,5%), con una mayor riqueza bacteriana en sujetos que mantenían el CAEM (índice Chao), una baja relación Firmicutes/Bacteroidetes, menor abundancia del género Clostridium y mayor asociación con una bacteria de la familia Christensenellaceae [familia asociada con la pérdida de peso en modelos de ratones, según Goodrich et al. (87)].
Los estilos de consumo de alimentos son importantes, sobre todo, para definir cuáles son los factores que permiten asegurar la resistencia ante patógenos epidémicos, en una población de determinada zona geográfica. Los estudios realizados en pacientes infectados con C. difficile, durante la epidemia que afectó a la población de los EE.UU. entre los años 2000 y 2003, sugirieron la posible relación entre el azúcar trehalosa presente en alimentos (frecuentemente utilizado en sushi, vegetales y helados como aditivo edulcorante protector frente a altas temperaturas) y la propagación de determinados ribotipos de esta bacteria. Utilizando modelos de ratón y las cepas aisladas de estos pacientes, se concluyó que la adopción y el uso generalizado del disacárido trehalosa en la dieta humana podría haber desempeñado un papel importante en la aparición de estas cepas epidémicas e hipervirulentas (88).
Actualmente, uno de los estilos de alimentación más extendido es el llamado estilo sushi factor, asociado con la presencia de una microbiota intestinal especializada en la digestión de algas marinas, estudiado a partir de las tradiciones japonesas (46). Los estudios realizados por Hehemann et al. (89) han demostrado que algunos genes (codificadores de la producción de enzimas hidrolíticas que actúan sobre polisacáridos de algas) presentes en Bacteroidetes de origen marino, han sido transferidos, probablemente, a Bacteroidetes presentes en el intestino de individuos japoneses. Estos géneros de Bacteroidetes marinos pudieron haber llegado a través del consumo de algas, como un mecanismo facilitador para el aprovechamiento energético en el intestino de estas poblaciones (89).
Estos modelos de alimentación se han ido exportando de una zona geográfica a otra, por lo que las diferencias culturales tradicionales en cuanto a la dieta van formando parte de nuevas culturas mixtas de alimentación. Por este motivo, su incorporación y adaptación de la microbiota a estos estilos permite hablar de la globalización de la microbiota (46).
Mah et al. (90) profundizaron en el conocimiento acerca de otros estilos de consumo de alimentos y condiciones socio-económicas a través de un estudio de caracterización microbiológica en heces de niños (3 años) que vivían en zonas rurales de Tailandia (Theppa), procedentes de familias de bajos ingresos económicos (la mayoría agricultores y granjeros) y de niños procedentes de la zona metropolitana de Singapur (con aceptables condiciones económicas). El estudio estaba encaminado a la profundización en el conocimiento acerca de la posible relación entre la alta prevalencia de enfermedades alérgicas en Singapur y el patrón de la microbiota intestinal (90).
Se realizaron aislamientos de cultivos, se identificaron y cuantificaron los microbios presentes en heces. En esta caracterización, las familias tailandesas tenían más hijos (una media de 4), consumían menos antibióticos y poseían una mayor ingesta de yogur. El nivel de rinitis (inflamación de fosas nasales sin padecer gripe) fue mayor que en los niños de zonas urbanas (90).
Los resultados demostraron que el consumo de agua en Singapur se producía a partir del agua tratada de grifo y sólo el 17,4% de las familias tailandesas tuvieron acceso a la red de agua (el resto se abastecían de fuentes naturales, pozos, estanques y lagos). Los niveles de bacterias lácticas, coliformes y Staphylococcus fueron mayores en niños tailandeses, donde la mayoría de las bacterias coliformes correspondieron a E. coli y K. pneumoniae, mientras que no se demostraron diferencias entre las poblaciones en cuanto a los niveles de Enterococcus. Los resultados no fueron conclusivos, pero sugirieron posibles diferencias en el patrón total de bacterias intestinales, entre poblaciones asiáticas (90).
Otro estudio metagenómico realizado en heces de niños sanos, con un rango de edad entre 8 y 11 años, procedentes de regiones con diferentes estilos de consumo de alimentos en Tailandia (regiones del noroeste y central) demostró que los niños del noroeste tenían un mayor consumo de carne (de pollo y vacuna), amplia variedad de fuentes de hidratos de carbono (fideos, arroz fermentado y batata), incluidas las verduras y las frutas, lo cual se correlacionó con una mayor abundancia de Lactobacillus, C. coccoides, E. rectale, Clostridium leptum, Prevotella y B. fragilis (91). Los niños de la zona central tenían preferencia por el arroz, consumo de cereales en el desayuno y leche de vaca, lo cual demostró una estrecha correlación con un menor contenido de los géneros anteriormente descriptos. Sin embargo, no se mostraron diferencias entre las poblaciones en cuanto al contenido de Bifidobacterium spp. y Enterobacteriaceae. Estos resultados sugirieron que un consumo frecuente de variedades de hidratos de carbono, fuentes de proteínas, frutas y verduras en los niños del norte puede ser uno de los factores favorables en el mantenimiento de la gran abundancia de especies bacterianas de ambos filos: Firmicutes y Bacteroidetes (91).
En el año 2018, el mundo entero se estremeció con la noticia de que un pequeño grupo de niños tailandeses había quedado atrapado en las profundidades de una cueva, durante varios días y fueron rescatados vivos. La pregunta general ante estos hechos fue: ¿cómo pudieron sobrevivir 12 niños (11-16 años) y su entrenador de fútbol atrapados por fuertes lluvias en la cueva durante 17 días, a una distancia de 4 km de la superficie, con el sólo abastecimiento de agua de las fuentes subterráneas contaminadas por las lluvias y sin ingerir alimentos? Todos fueron rescatados y se recuperaron satisfactoriamente (92) (93). Estos sucesos nos invitan a analizar qué posibles factores contribuyen a la supervivencia de sujetos procedentes de zonas geográficas económicamente desfavorecidas, pero con un estilo de alimentación y ejercitación que les podrían proteger ante situaciones de estrés. ¿Podría ser uno de estos factores su patrón de microbiota intestinal?
Epidemias y microbiota intestinal: ¿oportunistas?
Hasta aquí se han revisado algunos de los factores que pueden dar respuesta a las razones por las que el mantenimiento del equilibrio en la microbiota intestinal individual es fundamental para garantizar el equilibrio taxonómico y cuantitativo de la microbiota colectiva.
Sin lugar a dudas, los patógenos son oportunistas, aprovechan cualquier elemento que afecte el estado de portador y favorezca su mecanismo patogénico en el organismo.
El estado de portador fue ilustrado por Lawley et al. (94), a través de un estudio realizado en un modelo de ratones inmunocompetentes, los cuales presentaban el intestino colonizado por C. difficile en estado portador (la bacteria está presente pero no en estado virulento). En este estado, la bacteria expresaba bajos niveles de esporas y no se transmitían infecciones a los ratones que convivían durante el estudio. Para simular el estado de pacientes humanos hospitalizados, los ratones fueron tratados con antibióticos. El tratamiento desencadenó un estado altamente contagioso caracterizado por una reducción drástica en la diversidad de especies de la microbiota intestinal, con un incremento en el crecimiento de C. difficile y altos niveles de excreción de esporas. Una vez interrumpido el tratamiento con antibióticos, se logró la recuperación de la microbiota intestinal en los ratones inmunocompetentes y se suprimieron los altos niveles de C. difficile. Sin embargo, en aquellos ratones en los que se detectó una deficiencia en la vía de señalización de la respuesta inmune innata, se manifestó una enfermedad intestinal grave y el síndrome de disfunción multiorgánica que imita a la situación que se presenta en pacientes hospitalizados (94).
Las enfermedades bacterianas frecuentes oportunistas de mayor impacto son las causadas por V. cholerae, Shigella, Helicobacter pylori, Yersinia, Staphylococcus aureus y Clostridium perfringens. En los EE.UU., por ejemplo, se registran cerca de 18 000 casos de shigellosis cada año. Como muchos casos leves no se diagnostican, el número de casos puede ser veinte veces mayor, según el Centro de Control de Enfermedades de Atlanta, Estados Unidos (Centers for Disease Control and Prevention; CDC). En los países en desarrollo, esta enfermedad es común y está siempre presente en algunas comunidades (95).
Uno de los mecanismos de acción de estas bacterias oportunistas es el que opera a través de los sistemas de vesículas de membrana externa (VME), estructuras bacterianas que permiten el transporte de las enzimas proteolíticas, compartidas por otras bacterias, hacia la superficie de bacterias que carecen de estas funciones. En estos grupos de bacterias patógenas que se benefician, en cuanto al intercambio de enzimas y toxinas, se encuentran Shigella, Neisseria, Bacteroides, Pseudomonas, H. pylori, Vibrio, Salmonella, Brucella melitensis, C. jejuni, Aggregatibacter actinomycetemcomitans y Xenorhabdus nematophilus. La acción de toxinas está también asociada con algunos tipos de estas vesículas (96) y se transportan de manera natural formando parte de las mismas (97).
Un ejemplo ilustrativo de este tipo de colaboración interfilos en las bacterias patógenas colonizadoras, es el hecho de que la bacteria E. coli enterohemorrágica carece de enzimas hidrolasas que actúan sobre la mucina intestinal para producir fucosa (azúcar importante en la inducción de sus genes virulentos para la colonización en el intestino). Sin embargo, una especie de Bacteroides (B. thetaiotaomicron) dispone de esta batería enzimática (fucosidasas) que escinden la fucosa de la mucina intestinal (moco) en crecimiento. Este intercambio entre B. thetaiotaomicron y E. coli incrementa la disponibilidad de fucosa para esta última. Por lo tanto, el incremento de los niveles de Bacteroides puede favorecer la patogénesis enterobacteriana (98).
Es frecuente la infección por H. pylori como patógeno oportunista y puede considerarse de magnitudes epidémicas, a nivel mundial. Los mayores indicadores de prevalencia se refieren en Brasil (>70%), Rusia (>70%), Portugal (86,4%), Italia (56,2%), España (54,9%), Grecia (52,1%), Francia (46,9%), Tailandia (43,6%) y en menor medida (no obstante, considerable) en Alemania (35,3%), países escandinavos (35,5%), Reino Unido (35,5%), Noruega (30,7%), Australia (24,6%), Nueva Zelanda (24,0%), Suiza (18,9%) y países del Caribe (50- 70%). La adquisición de este patógeno está relacionada con edades tempranas de la niñez, en regiones donde los sistemas sanitarios y de aguas limpias son deficitarios (99). Estos datos demuestran que en los países desarrollados también es frecuente, probablemente, a causa de otros trastornos como el estrés y el deterioro de los patrones tradicionales de alimentación.
La transmisión de H. pylori puede darse también entre parejas, lo cual podría indicar que puede existir un estado portador, ya que pueden ser casos asintomáticos, en cuanto a sintomatología digestiva y negatividad de las pruebas serológicas (100).
Se conoce que la infección por H. pylori puede ser la causa de enfermedades del tracto gastrointestinal de tipo no neoplasia (enfermedad de úlcera peptídica, gastritis atrófica crónica) y de tipo neoplasia (adenocarcinoma de estómago, linfoma gástrico originario del crecimiento del tejido linfoide asociado a la mucosa) (101).
Los métodos diagnósticos son efectivos para la detección de este patógeno, según el estado del organismo humano, la medicación que han recibido previamente a las pruebas y la edad del paciente. La prueba del aliento o test de la ureasa (TU) es eficiente en los estados iniciales de la infección; sin embargo, en estados avanzados es necesaria la combinación de pruebas no invasivas e invasivas, como la exploración esofagogastroduodenoscopia con el correspondiente examen histopatológico de muestras de biopsia, la prueba de presencia de sangre en heces, PCR de las muestras y el estudio de cultivos bacterianos, entre otros (101).
El incremento de la prevalencia e incidencia de las enfermedades inflamatorias digestivas en Europa Occidental es alarmante, aunque es inferior al de otras enfermedades de gran impacto, como el cáncer, la diabetes y las enfermedades autoinmunes. La prevalencia en los cinco países europeos más poblados de Europa (Alemania, Francia, Reino Unido, Italia y España), EE.UU. y Japón ha aumentado un 2,8%, desde el año 2011 hasta la actualidad. Se estima que el crecimiento anual se podría acentuar hasta 2021, situándose en alrededor del 2,9%, cifra que equivale a casi tres millones y medio de casos diagnosticados (102).
La colitis ulcerosa es más frecuente que la enfermedad de Crohn, con un 67% y un 33%, respectivamente. Las mayores tasas de prevalencia e incidencia de la enfermedad del intestino irritable se encuentran en Europa Occidental y Norteamérica. Sin embargo, los países asiáticos, latinoamericanos y de Europa Oriental mantienen niveles más bajos. En cambio, los estudios en el continente africano son muy limitados y los datos disponibles son poco confiables (102).
Algunos autores plantean que en el futuro será necesario abordar estudios epidemiológicos previos, acerca del contenido de la microbiota intestinal, para poder emprender los sistemas de vacunación (103). Parker et al. (103) realizaron una revisión que demostró que la respuesta de las vacunas orales podría depender de varios tipos de bacterias de la microbiota intestinal, entre ellos, de Bacteroidetes, Bifidobacterium, Streptococcus y Lactobacillus.
Dado el escenario actual, frente a una de las pandemias más serias a nivel mundial a causa del nuevo coronavirus SARS-CoV-2, sería recomendable tener en cuenta los factores analizados y continuar con estrategias relacionadas con estudios epidemiológicos, diagnóstico y prevención, relacionados con la microbiota intestinal y su papel en la infección a causa de este tipo de virus.
Virus como oportunistas y posible colaboración con algunas bacterias. Algunos mecanismos homeostáticos implicados
La enfermedad por coronavirus 2019 (COVID-19) es una enfermedad infecciosa respiratoria emergente causada por el coronavirus que produce el síndrome respiratorio agudo severo 2 (conocido con las siglas SARS-CoV-2) extendido por el mundo, con connotación de pandemia. En sus mecanismos de invasión, el virus SARS-CoV-2 se une a los receptores de la enzima convertidora de angiotensina 2 para invadir a las células humanas y, estos receptores, se expresan, en gran medida en el epitelio intestinal y en el pulmonar (104).
A escasos meses de que se iniciase esta pandemia en el año 2020, Gu et al. (104) compartieron sus resultados con la comunidad científica, acerca de los hallazgos obtenidos en pacientes hospitalizados por COVID-19 (desde enero hasta marzo de 2020) y los compararon con los resultados previos obtenidos en pacientes hospitalizados por la gripe por influenza A estacional (H1N1), entre enero y marzo de 2018.
En este estudio se analizó la abundancia de algunos géneros bacterianos en heces, caracterizados taxonómicamente y cuantificados a través de métodos metagenómicos. Según estos autores, las manifestaciones y rutas de transmisión de H1N1 son similares a las de COVID-19. La inclusión de un grupo control de sujetos sanos (GCS) permitió proponer algunas bases teóricas para la diferenciación en el diagnóstico de estas dos enfermedades, sus posibles conexiones con algunos géneros bacterianos, que podrían en un futuro considerarse como marcadores del tipo de enfermedad, presentes en la microbiota intestinal, así como algunos elementos diferenciales en el proceso de homeostasis (104).
Los resultados de este estudio demostraron que la abundancia relativa de los filos Actinobacterias y Firmicutes disminuyó significativamente en el grupo H1N1 en comparación con los grupos COVID-19 y GCS, lo cual se debió, en gran parte, a una significativa reducción en las clases Actinobacteria, Erysipelothrichia y Clostridia (104).
El análisis a nivel de familias bacterianas mostró una reducción de los representantes bacterianos anaeróbicos Lachnospiraceae y Ruminococcaceae, productores de butirato, en los pacientes con H1N1. Este resultado se asoció con la reducción de la abundancia de Blautia, Agathobacter, Anaerostipes, Fusicatenibacter, Eubacterium hallii, Lachnospiraceae, Dorea, Faecalibacterium y Ruminococcus- 2. De manera similar, en los pacientes del grupo COVID- 19 se redujo dramáticamente la abundancia de la familia Ruminococcaceae y varios géneros de la familia Lachnospiraceae (Fusicatenibacter, Anaerostipes, Agathobacter, Lachnospiraceae no clasificada y E. hallii) al ser comparada con GCS. Sin embargo, el número de Streptococcus, Rothia, Veillonella y Actinomyces fue elevado en pacientes del grupo COVID-19, comparado con el de los grupos H1N1 y GCS (104).
Por lo tanto, se concluyó que el microbioma del grupo H1N1 estaba dominado por Enterococcus, Prevotella, Finegoldia y Peptoniphilus, mientras que en el microbioma del GCS predominaron Blautia, Romboutsia, Collinsella y Bifidobacterium. De ahí que los seis biomarcadores finales (Fusicatenibacter, Romboutsia, Anaerostipes, E. hallii, Ruminococcus y Blautia) fueron seleccionados para diferenciar el grupo H1N1 y GCS, mientras que cinco biomarcadores finales (Fusicatenibacter, Romboutsia, Intestinibacter, Actinomyces y Erysipelatoclostridium) fueron seleccionados para distinguir el grupo COVID-19 del GCS (104).
Por último, uno de los aportes de este trabajo fue el hecho de proponer posibles marcadores que diferencian el perfil de la microbiota en ambas enfermedades: siete biomarcadores finales (Streptococcus, Fusicatenibacter, Collinsella, Dorea, Agathobacter, E. hallii y Ruminococcus) y la connotación que puede suponer el aumento relativo de la abundancia de patógenos oportunistas, incluidos Streptococcus, Rothia, Veillonella, Erysipelatoclostridium y Actinomyces, en pacientes con COVID-19 (104).
En este análisis comparativo dentro del mismo grupo COVID-19, se demostró que no existían diferencias en cuanto a riqueza, diversidad y estructura de la microbiota bacteriana entre pacientes con COVID-19 (15 pacientes con categoría de severidad general y 15 pacientes con categoría severa). En todos los diferentes grados de severidad en la enfermedad, los biomarcadores se correlacionaron con los marcadores clínicos (PCR para identificación del ARN de SARS-CoV-2, procalcitonina, niveles del dímero D, citoquinas inflamatorias IL-2, IL-4 e IL-6). Como resultado, se encontró que existía una correlación positiva entre las citoquinas inflamatorias IL-2, IL-4 e IL-6 y la abundancia de algunos géneros de bacterias, presentes en pacientes con H1N1 (Finegoldia, Anaerococcus, Peptoniphilus, Intestinibacter y Prevotella) (104).
Se estima que Rothia contribuye a la patogenia de la neumonía, especialmente en personas inmunodeprimidas y pacientes con catéteres retenidos. En un estudio anterior realizado por estos autores (104), se sugirió que Streptococcus y Rothia estaban asociadas con la susceptibilidad a la infección pulmonar bacteriana secundaria, en pacientes con infección por el virus aviar H7N9 y que los cambios en el ambiente entérico e inmunológico, cuando Actinomyces estuviese presente, podrían agravar el daño causado por la enfermedad inflamatoria intestinal (104).
Otro elemento importante en la respuesta homeostática fue que los niveles de IL-2 e IL-4 aumentaron significativamente en los pacientes con H1N1 y correlacionaron positivamente con la abundancia de estas bacterias, probablemente porque la infección por influenza altera el perfil microbiano intestinal a través de un mecanismo dependiente de los interferones de tipo I, inducidos en el pulmón (104).
Algunas evidencias previas habían demostrado que la infección por influenza respiratoria provocaba lesiones pulmonares mediante la movilización de derivados pulmonares (CCR9 y células T CD4) hacia el intestino delgado y estimulaban la producción de interferón-γ por estas células (105).
La presencia de ARN del SARS-CoV-2 en muestras de heces de pacientes y el hecho de que la duración de esta molécula en las heces es más prolongada que su excreción por vías respiratorias, ha sido uno de los importantes aportes para confirmar la implicación del eje intestino-pulmón (104).
Una de las claves en el mecanismo de virulencia de este virus es la relacionada con los receptores ACE2 presentes en pulmones (abundantes en personas fumadoras), caracterizados como mono-carboxipeptidasas que convierten la hormona angiotensina II en un vasodilatador angiotensina (2) (3) (4) (5) (6) (7). Este último se une al receptor MAS1-R, que al parecer, juega un papel importante en muchas vías de respuestas antiinflamatorias de los mecanismos de protección tisular (105).
La interacción del virus SARS-CoV-2 con ACE2 de los neumocitos tipo 2, a través de su glicoproteína de espícula presente en la superficie viral (proteína S), activa la acción enzimática de la enzima receptor (mediada por el sistema de renina-angiotensina) sobre la proteína S. Dicha interacción genera mediadores peptídicos que regulan la entrada viral (regulada además, por una proteasa serino-dependiente transmembrana asociada a la superficie celular conocida como TMPSS2) (105).
El receptor ACE2 se expresa de manera abundante en el epitelio gastrointestinal e interviene en el reconocimiento del virus, cuando éste alcanza el sistema. La interacción del virus con los receptores durante el proceso de virulencia puede interferir en la absorción de nutrientes (como el aminoácido triptófano), que normalmente interactúan con ACE2. Con la reducción de la disponibilidad de estos receptores en la vía protectora de conversión de la angiotensina II, se altera la homeostasis intestinal y se producen síntomas similares a la gastroenteritis (104) (105).
La susceptibilidad a contraer COVID-19, detectada en personas afectadas por enfermedades del intestino inflamado, es evidente. Aunque la enfermedad manifiesta pequeños y medianos síntomas de resfriado en la mayoría de los pacientes, ésta conduce, frecuentemente, a complicaciones letales (neumonía progresiva, síndrome de estrés agudo respiratorio y falla orgánica a consecuencia de una hiperinflamación y el llamado síndrome de tormenta de citoquinas. Esto se debe a que la infección se produce por las dos vías mencionadas: pulmón e intestino (105).
La fusión y penetración de los componentes virales, a través de endosomas, inducen la liberación de quimioquinas por las células infectadas del hospedador, lo cual conduce a la acumulación de neutrófilos en el sitio de la infección. Los neutrófilos también liberan citoquinas y quimioquinas que atraen a otras células del sistema inmune (monocitos y linfocitos T). En este punto, es de destacar la respuesta inmune exagerada que se genera (más alta en los pacientes no sobrevivientes que en los sobrevivientes, en cuanto a los niveles altos de neutrófilos (105).
En los pacientes con COVID-19, la activación de las células T estuvo presente; de manera que se demostró que las células T CD4+ son importantes mediadoras de la inmunidad humoral protectora a través de la estimulación de células B productoras de anticuerpos antivirales específicos. Por otra parte, se demostró que las células T CD8+ son consideradas una de las claves en la actividad citotóxica y la protección local, ante la infección. Si estas respuestas no fuesen suficientemente efectivas en COVID-19, la llamada tormenta de citoquinas (IL-2, Il-6, IL-7, IL-8, TNF e IL-17), conduce a una linfohistiocitosis hemofagocítica secundaria en el estado grave de la enfermedad, con severas implicaciones en el sistema gastrointestinal (104).
Por otra parte, los niveles de anticuerpos IgA, IgG e IgM encontrados en los pacientes con COVID-19 fueron similares, tanto en los diagnosticados con categoría leve como en los de diagnóstico severo, lo cual sugirió que no hubo un deterioro general en la actividad de las células B ni en los niveles del complemento con el incremento de la severidad (105).
En algunos estudios realizados en pacientes graves, se detectó una disminución en la cantidad de linfocitos, mayor número de neutrófilos y una elevada cantidad relativa de neutrófilos/linfocitos, con una disminución del porcentaje de monocitos, eosinófilos y basófilos. Por lo tanto, se ha hecho énfasis en la neutrofilia como factor de riesgo para la progresión en la severidad y fallecimiento de los pacientes, así como en la elevación de los marcadores de inflamación, como procalcitonina, proteína C, dímero D y ferritina (105).
Algunos resultados obtenidos durante los primeros meses de la pandemia causada por SARS-CoV-2 han permitido postular posibles modelos homeostáticos involucrados en la infección y gravedad de la enfermedad, resumidos por Neurath (106).
Los desórdenes homeostáticos durante la infección por SARS-CoV-2 fueron investigados en otro estudio, a través de la comparación de los niveles de un número representativo de posibles componentes de la regulación anti y proinflamatoria, en pacientes hospitalizados que permanecían en la unidad de cuidados intensivos (UCI), pacientes no incluidos en UCI (grupo no UCI) y sujetos sanos (107).
En el grupo de pacientes UCI se detectaron los mayores valores de interleuquinas IL-1beta, IL-1RA, IL-2, IL-4, IL-6, IL-7, IL-8, IL-9, IL-10, IL-13 e IL-17, del factor de crecimiento de fibroblastos, del factor de estimulación colónica de granulocitos (G-CSF), del factor de estimulación colónica de macrófagos, de interferón-γ (IFN-γ), del TNF, del factor de crecimiento endotelial, de la proteína MCP-1 de monocitos, de la proteína inflamatoria de macrófagos (MIP-1α), del factor de crecimiento derivado de las plaquetas y de la proteína MIP-1β. En todos los casos se compararon con los de los sujetos sanos. Sin embargo, en estos pacientes no se mostraron cambios en los niveles de IL-5, IL-12 e IL-15 (107).
La comparación de estos patrones en los pacientes UCI (estado severo de la enfermedad) y no UCI (estado leve) demostró un incremento de IL-2, IL-7, IL-10, TNF, G-CSF, MCP-1 y MIP-1α, con la severidad de la enfermedad pero sin variación en los niveles de IL-6 (107).
En otro estudio similar al anterior, la combinación de los análisis del dímero D y los niveles de IL-6, permitió proponer la inclusión de IL-6 como biomarcador de COVID-19 (108).
Queda un largo camino por recorrer para establecer detalladamente el complejo mecanismo de estos virus en epidemias locales o extensivas en términos de pandemias. Sin embargo, se ha abordado con gran rapidez el conocimiento de la etiología de COVID-19, sus posibles vías de contagio, medidas efectivas para evitar la propagación y obtención de vacunas preventivas (en el año 2020, se han registrado 85 833 resultados en las bases de referencias PubMed de la Biblioteca Nacional Médica de Estados Unidos; https://www.ncbi.nlm.gov/ revisado: 23 de diciembre de 2020).
Los elementos analizados podrían explicar la susceptibilidad de personas con determinados factores de riesgo para contraer la infección por SARS-CoV-2 (afecciones respiratorias, trastornos cardiovasculares e hipertensión, diabetes, obesidad, cáncer, enfermedades inflamatorias digestivas, edades avanzadas, a los que se podría añadir estrés y probablemente un patrón de consumo de alimentos al estilo occidental.
La caracterización basada en las técnicas de medios de cultivo sigue siendo de gran utilidad. Sin embargo, los métodos metagenómicos, metabolómicos y metatranscriptómicos han abierto una nueva línea de posibilidades para la identificación del panel bacteriano, viral y de hongos, en cuanto a genes, proteínas constituyentes, moléculas de señalización, vías metabólicas e inmunológicas y sus funciones (109) (110) (111) (112) (113) (114).
De ahí la utilidad de la caracterización metagenómica de la taxonomía microbiana intestinal como criterio diagnóstico complementario y preventivo. Son tecnologías costosas, pero una vez establecidas, se amortizarían gracias a la información que brindan para la prevención de futuras enfermedades. La tecnología de la preparación de bibliotecas genómicas para el gen que codifica la subunidad 16S del ARNr de las bacterias (1500 pb) y la introducción de los métodos actuales de análisis computacional, hacen que el proceso se abarate de un año a otro. Una mayor fidelidad en la identificación se logra gracias a la amplificación de regiones hipervariables de este gen, que permite obtener fragmentos más pequeños (de alrededor de 500 pb) (111).
Existen diferentes plataformas (uBiome, American Gut, BIOHM, DayTwo, THRYVE, VIOME) para la caracterización de la taxonomía y contenido de todos los microorganismos presentes en el intestino (Tabla II), a través de la evaluación metagenómica en muestras de biopsias colorrectales y heces (115). Estos sistemas se basan en la autorrecolección de muestras y envío en condiciones adecuadas al centro de evaluación. Los resultados del estudio (que pueden tardar hasta 3 meses) son recibidos por los sujetos solicitantes, con la explicación requerida para que el médico especialista analice la efectividad de un tratamiento aplicado, cambios en la dieta y su efecto sobre la diversidad del microbioma. Algunas plataformas también incluyen recomendaciones, en cuanto a cambios en los estilos de consumo de alimentos (nutrición personalizada y proporción en los géneros bacterianos que garanticen el equilibrio en la microbiota) (115).
De momento, estas plataformas están distribuidas en los EE.UU., el Reino Unido y Australia, entre otros, pero no de forma generalizada en todo el mundo. Por lo tanto, sería ventajosa su ampliación hacia aquellos países donde se ha demostrado el papel de la microbiota en el desarrollo de enfermedades epidémicas, lo cual garantiza la caracterización de la tendencia en el patrón de la microbiota intestinal, a tal punto que se podrían llegar a predecir puntos críticos sociodemográficos de mayor susceptibilidad a determinados patógenos.
La experiencia adquirida durante la actual pandemia causada por el SARS-CoV-2, ha permitido desarrollar pruebas, tales como: el método de reacción en cadena de la polimerasa (PCR) para la detección de la presencia del ARN viral, a partir de muestras nasofaríngeas (116) y las tiras diagnósticas para detección de IgG/IgM, a partir de muestras de sangre tomadas 7 días después que las personas fueron infectadas por el virus (como control epidemiológico y de la evolución de los pacientes infectados) (117). De igual manera, se podrían implementar pruebas complementarias con la plataforma de tiras que permitan detectar la presencia de aquellos marcadores bacterianos presentes en heces, susceptibles a variación en este tipo de epidemias.
En la actualidad, no existen referencias disponibles acerca de la existencia de métodos rápidos de tiras o los conocidos como genosensores (118) (119) para detección múltiple de varios filos. Existen sistemas de microarrays, como el que se utiliza para la detección e identificación de enterobacterias causantes de diarrea (CLART®EnteroBac) (120) que facilitan la identificación de los principales marcadores microbianos presentes en heces causantes de diarreas.
Ya se está en condiciones de colocar al microbioma como una prueba para el diagnóstico complementario de las enfermedades, conceder el papel que merece a este órgano adicional que el ser humano posee, para el mantenimiento de la salud, la defensa contra las infecciones, el tratamiento contra el cáncer y la inflamación, al cual no se le prestó una atención significativa (121) (122).
Conclusiones
En poco tiempo se han acumulado nuevos conocimientos y se ha enriquecido el concepto de microbiota como algo esencial en la vida y la salud del ser humano y su importancia potencial en la lucha contra epidemias frente a agentes infecciosos, a otros trastornos como la obesidad, la diabetes, el cáncer, sus principales causas y efectos relacionados con el desbalance interno y en su relación con la ecología. Con las herramientas de la metagenómica estaremos mejor preparados para conocer más acerca de su equilibrio metabólico e inmunológico y alcanzar una salud estable.
Agradecimientos
Los autores agradecen a todos los profesionales bioquímicos, microbiólogos, médicos y epidemiólogos que continúan trabajando en el diagnóstico y prevención de enfermedades relacionadas con la microbiota intestinal. Al Dr. José Moreiro Socias y a la Sra. Begoña Alonso Zulueta, profesionales de Endocrinología y Nutrición, promotores de los estudios de microbiota intestinal, desde 2010.
Fuentes de financiación
El presente trabajo fue realizado sin haberse recibido una financiación específica.












 uBio
uBio