Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Revista argentina de microbiología
versión impresa ISSN 0325-7541versión On-line ISSN 1851-7617
Rev. argent. microbiol. v.37 n.2 Ciudad Autónoma de Buenos Aires abr./jun. 2005
Aplicación de PCR-RFLP para subtipificar Campylobacter jejuni
G. Giacoboni1*, M.G. Echeverría2, C. Perfumo3
1Laboratorio de Diagnóstico e Investigaciones Bacteriológicas, Facultad de Ciencias Veterinarias, Universidad Nacional de La Plata. 60 y 118 CC296 (B11900AVW) La Plata, Argentina. 2Cátedra de Virología CONICET; 3Cátedra de Patología Especial.
*Correspondencia. E-mail: giacoboni@fcv.unlp.edu.ar
RESUMEN
Diez cepas de Campylobacter jejuni aisladas de fetos porcinos abortados fueron identificadas por pruebas bioquímicas: 8 como C. jejuni biotipo II de Lior, y 2 como C. jejuni biotipo I. Para poder subtipificarlas se utilizó la técnica de reacción en cadena de la polimerasa (PCR) para amplificar el gen flaA y al producto obtenido se lo digirió con la enzima de restricción DdeI (RFLP). Se pudieron obtener 6 subtipos a partir de C. jejuni biotipo II, mientras que los dos aislamientos de biotipo I correspondieron a un mismo subtipo. Aunque existe una amplia variedad de técnicas de biología molecular que son aplicadas con fines epidemiológicos para Campylobacter, PCR-RFLP, demostró ser una técnica simple y accesible, capaz de subtipificar a C. jejuni.
Palabras clave: Campylobacter jejuni, subtipificación, PCR-RFLP
SUMMARY
PCR-RFLP for Campylobacter jejuni subtyping. Ten Campylobacter jejuni isolates, 8 identified as C. jejuni biotype II of Lior and 2 as C. jejuni biotipe I, were recovered from aborted pig fetuses. In order to discriminate among strains, restriction fragment length polymorphism (RFLP) using DdeI of polymerase chain reaction (PCR) products of flaA gen was used. C. jejuni biotype II strains could be diferenciated in 6 by PCR-RFLP, and one subtype was obtained from C. jejuni biotype I. Although there is great variability of molecular techniques applied to the Campylobacter epidemiological studies, PCR-RFLP demonstrated to be a simple and accessible technique to discriminate Campylobacter jejuni isolates.
Key words: Campylobacter jejuni, subtypes, PCR-RFLP
Desde que Vandamme en 1991 propuso a la familia Campylobacteraceae como superfamilia VI de acuerdo con los estudios de rRNA, una amplia variedad de subespecies se definieron tanto en el género Campylobacter como en el género Arcobacter (12). Las especies C. jejuni y C. coli son una de las causas de enteritis aguda en humanos, siendo los animales domésticos y silvestres, en particular las aves, los reservorios de C. jejuni, y los cerdos de C. coli (1). Los alimentos de origen animal (carne de aves, cerdo) hacen de esta zoonosis una enfermedad transmitida por alimentos (ETA), entre otras maneras de transmitirse al humano.
Estas bacterias tienen escasa actividad bioquímica. Al no fermentar los hidratos de carbono, se utilizan pruebas de tolerancia a diferentes temperaturas, la prueba de la hidrólisis del hipurato e hidrólisis del indoxil acetato, la sensibilidad a discos de 30 µg de ácido nalidíxico y cefalotina, y según la especie a identificar la tolerancia a diferentes concentraciones de sustancias como NaCl, glicina, etc. Son pocos los métodos fenotípicos (serotipificación, biotipificación, tipificación por fagos) que discriminen satisfactoriamente una especie o subespecie. Este inconveniente se exacerba porque todos ellos revelan una enorme diversidad (10).
Actualmente, la biología molecular aporta una amplia variedad de técnicas para subtipificar genotípicamente a Campylobacter spp. Algunas de ellas se basan en la digestión por enzimas de restricción de productos de PCR (PCR-RFLP) (4), en la amplificación al azar del ADN polimórfico (RAPD) (7), ribotipificación (3) o la electroforesis en geles de agarosa con campo pulsado (PFGE) (2, 8).
Dentro de la gran oferta de posibilidades de las técnicas de biología molecular, la elección depende del objetivo del trabajo y las posibilidades de poder acceder a ellas, pues requieren un equipamiento especial y altos costos en los elementos que se utilizan para realizarlas. A lo anterior habría que sumarle que no hay una prueba de oro incuestionable que se utilice como referencia, por lo que las variaciones en los resultados que se obtienen por las diferentes pruebas aplicadas dificultan su interpretación (13).
La técnica de PCR-RFLP para el gen flaA se basa en la amplificación del gen de la flagelina por PCR, seguida por la digestión por enzimas de restricción que generen fragmentos. La diferencia en la distribución de los sitios de restricción en los genes fla entre las cepas generará fragmentos de diferente tamaño y por lo tanto un modelo de banda diferente, que se visualizará cuando se los separa por electroforesis (4).
El objetivo de este trabajo fue aplicar una técnica de biología molecular accesible, con la que pudiéramos subtipificar las especies de Campylobacter jejuni de origen porcino obtenidas de diferentes establecimientos (cuatro), e identificadas previamente por pruebas bioquímicas.
A 10 cepas aisladas de abortos porcinos e identificadas bioquímicamente como C. jejuni I de Lior (dos cepas, denominadas F3 y F4), C. jejuni II de Lior (ocho cepas denominadas F1, F2, F5, F6, F8, F9, F10 y F11) (6) se les extrajo el ADN por el método de fenol/cloroformo/isoamilalcohol (5) y la cuantificación se realizó en espectrofotómetro. El ADN obtenido se diluyó con agua destilada estéril hasta obtener una concentración final de 20 ng/µl. Para la reacción en cadena de la polimerasa se utilizaron los cebadores para amplificar el gen flaA, obteniéndose un amplicon de 1700 bp. La secuencia de nucleótidos de los cebadores fueron: 1: 5'-GGA TTT CGT ATT AAC ACA AAT CGT GC y 2: 5'-CTG TAG TAA TCT TAA AAC ATT TTC. La reacción se llevó a cabo en un volumen final de 100 µl conteniendo 8 µl del ADN diluido. Los reactivos utilizados y la concentración final de la mezcla para PCR fueron: 1,5 mM MgCl2, 1µM de cada uno de los cebadores, 200 µM mezcla de cada uno de los dNTPs y 2,5 U de Taq polimerasa. Las condiciones de ciclado (Eppendorf Mastercicler Gradient) fueron: 94 ºC 1 min, y luego 45 ciclos de 94 ºC por 59 seg para la desnaturalización, para el alineamiento 52 ºC por 50 seg y para la extensión a 72 ºC durante 1 min 45 seg. Con un último ciclo de 5 min a 72 ºC. Cinco microlitros del producto se sembraron en un gel de agarosa de 0,7%, se tiñó con bromuro de etidio y se observó bajo transiluminador.
Cada uno de los productos obtenidos por PCR se precipitó con 2 volúmenes de etanol frío 99%, se lavó con etanol 70% y se resuspendió en 80 µl de buffer Tris-EDTA. Veinte µl del producto purificado se digirieron con la enzima DdeI a 37 ºC 24 horas y posteriormente se sembraron en gel de agarosa al 2%, el cual fue sometido a electroforesis por 4 horas a 90V. Se utilizó además un producto de PCR digerido con DdeI de un aislamiento de C. coli (denominado F7) obtenido también de abortos porcinos y biotipificado previamente como C. coli biotipo I de Lior y una cepa de C. jejuni ATCC 29.488.
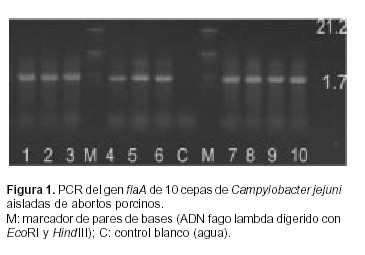
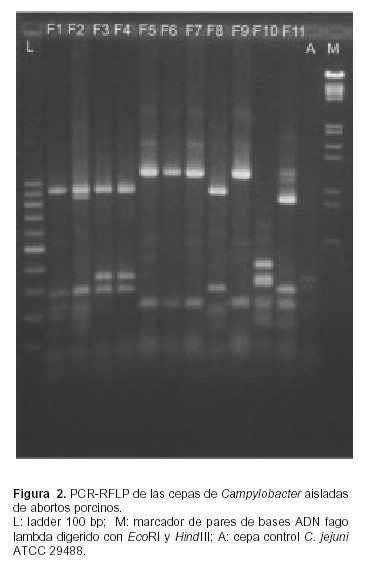
La reacción de PCR generó productos de 1700 bp de las 10 cepas procesadas (Figura 1). La digestión con la enzima DdeI mostró bandas de 100 a 1200 bp, diferenciándose distintos subtipos denominados A, B, C, D, E, F, G y H. Al subtipo A pertenece la cepa F1 (C. jejuni biotipo II de Lior); al subtipo B la cepa F2 (C. jejuni biotipo II de Lior); al subtipo C, F3 y F4 (C. jejuni biotipo I de Lior); al grupo D las cepas F5, F6 y F9 (C. jejuni biotipo II de Lior) y F7 (C. coli); al subtipo E, la F8 (C. jejuni biotipo II de Lior); la cepa F10 (C. jejuni biotipo II de Lior) al grupo F y la cepa F11 (C. jejuni biotipo II de Lior) al grupo G. La cepa de C. coli (F7) aislada de abortos porcinos del mismo muestreo, no pudo diferenciarse del resto de las cepas tipificadas como C. jejuni F5, F6 y F9 en el número de bandas obtenidas, incluyéndola en el grupo D. La cepa de control C. jejuni ATCC 29488 fue incluida en el grupo H (Figura 2).
Con la técnica de PCR-RFLP utilizando la enzima DdeI, se pudo diferenciar dentro de las especies identificadas como C. jejuni con el esquema de biotipificación de Lior, única herramienta utilizada en nuestro país como técnica para discriminar entre las especies de valor epidemiológico. La variabilidad genética del género Campylobacter ya ha sido descripta por otros autores, especialmente las cepas de origen porcino, utilizando PCR-RFLP digerido con otras enzimas (9) u otras técnicas como consenso intergénico repetitivo de enterobacteraceae (ERIC-PCR) (14) y ribotipificación (9).
La electroforesis de campo pulsado es la técnica de elección para estudios epidemiológicos en varias especies bacterianas. Sin embargo, los valores discriminatorios generados por ella, AFLP y RAPD son similares a los obtenidos por la evaluación del gen flaA por PCR-RFLP (4). La electroforesis por campo pulsado tiene el inconveniente de ser una técnica que está lejos de ser aplicada en laboratorios que no tienen acceso a equipamientos costosos. En especial, las bacterias de la familia Campylobacteraceae que no se buscan de rutina precisamente por requerir microaerofilia y convertirse éste en un factor limitante para su aislamiento. La subtipificación con PCR-RFLP con DdeI, podría ser una técnica útil en los comienzos de la diferenciación molecular de Campylobacter en nuestro país. Sin embargo, en nuestro estudio no pudimos diferenciar una cepa de C. coli aislada del mismo muestreo ni tampoco fue posible encontrar un patrón parecido al obtenido con la cepa de referencia C. jejuni ATCC 29.488. Esta técnica, como tantas otras aplicadas a Campylobacter están en vías de ser estandarizadas (4).
En este estudio, el poder de discriminación de la técnica fue aceptable y corroboró como lo hicieron otros autores (4, 11) que en la especie porcina pueden coexistir una gran variedad genotípica de Campylobacter (9).
Este trabajo fue presentado en el XVII Congreso Latinoamericano de Microbiología y X Congreso Argentino de Microbiología, realizado en la Ciudad Autónoma de Buenos Aires del 17 al 21 de octubre de 2004.
BIBLIOGRAFÍA
1. Blaser M, Berkowitz B (1979) Campylobacter enteritis: clinical and epidemiological features. Am. Inter. Med. 91: 179-185. [ Links ]
2. Champion C, Best E, Frost J (2002) Comparison of pulsedfield gel electrophoresis and amplified fragment length polymorphism techniques for investigating outbreaks of enteritis due to campylobacters J. Clin. Microbiol. 40: 2263-2265. [ Links ]
3. Fitzgerald C, Owen RJ, Stanley J (1996) Comprehensive ribotyping scheme for heat stable serotypes of Campylobacter jejuni. J. Clin. Microbiol. 34: 265-269. [ Links ]
4. Harrington C, Moran L, Ridley A, Newell D, Madden R (2003) Inter-laboratory evaluation of three flagellin PCR/RFLP methods for typing Campylobacter jejuni and C. coli: the CAMPYNET experience. J. Appl. Microbiol. 95: 1321-1333. [ Links ]
5. Lind L, Sjogren E, Melby K, Kaijser B (1996) DNA fingerprint and serotyping of Campylobacter jejuni isolates from epidemic outbreaks. J. Clin. Microbiol. 43: 892-896. [ Links ]
6. Lior H (1984) New extended biotyping scheme for Campylobacter jejuni, Campylobacter coli and Campylobacter laridis. J. Clin. Microbiol. 20: 636-640. [ Links ]
7. Madden R, Moran L, Scates P (1996) Sub-typing of animal and human Campylobacter spp. using RAPD. Lett. Appl. Microbiol. 23: 167-170. [ Links ]
8. Meinersmann R, PattonC, Evins G, Wachsmuth I, Fields P (2002) Genetic diversity and relationships of Campylobacter species and subspecies. Int. J. Syst. Evol. Microbiol. 52: 1789-1797. [ Links ]
9. Moore J, Lanser J, Heuzenroeder M, Rateliff R, Millar B, Madden R (2002) Molecular diversity of Campylobacter coli and C. jejuni isolated from pigs at slauther by flaA-RFLP analysis and ribotyping. J. Vet. Med. B. Infect. Dis. Vet. Public Health 49: 388-393. [ Links ]
10. Patton C, Wachsmuth I, Evins G, Kiehlbauch J, Plikaytis B, Troup N, et al. (1991) Evaluation of 10 methods to distin-guish epidemic-associated Campylobacter strains. J. Clin. Microbiol. 31: 1525-1530. [ Links ]
11. Petersen L, Newell D (2001) The ability of Fla-typing schemes to discriminate between strains of Campylobacter jejuni. J. Appl. Microbiol. 91: 217-224. [ Links ]
12. Vandamme P, De Ley J (1991) Proposal for a new family, Campylobacteraceae. Int. J. Syst. Bacteriol. 41: 451-455. [ Links ]
13. Wassenaarl T, Newell D (2000) Genotipyng of Campylobacter spp. Appl. Envirom. Microbiol. 66: 1-9. [ Links ]
14. Weijtens M, Reinders R, Urlings H, Van der Plas J (1999) Campylobacter infections in fattening pigs: excretion pattern and genetic diversity. J. Appl. Microbiol. 86: 63-70. [ Links ]
Recibido: 25/10/04
Aceptado: 23/6/05














