Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO  uBio
uBio
Share
Revista argentina de microbiología
Print version ISSN 0325-7541On-line version ISSN 1851-7617
Rev. argent. microbiol. vol.37 no.3 Ciudad Autónoma de Buenos Aires July/Sept. 2005
Tipificación molecular de Brucella abortus cepa 19 y su aplicación al control de biológicos
M.E. Pavan1, A. Nicola2, F. Grimoldi3, F. Cairó1, 3*
1 Biochemiq S.A. Viamonte 1365 piso 9, Ciudad Autónoma de Buenos Aires, Argentina; 2SENASA, Sector Brucelosis; 3Fac. Ciencias Veterinarias, Cátedra Enfermedades Infecciosas, Universidad de Buenos Aires, Chorroarín 280.
*Correspondencia. E-mail: mpavan@biochemiq.com; fcairo@biochemiq.com
RESUMEN
Brucella abortus es el agente etiológico de la brucelosis bovina. La cepa 19, utilizada en la elaboración de vacunas, puede ser identificada a través de una deleción en la región eri asociada con la sensibilidad al eritritol. Se optimizó un ensayo de PCR para caracterizar específicamente esta cepa. El método que describimos es un procedimiento rápido para identificar B. abortus y simultáneamente diferenciar la cepa 19 de otras cepas de B. abortus biovar 1. Hemos aplicado este ensayo para la detección de la cepa 19 en vacunas contra la brucelosis bovina elaboradas en Argentina. Los resultados indican que este método podría ser útil para el seguimiento de las cepas madres y semillas utilizadas en la producción industrial de esta vacuna. Esta metodología también contribuiría a la reducción del riesgo de la infección adquirida en el laboratorio y podría aplicarse como prueba de rutina para confirmar la presencia de B. abortus en vacunas no relacionadas.
Palabras clave: Brucella abortus, identificación, vacuna, diagnóstico, PCR, brucelosis
SUMMARY
Molecular characterization of Brucella abortus strain 19 and its application for controlling biologics. Brucella abortus is the etiological agent of bovine brucellosis. The strain 19 used in vaccine elaboration can be identified through a deletion in the eri region associated with its susceptibility to erythritol. We optimized a PCR assay for specific characterization of this strain. The method described here is a rapid procedure that enables identification of B. abortus, and simultaneous differentiation of the strain 19 from other B. abortus biovar 1 strains. We applied the assay to detect the strain 19 in vaccines against B. abortus produced in Argentina. The results show this method could be used to follow vaccine seed cultures of this strain. The methodology could also contribute to reduce the risk of a laboratory-acquired infection and could be of great help as a routine test for confirmation of B. abortus in non related vaccines.
Key words: Brucella abortus, identification, vaccine, diagnosis, PCR, brucellosis
INTRODUCCIÓN
Brucella abortus es uno de los principales agentes causantes de enfermedad en bovinos a nivel mundial. La infección con este organismo usualmente causa aborto y eventualmente puede producir enfermedad en humanos causando fiebre ondulante entre otros síntomas (7).
En Argentina y otros países el control de la brucelosis bovina se realiza mediante la vacunación de las terneras con una cepa atenuada de B. abortus biovar 1, la cepa 19 (fase lisa), y por segregación de los positivos a las pruebas serológicas confirmatorias.
Las técnicas bacteriológicas clásicas requieren un mínimo de cinco días para la identificación de cepas del género Brucella. Por tal motivo, internacionalmente se han hecho esfuerzos para identificar y diferenciar a nivel molecular a través de la reacción en cadena de la polimerasa (PCR) las seis especies del género Brucella y distintos biovares de B. abortus (1, 3) y para distinguir la cepa RB51, cepa atenuada, rugosa, utilizada como vacunal en otros países, de la cepa 19 de B. abortus (2, 3). Por otro lado, la cepa 19 puede diferenciarse de otras cepas de B. abortus por su inhabilidad para catabolizar eritritol, resultado de una deleción de 702 pares de bases (pb) en el locus eri. Esta deleción se ha utilizado para distinguir por PCR la cepa 19 de otras cepas de B. abortus biovar 1 (7). La OIE menciona que es posible identificar las cepas 19 y RB51 por PCR y remite a la bibliografía ya mencionada (5).
En estos trabajos, la cepa 19 de B. abortus se identifica indirectamente por la inhabilidad para amplificar una secuencia de ADN ubicada en el locus eri (1, 2, 3). Esto implica que cuando simultáneamente están presentes la cepa 19 y otras B. abortus (RB51 o patógenas), no es posible identificarla. Cuando la cepa 19 es identificada positivamente a través de un fragmento de amplificación, los datos aportados sobre las condiciones de amplificación son insuficientes (7).
El objetivo de este trabajo fue optimizar un método de PCR para la rápida tipificación a nivel molecular de la cepa 19, aplicarlo a la detección de esta cepa en vacunas contra la brucelosis bovina elaboradas en Argentina y determinar su sensibilidad para la detección de una contaminación con la cepa 19 de una vacuna bacteriana formolada, situación indeseada que puede ocurrir en plantas elaboradoras de vacunas.
MATERIALES Y MÉTODOS
Cepas y vacunas utilizadas
B. abortus biovar 1 cepa 19 (SENASA) y B. abortus biovar 1 cepa RB51 (SENASA) fueron crecidas en medio agar triptosa (Difco) a 37 °C. Vacunas contra la brucelosis bovina de seis laboratorios privados diferentes se adquirieron en las bocas de expendio (entes y distribuidoras) para su estudio.
Preparación del templado de ADN
Las vacunas se prepararon 10 veces concentradas con respecto a las instrucciones del laboratorio elaborador (200 µl agua bidestilada/dosis). Luego se resuspendieron 5 µl de cada vacuna en 100 µl de agua bidestilada en un tubo de 500 µl de pared delgada, se calentaron 10 min a 90 °C y enfriaron 1 min a 4 °C en un termociclador MJ Research con tapa caliente (PTC-150 con hot bonnet) para inactivar la muestra. Se centrifugó a 6.600 x g durante 1 min y el sobrenadante se tomó como el templado de ADN. Hemos probado que un calentamiento a 70 °C producía la liberación de suficiente ADN como para permitir su amplificación, sin mediar un paso previo de purificación. Sin embargo, se prefirió calentar las muestras a 90 °C con la finalidad de inactivarlas para reducir al mínimo su manipulación ya que la brucelosis es una zoonosis y una de las enfermedades más comunes adquiridas en el laboratorio.
Preparación del templado de ADN a partir de una vacuna formolada contaminada ex profeso
Un número decreciente de bacterias (B. abortus cepa 19) se agregó a un volumen fijo de vacuna contra Clostridium spp. La mezcla se incubó durante 18 horas a 37 °C. Al día siguiente se congeló a -20 °C y descongeló a 37 °C, y se dejó sedimentar durante 15 min a temperatura ambiente. El sobrenadante se centrifugó 1 min a 16.000 x g, las bacterias se lavaron 3 veces con dos volúmenes de agua para eliminar el formol, se resuspendieron 10x con respecto al volumen original y se continuó con la preparación del templado de ADN detallada previamente.
Oligonucleótidos utilizados en las distintas PCR
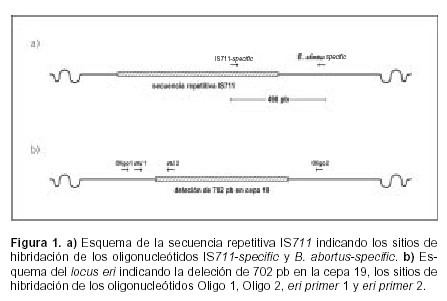
Se utilizaron los oligonucleótidos mencionados en los trabajos originales: IS711-specific 5' tgccgatcacttaagggccttcat (1), B. abortus-specific 5' gacgaacggaatttttccaatccc (1), eri primer 1 5' gcgccgcgaagaacttatcaa (2), eri primer 2 5' cgccatgttagcggcggtga (2), Oligo 1 5' ttggcggcaagtccgtcggt (7) y Oligo 2 5' cccagaagcga-gacgaaacg (7). El oligonucleótido IS711-specific hibrida en la secuencia repetitiva IS711, ésta es única y estable para todas las especies del género Brucella (1, 4, 6). El oligonucleótido B. abortus-specific hibrida en una región adyacente al extremo 3' del elemento IS711 que es específica para B. abortus. El fragmento de amplificación que se obtiene utilizando este par de oligonucleótidos es de 498 pb y permite diferenciar B. abortus de B. melitensis, B. canis, B. neotomae, B. ovis y B. suis (1), (Figura 1a). El eri primer 1 hibrida con una región del locus eri que es común a todas las cepas de todas las especies del género Brucella. El eri primer 2 hibrida dentro de la deleción de 702 pb que caracteriza a la cepa 19 (2, 3). Todas las cepas de B. abortus dan un fragmento de amplificación de 178 pb con este par de oligonucleótidos con excepción de la cepa 19 con la que no se obtiene un producto de amplificación porque el eri primer 2 no puede hibridar (Figura 1b). Los oligonucleótidos Oligo 1 y Oligo 2 hibridan en el locus eri, uno a cada lado de los extremos de la deleción encontrada en la cepa 19 (7). Este par de oligonucleótidos da un fragmento de amplificación de 361 pb con la cepa 19 (debido a la deleción) y de 1063 pb con todas las otras cepas de B. abortus (Figura 1, b).
Optimización de la PCR
El templado de ADN (5 µl) se agregó a la mezcla de reacción que contenía buffer Platinum Taq ADN polimerasa (20 mM Tris-HCl pH 8,4; 50 mM KCl), 1,5 mM MgCl2, 200 µM de cada dNTP, 2 unidades de Platinum Taq DNA polimerasa (Invitrogen), 0,5 µM de cada uno de los oligonucleótidos B. abortus-specific, IS711-specific y 1 µM de cada uno de los oligonucleótidos eri primer 1 y Oligo 2 (o 1 µM de Oligo 1 y Oligo 2 ), en un volumen final de 50 µl. Para optimizar este protocolo se hicieron varios ensayos de PCR en donde los oligonucleótidos utilizados fueron los que se indican en cada figura. El programa de amplificación optimizado fue: desnaturalización inicial a 94 °C durante 5 min seguida por 35 ciclos de 30 seg a 94 °C, 30 seg a 60 °C y 85 seg a 72 °C, y un ciclo final de 10 min a 72 °C. En los ensayos se incluyó un control negativo, donde el templado de ADN se reemplazó por agua, y un control positivo donde el templado de ADN se obtuvo a partir de un cultivo de B. abortus cepa 19. Todas las amplificaciones se realizaron en el termociclador MJ Research. Los productos de amplificación (8 µl) se analizaron por electroforesis en un gel 2,5% de agarosa en buffer TAE (40 mM Tris-acetato, 1 mM EDTA) durante 30 minutos. Los fragmentos se visualizaron por tinción con bromuro de etidio y luego se fotografiaron.
RESULTADOS
Puesta a punto del método de PCR
Las condiciones óptimas de amplificación se encontraron trabajando con los oligonucleótidos de a pares y luego haciendo diversas combinaciones con cuatro oligonucleótidos o una mezcla con los seis oligonucleótidos. Los esquemas de la figura 1 indican los sitios de hibridación de todos los oligonucleótidos utilizados.
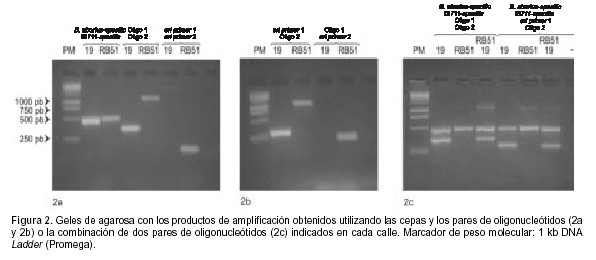
En la figura 2 se muestran los productos de amplificación para la cepa 19 y para la cepa RB51 que se eligió como representante del biovar 1 de B. abortus (para trabajar únicamente con cepas atenuadas). Con cada par de oligonucleótidos se obtuvo el fragmento del tamaño esperado según se mencionó anteriormente (Figura 2a).
La combinación del eri primer 1 con el Oligo 2 dio un fragmento de 297 pb para la cepa 19 y de aproximadamente 1000 pb para la cepa RB51 (Figura 2b). La combinación Oligo 1 con el eri primer 2 no dio fragmento con la cepa 19 y mostró un fragmento de 242 pb con la cepa RB51 (Figura 2b). En ambos casos los tamaños de los fragmentos de amplificación correspondieron con los esperados teóricamente.
Los oligonucleótidos B. abortus-specific y IS711-specific, que distinguen a la especie B. abortus, se combinaron con los otros oligonucleótidos, según se indica en la figura 2c, para diferenciar positivamente a la cepa 19 a través de un fragmento de amplificación, en un mismo tubo de reacción. La combinacion B. abortus-specific + IS711-specific + eri primer 1 + Oligo 2, así como la combinación B. abortus-specific + IS711-specific + Oligo 1 + Oligo 2 resultaron aptas para ser utilizadas en la misma reacción ya que se distinguen claramente los dos fragmentos de amplificación en un gel con alta concentración de agarosa. No pudieron utilizarse simultáneamente los seis oligonucleótidos debido a la generación de artefactos de bajo PM (menos de 250 pb).
Tipificación por PCR de la cepa de Brucella presente en las vacunas contra la brucelosis bovina producidas en Argentina
En todos los frascos de vacuna contra la brucelosis bovina producidos por distintos laboratorios privados se verificó la presencia de B. abortus cepa 19 ya que se obtuvo el fragmento de 498 pb específico para B. abortus y el de 297 pb específico para la cepa 19 (Figura 3). No se obtuvieron productos de amplificación de tamaños que correspondieran a otras cepas de B. abortus. Los controles positivos y negativos fueron satisfactorios.

Sensibilidad de la PCR optimizada para detectar contaminaciones con la cepa 19 de B. abortus
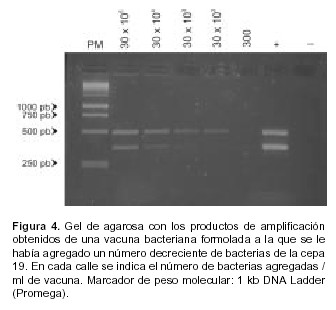
Un número decreciente de bacterias de la cepa 19 se agregaron ex profeso a un volumen fijo de una vacuna bacteriana formolada, con la idea de determinar la sensibilidad de la PCR optimizada para detectar dicha contaminación.
La figura 4 muestra que este protocolo fue útil para un amplio rango de contaminación con la cepa 19. El límite de detección hallado fue de 3000 bacterias contaminantes / ml de vacuna.
DISCUSIÓN
Hemos puesto a punto una PCR para tipificar a nivel molecular la cepa 19 de B. abortus, cepa utilizada en Argentina para la elaboración de vacunas contra la brucelosis bovina. El protocolo incluye un paso de inactivación de las bacterias durante la preparación del templado de ADN, con lo que se reducen a un mínimo los riesgos inherentes a la manipulación de esta cepa.
Es un método rápido que puede realizarse en el día y permite obtener una primera información fidedigna, mientras paralelamente pueden llevarse a cabo los métodos microbiológicos clásicos.
Es de destacar que esta PCR permite una detección doble, obteniéndose en un solo tubo de reacción un fragmento de amplificación específico para B. abortus y otro específico para la cepa 19. Con este método constatamos la presencia de la cepa 19 en todos los frascos de vacuna contra la brucelosis bovina analizados, y no encontramos otras cepas de B. abortus. Los vehículos de liofilización utilizados por los distintos laboratorios privados no han interferido en las reacciones de amplificación. Este método podría ser útil para el seguimiento de las cepas madres y semillas utilizadas en la producción industrial de esta vacuna, para detectar la contaminación con la cepa 19 de vacunas no relacionadas y para la identificación de cepas aisladas de muestras clínicas.
Agradecimientos: El presente trabajo fue realizado íntegramente con fondos aportados por Biochemiq S.A. El protocolo de PCR se identificó como BBA1.
BIBLIOGRAFÍA
1. Bricker BJ, Halling SM (1994) Differentiation of Brucella abortus bv. 1, 2, and 4, Brucella melitensis, Brucella ovis, and Brucella suis bv. 1 by PCR. J. Clin. Microbiol. 32: 2660-2666. [ Links ]
2. Bricker BJ, Halling SM (1995) Enhancement of the Brucella AMOS PCR assay for differentiation of Brucella abortus vaccine strains S19 and RB51. J. Clin. Microbiol. 33: 1640-1642. [ Links ]
3. Ewalt DR, Bricker BJ (2000) Validation of the abbreviated Brucella AMOS PCR as a rapid screening method for differentiation of Brucella abortus field strain isolates and the vaccine strains, 19 and RB51. J. Clin. Microbiol. 38: 3085-3086. [ Links ]
4. Halling SM, Tatum FM, Bricker BJ (1993) Sequence and characterization of an insertion sequence, IS711, from Brucella ovis. Gene 133:123-127. [ Links ]
5. Office International des Epizootes (2000) Leptospirosis. En: Manual of standards for diagnostic tests and vaccines. 4th edition. [ Links ]
6. Ouahrani S, Michaux S, Widada JS, Bourg G, Tournebize R, Ramuz M et al (1993) Identification and sequence analysis of IS6501, an insertion sequence in Brucella spp.: relationship between genomic structure and the number of IS6501 copies. J. Gen. Microbiol. 139: 3265-3273. [ Links ]
7. Sangari FJ, García-lobo JM, Agüero J (1994) The Brucella abortus vaccine strain B19 carries a deletion in the erythritol catabolic genes. FEMS Microbiol. Lett.121: 337-342. [ Links ]
Recibido 25/1/05
Aceptado 19/7/05














