Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO  uBio
uBio
Compartilhar
Revista argentina de microbiología
versão impressa ISSN 0325-7541versão On-line ISSN 1851-7617
Rev. argent. microbiol. v.38 n.2 Ciudad Autónoma de Buenos Aires ene./abr. 2006
Entorno genético de CTX-M-2 en aislamientos de Klebsiella pneumoniae provenientes de pacientes hospitalizados en Uruguay
R. Vignoli1, N. Cordeiro1, V. Seija1, F. Schelotto1, M. Radice2, J. Ayala3, P. Power2*, G. Gutkind2
1Departamento de Bacteriología y Virología, Instituto de Higiene, Facultad de Medicina, Universidad de la República, Alfredo Navarro 3051, Montevideo, Uruguay; 2Cátedra de Microbiología, Facultad de Farmacia y Bioquímica, Universidad de Buenos Aires, Junín 956, (1113) Ciudad Autónoma de Buenos Aires, Argentina; 3Centro de Biología Molecular «Severo Ochoa» CSIC-UAM, Campus de Cantoblanco, 28049 Madrid, España.
*Correspondencia. E-mail: ppower@ffyb.uba.ar
RESUMEN
Se estudiaron las cepas de Klebsiella pneumoniae K96005 y K13, productoras de la b-lactamasa de espectro extendido CTX-M-2, aisladas durante 1996 y 2003, respectivamente, de pacientes hospitalizados en Uruguay. Se realizó la caracterización del entorno génico del gen blaCTX-M-2 mediante mapeo por PCR y secuenciación de los amplicones. Los resultados revelan que en ambos aislamientos el gen codificante de dicha enzima se encuentra en un integrón complejo de clase 1, asociado a la presencia de orf513. El integrón identificado, cuyo arreglo de genes es aac(6’)-Ib, blaOXA-2, orfD, asociados a orf513-blaCTX-M-2, parece estar ampliamente diseminado en la región del Río de la Plata.
Palabras clave: orf513, integrón de clase 1, b-lactamasas de espectro extendido, blaCTX-M-2
ABSTRACT
Genetic environment of CTX-M-2 in Klebsiella pneumoniae isolates from hospitalized patients in Uruguay. We studied two CTX-M-2-producing Klebsiella pneumoniae clinical strains, K96005 and K13, isolated from hospitalized patients in Uruguay, during 1996 and 2003, respectively. The genomic surroundings of blaCTX-M-2 were characterized by PCR-mapping and DNA sequencing. Our results show that blaCTX-M-2 is included in a complex class-1 integron (InK13), associated with an orf513 in both isolates. The genetic array of the integron, aac(6’)-Ib, blaCTX-M-2, orfD (gene cassette region), associated with an orf513-blaCTX-M-2, seems to be widely disseminated over the Río de la Plata region.
Keywords: orf513, class 1 integron, extended-spectrum b-lactamases, blaCTX-M-2
La producción de b-lactamasas de espectro extendido (bLEE) en enterobacterias es un problema creciente en todo el mundo. Estas enzimas son capaces de inactivar a la mayoría de los antibióticos b-lactámicos (con la excepción de cefamicinas y carbapenemes).
El uso de antibióticos genera una presión de selección a favor de aquellas bacterias que son capaces de incorporar nuevo material genético, en particular genes que confieren resistencia. En este sentido, los integrones han jugado un papel preponderante en la captura, el rearreglo y la expresión de genes. De las nueve clases de integrones reconocidas hasta el momento, los inte-grones de clase 1 son los más frecuentemente asociados a resistencia en aislamientos clínicos (15).
La arquitectura de un integrón clásico de clase 1 consta de: i) un segmento conservado 5’ (5’-CS), que incluye el gen codificante de la integrasa de clase 1 (intI1); los promotores Pc y Pant, que dirigen la expresión de la integrasa IntI1 y de los genes en cassette, respectivamente; y un sitio attI1 de recombinación sitio específica; ii) un segmento conservado 3’ (3’-CS), compuesto básicamente por una copia trunca de qacED1, un gen que confiere resistencia marginal a compuestos de amonio cuaternario; y por el gen sul1, que confiere resistencia a sulfonamidas; y iii) una región variable (RV), que contiene los genes en cassette (15). Estos últimos carecen de promotor propio, pero se acompañan de un elemento de aproximadamente 59 pares de bases (59-be) o attC, de reconocimiento de la integrasa (15). Con esta arquitectura, los integrones son capaces de incorporar y expresar genes en cassette que, en general, codifican para resistencia a antibióticos.
Otros integrones derivados de los In6/In7, denominados integrones compuestos, han sido también descritos (9). Sobre la arquitectura clásica de un integrón de clase 1, se le agrega corriente abajo de 3’-CS un orf513, codificante de una hipotética recombinasa (12), algún gen de resistencia asociado a orf513 (carente del elemento 59-be) y una duplicación del 3’-CS (3’-CS2). Estos genes de resistencia asociados a orf513 no presentan un sitio de reconocimiento attC, pero sí un elemento de 26 a 28 bp que incluiría un sitio de reconocimiento de 17 bp para orf513 (12) y que, por analogía, llamaremos 26-be (Figura 1).

CTX-M-2 es la bLEE más frecuentemente detectada en Uruguay y Argentina (13, 17). Hace pocos años se ha descrito su ubicación en un integrón compuesto de clase 1 de estructura similar a la descrita en el párrafo anterior, el cual ha sido secuenciado a partir de aislamientos de Salmonella enterica serovar Infantis (InS21) (4), Morganella morganii (In116) (12) y Proteus mirabilis (In35) (1).
En Uruguay, los primeros informes de bLEE (año 1990) hacen referencia a enzimas derivadas de SHV en aislamientos de K. pneumoniae y E. coli (10). En 1991 se aislaron cepas de E. coli enteropatogénicas (EPEC) resistentes a oximino-cefalosporinas, que producen PER-2 y TEM-116 (18). Finalmente, corresponden al año 1996 los primeros aislamientos de K. pneumoniae con CTX-M-2 (17). Sin embargo, no han sido comunicados los entornos génicos de ninguna de ellas.
En este estudio, describimos el entorno genético de blaCTX-M-2 de un aislamiento de K. pneumoniae proveniente de un paciente hospitalizado durante el año 2003 en Uruguay (K13), comparándolo con uno de K. pneumoniae portador de CTX-M-2 de 1996 (K96005) (17).
Se estudiaron las cepas de K. pneumoniae K13 y K96005, ambas productoras de CTX-M-2. La primera fue seleccionada entre 11 aislamientos con idéntico perfil de multirresistencia, provenientes de hospitales de Montevideo, Canelones y Colonia, colectados durante 2003 (3). La segunda proviene de un estudio previo sobre aislamientos realizados en 1996 en Montevideo (17). Las cepas J53/pK13 y J53/pK05 fueron obtenidas mediante ensayos de conjugación, utilizando como cepa receptora E. coli J53 resistente a azida (Azr), con el objeto de determinar si los marcadores de resistencia son transferibles por elementos extracromosómicos.
Se determinó la susceptibilidad frente a una amplia variedad de antibióticos mediante paneles de microdilución (sistema Wider Fco. Soria Melguizo) y la concentración inhibitoria mínima (CIM) por dilución en agar, de acuerdo con las recomendaciones del CLSI (ex NCCLS) (8). Los resultados se muestran en la Tabla 1.

Luego de realizar un isoelectroenfoque analítico, los extractos crudos de ambas cepas, así como sus correspondientes transconjugantes, demostraron la presencia de tres bandas con actividad de b-lactamasas cuando se empleó ampicilina 500 µg/ml como sustrato revelador en un sistema iodométrico: pI aparentes de 5,4, 7,4-7,6 y 8,1. El revelado con ceftriaxona 1.000 µg/ml mostró una única banda de actividad con pI aparente de 8,1. La inhibición de las bLEE por clavulanato de litio (4 µg/ml) fue efectiva sobre dos de ellas (pI aparentes de 5,4 y 8,1) y solo parcial sobre la tercera (pI aparente de 7,4-7,6). Las actividades remanentes se revelaron con ampicilina 500 µg/ml.
Luego de amplificación por PCR se obtuvieron tres amplicones, de 0,85, 0,5 y 0,9 kb. Se utilizaron cebadores específicos de blaTEM (OTR3 y OTR4), blaOXA (OXA-2 A y OXA-2 B) y del grupo de blaCTX-M-2 (bla1 y bla2), respectivamente (ver Tabla 2); y ADN plasmídico.
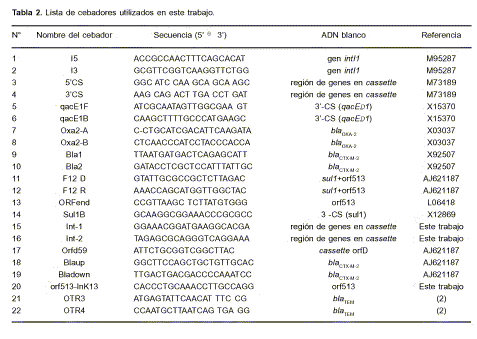
Los amplicones correspondientes a blaCTX-M-2 y blaOXA fueron secuenciados en ambos sentidos utilizando los mismos cebadores, en un secuenciador ABI PRISM 377. Para ambos aislamientos se confirmó la presencia de los genes blaCTX-M-2 y blaOXA2.
La detección de integrones de clase 1 se realizó mediante amplificación por PCR de las regiones conservadas 5’ (utilizando los cebadores I5 e I3) y 3’ (mediante los cebadores QacE 1F y Sul1B (ver Tabla 2). Los integrones fueron caracterizados de forma completa mediante mapeo por PCR y secuenciación de los amplicones, utilizando los cebadores descritos en la Tabla 2. Las combinaciones utilizadas, tanto para el mapeo como para la secuenciación, pueden verse en la Figura 1.
La secuenciación de los amplicones confirmó que el gen blaCTX-M-2 se encuentra en un integrón compuesto de clase 1 (Figura1), denominado InK13 en K. pneumoniae K13 (número de acceso AM040450), el cual presenta igual arquitectura que In116, InS21 (4, 12), e incluso, que el integrón de K. pneumoniae K96005, denominado InK05 (resultado no mostrado).
Con respecto a la región variable de genes en cassette de InK13, el arreglo aac(6’)-Ib - blaOXA-2 - orfD ha sido descrito, hasta el momento, únicamente para los integrones In116, InS21 e In35 de M. morganii, S. Infantis y P. mirabilis, respectivamente (1, 4, 12). Este trabajo aporta la primera descripción en K. pneumoniae.
Los genes involucrados en la resistencia observada poseen una identidad nucleotídica del 100% con los descritos en el integrón In116 (12), y son éstos aac(6’)-Ib, blaOXA-2, sul1 y blaCTX-M-2. La presencia de los genes blaCTX-M-2 y aac(6’)-Ib podrían explicar la resistencia a oximino-cefalosporinas y a aminoglucósidos, respectivamente.
La incorporación de genes bla en integrones permite agruparlos junto a otros que codifican para resistencia a diversos antibióticos, principalmente aminoglucósidos (1, 4, 12), en un entorno genético óptimo para su expresión. La diseminación de blaOXA-2 en K. pneumoniae, escasamente comunicada en la bibliografía (5), puede constituir un amplio reservorio de b-lactamasas relativamente resistentes a los inhibidores del tipo ácido clavulánico, que podrían derivar en bLEE resistentes a inhibidores, como fuera sugerido en Pseudomonas aeruginosa (11).
La secuencia del promotor Pant de intI1 en InK13 (regiones –35 [TTGACA] y –10 [TAAACT]) se presenta conservada con respecto a InS21 (número de acceso AJ311891), con una transversión T/A dos nucleótidos corriente abajo de la región –35, ausente en otras integrasas que han sido ingresadas en bases de datos. Se requieren estudios adicionales para determinar si esta modificación altera los niveles de expresión de los genes regulados por Pant.
El entorno génico corriente arriba del gen blaCTX-M-2 puede dividirse en tres zonas: una región inmediata de 506 bp, que abarca 266 bp con un 97% de similitud con respecto a una región homóloga corriente arriba de blaKLUA-1 (AJ272538) y que incluiría vestigios de su ubicación original en el cromosoma de Kluyvera ascorbata, y 240 bp con estricta identidad con la región común de integrones complejos derivados de In6/In7 (4, 12); una segunda región correspondiente a la hipotética recombinasa orf513, postulada como responsable de la escisión de blaCTX-M-2 desde su ubicación original (12) y, ubicado entre ambas regiones, se encuentra el elemento 26-be que contiene el sitio propuesto de reconocimiento de orf513 (AAAGGTGGTTTATACTTCCTATACCC). Este 26-be conserva un 100% de identidad corriente arriba de diversos genes de resistencia, como qnr (AY655485), blaCMY-1 (X92508) o dhfrX (AY049746), entre otros. Se postula que 26-be actuaría por sí mismo como un promotor para el gen de resistencia a quinolonas qnr (AY655485) mediante la secuencia TTTATA (7). Coincidentemente, 26-be comparte la secuencia AAAGGTGGTT con la secuencia de inserción ISEcp1 (16). Todos estos elementos sugieren que 26-be podría ser importante tanto para la movilización como para la expresión de genes de resistencia.
Para el caso de las b-lactamasas, tal movilización de genes ha permitido el reclutamiento de enzimas denominadas “cefotaximasas nacidas”, que no habrían evolucionado de precursores con menor espectro de actividad, como las derivadas de TEM y SHV (14). Los precursores de las enzimas del grupo CTX-M han sido encontrados, prácticamente inalterados, en el cromosoma de especies del género Kluyvera (6, 14), y han demostrado actividad bLEE al ser incluidos en un entorno favorable para su expresión (14).
Es interesante resaltar que los aislamientos estudiados (incluso K96005) corresponden a mediados de los años 90 en la región del Río de la Plata, al igual que los primeros aislamientos productores de CTX-M-2 estudiados en Argentina (1, 4, 12). Esta etapa podría corresponder a un período de diseminación regional de este tipo de integrones en diferentes especies. Sin embargo, estas estructuras del tipo de InK13 son relativamente estables, como lo señala el hecho de que se continúen detectando en aislamientos recientes. Por otro lado, su ubicación en un plásmido conjugativo contribuye a explicar, al menos en parte, la amplia diseminación interespecie de esta enzima tanto en Argentina como en Uruguay.
En este trabajo hemos descrito por primera vez el entorno genético de blaCTX-M-2 de cepas aisladas de pacientes hospitalizados en Uruguay. Los resultados obtenidos sugieren la diseminación regional de esta enzima, codificada en un integrón de clase 1 complejo.
Las secuencias parciales de los integrones InK13 e InK05 (de K. pneumoniae 96005) fueron depositadas en la base de datos EMBL bajo los números de acceso AM040450 y AM040449, respectivamente.
BIBLIOGRAFÍA
1. Arduino SM, Roy PH, Jacoby GA, Orman BE, Pineiro SA, Centron D. blaCTX-M-2 is located in an unusual class 1 integron (In35) which includes orf513. Antimicrob Agents Chemother 2002; 46: 2303-6. [ Links ]
2. Arlet G, Brami G, Décrè D, Flippo A, Gaillot O, Lagrange PH, et al. Molecular characterisation by PCR-restriction fragment length polymorphism of TEM b-lactamases. FEMS Microbiol Lett 1995; 134: 203-8. [ Links ]
3. Cordeiro N, Rodríguez A, Tarocco R, Seija V, Algorta G, Pedreira W, et al. VI Encuentro Nacional de Microbiología, 2003, Resumen 39, p. 64, Montevideo, Uruguay. [ Links ]
4. Di Conza J, Ayala JA, Power P, Mollerach M, Gutkind G. Novel class 1 integron (InS21) carrying blaCTX-M-2 in Salmonella enterica serovar Infantis. Antimicrob Agents Chemother 2002; 46: 2257-61. [ Links ]
5. Di Persio JR, Deshpande LM, Biedenbach DJ, Toleman MA, Walsh TR, Jones RN. Evolution and dissemination of extended-spectrum b-lactamase-producing Klebsiella pneumoniae: epidemiology and molecular report from the SENTRY Antimicrobial Surveillance Program (1997-2003). Diagn Microbiol Infect Dis 2005; 51: 1-7. [ Links ]
6. Humeniuk C, Arlet G, Gautier V, Grimont P, Labia R, Philippon A. b-Lactamases of Kluyvera ascorbata, probable progenitors of some plasmid-encoded CTX-M types. Antimicrob Agents Chemother 2002; 46: 3045-9. [ Links ]
7. Mammeri H, Van De Loo M, Poirel L, Martinez-Martinez L, Nordmann P. Emergence of plasmid-mediated quinolone resistance in Escherichia coli in Europe. Antimicrob Agents Chemother 2005; 49: 71-6. [ Links ]
8. National Committee for Clinical Laboratory Standards. Methods for dilution antimicrobial susceptibility tests for bacteria that grow aerobically; approved standard; Fifth edition, 2000; M7-A5. Wayne, Pa, USA. [ Links ]
9. Parsons Y, Hall RM, Stokes HW. A new trimethoprim resistance gene, dhfrX, in the In7 integron of plasmid pDGO100. Antimicrob Agents Chemother 1991; 35: 2436-9. [ Links ]
10. Pedreira W, Bazet C, López L. Nuevos mecanismos de resistencia bacteriana. Nuevas b-lactamasas plasmídicas de amplio espectro. Su importancia clínica y epidemiología actual. Arch Med Intern 1990; 12: 43-5. [ Links ]
11. Poirel L, Gerome P, De Champs C, Stephanazzi J, Naas T, Nordmann P. Integron-located oxa-32 gene cassette encoding an extended-spectrum variant of OXA-2 b-lactamase from Pseudomonas aeruginosa. Antimicrob Agents Chemother 2002; 46: 566-9. [ Links ]
12. Power P, Galleni M, Di Conza J, Ayala JA, Gutkind G. Description of In116, the first blaCTX-M-2-containing complex class 1 integron found in Morganella morganii isolates from Buenos Aires, Argentina. J Antimicrob Chemother 2005; 55: 461-5. [ Links ]
13. Quinteros M, Radice M, Gardella N, Rodríguez MM, Costa N, Korbenfeld D, et al. Extended-spectrum b-lactamases in Enterobacteriaceae in Buenos Aires, Argentina, public hospitals. Antimicrob Agents Chemother 2003; 47: 2864-9. [ Links ]
14. Rodríguez MM, Power P, Radice M, Vay C, Famiglietti A, Galleni M, et al. Chromosome-encoded CTX-M-3 from Kluyvera ascorbata: a possible origin of plasmid-borne CTX-M-1-derived cefotaximases. Antimicrob Agents Chemother 2004; 48: 4895-7. [ Links ]
15. Sabaté M, Prats G. Estructura y función de los integrones. Enferm Infecc Microbiol Clin 2002; 20: 341-5. [ Links ]
16. Saladin M, Cao VTB, Lambert T, Donay JL, Herrmann JL, Ould-Hocine Z, et al. Diversity of CTX-M b-lactamases and their promoter regions from Enterobacteriaceae isolated in three Parisian hospitals. FEMS Microbiol Lett 2002; 209: 161-8. [ Links ]
17. Vignoli R, Calvelo E, del Monte A, Ingold E, Power P, Radice M, et al. 9th International Congress on Infectious Diseases, 2000, Resumen 43021, p. 89, Buenos Aires, Argentina. [ Links ]
18. Vignoli R, Varela G, Mota MI, Cordeiro NF, Power P, Ingold E, et al. Enteropathogenic Escherichia coli strains carrying genes encoding the PER-2 and TEM-116 extended-spectrum b-lactamases isolated from children with diarrhea in Uruguay. J Clin Microbiol 2005; 43: 2940-3. [ Links ]
Recibido:14/11/05
Aceptado: 26/6/06














