Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO  uBio
uBio
Compartilhar
Revista argentina de microbiología
versão impressa ISSN 0325-7541versão On-line ISSN 1851-7617
Rev. argent. microbiol. v.38 n.3 Ciudad Autónoma de Buenos Aires jul./set. 2006
Comparación de diferentes métodos para identificar las especies del género Proteus
S. T. Castro1, C. R. Rodríguez1, B. E. Perazzi1, M. Radice2, M. Paz Sticotti3, H. Muzio3, J. Juárez1, G. Gutkind2, A. M. R. Famiglietti1, P. I. Santini†3, C. A. Vay1*
1Cátedra de Análisis Clínicos I, Laboratorio de Bacteriología, Departamento de Bioquímica Clínica, Facultad de Farmacia y Bioquímica, Hospital de Clínicas "José de San Martín", Universidad de Buenos Aires, Avda. Córdoba 2351 (1120) Ciudad Autónoma de Buenos Aires; 2Cátedra de Microbiología; 3Cátedra de Higiene y Sanidad, Facultad de Farmacia y Bioquímica, Universidad de Buenos Aires, Junín 956 (1113) Ciudad Autónoma de Buenos Aires, Argentina.
*Correspondencia. E-mail: cavay@ffyb.uba.ar
RESUMEN
Los objetivos de este trabajo fueron: a) identificar a nivel de especie aislamientos de Proteus siguiendo la combinación de los esquemas de Farmer y O'Hara; b) determinar la utilidad del sistema comercial API 20E y de un esquema reducido de pruebas (agar TSI y agar MIO: movilidad, indol y ornitina), comparar estos procedimientos con la metodología convencional y evaluar su sensibilidad y especificidad, y c) evaluar la utilidad del perfil proteico en la identificación de las distintas especies. Se estudiaron 205 aislamientos de Proteus spp. aislados en el período comprendido entre enero de 1998 y setiembre de 2004, recuperados de distintos materiales clínicos correspondientes a pacientes hospitalizados y ambulatorios atendidos en el Hospital de Clínicas. Los organismos fueron identificados mediante la metodología convencional, por el sistema API 20E y con un esquema reducido de pruebas; 48 de ellos fueron sometidos a un SDS-PAGE. API 20E identificó 79 de 87 aislamientos de P. mirabilis (90,8%), 103/103 del complejo P. vulgaris y 15/15 de P. penneri. Ocho aislamientos identificados como Proteus spp. resultaron ser P. mirabilis, al incluir una prueba adicional (maltosa). En la identificación, el esquema reducido coincidió en un 100% con la metodología convencional. A diferencia del sistema API 20E, el esquema reducido alcanza la correcta identificación de todas las especies en laboratorios de baja complejidad, sin la necesidad de pruebas adicionales. El perfil proteico permitió la correcta diferenciación de las tres especies, independientemente de las diferentes atipias de P. mirabilis.
Palabras clave: género Proteus, identificación
ABSTRACT
Comparison of different methods in order to identify Proteus spp. The objectives were: a) to identify Proteus strains to species level, following Farmer's and O'Hara's conventional biochemical reactions; b) to evaluate the sensitivity and specificity of both the API 20E method and a schema of reduced reactions (TSI and MIO agar: motility, indole and ornithine) comparing them with conventional methodology, and c) to evaluate the utility of SDS-PAGE (total proteins) in order to identify Proteus strains to species level. Two hundred and five Proteus spp. clinical isolates, were collected between January 1998 and September 2004, from inpatients and outpatients at Hospital de Clínicas. Strains were identified by means of conventional methodology, the API 20E method, and a schema of reduced reactions. SDS-PAGE (total proteins) was used in 48 out of the 205 strains. The API 20E method identified 79 out of 87 (90.8%) strains of P. mirabilis, 103 out of 103 P. vulgaris complex, and 15 out of 15 P. penneri. Eight strains of P. mirabilis were identified as Proteus spp., the acid production from maltose being necessary to identify them to species level. The schema of reduced reactions identified 205 out of 205 (100%) strains, that is, this schema of reduced reactions identified all the strains to species level without any additional tests, in marked contrast to the API 20E method. The SDS-PAGE (total proteins) identified the three species of the genus, even if the strains of P. mirabilis showed different biochemical reactions.
Key words: genus Proteus, identification
INTRODUCCIÓN
El género Proteus pertenece a la tribu Proteae de la familia Enterobacteriaceae, junto con los géneros Morganella y Providencia (2, 5). Recientemente, estudios de secuenciación de ARN ribosomal 16S y 5S han relacionado al género Plesiomonas con el género Proteus y lo han incorporado en la familia Enterobacteriaceae (14).
En la actualidad, el género está compuesto por 8 especies: Proteus mirabilis, Proteus vulgaris, Proteus penneri, Proteus hauseri, Proteus myxofaciens y las genomoespecies 4, 5 y 6. Las tres últimas y P. vulgaris sensu stricto forman el complejo P. vulgaris (9).
P. mirabilis y con menor frecuencia P. vulgaris y P. penneri han sido reconocidos como agentes etiológicos de distintos procesos infecciosos, tales como infección urinaria, infección quirúrgica, neumonías, bacteriemias, septicemias e infecciones óticas (5, 9). Asimismo, varios estudios mostraron la creciente implicancia del género en infecciones nosocomiales (5, 12).
La sensibilidad a los antibióticos ß-lactámicos difiere entre las distintas especies de este género. Proteus mirabilis es uno de los miembros más sensibles de la familia Enterobacteriaceae. Sin embargo, los perfiles de sensibilidad útiles para diferenciar los aislamientos salvajes de P. mirabilis (sensibles a ampicilina y cefalotina) de P. vulgaris y P. penneri (resistentes a ampicilina y cefalotina) no son del todo aplicables cuando las especies de Proteus adquieren resistencias a los antibióticos ß-lactámicos.
Los objetivos de este trabajo fueron:
– identificar a nivel de especie aislamientos pertenecientes al género Proteus siguiendo la combinación de los esquemas convencionales de Farmer y O'Hara (4, 9);
– evaluar la utilidad del sistema comercial API 20E y de un esquema reducido de pruebas (agar TSI y MIO: movilidad, indol y ornitina); compararlos con la metodología convencional y determinar su sensibilidad y especificidad;
– evaluar la utilidad del perfil de proteínas totales en la identificación de las especies de Proteus.
MATERIALES Y MÉTODOS
Microorganismos
Se estudiaron 205 aislamientos de Proteus spp., obtenidos en el período comprendido entre enero de 1998 y setiembre de 2004 de muestras clínicas de pacientes internados y ambulatorios atendidos en el Hospital de Clínicas "José de San Martín", de la Ciudad de Buenos Aires.
Los microorganismos fueron conservados en agar tripticasa soja a 4 °C y en leche descremada a –70 °C, y subcultivados en agar base Columbia (Britania, Buenos Aires, Argentina) con 5% de sangre y en agar EMB de Levine (Oxoid, Hamshere, England) para su posterior identificación.
Identificación bioquímica
El género Proteus fue identificado por su propiedad de invadir en agar base Columbia con sangre, y mediante las siguientes pruebas bioquímicas: fermentación de glucosa, sacarosa y lactosa, producción de sulfuro de hidrógeno y producción de gas a partir de glucosa, empleando en los tres casos agar TSI; desaminación de la lisina (agar LIA); hidrólisis de urea (medio Christensen); desaminación de la fenilalanina; hidrólisis de la gelatina y fermentación de la D-manosa. Las especies fueron identificadas sobre la base de las pruebas bioquímicas propuestas por Farmer et al. (4): producción de indol (agar MIO y SIM), descarboxilación de la ornitina (ODC por Möeller y agar MIO), y producción de ácido a partir de sacarosa y maltosa. En todos los casos se utilizaron productos Difco (Difco, Detroit, USA).
Se definió como complejo P. vulgaris a todos aquellos aislamientos de Proteus que produjeron indol, no descarboxilaron la ornitina y fermentaron la maltosa. Esta definición de complejo P. vulgaris, además de P. vulgaris sensu stricto y de las genomoespecies 4, 5 y 6, incluye a Proteus hauseri.
Las pruebas se incubaron a 35-37 °C durante 7 días, de acuerdo a los criterios de Farmer et al. (4), y los resultados fueron leídos a las 24 y a las 48 horas y a los 7 días, para la fermentación de hidratos de carbono y para la descarboxilación de la ornitina por Möeller. Las reacciones en agar TSI, LIA y fenilalanina fueron leídas a las 24 horas, en tanto que las del agar MIO, SIM y gelatina a las 48 horas.
Los microorganismos fueron identificados, además, por el sistema comercial API 20E (bioMérieux, Marcy l' Étoile, France), de acuerdo a la metodología descrita por el fabricante, y por un esquema reducido que incluyó: agar TSI y agar MIO (movilidad-indol-ornitina ).
Se analizó el perfil proteico de 48 cepas por SDS-PAGE, según metodología descrita por Laemmli et al. (7): 36 P. mirabilis, de los cuales 18 aislamientos presentaban características fenotípicas particulares (ornitina descarboxilasa negativa, no productores de sulfuro de hidrógeno o no invasores en agar sangre); 10 del complejo P. vulgaris y 2 P. penneri. Se compararon los resultados del esquema de Farmer et al. (4) (método de referencia) con los del sistema API 20E y los del esquema reducido. Se calculó sensibilidad, especificidad, valores predictivos positivo y negativo, y los correspondientes intervalos de confianza del 95% (EPI INFO 6.0).
RESULTADOS
Utilizando el esquema convencional, sobre un total de 205 aislamientos estudiados, 87 fueron identificados como P. mirabilis, 103 como pertenecientes al complejo P. vulgaris y 15 como P. penneri. Los resultados de la identificación de las especies de Proteus, según el esquema de Farmer et al. (4), se muestran en la Tabla 1.

El sistema comercial API 20E identificó 79 de los 87 (90,8%) aislamientos de P. mirabilis, todos los del complejo P. vulgaris y los correspondientes a P. penneri. Ocho aislamientos de P. mirabilis que no descarboxilaron la ornitina fueron identificados como Proteus spp. y requirieron la adición de la prueba de fermentación de maltosa para ser caracterizados como P. mirabilis (Tabla 2).

El esquema reducido identificó correctamente el 100% de los aislamientos, tanto los de P. mirabilis como los del complejo P. vulgaris y los de P. penneri (Tabla 3).

Los porcentajes de sensibilidad y especificidad, y los valores predictivos positivos y negativos del método comercial y del esquema reducido se muestran en la Tabla 4.
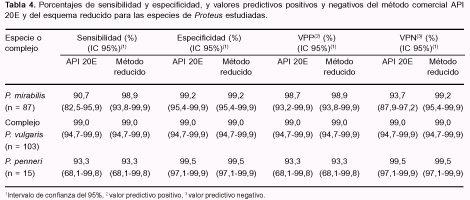
En la Figura 1 se observan los perfiles proteicos de algunos aislamientos de las diferentes especies de Proteus, donde cada especie muestra un electroferotipo particular.
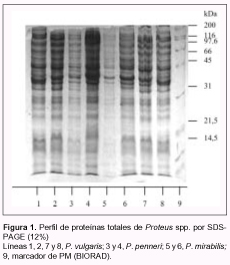
En la Figura 2 se muestran los perfiles proteicos de algunos aislamientos de P. mirabilis ODC positivos y ODC negativos, y de aislamientos de esta especie no productores de sulfuro de hidrógeno y no invasores en agar sangre, donde todos muestran idéntico electroferotipo.
DISCUSIÓN
El sistema API 20E identificó el 100% de las especies del complejo P. vulgaris y todos los aislamientos correspondientes a P. penneri a las 24 h, mientras que aquellos aislamientos de P. mirabilis ODC negativos fueron identificados correctamente a las 48-72 h, a partir de una prueba adicional (fermentación de maltosa). Estos resultados también coinciden con lo descrito por O' Hara et al. (10, 11). En este trabajo no se encontraron aislamientos de P. myxofaciens, puesto que este microorganismo, tal como lo describe la literatura, no ha sido hallado en muestras clínicas (1).
El esquema reducido permitió diferenciar todas las especies del género, incluidos los aislamientos de P. mirabilis que no descarboxilaron la ornitina, puesto que al no fermentar la sacarosa (TSI) pudieron ser diferenciados de P. penneri. Además, Farmer et al. (4) también demostraron que la totalidad de los 20 aislamientos de P. penneri estudiados fermentaron la sacarosa. El mismo resultado fue observado en este trabajo en los 15 aislamientos de P. penneri. Si bien Farmer et al. (4) describieron un 15% de aislamientos de P. mirabilis sacarosa positivos, esta característica no fue observada en nuestro estudio. De acuerdo con los resultados del trabajo de Farmer et al. (4), la probabilidad de encontrar un aislamiento de P. mirabilis sacarosa positivo y ornitina descarboxilasa negativo sería de 0,0015, por lo que el esquema mínimo propuesto en este estudio para la identificación de las especies resulta ser una alternativa válida. No obstante, sería conveniente confirmar la identidad de P. mirabilis ornitina descarboxilasa negativo con la prueba de fermentación de la maltosa.
Si bien en su esquema de identificación, el National Committee for Clinical Laboratory Standards (NCCLS) describe los aislamientos salvajes de P. mirabilis como sensibles a ampicilina (8), de acuerdo a resultados previamente publicados, en nuestro medio el 28% de los aislamientos fueron resistentes a aminopenicilinas y el 18% a cefalosporinas de primera generación debido a la presencia de una ß-lactamasa de amplio espectro del tipo TEM-1, por lo que se asemejan fenotípicamente a las especies de P. penneri y del complejo P. vulgaris, que poseen naturalmente una cefuroximasa cromosómica (13).
Por otra parte y también de acuerdo a resultados ya publicados, un 18% de los aislamientos de P. mirabilis fueron a su vez resistentes a las cefalosporinas de tercera generación, por la presencia de una ß-lactamasa de espectro extendido del tipo CTX-M-2, de manera que mostraron un fenotipo similar a los aislamientos de P. vulgaris y P. penneri hiperproductores de la ß-lactamasa cromosómica (13). Estos resultados apoyan la necesidad de identificar las especies teniendo en cuenta sus características fenotípicas. Si bien es una herramienta útil, el antibiotipo no puede ser utilizado como único elemento en la identificación.
En este estudio también se observó que los perfiles de proteínas totales resultan de utilidad para la diferenciación de las especies del género, independientemente de las distintas características fenotípicas presentes en P. mirabilis, puesto que los aislamientos ODC negativos, no productores de sulfuro de hidrógeno y no invasores, mostraron idéntico electroferotipo que los aislamientos de P. mirabilis con fenotipo habitual (3, 6). Por esta razón, dicha metodología resulta de utilidad como confirmatoria de la identidad en laboratorios especializados.
El esquema reducido resulta entonces interesante para la identificación de todas las especies de Proteus debido a su correlación con el esquema convencional, ya que identifica correctamente a todas ellas.
Agradecimientos: este trabajo fue realizado con el aporte del proyecto UBACyT B066 y el proyecto UBACyT B080.
BIBLIOGRAFÍA
1. Abbott SL. Klebsiella, Enterobacter, Citrobacter, Serratia, Plesiomonas and other Enterobacteriaceae. En: Murray PR, Baron EJ, Jorgensen JH, Pfaller MA, Yolken RH, editors. Manual of Clinical Microbiology. Washington D.C., ASM Press, 2003, p. 684-700. [ Links ]
2. Brenner DJ, Farmer JJ, Fanning GR, Steigerwalt AG, Klykken P, Wathen HG et al. Deoxyribonucleic acid relatedness of Proteus and Providencia species. Int J Syst Bacteriol 1978; 28: 269-82. [ Links ]
3. Costas M, Holmes B, Frith KA, Riddle C, Hawkey PM. Identification and typing of Proteus penneri and Proteus vulgaris biogroups 2 and 3, from clinical sources, by computarized analysis of electrophoretic protein patterns. J Appl Bacteriol 1993; 75: 489-98. [ Links ]
4. Farmer JJ, Davis BR, Hickman-Brenner FW, Mc. Whorter AC, Huntley-Carter GP, Asbury MA et al. Biochemical identification of new species and biogroups of Enterobacteriaceae isolated from clinical specimens. J Clin Microbiol 1985; 21: 46-76. [ Links ]
5. Gómez A, Baquero F, Nombela C. El género Proteus: aspectos microbiológicos y clínicos. Enf Inf Microbiol Clin 1991; 9: 567-75. [ Links ]
6. Holmes B, Costas M, Wood AC. Typing of Proteus mirabilis from clinical sources by computerized analysis of electrophoretic protein patterns. J Appl Bacteriol 1991; 71: 46-76. [ Links ]
7. Laemmli UK. Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature 1970; 227: 680-5. [ Links ]
8. National Committee for Clinical Laboratory Standards. Abbreviated identification of bacteria and yeast; approved guideline. Proteus spp, 2002; p. 5; M35-A. Wayne, Pa, USA. [ Links ]
9. O'Hara CM, Hickman-Brenner FW, Miller JM. Classification, identification and clinical significance of Proteus, Providencia, and Morganella. Clin Microbiol Rev 2000; 13: 534-46. [ Links ]
10. O'Hara CM, Rhoden DL, Miller JM. Reevaluation of the API 20E identification system versus conventional biochemicals for identification of members of the family Enterobacteriaceae: a new look at an old product. J Clin Microbiol 1992; 30: 123-5. [ Links ]
11. O'Hara CM, Tenover FC, Miller JM. Parallel comparison of accuracy of API 20E, Vitek GNI, MicroScan Walk / A way Rapid ID, and Becton Dickinson Cobas Micro ID-E/ NF for identification of members of the family Enterobacteriaceae and common gram-negative, non-glucose-fermenting bacilli. J Clin Microbiol 1993; 31: 3165-9. [ Links ]
12. Petras G, Bognar S. Origin and spread Pseudomonas aeruginosa, Proteus and Klebsiella in an infectious hospital. Acta Microbiol Acad Sci. Hung 1981; 28: 367-80. [ Links ]
13. Rodríguez C, Radice M, Perazzi B, Castro S, Juárez J, Santini P et al. Resistencia enzimática a betalactámicos en el género Proteus y evaluación de los fenotipos y genotipos de resistencia a cefalosporinas de tercera y cuarta generación en Proteus mirabilis. Enf Inf Microbiol Clin 2005; 23: 122-6. [ Links ]
14. Ruimy R, Breittmayer V, Elbaze P, Lafay B, Boussemart O, Gauthier M et al. Phylogenetic analysis and assesment of the genera Vibrio, Photobacterium, Aeromonas and Plesiomonas deduced from small-subunit rRNA sequences. Int J Syst Bacteriol 1994; 44: 416-26. [ Links ]
Recibido: 23/1/06
Aceptado: 17/7/06














