Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Revista argentina de microbiología
versión impresa ISSN 0325-7541versión On-line ISSN 1851-7617
Rev. argent. microbiol. v.38 n.4 Ciudad Autónoma de Buenos Aires oct./dic. 2006
Evaluación de dos técnicas de subtipificación molecular para el estudio de Pasteurella multocida
G. A. Leotta1,2*, I. Chinen1, G. B. Vigo2, J. Gugliada1, M. Rivas1
1Servicio Fisiopatogenia, Departamento Bacteriología, Instituto Nacional de Enfermedades Infecciosas - Administración Nacional de Laboratorios e Institutos de Salud “Dr. Carlos G. Malbrán”. Avda. Vélez Sarsfield 563 (1281) Ciudad Autónoma de Buenos Aires; 2Laboratorio de Diagnóstico e Investigaciones Bacteriológicas, Facultad de Ciencias Veterinarias, Universidad Nacional de La Plata, 60 y 118, cc: 296 (1900) La Plata, Pcia. de Buenos Aires. Argentina.
*E-mail: galeotta@anlis.gov.ar
RESUMEN
Se determinó la tipibilidad, la reproducibilidad y el poder discriminatorio de ERIC-PCR y ApaI-PFGE para establecer la relación genética de cepas de Pasteurella multocida. Se estudiaron 49 cepas de diferente origen, subespecie, biotipo, grupo capsular, serotipo somático y perfil de resistencia antimicrobiana. Por ERIC-PCR se establecieron 31 patrones, los que presentaron entre 10 y 14 bandas en un rango comprendido entre 0,2 y 1,2 kb. Por ApaI-PFGE se detectaron 37 patrones de restricción, los cuales presentaron entre 7 y 15 bandas bien definidas de 34 a 450 kb. La tipibilidad de ERIC-PCR fue del 100% (T=1) y la de ApaI-PFGE del 94% (T=0,94). La reproducibilidad de ambas técnicas fue del 100% (R=1); sin embargo, el poder discriminatorio de ERIC-PCR fue 93% (D=0,93) y el de ApaI-PFGE 98% (D=0,98). Mediante ambas técnicas fue posible agrupar las cepas con relación epidemiológica y diferenciar claramente las cepas no relacionadas. Se demostró el valor de ERIC-PCR y ApaI-PFGE para complementar estudios epidemiológicos, principalmente si las cepas en estudio son analizadas por ambas técnicas.
Palabras clave: Pasteurella multocida, subtipificación, PFGE, ERIC-PCR
ABSTRACT
Evaluation of two techniques of molecular subtyping to study Pasteurella multocida. Typeability, reproducibility, and discriminatory power of ERIC-PCR and ApaI-PFGE to establish the genetic relation of P. multocida strains were determined. Forty-nine strains of different source, biotype, capsular group, somatic serotype, and resistance to antimicrobials were studied. By ERIC-PCR, 31 patterns were defined with 10 to 14 bands in a rank of 0.2 and 1.2 kb. By ApaI-PFGE, 37 restriction patterns were established with 7 to 15 bands of 34 to 450 kb. Typeability was 100% (T=1) for ERIC-PCR, and 94% (T=0.94) for ApaI-PFGE. Reproducibility of both techniques was 100% (R=1). Discriminatory power was 93% (D=0.93) for ERIC-PCR, and 98% (D=0.98) for ApaI-PFGE. By using both techniques, epidemiologically related strains were grouped, and unrelated strains were clearly differentiated. The value of ERIC-PCR and ApaI-PFGE as complements to epidemiologic studies was demonstrated, especially when both techniques were used to analyze the strains.
Key words: Pasteurella multocida, subtypification, PFGE, ERIC-PCR .
INTRODUCCIÓN
Pasteurella multocida es el agente etiológico de diferentes enfermedades que padecen los animales de cría intensiva, entre las que se describen cólera aviar; neumonía y rinitis atrófica porcina (20); septicemia hemorrágica bovina; infecciones en conejos (2) y bronconeumonía enzoótica en bovinos, cabras y ovejas. La pasteurelosis es una enfermedad zoonótica y las infecciones más frecuentes en el hombre son ocasionadas por mordeduras o arañazos de perros y gatos (6, 12).
En Argentina se describieron diferentes presentaciones de pasteurelosis en animales domésticos (7) y en humanos (16), aunque no se realizaron estudios epidemiológicos sobre las poblaciones afectadas, ni se realizó la subtipificación de P. multocida.
Los aislamientos de P. multocida presentan linajes genéticos divergentes (19). Esta divergencia evolutiva refleja la acumulación de mutaciones no letales al azar, como sustitución de un par de bases, deleción de un gen individual o adquisición de ADN de otras especies microbianas. Con el desarrollo de técnicas moleculares fue posible detectar con precisión alteraciones genéticas mínimas. En general, estas diferencias genéticas son compartidas por aislamientos relacionados epidemiológicamente e indican un posible origen común (23).
Los primeros trabajos sobre subtipificación de P. multocida se realizaron mediante la caracterización de uno o varios marcadores fenotípicos (5, 15). Las técnicas de subtipificación fenotípica utilizadas con mayor frecuencia son: a) biotipificación, b) serotipificación somática y capsular, c) sensibilidad a los antimicrobianos y d) técnicas basadas en la separación de proteínas (3). Sin embargo, las técnicas de subtipificación fenotípica presentan algunas limitaciones, como por ejemplo baja reproducibilidad y bajo poder discriminatorio (4).
Las técnicas de subtipificación genotípica utilizadas para el estudio de P. multocida son: a) análisis de ADN plasmídico, b) análisis con endonucleasas de restricción (REA), c) ribotipificación, d) técnicas basadas en la reacción en cadena de la polimerasa (PCR), e) macrorrestricción, f) amplificación de fragmentos polimórficos largos (AFLP) y g) tipificación de secuencias multilocus (MLST).
Entre las técnicas de subtipificación molecular se encuentran aquellas basadas en PCR, como las secuencias repetitivas intergénicas de consenso de las enterobacterias (ERIC-PCR) (1) y las técnicas basadas en macrorrestricción, como la electroforesis en campos pulsados (PFGE) (9), considerada como técnica de oro para la subtipificación de numerosas especies bacterianas (23). Sin embargo, ninguna de las técnicas de subtipificación fue considerada “técnica de oro” para el estudio de P. multocida.
El objetivo del trabajo fue determinar la tipibilidad, la reproducibilidad y el poder discriminatorio de ERIC-PCR y ApaI-PFGE para establecer la relación genética de cepas de P. multocida.
MATERIALES Y MÉTODOS
Cepas
Se estudiaron 49 cepas de P. multocida de diferente origen: a) 17 cepas de referencia, provistas por el Dr. M. J. Wolcott (USGS-National Wildlife Health Center, Madison, Wisconsin, EE.UU.) y el Dr. P. J. Blackall (Queensland Poultry Research and Development Centre, Animal Research Institute, Moorooka, Australia); b) 11 cepas aisladas de gallinas reproductoras con cólera aviar crónico, 9 aisladas en Argentina, una en Uruguay y la otra en Australia (provista por el Dr. P. J. Blackall, Queensland Poultry Research and Development Centre, Animal Research Institute, Moorooka, Australia); c) 10 cepas aisladas de cerdos en la provincia de Buenos Aires; d) 8 cepas aisladas de humanos (Servicio Bacteriología Especial, INEI-ANLIS “Dr. Carlos G. Malbrán”) y e) 3 cepas aisladas de aves antárticas muertas por cólera aviar. Las 49 cepas fueron identificadas por técnicas fenotípicas y genotípicas descritas previamente (15).
La técnica de ERIC-PCR fue realizada según Amonsin et al. (1). La extracción y purificación de ADN genómico se realizó con el kit Wizard™ (Promega, Wisconsin, EE.UU.). Los oligo-nucleótidos utilizados para generar los perfiles de ADN fueron: ERIC1R (5’ ATGTAAGCTCCTGGGGATTCAC) y ERIC2 (5’ AAGTAAGTGAC TGGGGTGAGCG) (Biodynamics, Florida, EE.UU.). Los fragmentos de ADN obtenidos fueron separados por electroforesis en geles de agarosa al 2% (Invitrogen Life Technologies, Brasil) con bromuro de etidio (2 µg/ml) (Promega). La corrida electroforética fue realizada en buffer tris acetato EDTA 1X durante 3,5 h a 80 volts, y el marcador de peso molecular utilizado fue 1 kb DNA Ladder (Promega).
La técnica de PFGE fue realizada según Gunawardana et al. (9), con algunas modificaciones. Los bloques de agarosa que contenían el ADN de cada cepa fueron digeridos con 40 U de la enzima de restricción ApaI (Promega). El marcador de peso molecular utilizado fue Lambda Ladder PFGE Marker (New England BioLabs, Madison, EE.UU.). Los fragmentos de ADN fueron separados en un gel de agarosa al 1,2% (Pulsed Field Certified Agarose, BioRad, Hercules, CA, EE.UU.) en buffer tris borato EDTA 0,5X a 14 °C, en una cámara de electroforesis CHEF-DR III System (BioRad). El tiempo de corrida fue de 22 h, con un voltaje constante de 170 V se utilizó un pulso lineal rampeado de 1-30 seg. La tinción del gel se realizó con 0,5 µg/ml de solución de bromuro de etidio (Promega).
Análisis de los perfiles moleculares
Los perfiles moleculares obtenidos por ERIC-PCR y ApaI-PFGE fueron fotografiados con el equipo Kodak Digital Science 1D™ y analizados con el software BioNumerics versión 3.5 (Applied Maths, Kortrijk, Belgium). Para optimizar el análisis de los perfiles obtenidos por ERIC-PCR se estableció un rango comprendido entre 0,2 y 1,2 kb, ya que las bandas de mayor peso molecular no fueron reproducibles. La relación entre los perfiles fue estimada mediante la proporción de bandas compartidas, aplicando el coeficiente de Dice y generando dendrogramas basados en el método Unweighted Pair Group Method with Arithmatic Mean (UPGMA). Los patrones moleculares obtenidos por ERIC-PCR fueron agrupados en clusters con una similitud superior al 85%, y los patrones moleculares obtenidos por ApaI-PFGE fueron agrupados en clusters con una similitud superior al 80%.
Evaluación de las técnicas de subtipificación
Se determinó la tipibilidad, la reproducibilidad y el poder discriminatorio de cada una de las técnicas utilizadas (11).
Tipibilidad
Se consideraron los resultados obtenidos con las 49 cepas analizadas. Se utilizó la siguiente fórmula: T = Nt/N, donde Nt es el número de cepas tipificables y N el número de aislamientos analizados.
Reproducibilidad
Se consideraron los resultados obtenidos en 3 ensayos independientes con cada una de las 49 cepas analizadas. Se utilizó la siguiente fórmula: R = Nt/N, donde Nt es el número de aislamientos con el mismo patrón o tipo en análisis repetidos, y N el número de aislamientos analizados
Poder discriminatorio
Se consideraron los resultados obtenidos con las 45 cepas que pudieron ser analizadas con ambas técnicas de subtipificación (22). El poder discriminatorio (D) de las técnicas fue determinado mediante el índice de Simpson, según la siguiente fórmula:
![]()
Donde N es el número total de aislamientos estudiados, xj el número de aislamientos en el tipo j y s el número de tipos diferentes
RESULTADOS
Cepas
La subespecie, el origen y el año de aislamiento de las cepas se presentan en la Tabla 1. El biotipo, tipo capsular, serotipo somático y perfil de resistencia antimicrobiana de las 49 cepas estudiadas se presentan en las Figuras 1 y 2. Cinco cepas de referencia fueron biotipo 9 y 10, no descritos en las cepas aisladas en Argentina (15), mientras que las 11 cepas restantes fueron biotipo 3 y 5. Las cepas de Pasteurella multocida subsp. gallicida NADC P-2225 (S14) y ATCC 51689 se destacaron por no fermentar la xilosa (biotipo 9), al igual que las cepas de Pasteurella multocida subsp. multocida NADC P-2192 (S6), NADC P-1997 (S7) y NADC P-1573 (S12) (biotipo 10). Con respecto al perfil de resistencia a los antimicrobianos, las cepas NADC P-2100 (S10), NADC P-903 (S11) y NADC P-2237 (S15) fueron resistentes a tiamulina, la cepa S7 fue resistente a tiamulina y estreptomicina, y la cepa aislada de gallinas reproductoras en Uruguay (G3) fue resistente a enrofloxacina.
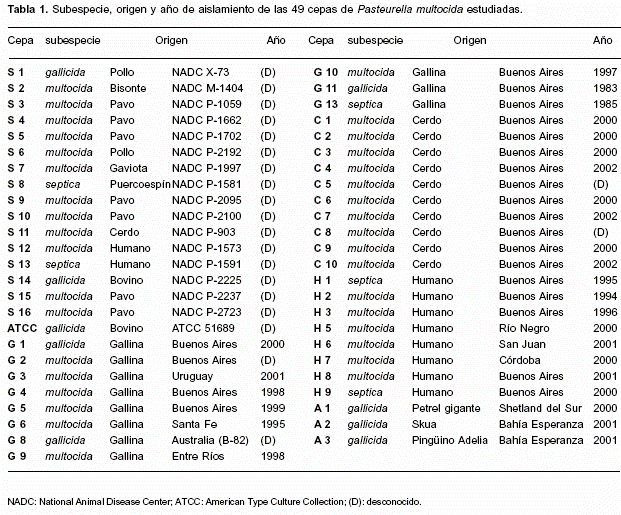
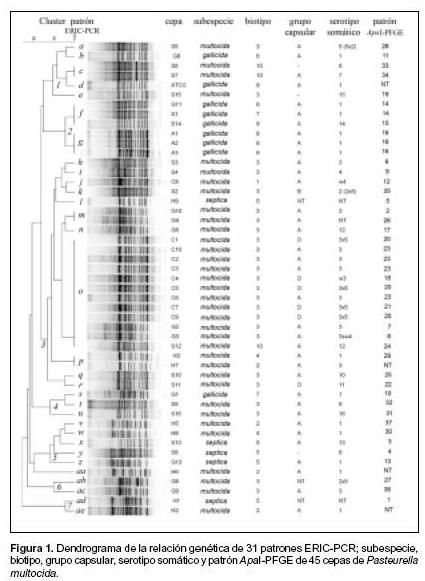
ERIC-PCR
Sobre un total de 49 cepas de P. multocida analizadas, se establecieron 31 patrones ERIC-PCR. Los patrones presentaron de 10 a 14 bandas en un rango comprendido entre 0,2 y 1,2 kb, y fueron agrupados en 7 clusters (1-7). En el cluster 1 agruparon 5 patrones (a-e) con un 89,6% de similitud, correspondientes a cinco cepas aisladas de aves y una de bovino. Una de estas cepas fue aislada en Australia (G8) y las otras 5 fueron de referencia (S5, S6, S7, S15, y ATCC). Dos cepas de P. multocida subsp. multocida (S6 y S7) presentaron el mismo patrón (c). En el cluster 2 agruparon 2 patrones (f y g) con una similitud del 95,6%, correspondientes a 6 cepas de P. multocida subsp. gallicida. Dos cepas de referencia (S1 y S14) y una cepa aislada de gallina (G11) presentaron el mismo patrón (f), y 3 cepas aisladas en la Antártida (A1, A2, y A3) fueron indistinguibles. En el cluster 3 agruparon 23 cepas de P. multocida subsp. multocida y una de P. multocida subsp. septica (H9), distribuidas en 11 patrones (h-r) con una similitud del 86,9%. En este cluster agruparon todas las cepas aisladas de cerdos (C1-C10), 6 cepas de referencia (S2, S3, S4, S10, S11, y S12), 4 cepas aisladas de gallinas (G2, G3, G4, y G10) y 3 cepas aisladas de humanos en Argentina (H3, H7, y H9). Entre las cepas con idéntico patrón, 9 (patrón o) fueron aisladas de cerdos en Buenos Aires entre los años 2000 y 2002. Dos cepas con idéntico patrón (m) fueron aisladas de gallinas en Buenos Aires en 1997 (G10) y 1998 (G4). Otras 2 cepas con idéntico patrón (p) fueron aisladas de humanos en Buenos Aires, en 1996 (H3), y en Córdoba, en 2000 (H7). En el cluster 4 agruparon 3 patrones (s-u) con un 88,3% de similitud, correspondientes a 3 cepas de P. multocida tipo A de origen aviar, de las cuales 2 fueron cepas de referencia (S9, S16) y una fue aislada en Buenos Aires (G1). En el cluster 5 agruparon 5 patrones (v-z) con una similitud del 88,2%, y cada patrón representó a una cepa. En el cluster 6 agruparon 2 cepas de P. multocida subsp. multocida biotipo 3, aisladas de gallinas en Santa Fe, en 1995 (G6), y en Entre Ríos, en 1998 (G9). Los patrones obtenidos con estas 2 cepas presentaron una similitud del 90%. En el cluster 7 agruparon 2 cepas identificadas como P. multocida subsp. multocida (H2) y P. multocida subsp. septica (H1), aisladas de humanos en Buenos Aires en 1994 y 1995, respectivamente. Los patrones obtenidos con estas 2 cepas presentaron una similitud del 95,6 %. El patrón aa, correspondiente a la cepa H6, no agrupó en ningún cluster por presentar una similitud < 85%. En la Figura 1 se presenta el dendrograma generado a partir de los resultados obtenidos por ERIC-PCR.
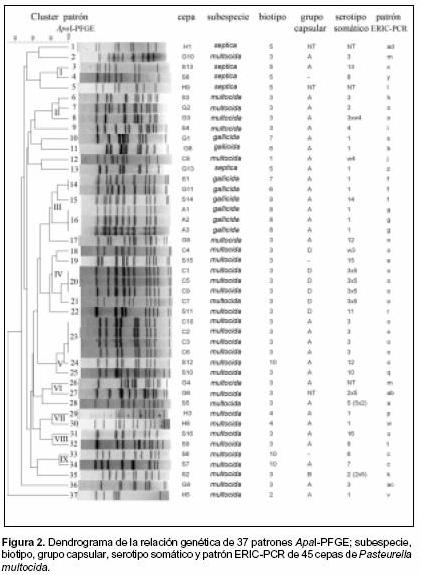
ApaI-PFGE
Sobre un total de 45 cepas de P. multocida analizadas, se establecieron 37 patrones de restricción, los cuales presentaron entre 7 y 15 bandas bien definidas, de 34 a 450 kb. Veintitres patrones fueron agrupados en 9 clusters. La cepa de referencia ATCC 51689 y las cepas H2, H6 y H7 no se incluyeron en el análisis debido a que no fueron tipificables por ApaI-PFGE, porque se degradó su ADN en 3 oportunidades, inclusive al utilizar tiourea en el buffer de corrida. En el cluster I agruparon 2 patrones (3 y 4) con un 87,5% de similitud, correspondientes a 2 cepas de P. multocida subsp. septica biotipo 5 (S8 y S13). En el cluster II agruparon 3 patrones (6, 7 y 8), correspondientes a 3 cepas de P. multocida subsp. multocida A3 de origen aviar (S3, G2 y G3). Entre estos patrones se observó una similitud del 81,9%, y se diferenciaron las cepas G2 y G3, que tuvieron el mismo patrón ERIC-PCR (o). En el cluster III agruparon 6 cepas de P. multocida subsp. gallicida tipo A distribuidas en 3 patrones (14, 15 y 16), con un 81,3% de similitud. La cepa S14 (patrón 15) pudo diferenciarse de las cepas S1 y G11 (patrón 14) con las cuales presentaba el mismo patrón de ERIC-PCR (f). En el cluster III también agruparon las cepas aisladas en la Antártida (A1, A2 y A3), indistinguibles por ambas técnicas. En el cluster IV agruparon 6 cepas de P. multocida subsp. multocida biotipo 3 tipo D, las que fueron distribuidas en 4 patrones (18, 19, 20 y 21) con un 82% de similitud. Cinco cepas agrupadas en este cluster fueron aisladas de cerdos (C1, C4, C5, C7 y C9). En el cluster V agruparon 5 cepas de P. multocida subsp. multocida biotipo 3 grupo capsular A, distribuidas en 3 patrones (23, 24 y 25), con una similitud del 86,9%. En el cluster VI agruparon 2 patrones (26 y 27), con una similitud del 81,4%. Ambos patrones correspondieron a 2 cepas de P. multocida subsp. multocida biotipo 3 aisladas de gallinas en Buenos Aires, en 1998 (G4), y en Santa Fe, en 1995 (G6). Por ERIC-PCR, estas cepas fueron agrupadas en diferentes clusters (3 y 6). En el cluster VII agruparon 2 patrones (29 y 30) con una similitud del 81,8%, correspondientes a 2 cepas (H3 y H8) de P. multocida subsp. multocida aisladas de humanos en Buenos Aires en 1996 y 2001. Por ERIC-PCR estas cepas fueron agrupadas en diferentes clusters (3 y 5). En el cluster VIII agruparon 2 patrones (31 y 32) con una similitud del 82,3%, correspondientes a 2 cepas de referencia de P. multocida subsp. multocida biotipo 3 grupo capsular A, aisladas de pavos (S9 y S16) y agrupadas en el mismo cluster (4) por ERIC-PCR. En el cluster IX agruparon 2 patrones (33 y 34) con una similitud del 90%, correspondientes a 2 cepas de P. multocida subsp. multocida biotipo 10, agrupadas en el mismo cluster (c) por ERIC-PCR. Los patrones 1, 2, 5, 9, 10, 11, 12, 13, 17, 22, 28, 35, 36 y 37 tuvieron un porcentaje de similitud inferior al 80%, por lo que no fueron agrupados en ningún cluster. En la Figura 2 se presenta el dendrograma generado a partir de los resultados obtenidos por ApaI-PFGE.
Evaluación de las técnicas de subtipificación
Al evaluar las técnicas de subtipificación, se demostró que la tipibilidad de ERIC-PCR fue del 100% (T=1) y la tipibilidad de ApaI-PFGE fue del 94% (T=0,94). La reproducibilidad de ambas técnicas fue del 100% (R=1). Sin embargo, el poder discriminatorio de ERIC-PCR fue 93% (D=0,93) y el de ApaI-PFGE 98% (D=0,98).
DISCUSIÓN
Existe una gran controversia acerca de cuál es la técnica de subtipificación molecular más adecuada para el estudio de P. multocida. Snipes et al. (21) y Fussing et al. (8) obtuvieron buenos resultados al utilizar HhaI-REA y HindI-ribotipificación, respectivamente. Sin embargo, Blackall y Miflin (4) demostraron que el poder discriminatorio de HhaI-REA (D=0,86) y HpaII-ribotipificación (D=0,89) fue más bajo que el valor mínimo recomendado por Hunter y Gaston (11). Mediante la subtipificación de cepas por RAPD-PCR y AFLP, Huber et al. (10) demostraron que un lote de vacunas vivas originó varios brotes de cólera aviar en aves domésticas. Si bien los autores no evaluaron el poder discriminatorio de las técnicas, demostraron la importancia de la subtipificación molecular de P. multocida en investigaciones epidemiológicas. En algunos trabajos se evaluó el poder discriminatorio de técnicas basadas en PCR, por ejemplo RAPD-PCR (D=0,86), ERIC-PCR (D=0,89), AFLP (D=0,93), y REP-PCR (D=0,95) (1, 9, 17). Townsend et al. (24) demostraron la utilidad de PFGE en estudios de epidemiología molecular, al diferenciar cepas indistinguibles por serotipificación, perfil proteico y ribotipificación. Asimismo, algunos autores obtuvieron mejores resultados al subtipificar cepas de P. multocida por ApaI-PFGE que al hacerlo por ribotipificación, REA, RAPD y REP-PCR (9, 13, 18). Inclusive, se demostró que ApaI-PFGE es una técnica que presenta buena reproducibilidad en diferentes laboratorios (18).
En el presente trabajo se subtipificaron 49 cepas de P. multocida de diferente origen, y se obtuvieron 7 clusters por ERIC-PCR y 9 clusters por ApaI-PFGE. Los clusters obtenidos por ambas técnicas agruparon patrones según origen, subespecie, grupo capsular y serotipo somático. Las cepas provenientes de la Antártida fueron aisladas en el contexto de un estudio epidemiológico de cólera aviar (14). Las cepas aisladas de cerdo presentaron mayor homología por ambas técnicas que las cepas de otros orígenes, posiblemente porque estuvieron relacionadas en tiempo y lugar. Por ejemplo, por ERIC-PCR las 10 cepas aisladas de cerdos fueron agrupadas en el mismo cluster (3) y 9 cepas presentaron el mismo patrón (o); por ApaI-PFGE estas cepas fueron agrupadas en 2 clusters, excepto la cepa C8 (< 80% de similitud), diferenciándose en 5 patrones (12, 18, 20, 21 y 23). Mediante ambas técnicas fue posible agrupar cepas con relación epidemiológica y diferenciar las cepas sin relación epidemiológica. Algunas cepas no relacionadas epidemiológicamente (H3, H8, G4 y G6) agruparon en diferentes clusters al ser analizadas por ambas técnicas de subtipificación, por ello es muy importante utilizar estas técnicas en el contexto de estudios epidemiológicos.
ApaI-PFGE (D=0,98) presentó mayor poder discriminatorio que ERIC-PCR (D=0,93). Según Hunter y Gaston (11), el poder discriminatorio que debe presentar una técnica de subtipificación molecular debe ser al menos del 90% (D=0,90). Para cumplir con el 5% de probabilidad aceptable (error tipo I), el valor ideal de D debe ser > 0,95, aunque las técnicas de subtipificación que presentan un poder discriminatorio < 0,95 pueden ser utilizadas en combinación con otras técnicas para llegar al valor de D=0,95 (22). ApaI-PFGE presentó un valor ideal de D y por lo tanto podría utilizarse como única técnica, sin embargo 4 cepas no pudieron ser subtipificadas por esta técnica. Por el contrario, al considerar el poder discrimi-natorio obtenido con ERIC-PCR, podemos inferir que debería utilizarse en combinación con otra técnica de subtipificación; sin embargo, por ERIC-PCR fue posible diferenciar y agrupar en 4 clusters diferentes las cepas no tipificables por ApaI-PFGE.
ERIC-PCR y ApaI-PFGE presentan ventajas y desventajas. Así por ejemplo, ApaI-PFGE tiene un gran poder discriminatorio y buena reproducibilidad, pero es una técnica costosa, por lo tanto su utilización se restringe a los laboratorios de referencia. ERIC-PCR presenta menor poder discriminatorio, pero es menos costosa y el equipamiento necesario para su realización puede ser utilizado con otros fines, como por ejemplo para diagnóstico microbiológico.
En el presente trabajo ERIC-PCR presentó mayor tipibilidad (T=1) que ApaI-PFGE (T=0,94), ApaI-PFGE presentó mayor poder discriminatorio (D=0,98) que ERIC-PCR (D=0,93), y ambas técnicas tuvieron un 100% (R=1) de reproducibilidad. Se demostró el valor de las dos técnicas de subtipificación para complementar estudios epidemiológicos, principalmente si se usan combinadas. Tanto ERIC-PCR como ApaI-PFGE pueden utilizarse para reconocer brotes de pasteurelosis, establecer el origen de los mismos, identificar las vías de transmisión, controlar la diseminación de cepas con resistencia a antibióticos y vigilar los programas de vacunación. Sin embargo, sería apropiado evaluar nuevas técnicas para la subtipificación molecular de cepas de P. multocida aisladas en la Argentina, entre las que se podría incluir la tipificación de secuencias multilocus (MLST) o multi-locus variable-number tandem repeat analysis (MLVA).
Agradecimientos: los autores agradecen al Dr. M. J. Wolcott (USGS-National Wildlife Health Center, Madison, Wisconsin, EE.UU.) por colaborar en la serotipificación de las cepas y proveer las cepas de referencia; al Dr. P. J. Blackall (Queensland Poultry Research and Development Centre, Animal Research Institute, Moorooka, Australia) por proveer la cepa ATCC 51689 y una cepa aislada de gallina (B-82); y al Servicio Bacteriología Especial, Departamento Bacteriología, INEI - ANLIS “Dr. Carlos G. Malbrán” por proveer las cepas de origen humano.
BIBLIOGRAFIA
1. Amonsin A, Wellehan JFX, Li LL, Laber J, Kapur V. DNA fingerprinting of Pasteurella multocida recovered from avian sources. J Clin Microbiol 2002; 40: 3025-31. [ Links ]
2. Biberstein EL. Pasteurella. En: Biberstein EL, Zee YC, editors. Review of Veterinary Microbiology. Oxford, Blackwell Scientific Publication, 1990, p. 175-80. [ Links ]
3. Blackall PJ, Fegan N, Chew GTI, Hampson DJ. A study of the use of multilocus enzyme electrophoresis as a typing tool in fowl cholera outbreaks. Avian Pathol 1999; 28: 195-8. [ Links ]
4. Blackall PJ, Miflin JK. Identification and typing of Pasteurella multocida: a review. Avian Pathol 2000; 29: 271-87. [ Links ]
5. Brogden KA, Packer RA. Comparison of Pasteurella multocida serotyping systems. Am J Vet Res 1979; 40: 1332-5. [ Links ]
6. Brue C, Chosidow O. Pasteurella multocida wound infection and cellulitis. Int J Dermathol 1994; 33: 471-3. [ Links ]
7. De Diego AI. Guía para el estudio de las enfermedades infecciosas de los animales (aves y mamíferos). Buenos Aires, Argentina. Talleres Gráficos Farro, 1974, p. 752. [ Links ]
8. Fussing V, Nielsen JP, Bisgaard M, Meyling A. Development of a typing system for epidemiological studies of porcine toxin producing Pasteurella multocida ssp. multocida in Denmark. Vet Microbiol 1999; 65: 61-74. [ Links ]
9. Gunawardana GA, Townsend KM, Frost AJ. Molecular characterization of avian Pasteurella multocida isolates from Australia and Vietnam by REP-PCR and PFGE. Vet Microbiol 2000; 72: 97-109. [ Links ]
10. Huber BS, Allred DV, Carmen JC, Frame DD, Whiting DG, Cryan JR, et al. Random amplified polymorphic DNA and amplified fragment length polymorphism analyses of Pasteurella multocida isolates from fatal fowl cholera infections. J Clin Microbiol 2002; 40: 2163-8. [ Links ]
11. Hunter PR, Gaston MA. Numerical index of the discriminatory ability of typing systems: an application of Simpson’s Index of Diversity. J Clin Microbiol 1988; 26: 2465-6. [ Links ]
12. Kehrenberg C, Salmon SA, Watts JL, Schwarz S. Tetracycline resistance genes in isolates of Pasteurella multocida, Mannheimia haemolytica, Mannheimia glucosida and Mannheimia varigena from bovine and swine respiratory disease: intergenic spread of the tet(H) plasmid pMHT1. J Antimicrob Chemother 2001; 48: 631-40. [ Links ]
13. Lainson FA, Aitchison KD, Donachie W, Thomson JR. Typing of Pasteurella multocida isolated from pigs with and without porcine dermatitis and nephropathy syndrome. J Clin Microbiol 2002; 40: 588-93. [ Links ]
14. Leotta GA, Chinen I, Vigo G, Pecoraro M, Rivas M. Outbreaks of avian cholera in Hope Bay, Antarctica. J Wildl Dis 2006; 42: 259-70. [ Links ]
15. Leotta GA, Vigo GB, Chinen I, Prieto M, Callejo R, Rivas M. Identificación, biotipificación y caracterización de cepas de Pasteurella multocida aisladas en Argentina. Rev Argent Microbiol 2006; 38:125-9. [ Links ]
16. Martinelli IV, Diez RS, Gutiérrez MA, Grinberg A, Bonazzi S, Predari SC. Neumonía por Pasteurella multocida subespecie multocida. XVII Congreso Latinoamericano de Microbiología. X Congreso Argentino de Microbiología, 2004, Resumen C-200, ed. electrónica, Ciudad Autónoma de Buenos Aires, Argentina. [ Links ]
17. Paixao R, Moreno AM, Doto DS, Baccaro MR, Ferreira AJP, Gomes CR, et al. Genotypic characterization of P. multocida strains using RAPD and AFLP. 18th IPVS Congress 2004, vol 1, p. 2, Hamburg, Germany. [ Links ]
18. Pedersen K, Dietz HH, Jorgensen JC, Christensen TK, Bregnballe T, Andersen TH. Pasteurella multocida from outbreaks of avian cholera in wild and captive birds in Denmark. J Wildl Dis 2003; 39: 808-16. [ Links ]
19. Petersen KD, Christensen H, Bisgaard M, Olsen JE. Genetic diversity of Pasteurella multocida fowl cholera isolates as demonstrated by ribotyping and 16S rRNA and partial atpD sequence comparisons. Microbiol 2001; 147: 2739-48. [ Links ]
20. Radostits OM, Gay CC, Blood DC, Hinchcliff KW. A Textbook of the Diseases of Cattle, Sheep, Pigs, Goats, and Horses. Philadelphia, Saunders, 2000, p. 829-67. [ Links ]
21. Snipes KP, Hirsh DC, Kasten RW, Carpenter TE, Hird DW, McCapes RH. Homogeneity of characteristics of Pasteurella multocida isolated from turkeys and wildlife in California, 1985-88. Avian Dis 1990; 34: 315-20. [ Links ]
22. Struelens MJ and the Members of the European Study Group on Epidemiological Markers (ESGEM), of the European Society for Clinical Microbiology and Infectious Diseases (ESCMID). Consensus guidelines for appropiate use and evaluation of microbial epidemiologic typing systems. Clin Microbiol Infect 1996; 1: 2-11. [ Links ]
23. Tenover FC, Arbeit RD, Goering RV, Mickelsen PA, Murray BE, Persing DH, et al. Interpreting chromosomal DNA restriction patterns produced by pulsed-field gel elec-trophoresis: criteria for bacterial strain typing. J Clin Microbiol 1995; 33: 2233-9. [ Links ]
24. Townsend KM, Dawkins HJ, Papadimitriou JM. Analysis of haemorrhagic septicaemia-causing isolates of Pasteurella multocida by ribotyping and field alteration gel electrophoresis (FAGE). Vet Microbiol 1997; 57: 383-95. [ Links ]
Recibido: 4/09/06
Aceptado: 10/11/06














