Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Revista argentina de microbiología
versión impresa ISSN 0325-7541versión On-line ISSN 1851-7617
Rev. argent. microbiol. v.40 n.1 Ciudad Autónoma de Buenos Aires ene./mar. 2008
Análisis in silico de la capacidad de dos técnicas de PCR para la detección del gen stx.
L. Galli1, 2*, G. A. Leotta1, 2, M. J. Gugliada1, M. Rivas1
1Servicio Fisiopatogenia, Departamento Bacteriología, Instituto Nacional de Enfermedades Infecciosas - ANLIS "Dr. Carlos G. Malbrán". Av. Vélez Sarsfield 563, 1281 Ciudad Autónoma de Buenos Aires, Argentina.
2Consejo Nacional de Investigaciones Científicas y Técnicas.
* Correspondencia: E-mail: lgalli@anlis.gov.ar
RESUMEN
Escherichia coli productor de toxina Shiga es un patógeno emergente cuyo principal factor de virulencia son las toxinas Shiga (Stx), codificadas por los genes stx. Estas toxinas se clasifican en 6 tipos (1, 2, 2c, 2d, 2e y 2f) que agrupan a 22 variantes. En Argentina se validaron dos técnicas de PCR para la detección de los genes stx, PCR-MK y PCR múltiple. Los objetivos del trabajo fueron analizar mediante el uso de herramientas bioinformáticas la capacidad de dichas técnicas para detectar las variantes del gen stx y demostrar experimentalmente la amplificación de 8 variantes stx. Se recopilaron 25 secuencias nucleotídicas de la base de datos GenBank correspondientes a 21 variantes de stx. Se utilizó el programa BLAST 2 sequences para analizar la complementariedad de las bases nucleotídicas entre las secuencias de las variantes y las secuencias de los cebadores utilizados en las PCR estudiadas. La técnica de PCR-MK permite detectar los tipos stx1, stx2, stx2c, stx2d y stx2f, aunque no permite detectar el tipo stx2e y tres variantes del tipo stx2c. La PCR múltiple permite detectar los tipos stx1, stx2, stx2c, stx2d, pero no los tipos stx2e y stx2f. Se demostró experimentalmente que ambas técnicas de PCR son apropiadas para la detección de las variantes que están asociadas a enfermedad grave en el hombre.
Palabras clave: Escherichia coli; Toxina Shiga; Stx; PCR; Bioinformática
ABSTRACT
In silico analysis of the capability of two polimerase chain reaction techniques for stx gene detection. Shiga toxin-producing Escherichia coli is an emergent pathogen, being the Shiga toxin (Stx) the main virulence factor. These toxins are classified into 6 types (1, 2, 2c, 2d, 2e and 2f) and 22 variants. In Argentina, two PCR for stx gene detection, PCR-MK and multiplex-PCR, were validated. The aim of this work was to analyze, by using bioinformatic tools, the stx variants that could be amplified by these PCRs, and to experimentally show the amplification of 8 stx variants. Twentyfive nucleotide sequences were collected from GenBank corresponding to 21 stx variants. The BLAST 2 sequences program was used to analyze the complementarities between the nucleotide sequence of the variants and the primers corresponding to the PCR studied. PCR-MK could detect types stx1, stx2, stx2c, stx2d and stx2f, but not type stx2e and three type stx2c variants. On the other hand, the multiplex-PCR could detect types stx1, stx2, stx2c, stx2d, but not stx2e and stx2f types. It was experimentally determined that both PCRs can detect those variants that cause severe disease in humans.
Key words: Escherichia coli; Shiga toxin; Stx; PCR; Bioinformatics
Escherichia coli productor de toxina Shiga (STEC) es un patógeno emergente transmitido por alimentos, que se asocia a casos esporádicos y a brotes de diarrea, colitis hemorrágica y síndrome urémico hemolítico (SUH). En Argentina, el SUH post-entérico asociado a STEC es endémico. Este grupo bacteriano se ha aislado del intestino de numerosas especies animales, aunque se considera que el principal reservorio son los rumiantes en general, y el ganado bovino en particular (5). La fuente de infección más importante para el hombre son los alimentos cárnicos, especialmente la carne molida y los subproductos crudos o insuficientemente cocidos (5). Otras fuentes de infección son la contaminación cruzada, el contacto directo del hombre con los animales y la transmisión persona a persona (12).
Escherichia coli O157:H7 es el prototipo de más de 200 serotipos que comparten el mismo potencial patogénico, cuyo principal factor de virulencia son las toxinas Shiga (Stx) (10). Estas toxinas poseen estructura de subunidades AB5 y están codificadas por bacteriófagos insertos en el cromosoma bacteriano. Según Scheutz y Strockbine (13), las toxinas Shiga se clasifican en 6 tipos, 1, 2, 2c, 2d, 2e y 2f, los que agrupan a 22 variantes stx. Esta clasificación se basa en la variabilidad antigénica, diferencias respecto de su toxicidad en tejidos celulares y animales, capacidad para ser activadas por elastasa de ratón y diferencias en las secuencias aminoacídicas o nucleotídicas (3). Las cepas STEC de origen humano, animal o de alimentos pueden producir diferentes tipos de Stx.
Se han desarrollado varias metodologías para la detección de Stx y numerosas técnicas basadas en la reacción en cadena de la polimerasa (PCR) para detectar los genes stx (1, 4, 11). En Argentina se validaron dos técnicas de PCR para la detección de los genes stx, PCR-MK (7) y PCR múltiple (6). La primera de ellas se utiliza para detectar STEC en alimentos cárnicos; se la usa como tamizaje a partir de caldo de enriquecimiento, ya que posee un control interno de amplificación competitivo. La PCR múltiple es utilizada como tamizaje para la detección de las variantes stx1, stx2, y rfbO157 a partir de medios sólidos y como técnica confirmatoria de las cepas aisladas. Si bien estas técnicas se utilizan de rutina para el análisis de muestras clínicas, de alimentos, de animales y ambientales (2, 12, 14), no se conoce con exactitud cuáles son las variantes stx que amplifican. Los objetivos del trabajo fueron: a) realizar un análisis in silico de la capacidad de las técnicas de PCR-MK y PCR múltiple para detectar las distintas variantes de los genes stx, mediante el uso de computadoras y herramientas bioinformáticas, y b) demostrar experimentalmente la amplificación de 8 variantes stx.
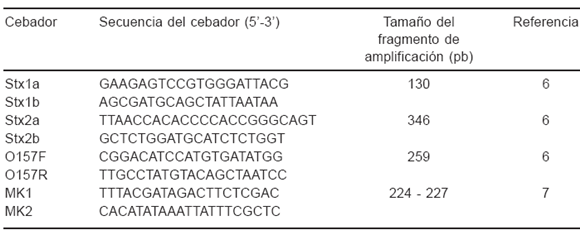
En la técnica de PCR-MK se utilizan los cebadores MK1 y MK2, que amplifican una región conservada de 227 pb de stx1 y de 224 pb de stx2 (7). En la PCR múltiple se utilizan tres pares de cebadores para amplificar un fragmento de 130 pb del gen correspondiente a la subunidad B de stx1 (6), un fragmento de 346 pb del gen correspondiente a la subunidad A de stx2 (6), y un fragmento de 259 pb del gen rfbO157 (6). En la Tabla 1 se presentan las secuencias de cada uno de los cebadores utilizados en ambas PCR.
Tabla 1. Secuencias de los cebadores utilizados en las técnicas de PCR-MK y PCR múltiple.
Para realizar el análisis in silico, se recopilaron 25 secuencias nucleotídicas correspondientes a 21 variantes stx (13). Las secuencias se obtuvieron de la base de datos GenBank (www.ncbi.nlm.nih.gov/Genbank), a excepción de las variantes stx2-O22 y stx2-O157-TK-51 que se obtuvieron de la publicación de Lin y col. (8). La secuencia nucleotídica correspondiente a la variante stx1-O103 no fue hallada en GenBank ni en el trabajo original donde se ópor primera vez esta variante (9).
Se utilizó el programa BLAST 2 sequences 2.2.15 (www.ncbi.nlm.nih.gov/BLAST) para determinar la complementariedad de las bases nucleotídicas entre las secuencias de las variantes stx y las secuencias de los cebadores utilizados en las técnicas de PCR-MK y múltiple.
Para demostrar experimentalmente la amplificación de las variantes stx, se utilizaron las siguientes cepas de E. coli: FP1009/02 (O157:H7, stx1), FP590/04 (ONT:NM, stx1-OX3), FP207/02 (O111:NM, stx1, stx2), E32511 (O157:NM, stx2c), HH8 (O145:H4, stx2-O48), FP656/04 (O146:H28, stx2-O118), FP149/02 (O174:H21, stx2d2), y II9 (O113:H4, stx2d3). Entre éstas se encuentran cepas de referencia y cepas aisladas de casos clínicos o de animales en Argentina. Estas cepas fueron analizadas mediante las técnicas de PCR-MK y PCR múltiple según los protocolos validados previamente (6, 7).
Al comparar cada una de estas secuencias con las correspondientes a los cebadores MK1 y MK2, se estableció que la técnica de PCR-MK puede amplificar 17 de 21 variantes y 20 de 25 secuencias. Las variantes stx2-OX3/a-031, stx2-OX3/b-031, stx2-O111-PH y stx2f no presentan secuencia homóloga para los cebadores MK y, por lo tanto, no pueden ser amplificadas por esta técnica. En el caso de la PCR múltiple, se pueden amplificar 19 de 21 variantes y 21 de 25 secuencias. Las variantes stx2e y stx2f no pueden ser amplificadas por esta PCR, ya que no presentan secuencias homólogas para los cebadores Stx2. En la Tabla 2 se presenta la designación de los tipos stx, las variantes stx, el microorganismo de origen, el número de base inicial y final del gen que se amplificará por PCR -MK y PCR múltiple, la designación previa o sinónimos para los genes de las toxinas y el número de acceso al GenBank. Las 8 cepas STEC portadoras de las variantes stx1, stx1-OX3, stx2, stx2c, stx2-O48, stx2-O118, stx2d2 y stx2d3 fueron positivas por ambas técnicas de PCR. En la figura 1 se presentan los resultados obtenidos al analizar estas 8 variantes stx por las técnicas de PCR-MK y PCR múltiple.
Tabla 2. Resultado del análisis in silico.
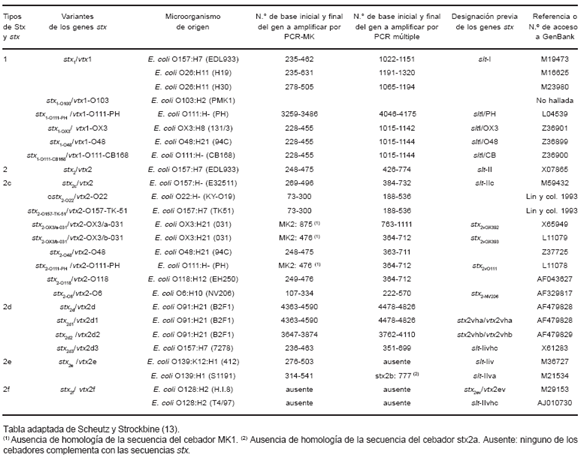

Figura 1. Resultados obtenidos al analizar 8 cepas de Escherichia coli productor de toxina Shiga mediante las técnicas de PCR-MK y PCR múltiple para la detección de los genes stx. A) Resultados de la PCR-MK. Calle 1: FP1009/02 (stx1), calle 2: FP590/04 (stx1-OX3), calle 3: FP207/02 (stx1, stx2), calle 4: E32511 (stx2c), calle 5: HH8 (stx2-O48), calle 6: FP656/04 (stx2-O118), calle 7: FP149/02 (stx2d2), calle 8: II9 (stx2d3), calle 9: EDL933 (control positivo: stx1, stx2), calle 10: ATCC 25922 (control negativo), calle 11: IAC (control interno de amplificación), calle 12: control de reactivos, calle 13: marcador de peso molecular Cienmarker (Biodynamics). B) Resultados de la PCR múltiple. Calle 1: FP1009/02 (stx1), calle 2: FP590/ 04 (stx1-OX3), calle 3: FP207/02 (stx1, stx2), calle 4: E32511 (stx2c), calle 5: HH8 (stx2-O48), calle 6: FP656/ 04 (stx2-O118), calle 7: FP149/02 (stx2d2), calle 8: II9 (stx2d3), calle 9: EDL933 (control positivo: stx1, stx2), calle 10: ATCC 25922 (control negativo), calle 11: control de reactivos, calle 12: marcador de: peso molecular Cienmarker (Biodynamics).
En la última década, se describieron diferentes metodologías para la detección de STEC a partir de muestras clínicas, de animales, de alimentos y ambientales (2, 12, 14). Sin embargo, sólo algunas de ellas utilizan técnicas de PCR como tamizaje (4). A excepción del serotipo O157:H7, la gran diversidad de serotipos de STEC asociados a enfermedades que afectan al hombre impide la utilización de medios altamente selectivos y diferenciales, como así también de condiciones de incubación restrictivas.
Debido a que es necesario utilizar técnicas basadas en la detección del principal factor de virulencia de STEC (1), es importante que estas técnicas detecten la mayor cantidad de variantes Stx. Las técnicas de PCR-MK y PCR múltiple fueron estandarizadas y validadas para mejorar la detección de STEC en Argentina. Mediante un análisis in silico, en este trabajo se demostró que sobre 22 variantes stx (13) las técnicas de PCR-MK y PCR múltiple pueden amplificar 17 y 19 variantes, respectivamente. Además, se confirmó que ambas técnicas son apropiadas para la detección de las variantes stx asociadas con mayor frecuencia a enfermedades severas que afectan al hombre. Sin embargo, mediante el uso de la técnica de PCR-MK no es posible amplificar 3 variantes del tipo stx2c (stx2-OX3/a-031, stx2-OX3/b-031, y stx2-O111-PH) ni el tipo stx2f, y la técnica de PCR múltiple no permite amplificar los tipos stx2e y stx2f.
Las variantes stx2-OX3/a-031, stx2-OX3/b-031, y stx2-O111-PH fueron aisladas de ovejas y en el hombre estuvieron asociada s a casos de diarreas no complicadas y a pacientes asintomáticos, pero no a enfermedad severa (13). El tipo stx2e está asociado a la enfermedad de los edemas del cerdo, y el tipo stx2f fue aislado de heces de paloma. Si bien stx2e fue aislado de casos de diarrea en humanos, estos dos tipos de Stx no fueron descritos en casos de enfermedad severa en la Argentina (3).
Mediante el an álisis in silico y el estudio de cepas STEC portadoras de 8 variantes stx diferentes, se demostró que las técnicas de PCR-MK y PCR múltiple permiten detectar las variantes stx de impacto en salud pública. Por lo tanto, para la búsqueda de STEC en alimentos se recomienda utilizar la PCR-MK como tamizaje a partir del caldo de enriquecimiento, y la PCR múltiple como técnica de confirmación. En el análisis de muestras clínicas se recomienda utilizar la técnica de PCR múltiple, ya que el tamizaje se realiza a partir de un medio sólido y, además, diferencia los genes stx1, stx2 y rfbO157.
1. Bastian SN, Carle I, Grimont F. Comparison of 14 PCR systems for the detection and subtyping of stx genes in Shiga toxin-producing Escherichia coli. Res Micobiol 1998; 149: 457-72. [ Links ]
2. Bentancor A, Rumi MV, Gentilini MV, Sardoy C, Irino K, Agostini A, et al. Shiga toxin-producing and attaching and effacing Escherichia coli in cats and dogs in a high hemolytic uremic syndrome incidence region in Argentina. FEMS Microbiol Lett 2007; 267: 251-6. [ Links ]
3. Bettelheim KA, Beutin L. Rapid laboratory identification and characterization of verocytotoxigenic (Shiga toxin-producing) Escherichia coli (VETC/STEC). J Appl Microbiol 2003; 95: 205-17. [ Links ]
4. Capps KL, McLaughlin EM, Murray AWA, Aldus CF, Wyatt GM, Peck MW, et al. Validation of three rapid screening methods for detection of verotoxin-producing Escherichia coli in foods: interlaboratory study. J AOAC Int 2004; 87: 68-77. [ Links ]
5. Caprioli A, Morabito S, Brugere H, Oswald E. Enterohaemorrhagic Escherichia coli: emerging issues on virulence and modes of transmission. Vet Res 2005; 36: 289-311. [ Links ]
6. Leotta GA, Chinen I, Epszteyn S, Miliwebsky E, Melamed IC, Motter M, et al. Validación de una técnica de PCR múltiple para la detección de Escherichia coli productor de toxina Shiga. Rev Argent Microbiol 2005; 37: 1-10. [ Links ]
7. Leotta GA. Validación de una técnica de reacción en cadena de la polimerasa (PCR) para la detección de Escherichia coli productor de toxina Shiga en alimentos cárnicos (tesis). Buenos Aires. ANLIS "Dr. Carlos G. Malbrán" y Universidad Nacional de San Martín; 2006. [ Links ]
8. Lin ZH, Kurazono S, Yamasaki S, Takeda Y. Detection of various variant verotoxin genes in Escherichia coli by polymerase chain reaction. Microbiol Immunol 1993; 37: 543-8. [ Links ]
9. Mariani-Kurkdjian P, Denamur E, Milon A, Picard B, Cave H, Lambert-Zechovsky N, et al. Identification of a clone of Escherichia coli O103:H2 as a potential agent of hemolytic uremic syndrome in France. J Clin Microbiol 1993; 31: 296-301. [ Links ]
10. Microbionet. Bettelheim KA: Serotypes of verotoxin-producing Escherichia coli reported in the literature apart from those belonging to serogroup O157. Disponible en: http:// www.microbionet.com.au/frames/feature/vetc/intro. [ Links ]
11. Paton A, Paton J. Detection and characterization of Shiga toxigenic Escherichia coli by using Multiplex PCR assays for stx1, stx2, eaeA, enterohemorrhagic E. coli hlyA, rfbO111, and rfbO157. J Clin Microbiol 1998; 36: 598-602. [ Links ]
12. Rivas M, Miliwebsky E, Chinen I, Deza N, Leotta GA. Epidemiología del síndrome urémico hemolítico en Argentina. Diagnóstico del agente etiológico, reservorios y vías de transmisión. Medicina (Buenos Aires) 2006; 66: 27-32. [ Links ]
13. Scheutz F, Strockbine NA. Escherichia. En: Garrity GM, Brenner DJ, Krieg NR, Staley JT, editors. Bergey's Manual of Systematic Bacteriology NY, Springer, 2005, p. 607-24. [ Links ]
14. Tanaro JD, Leotta GA, Lound LH, Galli L, Ledri SE, Carbonari CC, et al. Detección de Escherichia coli productor de toxina Shiga en muestras de materia fecal bovina y agua ambiental. III Congreso Argentino de Microbiología de Alimentos, 2006. Resumen C6, p. 107, Ciudad Autónoma de Buenos Aires, Argentina. [ Links ]
Recibido: 31/07/07
Aceptado: 20/11/07














