Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Revista argentina de microbiología
versión impresa ISSN 0325-7541versión On-line ISSN 1851-7617
Rev. argent. microbiol. v.40 n.4 Ciudad Autónoma de Buenos Aires oct./dic. 2008
Aislamiento de Candida dubliniensis en distintos materiales clínicos. Análisis de métodos fenotípicos de diferenciación con Candida albicans
G. Pineda1, 2, K. Scollo3, G. Santiso1, E. Lehmann1, A. Arechavala1*
1Unidad Micología. Hospital de Infecciosas "F. J. Muñiz". Uspallata 2272 (1282) Ciudad Autónoma de Buenos Aires;
2Sección Microbiología Hospital Austral, Pilar, Provincia de Buenos Aires;
3Departamento de Diagnóstico, Instituto Nacional de Parasitología "Dr. Mario Fatala Chaben", Av. Paseo Colón 568, Ciudad Autónoma de Buenos Aires. Argentina
*Correspondencia. E-mail: hmmicologia@intramed.net
RESUMEN
Con el objeto de estimar la frecuencia de aislamientos de Candida dubliniensis en materiales clínicos en el Hospital de Infecciosas F. J. Muñiz, se identificaron 388 levaduras entre setiembre de 2005 y agosto de 2007. Doscientos doce aislamientos presentaban color verde en CHROMagar® y producían tubos germinativos y clamidoconidias en agarleche. Para diferenciar cuales de ellos correspondían a Candida albicans o a C. dubliniensis, se utilizaron distintos métodos fenotípicos y se evaluó la utilidad de cada técnica a fin de proponer un algoritmo de identificación simple, económico y confiable. Se estudió el color en 2 medios con sustratos cromogénicos, la producción de clamidoconidias en medios de Staib, agar tomate-zanahoria y agar-tabaco; en este último medio también se evaluaron las características macromorfológicas de las colonias; se evaluó la presencia de actividad lipolítica (medio-opacidad), capacidad de desarrollo a 45 °C y asimilación de D-xilosa. El 6,1% (13/212 aislamientos) correspondió a C. dubliniensis (3,3% del total de levaduras). No se pudo diferenciar entre ambas especies por el color en los medios cromogénicos usados. Las pruebas que resultaron más sensibles y específicas fueron crecimiento a 45 °C, asimilación de D-xilosa, color y desarrollo en agar-tabaco. C. albicans produjo clamidoconidias en los 3 medios diferenciales, entre 11,6% y 15,1% de los casos. La presencia de lipasas se evidenció en el 95,6% de C. albicans pero 2 de las 13 cepas de C. dubliniensis también presentaron halo de opacidad. Consideramos que se deben usar, al menos, 3 métodos diferentes para discriminar entre estas levaduras ya que ninguna prueba es absolutamente sensible o específica.
Palabras clave: C. dubliniensis; C. albicans; Identificación fenotípica.
ABSTRACT
Isolation of Candida dubliniensis in different clinical samples. Analysis of phenotypical methods to differenciate from Candida albicans. In order to estimate the frequence of Candida dubliniensis in clinical samples in F. J. Muñiz Infectious Diseases Hospital, a total of 388 yeasts from September 2005 to August 2007. There were 212 isolates which presented a green color on CHROMagar® Candida medium and produced germ tubes and chlamidoconidiae in milk-agar; so as to distinguish whether they corresponded to Candida albicans or C. dubliniensis, different phenotypical methods were utilized. It was also evaluated the usefulness of each one in order to suggest a simple, economic and reliable identification algorithm. Each isolate was subcultured in two chromogenic media and then, the following determinations were done: chlamidospores production in Staib-agar, tomato-carrot-agar and tobacco-agar, colonies macromorphology was also studied in the last medium; opacity-test in Tween 80-CaCl2 agar (lipase activity), growing capacity at 45 °C, and D-xylose assimilation. Thirteen strains (6.1%) corresponded to C. dubliniensis. The difference in color between both species on chromogenic media was not so stressed as it is pointed out in some works. The more specific and sensitive tests were the ability to grow at 45 °C, D-xylose assimilation, color and macroscopic appearance in tobacco-agar. Between 11.6% and 15.1% of C. albicans strains produced chlamidoconidiae in the 3 differential media tested. The opacity halo (lipase) was evident in 95.6% of C. albicans isolates but 2 out of 13 C. dubliniensis also presented precipitation halo. We consider that at least 3 different phenotypical methods should be used to distinguish properly these two species since none of the tests is absolutely sensitive or specific.
Key words: C. dubliniensis; C. albicans; Phenotypical identification.
INTRODUCCIÓN
En 1995, Sullivan et al. describieron una nueva especie de Candida, asociada con lesiones orales de pacientes infectados con el virus de la inmunodeficiencia humana, a la que denominaron Candida dubliniensis y que se encuentra relacionada filogenéticamente con Candida albicans (8, 48, 49).
Inicialmente se aisló de candidiasis orofaríngeas de pacientes con SIDA, pero luego se han publicado numerosos trabajos donde señalan su presencia en todo tipo de muestras clínicas, que incluyen hemocultivos y en enfermos con o sin infección por el VIH (8, 29, 31, 43, 48, 50).
Ambas especies comparten características morfológicas, fisiológicas y presentan patrones bioquímicos similares (49, 50), lo que hace dificultosa la diferenciación entre las mismas y algunos aislamientos pueden haber sido identificados erróneamente. Varios investigadores han realizado estudios retrospectivos para reubicar cepas previamente catalogadas como C. albicans (17, 28, 32).
Las dos presentan un color similar en medios con sustratos cromogénicos (5, 11, 20, 21), tienen la capacidad de formar tubos germinativos y clamidoconidias en los medios habituales (agar leche, agar harina de maíz con Tween 80). Sin embargo, una de las características que llamó la atención desde un principio es la capacidad de desarrollar resistencia frente a fluconazol que tiene C. dubliniensis (20, 26, 30, 31, 44).
Por otra parte, ya se han desarrollado varios métodos basados en las características genotípicas que permitirían identificar adecuadamente a esta nueva especie de Candida (1, 3, 9, 10, 24, 30, 35, 47, 49) pero no todos los laboratorios de diagnóstico micológico cuentan con esta tecnología.
También se han descrito numerosas técnicas para evidenciar las características fenotípicas diferenciales entre estas dos levaduras. Entre ellas podemos citar la variación de color en medios con sustratos cromogénicos (CHROMagar Candida, Candida ID2), la capacidad de desarrollo a distintas temperaturas, los patrones de asimilación de carbohidratos, el color, el aspecto de las colonias y la producción de clamidoconidias en diferentes sustratos, la producción de un halo de opacidad en medios con Tween 80 y CaCl2, la asimilación de D-xilosa o la producción de β glucosidasa, la inmunofluorescencia con anticuerpos monoclonales, la coagregación con Fusobacterium nucleatum, identificación mediante electroforesis en gel de almidón y SDS-PAGE, etc. (2, 4, 6, 11- 16, 20, 22, 23, 25, 33, 34, 36, 37, 38, 41-43, 48-50).
En nuestro país no existe aún mucha información acerca de la prevalencia de C. dubliniensis en distintas instituciones asistenciales. El Hospital F. J. Muñiz de la ciudad de Buenos Aires es un centro especializado en enfermedades infecciosas y allí se atiende una elevada proporción de enfermos VIH +. Desde hace una década, en la Unidad Micología de este establecimiento se agregaron a las determinaciones de rutina para la identificación de C. albicans algunas técnicas que permitan diferenciar además C. dubliniensis, tales como el uso del medio de Staib (2, 46), la asimilación de xilosa y la capacidad de desarrollo a 45 °C (13, 35). Sin embargo, se encontró que varios aislamientos de C. albicans producían clamidoconidias en el medio de Staib, aunque las otras características correspondían a dicha especie, y esto nos llevó a implementar otras técnicas fenotípicas a fin de encontrar un algoritmo de diferenciación sencillo, económico y seguro.
En este trabajo nos propusimos conocer la proporción de aislamientos de C. dubliniensis en las muestras clínicas que se reciben en la Unidad Micología de este hospital y evaluar la batería de métodos fenotípicos que se utilizan en el Servicio para la discriminación entre ambas levaduras y determinar cuáles son los más fiables.
MATERIALES Y MÉTODOS
Cepas
De un total de 388 aislamientos, se incluyeron 212 que correspondían a levaduras compatibles con las especies en estudio. Las mismas provenían de los siguientes materiales clínicos: exudados orofaríngeos 48, materiales respiratorios 51, flujos vaginales 86, orinas 6 y uñas de mano 21. Además se utilizaron como control 5 cepas de colección: 3 de C. albicans (ATCC 64458, ATCC 90028 y ATCC 90029) y 2 de C. dubliniensis (3949 y 11473 cedidas gentilmente por la Cátedra de Microbiología de la Facultad de Medicina y Odontología de la Universidad del País Vasco). Todas fueron subcultivadas en medio de Sabouraud sin antibióticos 24 horas previas a la realización de las pruebas. A todas aquellas con alguna característica compatible con C. dubliniensis se las tipificó, además, mediante API ID 32C (bioMérieux, L'Étoile, Francia) y amplificación del ADN específico por PCR con los iniciadores descriptos por Donnelly et al. (9) (datos no mostrados).
Siembra en medios con sustratos cromogénicos (21)
Se realizó una suspensión de las levaduras en solución fisiológica y se sembraron placas con CHROMagar Candida® (CHROMagar Company, París, Francia) y medio con sustratos cromogénicos de Oxoid® (Cromogenic Candida). Se incubaron a 30 °C durante 72 horas. Al cabo de ese lapso se consignó el color desarrollado y el aspecto de la colonia. Una colonia aislada se repicó en medio de Sabouraud sin antibióticos y se incubó a 30 °C durante 24 horas para realizar todas las pruebas de identificación.
Formación de tubos germinativos (TG) y clamidoconidias (CL) en agar leche (18)
Se preparó el agar leche de acuerdo con el método explicado en otros trabajos (7) y se lo colocó sobre portaobjetos estériles en cámara húmeda. Se sembró una estría por incisión en el medio y se cubrió con un cubreobjetos estéril. Se incubó a 37 °C durante 3 horas y se realizó la lectura microscópica con 200X y 400X para observar la presencia de TG, luego se volvió a incubar a 30 °C durante 48 horas para visualizar la formación de CL (Figura 1). Este no fue uno de los métodos diferenciales, sino que se utilizó como criterio de inclusión de las levaduras compatibles con ambas especies.

Figura 1. Clamidoconidias terminales de C. albicans en agar leche, luego de 48 h de incubación, 400X.
Formación de CL en medio de Staib (44)
La siembra se realizó sobre portaobjetos de la misma forma que en el agar-leche. Se incubó a 28 °C durante 48 horas y se observó microscópicamente la presencia de CL.
Detección de actividad esterasa en medio de opacidad
Se utilizó el medio descrito por Slikfin et al. (45) con Tween 80 al 1% y CaCl2 al 0,1%. Se sembró en forma de círculo de 1 cm de diámetro y se incubó a 30 °C durante 72 horas. Se observó la presencia de un halo de opacidad alrededor de la colonia (Figura 2).
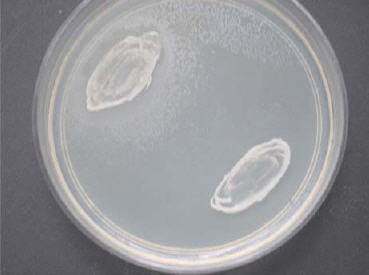
Figura 2. Presencia de halo de opacidad alrededor de la colonia de C. albicans.
Desarrollo en agar tabaco (19)
Se preparó de acuerdo con la técnica de Khan et al. pero en lugar de cigarrillos se utilizó tabaco para pipa marca Exceter® (Nobleza-Piccardo S. R. L., Argentina). Las placas se sembraron de igual forma que el agar-opacidad y se incubaron a 30 °C durante 48-72 h y se analizó la macromorfología de las colonias: la presencia de borde (regular o festoneado), el color (blanco o dorado) y el aspecto (liso o rugoso) (Figura 3a) y la formación de CL a 200X y 400X (Figura 3b).

Figura 3a. Color de las colonias de C. albicans y C. dubliniensis en agar-tabaco.
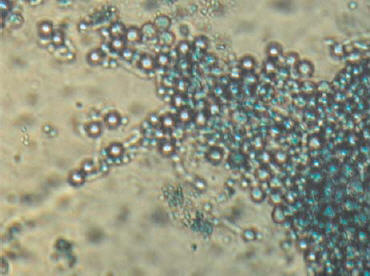
Figura 3b. Aspecto microscópico de C. dubliniensis en agar-tabaco con presencia de conglomerados de clamidoconidias, 400X.
Desarrollo a 45 °C (35)
Se realizó la siembra en medio de Sabouraud sin antibióticos y se incubó a 45 °C durante 24 h.
Formación de CL en medio de tomate-zanahoria
Se utilizó el medio descrito por Alves et al. (4) modificado. Se preparó un extracto con 20 g de zanahoria rallada y 20 g de tomate picado y sin semillas, se hirvieron durante 1 hora en 1 litro de agua, se filtró por gasa y se llevó nuevamente a volumen de 1 litro, se agregaron 20 g de agar, se disolvió y se autoclavó durante 15 min a 1 atm. de presión. Se vertió el medio sobre portaobjetos (3 ml) y se sembró por incisión, se colocó un cubreobjetos estéril y se incubó en cámara húmeda a 30 °C durante 48 h. Se observó microscópicamente, la presencia de CL a 200 X y 400 X.
Utilización de D-xilosa (10, 13)
Se realizó una suspensión con una turbidez equivalente al N° 2 de la escala de McFarland en 0,5 ml de solución fisiológica de un repique de 24 h en agar Sabouraud a 37 °C. Se agregó una tableta de D-xilosa Rosco® (Rosco, Dinamarca) y se incubó a 37 °C durante 3 horas. El viraje al amarillo indica la asimilación de este azúcar.
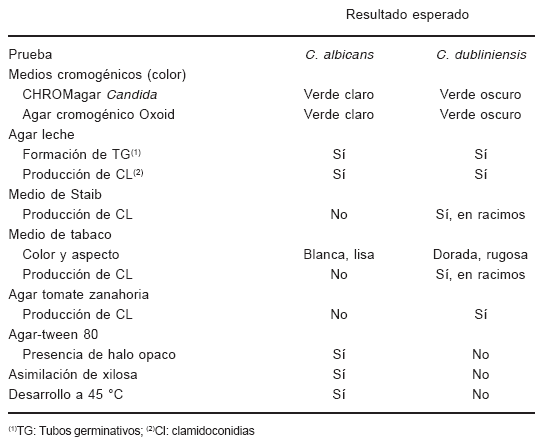
En la Tabla 1 se señalan las características que corresponden a cada especie y que se verificaron, además, en las 5 cepas control.
Tabla 1. Características fenotípicas correspondientes a C. albicans y C. dubliniensis en los distintos métodos ensayados
RESULTADOS
El agar-leche demostró ser sumamente sensible para la identificación de este grupo de Candida ya que se observaron TG en el 93,8% y formaron CL el 100% de los 212 aislamientos. Este medio siempre fue utilizado en primera instancia antes de iniciar la diferenciación entre las 2 especies.
Los resultados de las distintas pruebas diferenciales referentes a los aspectos macro y microscópicos de las colonias y características bioquímicas con los distintos perfiles fenotípicos están consignados en la Tabla 2.
Tabla 2.
Distribución de los aislamientos según los resultados de las distintas pruebas realizadas
Los 212 aislamientos estudiados así como las cepas control presentaron color verde en los dos medios con sustrato cromogénico, sin embargo las tonalidades del mismo no permitieron el diagnóstico presuntivo de una u otra especie. CHROMagar fue mejor en la identificación presuntiva de C. albicans (98,0% vs. 77,4% presentaron color verde claro) en tanto que Agar cromogénico de Oxoid detectó mejor a C. dubliniensis (4 cepas vs. 9 cepas de color verde oscuro).
En el medio de Staib no se analizó el aspecto macroscópico del desarrollo de ambas especies. Las diferencias en aspecto y color de las colonias fueron muy notorias en el medio de tabaco (100% de C. dubiniensis fueron doradas y rugosas y 98,5% de C. albicans fueron blancas y lisas).
La producción de CL en todos los aislamientos de C. dubliniensis de muestras clínicas así como de las cepas control se obtuvo en los 3 medios diferenciales y, en la mayor parte de casos, se presentaban en forma de racimos. Sin embargo, se observaron también en 23 (11,6%) en medio de Staib, 30 (15,1%) en agar tabaco y 24 (12,1%) en tomate-zanahoria, de los correspondientes a C. albicans.
C. albicans desarrolló a 45 °C en el 99,0% de los casos (197 aislamientos) y asimilaron D-xilosa 195 de ellos (98,0%). Por otra parte sólo un aislamiento de C. dubliniensis desarrolló a 45 °C y otro fue capaz de utilizar D-xilosa. La actividad de lipasa se evidenció en el medio con Tween 80 y cloruro de calcio en 190 (95,6%) aislamientos de C. albicans y no se encontró esta actividad enzimática en 11/13 (84,6%) de C. dubliniensis.
DISCUSIÓN
Los medios con sustratos cromogénicos son de gran ayuda para la identificación presuntiva de distintas levaduras, a la vez que permiten reconocer la existencia de infecciones por más de una especie simultáneamente.
Sin embargo, las colonias se presentan en una gama de color determinado y la caracterización del mismo depende del observador. Por este motivo no siempre es sencillo discriminar entre variaciones de tonalidades de un color para asignar solamente con esto la correspondencia con la especie aislada.
En numerosos trabajos se señala que C. dubliniensis produce colonias de color verde más oscuro que C. albicans en el medio de CHROMagar Candida (10, 12, 43) y lo mismo sucedería en el agar cromogénico de Oxoid. Sin embargo, en varias investigaciones se señala que no siempre se evidencia la diferencia de color y que basarse solamente en este aspecto puede llevar a confusión (5, 23, 48). En nuestra experiencia, ni las cepas control ni los aislamientos clínicos mostraron diferencias de tonalidad que permitieran diferenciar unas de otras. No hemos utilizado Candida ID2 (bioMérieux, Francia) que, de acuerdo con Eraso et al. (11) permite discriminar mejor entre ambas que los medios cromogénicos utilizados por nosotros.
La producción de CL en racimo en algunos medios como los que contienen tabaco (19), ácido cafeico (2), agar tomate (4), medio de Staib (46) y también el medio con extracto de semillas de girasol es considerada por varios autores como métodos muy buenos para diferenciar entre ambas especies. En este trabajo encontramos que entre 11,6% y 15,1% de las cepas de C. albicans, según el medio utilizado, produjeron CL más o menos abundantes. Por otra parte el agar-tabaco demostró que todos los aislamientos de C. dubliniensis presentaron colonias de aspecto rugoso y color dorado y formaban CL en gran cantidad, en tanto 195 (98,0%) cepas de C. albicans fueron blancas y lisas independientemente de su capacidad de formar CL en este medio.
Los resultados obtenidos con la siembra a 45 °C y la asimilación de D-xilosa fueron altamente satisfactorios para la identificación de C. albicans, aunque debe tenerse en cuenta la posibilidad de falso-positivos y falso-negativos con estas técnicas (13). La formación de halo de precipitación en el medio de opacidad demostró muy buena sensibilidad y especificidad para esta especie, cuando se usó CaCl2 al 0,1%, ya que la visualización con una concentración menor es dificultosa (45).
Con respecto a la proporción de aislamientos de C. dubliniensis en nuestro laboratorio, en el transcurso de dos años demostró que es de 13/212 (6,1%) de los que se estudiaron; esta proporción es superior a la encontrada por Ahmad en Kuwait (1) y a la descripta por Odds et al. (32) en un trabajo retrospectivo donde encuentran un 2,1% de aislamientos provenientes de distintas partes del mundo y desde la década del 70, o la del trabajo de Jabra-Rizk et al. que encuentran 1,2% en aislamientos de la Universidad de Maryland (17). Por otra parte, Mata et al. encontraron 14,4% entre 111 cepas previamente identificadas como C. albicans en Venezuela (27). En nuestro país hay pocas comunicaciones acerca de la prevalencia de esta especie (40). López et al. (23) no detectaron ninguna en 44 muestras de mucosa oral en la ciudad de Rosario con la utilización de métodos fenotípicos y moleculares, tampoco fueron aisladas en exudados vaginales por Buscemi et al (7) ni por Rodero et al (39) en candidiasis nosocomiales. Los aislamientos de nuestro trabajo correspondieron a pacientes VIH + en 10 casos, uno VIH - y no se conocía el status serológico en dos pacientes de consultorios externos y los materiales de donde provenían fueron: lesiones orofaríngeas 7, secreciones respiratorias 5 y 1 orina. Estos datos concuerdan con las localizaciones más frecuentes descritas en distintas investigaciones (1, 7, 17, 26, 32, 49).
Consideramos que el uso combinado de al menos 3 técnicas de diferenciación fenotípica, puede ser una alternativa válida para todos aquellos laboratorios que no cuentan con metodologías moleculares de diagnóstico. De acuerdo con los resultados de esta investigación encontramos que las características macro y micromorfológicas en el medio de tabaco, el desarrollo a 45 °C y asimilación de D-xilosa, son las más adecuadas para este propósito ya que permiten distinguir entre las dos especies de forma confiable, simple y económica.
1. Ahmad S, Mocadas E, Al-Sweih N, Kan ZU. Phenotypic and molecular characterization of Candida dubliniensis isolates from clinical specimens in Kuwait. Medical principles and Practice 2005; 14 (Suppl 1): 77-83. [ Links ]
2. Al Mosaid A, Sullivan D, Salkin IF, Sanley D, Coleman DC. Differentiation of Candida dubliniensis from Candida albicans on Staib agar and caffeic acid-ferric citrate agar. J Clin Microbiol 2001; 39: 323-7. [ Links ]
3. Alonso-Vargas R, Garaizar J, Pontón J, Quindós G. Utility of Random Amplified Polymorphic DNA in the discrimination between Candida albicans and Candida dubliniensis. Rev Iberoam Micol 2000; 17: 10-3. [ Links ]
4. Alves SH, de Loret ES, Linares CE, Silveira C, Scheid LA, Pereira DB, et al. Comparison among tomato juice agar with other three media for differentiation of Candida dubliniensis from Candida albicans. Rev Inst Med Trop Sao Paulo 2006; 48: 119-21. [ Links ]
5. Ballesté R, Arteta Z, Fernández N, Mier C, Mousqués N, Xavier B, et al. Evaluación del medio cromógeno CHROMagar Candida TM para la identificación de levaduras de interés médico. Rev Méd Urug 2005; 21: 186-93. [ Links ]
6. Bikandi J, San Millan R, Moragues MD, Cebas G, Clarke M, Coleman DC, et al. Rapid identification of Candida dubliniensis by indirect immunofluorescence based on differential localization of antigen on C. dubliniensis blastospores and Candida albicans germ tubes. J Clin Microbiol 1998; 36: 2428-33. [ Links ]
7. Buscemi L, Arechavala A, Negroni R. Estudio de las vulvovaginitis agudas en pacientes adultas, sexualmente activas, con especial referencia a la candidiasis, en pacientes del hospital de infecciosas Francisco J. Muñiz. Rev Iberoam Micol 2004; 21: 177-81. [ Links ]
8. Coleman DC, Sullivan DJ, Bennet, DE, Moran GP, Barry HJ, Shanley DB. Candidiasis the emergence of a novel species, Candida dubliniensis. AIDS, 1997; 11: 557-67. [ Links ]
9. Donnelly SM, Sullivan DJ, Shanley DB, Coleman DC. Phylogenetic analysis and rapid identification of Candida dubliniensis based on analysis of ACT1 intron and exon sequences. Microbiology 1999; 145: 1871-82. [ Links ]
10. Ellepola AN, Hurst SF, Elie CM, Morrison CJ. Rapid and unequivocal differentiation of Candida dubliniensis from other Candida species using species-specific DNA probes: comparison with phenotypic identification methods. Oral Microbiol Immunol 2003; 18: 379-88. [ Links ]
11. Eraso E, Sahand IH, Villar-Vidal M, Marcos C, Moragues D, Madariaga L, et al. Usefulness of Candida ID2 agar for the presumptive identification of Candida dubliniensis. Med Mycol 2006; 44: 611-5. [ Links ]
12. Freydiere AM, Guinet R, Bkoiron P. Yeast identification in the clinical microbiology laboratory: phenotypical methods. Med Mycol 2001; 39: 9-33. [ Links ]
13. Gales AC, Pfaller MA, Houston AK, Joly S, Sullivan DJ, Coleman DC, et al. Identification of Candida dubliniensis based on temperature and utilization of xilose and alphamethil-D-glucoside as determined with the API 20CAUX and Vitek YBC systems. J Clin Microbiol 1999; 37: 3804-8. [ Links ]
14. Guelfand L, Grisolía P, Bozzano C, Kaufman S. Comparación de métodos para la identificación de las levaduras más frecuentes en el laboratorio de microbiología clínica. Rev Argent Microbiol 2003; 35: 49-53. [ Links ]
15. Giusiano G, Mangiaterra M. Diferenciación e identificación presuntiva rápida de levaduras con el medio Chromagar Candida. Rev Argent Microbiol 1998; 30: 100-3. [ Links ]
16. Jabra-Rizk MA, Falkler Jr WA, Merz WG, Kelley JL, Baqui AAMA, Meiller TF. Candida dubliniensis and Candida albicans display surface variations consistent with observed intergeneric coaggregation. Rev Iberoam Micol 1999; 16: 8-14. [ Links ]
17. Jabra-Rizk MA, Falkler Jr WA, Merz WG, Baqui AAMA, Kelley JL. Retrospective identification and characterization of Candida dubliniensis isolates among Candida albicans clinical laboratory isolates from human immunodeficiency virus (HIV)-infected and non-HIV-infected individuals. J Clin Microbiol 2000; 38: 2423-6. [ Links ]
18. Jitsuron S, Klamsini S, Pattararagron N. Milk medium for germ tube and chlamydoconidiae production by Candida. Mycopathologia, 1993; 123: 95-8. [ Links ]
19. Khan ZU, Ahmad S, Mocadas E, Chady R. Tobacco agar, a new medium for differentiating Candida dubliniensis from Candida albicans. J Clin Microbiol 2004; 42: 4796-8. [ Links ]
20. Kirkpatrick WR, Revankar SG, McAtee RK, López-Ribot JL, Fothergill AW, McCarthy DI, et al. Detection of Candida dubliniensis in oropharyngeal samples from human immunodeficiency virus infected patients in North America by primary CHROMagar Candida screening and susceptibility testing of isolates. J Clin Microbiol 1998; 36: 3007-12. [ Links ]
21. Koehler AP, Chu KC, Honang ET, Cheng AF. Simple, reliable, and cost-effective yeast identification scheme for the clinical laboratory. J Clin Microbiol 1999; 37: 422-6. [ Links ]
22. Linares Sicilia MJ, Solís Cuesta F. Identificación de levaduras. En: Pemán J, Martín-Mazuelos E, Rubio Calvo MC: Guía Práctica de Identificación y Diagnóstico en Micología Clínica. Rev Iberoam Micol 2001; p. 11-1-18. [ Links ]
23. López C, Giro L, Ramos L, Ramadán S, Bulacio L. Comparación de diferentes métodos para la identificación de especies del género Candida. Rev Argent Microbiol 2005; 37: 16-21. [ Links ]
24. Mannarelli BM, Kurtzman CP. Rapid identification of Candida albicans and other human pathogenic yeasts by using short oligonucleotides in a PCR. J Clin Microbiol 1998; 36: 1634-41. [ Links ]
25. Marot-Leblond A, Beucher B, David S, Nail-Billard S, Roblert R. Development and evaluation of a rapid latex agglutination test using a monoclonal antibody to identify Candida dubliniensis colonies. J Clin Microbiol 2006; 44: 138-42. [ Links ]
26. Martínez M, López-Ribot JL, Kirkpatrick WR, Coco BJ, Bachmann SP, Patterson TF. Replacement of Candida albicans with C. dubliniensis in human immunodeficiency virus-infected patients with oropharyngeal candidiasis treated with fluconazole. J Clin Microbiol 2002; 40: 3135-9. [ Links ]
27. Mata Essayag S, Hartung de Capriles C, Sánchez L, Gallardo S, Pérez C, Colella MT, et al. Aislamiento de Candida dubliniensis en Venezuela. Antibiot Infecc 2002; 40: 165-70. [ Links ]
28. Meis JF, Verduyn Lunel FM, Verweij PE, Voss A. One-year prevalence of Candida dubliniensis in a Dutch University. J Clin Microbiol 2000; 38: 3139-40. [ Links ]
29. Meis JF, Ruhnke M, De Pauw BE, Odds FC, Siegert W, Verweij PE. Candida dubliniensis candidemia in patients with chemotherapy-induced neutropenia and bone marrow transplantation. Emerg Infec Dis 1999; 5: 150-3. [ Links ]
30. Meyer W, Maszewska K, Sorrell TC. PCR fingerprinting: a convenient molecular tool to distinguish between Candida dubliniensis and Candida albicans. Med Mycol 2001; 39: 185-93. [ Links ]
31. Moran GP, Sullivan DJ, Herman MC, McCreary CE, Harrington BJ, Shanley DB, et al. Antifungal drug susceptibilities of oral Candida dubliniensis isolates from human immunodeficiency virus (HIV)-infected and non-HIV-infected subjects and generation of stable fluconazole-resistant derivatives in vitro. Antimicrob Agents Chemother 1997; 41: 617-23. [ Links ]
32. Odds FC, Van Nuffel L, Dams G. Prevalence of Candida dubliniensis isolates in a yeast stock collection. J Clin Microbiol 1998; 36: 2869-73. [ Links ]
33. Pincus DH, Coleman DC, Pruitt WR., Padhye AA, Salkin IF, Geimer M, et al. Rapid identification of Candida dubliniensis with commercial yeast identification systems. J Clin Microbiol 1999; 37: 3533-9. [ Links ]
34. Pincus DH, Orenga S, Chatellier S. Yeast identification past, present, and future methods. Med Mycol 2007; 45: 97-121. [ Links ]
35. Pinjon E, Sullivan D, Salkin I, Shanley D, Coleman D. Simple, inexpensive reliable method for differentiation of Candida dubliniensis from Candida albicans. J Clin Microbiol 1998; 36: 2093-5. [ Links ]
36. Polackeck L, Melamed M, Bercovier H, Salkin IF. b-glucosidase in Candida albicans and its application in yeast identification. J Clin Microbiol 1987; 25: 907-10. [ Links ]
37. Quindós G, Alonso-Vargas R, Helou S, Arechavala A, Martín-Mazuelos E, Negroni R. Evaluación micológica de un nuevo medio de cultivo cromógeno (Candida ID) para el aislamiento e identificación presuntiva de C. albicans. Rev Iberoam Micol 2001; 18: 23-8. [ Links ]
38. Quindós G, Pontón J. Candidiasis de la cavidad oral: etiología, patogenia y diagnóstico de laboratorio. Medicina Oral 1996; 1: 85-95. [ Links ]
39. Rodero L, Davel G, Córdoba S, Soria M, Canteros C, Hochenfellner F, et al. Estudio multicéntrico sobre candidiasis nosocomial en la República Argentina. Rev Argent Microbiol 1999; 31: 114-9. [ Links ]
40. Rodero L, Losso M, Cantero C, Hochenfellner F, Davel G. Candida dubliniensis: primer aislamiento en Argentina. Rev Argent Microbiol 1998; 30: 39-41. [ Links ]
41. Rosa EAR, Rosa RT, Boriollo MFG, Bernardo WLC, Hofling JF. Oral Candida albicans and Candida dubliniensis differentiation by multilocus enzyme electrophoresis and sodium dodecylsulphate-polyacrylamide gel electrophoresis. Rev Argent Microbiol 2003; 35: 24-8. [ Links ]
42. Salkin IF, Pruitt WR, Padhye AA, Sullivan DJ, Coleman DC, Pincus DH. Distinctive carbohydrate assimilation profiles used to identify the first clinical isolates of Candida dubliniensis recovered in the United States. J Clin Microbiol 1998; 36: 1467. [ Links ]
43. Schoofs A, Odds FC, Colebundess R, Ieren M, Goosserss H. Use of specialized isolation media for recognition and identification of Candida dubliniensis from HIV-infected patients. Eur J Clin Microbiol Infect Dis 1997; 16: 296-300. [ Links ]
44. Serda Kantarcroglu A, Yücel A. The presence of fluconazoleresistant Candida dubliniensis strains among Candida albicans isolates from immunocompromised or otherwise debilitated HIV-negative Turkish patients. Rev Iberoam Micol 2002; 19: 44-8. [ Links ]
45. Slifkin M. Tween 80 opacity test responses of various Candida species. J Clin Microbiol 2000; 38: 4626-8. [ Links ]
46. Staib F, Morschauser J. Chlamydospore formation on Staib agar as a species-specific characteristic of Candida dubliniensis. Mycoses 1999; 42: 521-4. [ Links ]
47. Steffan P, Vazquez JA, Boikov D, Xu C, Sobel JD, Akins RA. Identification of Candida species by randomly amplified polymorphic DNA fingerprinting of colony lysates. J Clin Microbiol 1997; 35: 2031-9. [ Links ]
48. Sullivan D, Coleman D. Candida dubliniensis: Characteristics and identification. J Clin Microbiol 1998; 36: 329-34. [ Links ]
49. Sullivan DJ, Westerneng TJ, Haynes KA, Bennet DE, Coleman DC. Candida dubliniensis sp. nov., phenotypic and molecular characterization of a novel species associated with oral candidiasis in HIV-infected individuals. Microbiology 1995; 141: 1507-21. [ Links ]
50. Tintelnot K, Haase G, Seibold M, Bergmann F, Staemmler M, Franz T, et al. Evaluation of phenotypic markers for selection and identification of Candida dubliniensis. J Clin Microbiol 2000; 38: 1599-608. [ Links ]
Recibido: 27/11/07
Aceptado: 05/08/08.














