Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Revista argentina de microbiología
versión impresa ISSN 0325-7541versión On-line ISSN 1851-7617
Rev. argent. microbiol. v.40 n.4 Ciudad Autónoma de Buenos Aires oct./dic. 2008
Caracterización fenotípica y genotípica de la resistencia a imipenem en Pseudomonas aeruginosa aisladas en un hospital de Buenos Aires
D. Cejas1, M. Almuzara2, G. Santella1, A. Tuduri2, S. Palombarani2, S. Figueroa2, G. Gutkind1, M. Radice1*.
1Cátedra de Microbiología, Facultad de Farmacia y Bioquímica, Universidad de Buenos Aires; Junín 954, (1113) Buenos Aires;
2Laboratorio de Bacteriología, Hospital Interzonal General de Agudos "Eva Perón", San Martín, Provincia de Buenos Aires. Argentina.
*Correspondencia. E-mail: mradice@ffyb.uba.ar
RESUMEN
En el presente estudio, que tuvo por objeto analizar los mecanismos involucrados en la resistencia a carbapenemes, se incluyeron 129 aislamientos de Pseudomonas aeruginosa recuperados durante el año 2006 en el Hospital "Eva Perón" de la Provincia de Buenos Aires. La caracterización fenotípica y genotípica de la resistencia permitió reconocer la presencia de metalo-b-lactamasas (MBL) en el 14% de esos aislamientos. En todos ellos se identificó la presencia de la enzima IMP-13; sin embargo, algunos aislamientos resultaron sensibles a carbapenemes de acuerdo con los puntos de corte establecidos por el CLSI e incluso con las sugerencias de la Subcomisión de Antimicrobianos de SADEBAC, AAM. El ensayo de detección fenotípica de MBL de sinergia con doble disco resultó útil en este estudio. Sólo aquellos aislamientos productores de IMP-13 que a su vez presentaron alteraciones en las proteínas de membrana externa resultaron completamente resistentes a imipenem. Los aislamientos productores de MBL correspondieron a varios tipos clonales, lo cual sugiere no sólo la diseminación de una cepa resistente, sino también la diseminación horizontal de este mecanismo de resistencia entre clones diferentes.
Palabras clave: Carbapenemasas; Metalo-b-lactamasas; Resistencia a carbapenemes.
ABSTRACT
Phenotypic and genotypic characterization of imipenem-resistant Pseudomonas aeruginosa isolated in Buenos Aires. From 129 P. aeruginosa isolated at a health care centre located in Buenos Aires (Hospital "Eva Perón"), 14% produced IMP-13. Although 18 isolates were metallo-b-lactamases (MBL) producers, only those isolates that displayed altered outer membrane protein profiles correlated with the resistant category according to CLSI or even Subcomisión de Antimicrobianos, SADEBAC, AAM. Phenotypic screening of metallo-b-lactamases proved to be appropriate for detecting MBL producing isolates. IMP-13 producing isolates corresponded to at least five different clonal types, which not only suggests the dissemination of the resistant strain but also of the resistant marker.
Key words: Carbapenemases; Metallo-b-lactamases; Carbapenem resistance.
INTRODUCCIÓN
Pseudomonas aeruginosa es uno de los patógenos recuperados con más frecuencia en infecciones hospitalarias. Las infecciones causadas por este microorganismo suelen resultar complicadas y están asociadas a un alto índice de mortalidad. Los carbapenemes constituyen la opción terapéutica más utilizada en cuadros severos producidos por bacilos gram-negativos no fermentadores, en los cuales es frecuente la multirresistencia a otros antibióticos. La resistencia a carbapenemes en P. aeruginosa ha sido descrita en diversas partes del mundo y se considera un problema terapéutico creciente (7, 30); en nuestro país se ha documentado en el 26% de los aislamientos (31).
P. aeruginosa puede adquirir fácilmente resistencia a la mayoría de las drogas de utilización clínica a través de mutaciones en los genes codificantes o reguladores de los mecanismos involucrados en su resistencia natural (12, 14, 18, 19, 24), o a través de la adquisición de determinantes genéticos de enzimas con capacidad de hidrolizar antibióticos (9). Las mutaciones que conducen a la pérdida de la proteína OprD contribuyen a la resistencia a imipenem y a la disminución de la sensibilidad a antibióticos (29), mientras que aquellas que determinan el incremento en la expresión de los sistemas de eflujo de tipo Mex pueden conferir resistencia o sensibilidad reducida a los b-lactámicos, excepto a imipenem (4, 20, 21). Por su parte, la hiperproducción de enzimas de tipo AmpC se asocia a la resistencia a penicilinas y cefalosporinas (13).
La adquisición de genes codificantes de metalo-b- lactamasas (MBL) constituye un mecanismo de resistencia con alta implicancia epidemiológica debido a su capacidad de diseminación horizontal (36). Estas enzimas pertenecen a la clase molecular B de Ambler (1) y se caracterizan por presentar un amplio perfil de sustratos, que incluye cefalosporinas de espectro extendido (cefotaxima, ceftacidima y cefepime) y carbapenemes (imipenem y meropenem), pero no hidrolizan monobactamas (aztreonam); no son afectadas por los inhibidores de uso clínico y su actividad es susceptible a quelantes como el EDTA (25). Se conocen diversos grupos de MBL: IMP, VIM, SPM, GIM-1, SIM-1 y AIM-1. Las enzimas de tipo IMP y VIM constituyen las más ampliamente diseminadas en el mundo (5, 30, 36, 38). Éstas han sido descritas principalmente en P. aeruginosa y otros microorganismos no fermentadores, y con menor frecuencia en enterobacterias. Los genes codificantes de MBL, a excepción de spm-1, se encuentran como genes en cassette reclutados en integrones de clase 1 o clase 3, que pueden, a su vez, estar localizados en plásmidos y transposones; esto contribuiría a la amplia diseminación de estos genes de resistencia (36).
A pesar de que en otras partes del mundo han sido ampliamente documentadas, en nuestro país las MBL se detectaron recién en el año 2002 (27), y hasta la fecha fueron informadas las enzimas VIM-2, VIM-11, IMP- 13 y SPM-1 (3, 6, 26, 28).
Estudios previos han demostrado que alteraciones en la expresión de la proteína OprD y la sobreexpresión de las bombas de eflujo de la familia RND (resistant-nodulation- cell division) en cepas productoras de MBL incrementan los niveles de resistencia a imipenem y meropenem, respectivamente (17, 37).
En este trabajo se analizaron los mecanismos responsables de la resistencia o la sensibilidad disminuida a carbapenemes en P. aeruginosa aisladas de pacientes internados en el Hospital Interzonal General de Agudos "Eva Perón" de la Provincia de Buenos Aires durante el año 2006. Se intentó caracterizar fenotípica y genotípicamente las MBL presentes y las posibles alteraciones en la membrana externa, para analizar la correlación entre los mecanismos detectados y los respectivos fenotipos de resistencia.
MATERIALES Y MÉTODOS
Microorganismos
Se incluyeron aislamientos de P. aeruginosa recuperados de pacientes internados en diferentes servicios del Hospital "Eva Perón" de la Provincia de Buenos Aires, durante el año 2006, que presentaron resistencia a ceftacidima y disminución de la sensibilidad poblacional a imipenem, determinadas por el ensayo de difusión en agar según el CLSI. Se entiende como disminución de la sensibilidad poblacional a la presencia de halos de inhibición que, si bien no resultan inferiores a los puntos de corte de resistencia del CLSI, se alejan de los obtenidos para la población salvaje (16).
Determinación de la sensibilidad a antimicrobianos
Los ensayos de sensibilidad por difusión en agar y la determinación de la concentración inhibitoria mínima (CIM) por dilución en medio sólido se realizaron según las recomendaciones del CLSI (22, 23) y de la Subcomisión de Antimicrobianos, SADEBAC-AAM (33). Se ensayaron los siguientes antibióticos: ceftacidima (CAZ), ceftacidima/clavulánico (CAC), piperacilina/ tazobactam (PTZ), imipenem (IMI) y meropenem (MER) (Laboratorio Britania); se incluyeron discos de EDTA (1 μmol). Los discos conteniendo antibióticos fueron ubicados de acuerdo con el esquema sugerido por la Subcomisión de Antimicrobianos. La CIM se ensayó para los siguientes antibióticos dentro de los rangos que se indican en μg/ml: piperacilina (PIP) (1-128), PTZ (1-128), CAZ (16-1024), cefepime (FEP) (16-1024), aztreonam (AZT) (0,5-128), IMI (0,5-32), MER (1-128), amikacina (AKN) (0,5-128), gentamicina (GEN) (0,5-128), ciprofloxacina (CIP) (0,5-128).
Detección fenotípica de metalo-b-lactamasas
Se realizaron ensayos de sinergia de doble disco empleando EDTA (1 μmol) como inhibidor de las MBL (26), sobre la base del ensayo de sinergia descrito inicialmente por Arakawa et al. (2). Para ello se inocularon las placas según las recomendaciones del CLSI para la prueba de difusión, y se colocaron discos conteniendo EDTA (1 μmol) y discos comerciales conteniendo CAZ y con IMP a 15 mm de distancia, centro a centro del primero. Un agrandamiento en el halo de inhibición del disco con antibiótico en la zona adyacente al disco con EDTA fue interpretado como posible presencia de MBL.
Detección del perfil de sustratos de las b-lactamasas
Se obtuvieron extractos enzimáticos crudos por ruptura ultrasónica de los cultivos bacterianos. Se evaluó el perfil de hidrólisis de sustratos por el método iodométrico en agar, utilizando como sustratos ampicilina (AMP) (500 μg/ml), CAZ (1000 μg/ml) e IMI (1000 μg/ml) (32).
Determinación del perfil de proteínas de membrana externa (OMP)
La extracción de porinas se realizó según el método de Hancock y Poxton (10) y los perfiles fueron resueltos mediante electroforesis en geles de poliacrilamida al 12% en condiciones desnaturalizantes (SDS-PAGE), empleando un marcador de peso molecular de proteínas (Bio-Rad). Se seleccionaron para este ensayo los siguientes aislamientos, que presentaron distintos niveles de resistencia a carbapenemes y fueron productores o no productores de MBL: aislamiento 145, productor de IMP-13 (CIMIMI >32 μg/ml); aislamiento 152, productor de IMP-13 (CIMIMI = 8 μg/ml); aislamiento 208, productor de IMP-13 (CIMIMI = 8 μg/ ml); aislamiento 255, no productor de MBL (CIMIMI = 16 μg/ml). Se incluyó en el ensayo la cepa de P. aeruginosa ATCC 27853 como control.
Detección de la presencia de genes codificantes de MBL
Se realizó un ensayo de Multiplex-PCR empleando como molde ADN total (8). Se emplearon los siguientes pares de oligonucleótidos (5'-3'): VIMG-F: TCT ACA TGA CCG CGT CGT CTG TC, VIM-R mult: CGA ATG CGC AGC ACC AG; IMPA-F: CTA CCG CAG CAG AGT CTT TGC, IMP-R mult: CCA AAC YAC TAS GTT ATC T; SPM-F mult: AAA ATC TGG GTA CGC AAA CG, SPM-R mult: ACA TTA TCC GCT GGA ACA GG; GIMF mult:TCG ACA CAC CTT GGT CTG AA, GIM-R mult: AAC TTC CAA CTT TGC CAT GC; SIM-F mult: TAC AAG GGA TTC GGC ATC G, SIM-R mult: TAA TGG CCT GTT CCC ATG TG. Los reactivos empleados fueron de Invitrogen (EE.UU.). Se empleó un termociclador BIOMETRA T-Gradient (Alemania) y la siguiente reacción de amplificación: desnaturalización 94 °C, 5 minutos; seguida de 36 ciclos de: desnaturalización 94 °C, 30 segundos; hibridación 52 °C, 40 segundos; amplificación 72 °C, 50 segundos; y un período final de elongación 72 °C, 5 minutos. Los fragmentos esperados fueron: imp: 188 pb, vim: 390 pb, spm: 271 pb, gim: 477 pb, sim: 570 pb.
Determinación de la localización de los genes codificantes de MBL
Se amplificó por PCR la región variable de integrones de clase 1 a partir de DNA total. Se emplearon los siguientes oligonucleótidos (5'-3'): 5' C: GGC ATC CAA GCA GCA AGC, 3'C: AAG CAG ACT TGA CCT GAT. Los reactivos empleados fueron de Invitrogen (USA). Se empleó un termociclador BIOMETRA T-Personal (Alemania) y la siguiente reacción de amplificación: desnaturalización a 95 °C, 5 minutos; seguida de 30 ciclos de desnaturalización a 95 °C, 1 minuto; hibridación a 52 °C, 1 minuto; amplificación a 72 °C, 2 minutos; y un período final de elongación a 72 °C, 20 minutos. Se empleó un kit de Bioneer (EE.UU.) para purificar las bandas correspondientes a las regiones variables. Se realizó una PCR empleando cebadores específicos para imp, utilizando como molde el amplicón correspondiente a las regiones variables. Se emplearon los siguientes oligonucleótidos (5'-3'): IMP-AF: GAA GGC GTT TAT GTT CAT ACT T, IMP-AR: GTT TTG CCT TAC CAT ATT TGG A. Los reactivos empleados fueron de Invitrogen (EE.UU.). Se empleó un ciclador BIOMETRA T-Personal (Alemania) y la siguiente reacción de amplificación: desnaturalización 95 °C, 5 minutos; seguida de hibridación 52 °C, 15 minutos, con el agregado de la Taq polimerasa a los 6 minutos; y amplificación 72 °C, 6 minutos; seguida de 29 ciclos de desnaturalización 95 °C, 1 minuto; hibridación a 52 °C, 1 minuto; amplificación 72 °C, 1 minuto; y un período final de elongación 72 °C, 20 minutos. El fragmento esperado fue de 0,4 kb.
Secuenciación de los productos amplificados
Las secuencias nucleotídicas de los distintos fragmentos amplificados fueron determinadas por el servicio de secuenciación de Macrogen (Corea), empleando el sistema ABI Prism DNA 3700. Se usó la herramienta Blast (Basic Local Alignment Search Tool) del NCBI (National Center for Biotechnology Information) para comparar con las secuencias génicas depositadas en base de datos (http://www.ncbi.nlm.nih.gov/blast/).
Tipificación molecular de los aislamientos productores de MBL
Se amplificaron por PCR las secuencias repetitivas del DNA: REP-PCR, ERIC-PCR a partir de DNA total (34). Se emplearon los siguientes oligonucleótidos (5'-3'): REP-1: IIIGCGCCGICATCAGGC, REP-2: ACGTCTTATCAGGCCTAC; ERIC-1: CACTTAGGGGTCCTCAATGTA, ERIC-2: AAGTAAGTGACTGGGGTGAGCG. Los reactivos empleados fueron de Invitrogen (EE.UU.). Se empleó un ciclador Biometra T-Personal (Alemania) y las siguientes condiciones de amplificación: desnaturalización a 94 °C, 2 minutos; seguida de 30 ciclos de: desnaturalización a 94 °C, 30 segundos; hibridación a 50 °C, 1 minuto; amplificación a 72 °C, 4 minutos; y un período de extensión final a 72 °C, 7 minutos. El patrón de bandas fue visualizado con 0,5 μg/ml de bromuro de etidio. La matriz de similitud fue realizada empleando el programa TREECON y se construyeron dendrogramas basados en el algoritmo UPGMA (unweighted pair group method arithmetic averages).
RESULTADOS
Durante el año 2006 se recolectaron un total de 129 aislamientos de P. aeruginosa, de los cuales 36 resultaron resistentes a ceftacidima. En 18 de ellos se sospechó la presencia de MBL en el ensayo de difusión realizado según las recomendaciones de la Subcomisión de Antimicrobianos, ya que se observó sinergia entre los carbapenemes y el disco que contenía EDTA (Figura 1a). Los resultados fueron concordantes con el ensayo de detección fenotípica de MBL (Figura 1b). Estos aislamientos resultaron también resistentes a CAC, FEP, AZT, AKN, GEN y CIP (Tabla 1), y hubo concordancia de los resultados obtenidos en los ensayos por dilución y por difusión. Todos fueron sensibles a PTZ en el ensayo por difusión, mientras que dos presentaron resistencia cuando se determinó la CIM a esta combinación. Sin embargo, no se observó buena correlación en los resultados de sensibilidad a los carbapenemes entre los ensayos por dilución y por difusión: 8 aislamientos que fueron sensibles a imipenem en el ensayo por difusión resultaron de sensibilidad intermedia en el ensayo por dilución (Tabla 1). Esta discordancia fue observada en trabajos previos (26), en especial para FEP.
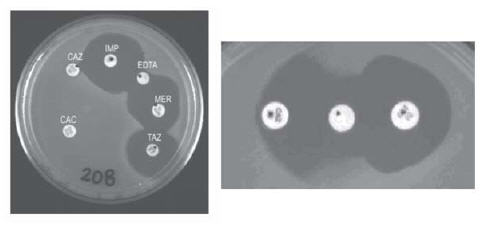
Figura 1. a) ensayo de sensibilidad a antimicrobianos por difusión en agar correspondiente al aislamiento 208 empleando el esquema sugerido por la Subcomisión de Antimicrobianos de SADEBAC. CAZ: ceftacidima, IMI: imipenem, MER: meropenem, TAZ: piperacilinatazobactama, CAC: ceftacidima-clavulánico. b) ensayo de detección fenotípica de MBL por el método de doble disco empleando EDTA (1 μmol), correspondiente al aislamiento 208.
Tabla 1.
Datos y perfil de sensibilidad de los aislamientos productores de MBL
Por otro lado, se observó asociación en la resistencia a los distintos carbapenemes ensayados, ya que los aislamientos que fueron resistentes a meropenem también lo fueron a imipenem. Sólo 7 de los 18 aislamientos resultaron resistentes a imipenem en los ensayos de sensibilidad por difusión de acuerdo con los puntos de corte del CLSI. Incluso si se tiene en cuenta lo sugerido por la Subcomisión de Antimicrobianos acerca de que los halos menores o iguales a 21 mm podrían ser considerados como sospechosos de producir MBL, sólo 11 de los 18 aislamientos quedarían incluidos con este criterio.
La sensibilidad a AZT en microorganismos resistentes a carbapenemes resulta un buen indicador de la presencia de MBL; sin embargo, la resistencia a dicho agente no descarta su presencia, dado que la resistencia a AZT podría deberse a otros mecanismos no analizados en el presente estudio (26).
Los extractos enzimáticos correspondientes a esos 18 aislamientos fueron activos frente a AMP, CAZ e IMI; la hidrólisis de este último indica claramente la presencia de una carbapenemasa.
La detección de genes codificantes de MBL por Multiplex-PCR reveló la presencia de imp en los 18 aislamientos que presentaron sinergia con EDTA, mientras que la detección de genes codificantes de MBL fue negativa en el resto de los aislamientos, lo que indica que los ensayos de detección fenotípica resultaron adecuados. La amplificación por PCR del segmento variable de integrones de clase 1 permitió evidenciar la presencia de al menos una región variable de 1,5 kb en todos los casos. Se confirmó la localización de imp en dicho fragmento por PCR anidada. La secuenciación de los amplicones de 1,5 kb permitió identificar la presencia del gen imp-13 como primer gen en cassette, seguido del gen codificante de una enzima modificadora de aminoglucósidos de tipo acetilante, aacA4 (Figura 2).
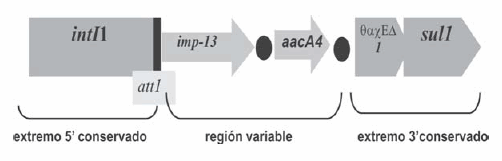
Figura 2. Entorno genético de imp-13. intl1: gen codificante de la integrasa, att1: sitio de recombinación específica, imp-13: gen codificante de la MBL IMP-13, aacA4: gen codificante de la enzima modificadora de aminoglucósidos de tipo acetilante, qacΔ1: gen de resistencia a compuestos de amonio cuaternario, sul1: gen de resistencia a sulfamidas.
Al comparar los perfiles de OMP de los aislamientos productores de IMP-13, se observó en el aislamiento resistente a imipenem la desaparición de una banda proteica de aproximadamente 46 kDa, presente en los aislamientos que no mostraron resistencia a imipenem y en la cepa control (Figura 3). De acuerdo con lo descrito en la bibliografía (24), esta banda correspondería a una porina que permite la entrada de imipenem en P. aeruginosa (OprD), aunque sería necesario contar con anticuerpos específicos para poder confirmarlo. Por otro lado, en el perfil correspondiente al aislamiento de P. aeruginosa 255 (no productor de MBL), se observa la desaparición de la misma banda proteica (Figura 3); este aislamiento presenta una CIM de IMI de 16 μg/ml. Estos resultados permitirían confirmar que la disminución en la expresión de una porina en los aislamientos productores de IMP-13 conduciría a francas resistencias a imipenem, mientras que la producción de esta enzima en presencia de patrones no alterados de porinas no sería suficiente para elevar la CIM de imipenem por encima de los puntos de corte de resistencia.
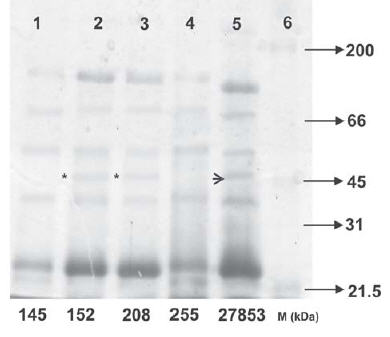
Figura 3. Perfil de OMP en un gel de poliacrilamida al 12%. Calle 1: aislamiento 145 productor de IMP-13 resistente a IMI, calle 2: aislamiento 152 productor de IMP-13 de resistencia intermedia a IMI, calle 3: aislamiento 208 productor de IMP-13 de resistencia intermedia a IMI, calle 4: aislamiento 255 no productor de MBL resistente a IMI, calle 5: cepa ATCC 27853, calle 6: marcador de peso molecular de proteínas.Los asteriscos entre las calles 1, 2 y 3 y la flecha entre las 4 y 5 indican la banda de 46 kDa.
A través de los ensayos de tipificación molecular se pretendió analizar si los aislamientos productores de IMP- 13 eran el resultado de la diseminación de un único clon o si existiría más de una línea clonal. En el dendrograma obtenido por ERIC-PCR se observaron tres grupos, E1, E2 y E3, el primero integrado por 15 aislamientos que no pudieron ser discriminados entre sí (Figura 4). Cuando se analizó el dendrograma obtenido por REP-PCR se observó la presencia de al menos 5 grupos (R1 a R5), algunos de ellos con aislamientos que presentaron entre sí más de un 90% de similitud (Figura 4). Combinando los resultados obtenidos con ambas técnicas se definieron los diferentes grupo clonales que se muestran en la Tabla 1.
Figura 4.
Tipificación de los aislamientos productores de IMP-13; a) REP-PCR, b) ERIC-PCR.
DISCUSIÓN
La prevalencia de aislamientos de P. aeruginosa productores de MBL en el Hospital "Eva Perón" durante el año 2006 fue de 14%, y éstos fueron recuperados en su mayoría de pacientes internados en la Unidad de Cuidados Intensivos (14/18 aislamientos). Pese a ser productores de MBL, sólo 7 de ellos resultaron clínicamente resistentes a carbapenemes. Considerando las recomendaciones de la Subcomisión de Antimicrobianos se logró incrementar las detecciones, pero éstas no resultaron adecuadas para todos los microorganismos ensayados, ya que 7 aislamientos productores de IMP-13 presentaron halos de inhibición de IMI mayores al punto de corte sugerido. Esto constituye un llamado de atención, el cual debería ser corroborado con un número mayor de aislamientos. Si bien dichos aislamientos fueron categorizados como sensibles, presentaron halos de inhibición entre 22 y 24 mm, los cuales se alejan de los halos observados para la población de P. aeruginosa no productora de mecanismos de resistencia (halos de imipenem de 28 a 32 mm). Por otro lado, la CIM de IMI de dichos aislamientos fue de 8 μg/ml (nivel intermedio), lo cual sugiere que la determinación de la CIM resultaría más adecuada en la detección de microorganismos que presentan mecanismos de resistencia.
Si bien ha sido descrito que la participación de mecanismos de resistencia tales como la pérdida de OprD o la hiperproducción de sistemas de eflujo elevan los niveles de CIM de carbapemes en cepas productoras de MBL (11, 17, 35), en este trabajo la alteración en dicha porina resultó necesaria para conferir resistencia fenotípica a imipenem. La producción de IMP-13 parecería requerir de la presencia simultánea de otros mecanismos. Así, los aislamientos productores de dicha enzima en los que se observó una disminución en la expresión de OprD presentaron franca resistencia a imipenem, fácilmente detectable en los ensayos de sensibilidad. Si bien esto no fue analizado en el presente estudio, es probable que la resistencia a meropenem esté asociada a la sobreexpresión de MexEF-OprN, ya que existe correlación entre la sobreexpresión de esta bomba y la disminución en la expresión de OprD (15, 29).
La ocurrencia de aislamientos productores de MBL en este hospital parece deberse no sólo a la diseminación de algunas cepas multirresistentes, sino también a la diseminación horizontal del marcador de resistencia. Considerando que algunos aislamientos productores de IMP- 13 resultan indistinguibles clonalmente por ambos métodos empleados (E1-R1) y que algunos de ellos presentan sensibilidad disminuida a carbapenemes mientras que otros presentan francas resistencias (por déficit de OprD), es posible pensar en la selección de estos últimos a partir de los primeros; por ello resultaría conveniente la inclusión de los ensayos de detección fenotípica de MBL en todas las P. aeruginosa que presenten sensibilidad disminuida a carbapenemes.
Agradecimientos: este trabajo fue parcialmente financiado con subsidios de la ANPCyT y UBACyT a Gabriel Gutkind.
1. Ambler R, Coulson A, Frère JM, Ghuysen JM, Joris B, Forsman M, et al. A standard numbering scheme for the class a b-lactamases. Biochem J 1991; 276: 269-72. [ Links ]
2. Arakawa Y, Shibata N, Shibayama K, Kurokawa H, Yagi T, Fujiwara H, et al. Convenient test for screening metallobeta-lactamase-producing gram-negative bacteria by using thiol compounds. J Clin Microbiol 2000; 38: 40-3. [ Links ]
3. Bucca R, Mastroberti M, Centron D, Jorda Vargas L, Kovensky J. Emergencia de metalo-b-lactamasas en Pseudomonas spp. provenientes de infecciones en pacientes quemados. Congreso SADEBAC 2006, 2006, p. 16560. [ Links ]
4. Cavallo J, Hocquet D, Plesiat P, Fabre R, Roussel-Delvallez M. Susceptibility of Pseudomonas aeruginosa to antimicrobials: a 2004 French multicentre hospital study. J Antimicrob Chemother 2007; 59: 1021-4. [ Links ]
5. Cornaglia G, Akova M, Amicosante G, Canton R, Cauda R, Docquier JD, et al. Metallo-beta-lactamases as emerging resistance determinants in gram-negative pathogens: open issues. Int J Antimicrob Agents 2007; 29: 380-8. [ Links ]
6. Cuirolo A, Radice M, Gutkind G. Molecular characterization of Imp-13 producing Pseudomonas aeruginosa. 46th. ICAAC, 2006, Abstract C2-415, pag. 109, San Francisco, USA. [ Links ]
7. Docquier J, Luzzaro F, Amicosante G, Toniolo A, Rossolini GM. Multidrug-resistant Pseudomonas aeruginosa producing Per-1 extended-spectrum serine-beta-lactamase and Vim-2 metallo-beta-lactamase. Emerg Infect Dis 2001; 7: 910-1. [ Links ]
8. Ellington M, Kistler J, Livermore D, Woodford N. Multiplex PCR for rapid detection of genes encoding acquired metallobeta-lactamases. J Antimicrob Chemother 2007; 59: 321-2. [ Links ]
9. Gutierrez O, Juan C, Cercenado E, Navarro F, Bouza E, Coll P, et al. Molecular epidemiology and mechanisms of carbapenem resistance in Pseudomonas aeruginosa isolates from Spanish hospitals. Antimicrob Agents Chemother 2007; 51: 4329-35. [ Links ]
10. Hancock I, Poxton I. Bacterial Cell Surface Techniques. First ed. 1988. John Wiley and Sons. [ Links ]
11. Henrichfreise B, Wiegand I, Pfister W, Wiedemann B. Resistance mechanisms of multiresistant Pseudomonas aeruginosa strains from Germany and correlation with hypermutation. Antimicrob Agents Chemother 2007; 51: 4062-70. [ Links ]
12. Hogan D, Kolter R. Why are bacteria refractory to antimicrobials? Current Opinion in Microbiology 2002; 5: 472-7. [ Links ]
13. Juan C, Macia M, Gutierrez O, Vidal C, Perez J, Oliver A. Molecular mechanisms of beta-lactam resistance mediated by AmpC hyperproduction in Pseudomonas aeruginosa clinical strains. Antimicrob Agents Chemother 2005; 49: 4733-8. [ Links ]
14. Köhler T, Michea-Hamzehpour M, Epp S, Pechere J-C. Carbapenem activities against Pseudomonas aeruginosa: respective contributions of OprD and efflux systems. Antimicrob Agents Chemother 1999; 43: 424-7. [ Links ]
15. Kolayli F, Karadenizli A, Savli H, Ergen K, Hatirnaz O, Balikci E, et al. Effect of carbapenems on the transcriptional expression of the OprD, oprM and oprN genes in Pseudomonas aeruginosa. J Med Microbiol 2004; 53: 915-20. [ Links ]
16. Livermore DM. b-lactamases in laboratory and clinical resistance. Clin Microbiol Rev 1995; 8: 557-84. [ Links ]
17. Maniati M, Ikonomidis A, Mantzana P, Daponte A, Maniatis A, Pournaras S. A highly carbapenem-resistant Pseudomonas aeruginosa isolate with a novel blavim-4/blap1b integron overexpresses two efflux pumps and lacks oprD. J Antimicrob Chemother 2007; 60: 132-5. [ Links ]
18. Maseda H, Yoneyama H, Nakae T. Assignment of the substrate-selective subunits of the MexEF-OprN multidrug efflux pump of Pseudomonas aeruginosa. Antimicrob Agents Chemother 2000; 44: 658-64. [ Links ]
19. Masuda N, Sakagawa E, Ohya S. Outer membrane proteins responsible for multiple drug resistance in Pseudomonas aeruginosa. Antimicrob Agents Chemother 1995; 39: 645-9. [ Links ]
20. Masuda N, Sakagawa E, Ohya S, Gotoh N, Tsujimoto H, Nishino T. Substrate specificities of MexAB-OprM, MexCD-OprJ, and MexXY-OprM efflux pumps in Pseudomonas aeruginosa. Antimicrob Agents Chemother 2000; 44: 3322-7. [ Links ]
21. Mesaros N, Glupczynski Y, Avrain L, Caceres N, Tulkens P, Van Bambeke F. A combined phenotypic and genotypic method for the detection of Mex efflux pumps in Pseudomonas aeruginosa. J Antimicrob Chemother 2007; 59: 378-86. [ Links ]
22. National Committee for Clinical Laboratory Standards. Methods for dilution antimicrobial susceptibility tests for bacteria that grow aerobically; approved standard. Sixth edition. M7-A6. Performance standards for antimicrobial susceptibility testing. Fourteenth informational supplement. Wayne, Pa., 2004. [ Links ]
23. National Committee for Clinical Laboratory Standards. Performance standards for antimicrobial disk susceptibility tests; approved standard - Eighth edition - M2-A8, Performance standards for antimicrobial susceptibility testing. Fourteenth informational supplement. Wayne, Pa., 2004. [ Links ]
24. Ochs M, McCusker M, Bains M, Hancock R. Negative regulation of the Pseudomonas aeruginosa outer membrane porin OprD selective for imipenem and basic amino acids. Antimicrob Agents Chemother 1999; 43: 1085-90. [ Links ]
25. Page M I. The reactivity of beta-lactams, the mechanism of catalysis and the inhibition of beta-lactamases. Curr Pharm Des 1999; 5: 895-913. [ Links ]
26. Pagniez G, Radice M, Cuirolo A. Rodríguez O, Rodríguez H, Vay C, et al. Prevalencia de metalo-b-lactamasas en Pseudomonas aeruginosa resistentes a carbapenemes en un Hospital Universitario de Buenos Aires. Rev Argent Microbiol 2006; 38: 33-7. [ Links ]
27. Pagniez G, Radice M, Amoroso A, Famiglietti A, Gutkind G. Class I integron containing a new variant of Vim-2 metallo-beta-lactamase in Pseudomonas aeruginosa. 44 th. ICAAC, 2004, Abstract C1-293, p. 65, Washington, USA. [ Links ]
28. Pasteran F, Faccone D, Petroni A, Rapoport M, Galas M, Vazquez M, et al. Novel variant [bla(vim-11)] of the metallobeta-lactamase bla(vim) family in a Ges-1 extendedspectrum-beta-lactamase-producing Pseudomonas aeruginosa clinical isolate in Argentina. Antimicrob Agents Chemother 2005; 49: 474-5. [ Links ]
29. Quale J, Bratu S, Gupta J, Landman D. Interplay of efflux system, AmpC, and oprD expression in carbapenem resistance of Pseudomonas aeruginosa clinical isolates. Antimicrob Agents Chemother 2006; 50: 1633-41. [ Links ]
30. Queenan A, Bush K. Carbapenemases: the versatile betalactamases. Clin Microbiol Rev 2007; 20: 440-58. [ Links ]
31. Quinteros M, Radice M, Famiglietti A, Marín M, Nicola F, Casellas J M, et al. Análisis de la sensibilidad a los antimicrobianos en los aislamientos de pacientes internados. Años 2004-2005. http://www.aam.org.ar/novedades/ 2004-2005.pdf 2006. [ Links ]
32. Rossi A, Lopardo H, Woloj M, Picandet A, Mariño M, Galas M, et al. Non-typhoid Salmonella spp resistant to cefotaxime. J Antimicrob Chemother 1995; 36: 697-702. [ Links ]
33. Subcomisión de Antimicrobianos, SADEBAC, AAM. Caracterización fenotípica de la resistencia a los b-lactámicos en Pseudomonas aeruginosa y Acinetobacter spp. http://www. aam.org.ar/novedades/consenso%20BNNF%20 Anexo. pdf [ Links ]
34. Syrmis M, O'Carroll M, Sloots T, Coulter C, Wainwright C, Bell S, et al. Rapid genotyping of Pseudomonas aeruginosa isolates harboured by adult and paediatric patients with cystic fibrosis using repetitive-element-based PCR assays. J Med Microbiol 2004; 53: 1089-96. [ Links ]
35. Tam V, Chang K, LaRocco M, Schilling A, McCauley K, Poole K, et al. Prevalence, mechanisms, and risk factors of carbapenem resistance in bloodstream isolates of Pseudomonas aeruginosa. Diagn Microbiol Infect Dis 2007; 58: 309-14. [ Links ]
36. Walsh T, Toleman M, Poirel L, Nordmann P. Metallo-betalactamases: The quiet before the storm? Clin Microbiol Rev 2005; 18: 306-325. [ Links ]
37. Wang C, Mi Z H. Imipenem-resistant Pseudomonas aeruginosa producing Imp-1 metallo-beta-lactamases and lacking the outer-membrane protein oprD. J Med Microbiol 2006; 55: 353-4. [ Links ]
38. Yong D, Ritchie B, Pratt R, Toleman M, Walsh T. A novel sub-group metallo-b-lactamase (MBL), Aim-1 emerges in Pseudomonas aeruginosa (Psa) from Australia. 47 th ICAAC, 2007, abstract C1-593, p. 75, Chicago, USA. [ Links ]
Recibido: 27/05/08
Aceptado: 07/10/08.














