Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO  uBio
uBio
Share
Revista argentina de microbiología
Print version ISSN 0325-7541On-line version ISSN 1851-7617
Rev. argent. microbiol. vol.41 no.1 Ciudad Autónoma de Buenos Aires Jan./Mar. 2009
Caracterización del gen de la citotoxina vacuolizante de Helicobacter pylori a partir de biopsias gástricas de pacientes residentes en Tolima, Colombia
A. M. López1, M. P. Delgado1*, C. Jaramillo1, A. Amézquita1, G. Parra2, M. M. Echeverry2
1Universidad de los Andes, Carrera 1 N° 18-10 (Of 210) Bogotá, Colombia;
2Universidad del Tolima, B. Santa Helena A.A. 546 - Ibagué, Colombia.
*Correspondencia. E-mail: mdelgado@uniandes.edu.co
RESUMEN
Helicobacter pylori es una bacteria que coloniza la mucosa gástrica de los humanos. Este microorganismo produce una citotoxina vacuolizante conocida como VacA y codificada por el gen vacA, el que se considera un factor de virulencia importante. Las cepas de H. pylori con diferentes alelos de vacA exhiben una gran variedad de fenotipos, algunos de los cuales han sido asociados con enfermedades gastroduodenales. El presente estudio pretende aportar datos sobre la prevalencia de H. pylori y de los genotipos de vacA en pacientes residentes en Tolima (Colombia), así como determinar la relación entre estos datos y el desarrollo de diferentes patologías gastroduodenales. Se incluyeron en este análisis 73 pacientes con diferentes patologías gástricas. Con el ADN total extraído de cada biopsia, se determinó la presencia de la bacteria mediante la amplificación de un fragmento específico del gen 16S ADNr. También se realizó la genotipificación del gen vacA por PCR. De las 50 muestras genotipificadas, el 52% mostró el alelo vacA s1m1, el 42% el alelo vacA s2m2, el 4% el s1m2 y el 2% los alelos s1,s2,m1,m2. Se evidenció una mayor sensibilidad en la detección de H. pylori por medio del gen vacA que por el gen 16S ADNr. En la población evaluada no se encontró asociación entre el genotipo de vacA y la presencia de las distintas patologías incluidas en este estudio.
Palabras clave: Helicobacter pylori; vacA; Detección; Tolima; Colombia.
ABSTRACT
Characterization of the Helicobacter pylori vacuolating cytotoxin gene in gastric biopsy specimens from patients living in Tolima, Colombia. Helicobacter pylori successfully colonizes the gastric niche. These bacteria produce a vacuolating cytotoxin known as VacA, which is codified by the vacA gene. This protein represents an important virulence factor. H. pylori strains have different vacA alleles, which show a variety of phenotypes that have been associated with gastrointestinal diseases. The aim of this study was to generate data about the prevalence of H. pylori and the vacA genotype in Tolima (Colombia) residents, and to evaluate if there exists a relationship between these data and the development of different gastrointestinal pathologies. Seventy three patients with different pathologies were included. The DNA extracted from biopsy specimens was analyzed and the presence of bacteria was determined by amplifying a fragment of the 16 rDNA gene. The vacA genotype was also determined by PCR. Fifty-two percent out of the 50 genotyped samples showed vacA s1m1 allele, 42% vacA s2m2, 4% s1m2, and 2% s1,s2,m1,m2. A higher sensitivity for the detection of H. pylori was evidenced by amplifying the vacA gene rather than the 16S rDNA gene. No association was found between the vacA genotype and the gastrointestinal diseases included in the study.
Key words: Helicobacter pylori; vacA; Detection; Tolima; Colombia.
INTRODUCCIÓN
La infección por Helicobacter pylori ocurre en todo el mundo, pero su prevalencia varía en gran medida entre países y entre poblaciones de un mismo país (25, 33). La prevalencia en los adultos de mediana edad es del 80% en los países en vías de desarrollo, mientras que en los países desarrollados es del 20 al 50% (25, 33).
La mayoría de las personas, una vez infectadas, pueden permanecer sin desarrollar ningún tipo de sintomatología. No obstante, el curso típico de la enfermedad en pacientes infectados comienza con una gastritis crónica superficial que, eventualmente, progresa a gastritis atrófica. Este progreso parece ser un evento clave en la cascada celular que resulta, finalmente, en el desarrollo de carcinoma gástrico (21, 26).
H. pylori posee la habilidad de producir la citotoxina vacuolizante VacA, que es el factor de virulencia responsable de la formación de vacuolas en las células epiteliales gástricas (2, 6, 12, 15, 16). La proteína VacA, de aproximadamente 95 kDa, es codificada por el gen vacA y produce diferentes efectos que pueden contribuir a la persistencia de H. pylori en el nicho gástrico. Esta citotoxina forma poros en las membranas de las células epiteliales y permite la salida de aniones y urea; esto es importante pues la hidrólisis de la urea, catalizada por la ureasa de H. pylori, protege a la bacteria de la acidez gástrica. Esta proteína también favorece la labilidad de las uniones epiteliales, lo que hace posible el paso de los nutrientes a través de la barrera mucosa en beneficio de la bacteria (6, 10, 24, 33). Asimismo, se ha sugerido que VacA tiene efecto en la supresión de la respuesta inmune específica mediante: i) la inhibición de la maduración de los fagosomas en los macrófagos, ii) la inhibición selectiva de la presentación de los antígenos a las células T, y iii) el bloqueo de la proliferación de dichas células (6, 7, 10, 19, 20, 31, 33, 34), entre otros efectos. Igualmente, se ha visto que induce cambios en el citoesqueleto, apoptosis (6, 10, 13, 20, 33), y supresión de la proliferación y migración de las células epiteliales (6, 10, 20, 33). Las cepas de H. pylori con diferentes alelos de vacA exhiben una gran variedad de fenotipos, probablemente relacionados con diversas enfermedades gastroduodenales. Este gen tiene dos regiones diferenciadas, una de 0,5 kb que corresponde a la región de una secuencia señal, denominada vacA s, y otra región de 0,73 kb que corresponde a la región media, conocida como vacA m (6, 10, 33).
La región señal del gen vacA codifica para un péptido señal y para el extremo amino de la toxina VacA procesada. Respecto de esta región se conocen dos tipos: s1 y s2. El tipo s1 es completamente activo, mientras que el tipo s2 tiene un extremo amino corto que bloquea la formación de vacuolas y disminuye la formación de poros en las membranas eucariotas (6, 10, 12, 17, 18, 33). Las cepas vacA s2 son raramente aisladas de pacientes con úlceras pépticas o adenocarcinoma gástrico (6, 10, 12, 17, 33).
La región media de vacA codifica para parte del dominio de unión de la toxina a la célula. Sobre la base de la secuencia de nucleótidos de esta región, las cepas se clasifican en dos familias de alelos: el alelo tipo m1 para las cepas de H. pylori que tienen actividad citotóxica y el alelo m2 para las cepas que carecen de ésta (3).
Las formas s1/m2 interactúan y vacuolizan un rango menor de células e inducen un menor daño que las formas s1/m1. Sin embargo, también actúan eficientemente en la formación de poros en las membranas e incrementan la permeabilidad celular (6, 10, 12, 14, 33). En general, se ha visto que las cepas vacA s1/m1 están asociadas con patologías graves, como el carcinoma gástrico (6, 10, 12, 33). No obstante, los datos son contradictorios, por lo que resulta de gran utilidad realizar estudios que amplíen la información, no sólo a una escala regional, sino también mundial, para tratar de desarrollar sistemas de reconocimiento de los grupos de alto riesgo dentro de poblaciones con altos índices de infección por H. pylori, como la colombiana.
En este estudio se pretende: i) aportar datos sobre la prevalencia de H. pylori y sobre el genotipo de vacA en un grupo de pacientes con diferentes patologías gástricas; y ii) investigar la relación entre la presencia de la bacteria o el genotipo de vacA con las patologías gástricas identificadas.
MATERIALES Y MÉTODOS
Pacientes
En el estudio se incluyeron 73 pacientes, 44 mujeres y 29 hombres, con una edad promedio de 55 años (rango, 19 a 79 años), que asistieron a la unidad de endoscopia de la Clínica Manuel Elkin Patarroyo del Instituto de Seguros Sociales (ISS) y a un consultorio privado de gastroenterología en Ibagué, Colombia. A todos los individuos participantes, quienes consintieron de forma escrita su participación en este proyecto, se les había indicado la realización de la endoscopia alta como parte del estudio de un cuadro de dispepsia. Las muestras consistieron, en promedio, de tres especímenes de biopsia del tejido gástrico para el análisis histopatológico y de un fragmento de tejido del antro gástrico que fue sometido a los análisis moleculares.
Criterios de exclusión
Se excluyeron los pacientes con cáncer que habían recibido radioterapia o quimioterapia durante los seis meses previos a la toma de la muestra, los pacientes con enfermedades cardiovasculares y respiratorias, y aquellos que recibieron tratamiento con antiácidos doce horas antes del procedimiento, con bloqueadores de la bomba de protones e H2 quince días antes de la endoscopia o con antibióticos durante los tres meses previos a la toma de muestra.
Coloraciones
La coloración de hematoxilina-eosina fue empleada para la realización del análisis histológico, mientras que la coloración de Giemsa fue utilizada para la detección de H. pylori en el tejido.
Extracción del ADN total
La extracción de ADN a partir de un fragmento de tejido del antro gástrico se realizó mediante el equipo comercial AquaPure Genomic DNA (BIORAD®, EE. UU. ), siguiendo las recomendaciones del fabricante.
Detección molecular de H. pylori
La detección de H. pylori se realizó mediante la amplificación de un fragmento del gen 16S ADNr específico de la especie, utilizando los cebadores descritos por Thoreson AC et al. (35) (Tabla 1). La reacción se realizó en un volumen final de 25 μl, que contenía: 50 mM de KCl; 20 mM de Tris HCl pH 8,4; 2,5 mM de MgCl2; 0,2 mM de cada desoxinucleótido trifosfato; 1 pm/μl de cada cebador (ACT-1 y ACT-2); 1,25 U de Taq polimerasa (InvitrogenTM, EE. UU.); y aproximadamente 90 ng/μl de ADN genómico. El programa de amplificación incluyó un primer paso de desnaturalización a 94 °C durante 4 min, seguido de 38 ciclos (94 °C/1 min, 52 °C/1 min y 72 °C/1 min); con una extensión final a 72 °C durante 10 min.
Para descartar la posible inhibición de la reacción, las muestras que resultaron negativas en la PCR para el gen 16S ADNr o para el gen vacA, o para ambos, se analizaron mediante la amplificación de un fragmento del gen de la β-globina humana (Tabla 1) (32) y de una región del gen IL-1B humano (Tabla 1) (28). Para la amplificación de la región del gen de la β-globina, la reacción se llevó a cabo en un volumen final de 25 μl, y la mezcla de reacción contenía: 50 mM de KCl; 20 mM de Tris HCl pH 8,4; 3,0 mM de MgCl2; 0,2 mM de cada desoxinucleótido trifosfato; 1 pm/μl de cada cebador; 1,25 U de Taq polimerasa; y alrededor de 60 ng/μl de ADN genómico. El programa de amplificación incluyó una desnaturalización inicial a 94 °C durante 5 min, seguida de 35 ciclos (94 °C/30 s, 57,5 °C/45 s y 72 °C/45 s) y, finalmente, de un paso de extensión a 72 °C durante 5 min.
Tabla 1. Cebadores usados en el estudio
Para la amplificación de un fragmento de la región del gen IL1B, la reacción se realizó en un volumen final de 25 μl, que contenía: 50 mM de KCl; 20 mM de Tris HCl pH 8,4; 2.5 mM de MgCl2; 0,2 mM de cada desoxinucleótido trifosfato; 1 pm/μl de cada iniciador; 1,25 U de Taq polimerasa; y 60 ng/μl de ADN genómico. El programa de amplificación consistió en una desnaturalización inicial a 94 °C durante 5 min, seguida de 35 ciclos (94 °C/1 min, 55 °C/1 min y 72 °C/1 min) y, finalmente, de una extensión a 72 °C durante 7 min.
Tipificación del gen vacA de H. pylori
La PCR para la genotipificación del gen vacA incluyó a los cebadores VA1-F y VA1-R para identificar a los alelos s1 y s2 de la región señal (Tabla 1) (3, 37), y a los cebadores VAG-F y VAG-R para diferenciar a los alelos m1 y m2 de la región media por diferencias en el tamaño del amplicón (Tabla 1) (3, 37). En ambos casos la reacción se realizó en un volumen final de 25 μl, que contenía: 50 mM de KCl; 20 mM de Tris HCl pH 8,4; 3,0 mM de MgCl2; 0,2 mM de cada desoxinucleótido trifosfato; 1 pm/μl de la mezcla de cebadores; 1,25 U de Taq polimerasa; y 60 ng/ μl de ADN genómico. El programa de amplificación consistió en una desnaturalización inicial a 94 °C durante 4 min, seguida por 35 ciclos [95 °C/1 min, 59 °C/1 min (región s) o 52 °C/1 min (región m), y 72 °C/1 min]. Los productos fueron visualizados mediante UV en geles de agarosa al 2% para la región señal y al 1,5% para la región media.
Diseño del estudio y análisis estadístico
La presente investigación, que corresponde a un estudio observacional de tipo descriptivo y de corte transversal, incluyó la determinación de la sensibilidad (S), la especificidad (E), el valor predictivo positivo (VPP) y el valor predictivo negativo (VPN) de las técnicas moleculares, usando la histología como técnica de referencia (23). El índice Kappa fue calculado con el fin de evaluar la concordancia entre los dos métodos empleados para la detección de H. pylori (examen histopatológico y PCR).
Por medio del programa SPSS versión 14,0, se realizaron modelos de regresión multinomial y de regresión ordinal con el fin de detectar la posible asociación entre la presencia de H. pylori, el genotipo de vacA identificado, la edad o el sexo de los pacientes y el desarrollo de las diferentes patologías incluidas en el estudio. En ambos casos se determinó que la diferencia era significativa si p<0,05.
Aspectos éticos
El presente estudio fue aprobado por los Comités de Ética de la Universidad de los Andes y de la Universidad del Tolima.
RESULTADOS
El análisis histológico de las muestras de los 73 pacientes mostró que el 76,7% padecía gastritis no atrófica, el 5,5% gastritis atrófica, el 8,2% metaplasia y el 2,7% carcinoma gástrico. En el 6,8% de los pacientes no se detectó patología.
La prevalencia de infección con H. pylori fue de 58,9% (43/73) por medio de la PCR para la detección del fragmento específico de la especie del gen 16S ADNr, y de 67,1% (49/73) cuando la detección se realizó aplicando el análisis histológico. El índice Kappa calculado fue de 0,76, lo que indica una buena concordancia entre ambos métodos. Para esta técnica se obtuvieron valores de S = 86%, E = 96% y VPP y VPN de 2 y 77%, respectivamente.
Con respecto a la caracterización del gen vacA, se genotipificaron 50 de las 73 muestras (Tabla 2). El 52% eran vacA s1m1, el 42% vacA s2m2, el 4% s1m2 y el 2% s1, s2, m1, m2. Este último resultado da indicios de infección con múltiples cepas de H. pylori, por lo que esta muestra se excluyó al realizar los modelos de regresión. La concordancia fue excelente (0,88) entre el método de PCR usado para la detección del gen vacA y el análisis histológico. La detección del gen vacA mostró S = 98%, E = 92%, y VPP = VPN = 96%.
Tabla 2. Frecuencia de genotipos de vacA (total de muestras analizadas, n = 50)
En aquellas muestras que resultaron negativas al realizar la detección molecular de H. pylori se amplificó exitosamente un fragmento del gen de la β-globina y de la interleuquina 1 β-humana.
La Figura 1 muestra las combinaciones de los resultados posibles (histología, detección del gen 16S ADNr y genotipificación de vacA) y la frecuencia de las muestras obtenidas en cada una de las categorías. Es posible apreciar que no siempre hubo concordancia entre los tres métodos. La regresión ordinal no mostró asociación entre el genotipo de vacA (s1m1 > s2m1 > s2m2) o la presencia de H. pylori (presente > ausente) y el desarrollo de diferentes patologías gastroduodenales (cáncer > metaplasia > gastritis atrófica > gastritis no atrófica), para un p > 0,05 (datos no mostrados). Al realizar la regresión multinomial para evaluar el efecto de cada una de las variables analizadas en forma independiente, tampoco se encontró asociación entre éstas (Figura 2).
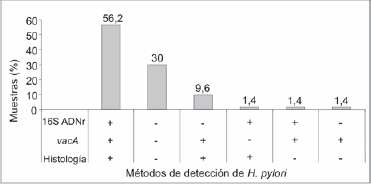
Figura 1. Comparación de tres métodos de detección de H. pylori. La presencia de H. pylori fue evaluada mediante la amplificación de un fragmento del gen 16S ADNr, la genotipificación del gen vacA y por histología. La figura muestra las diferentes combinaciones que se obtuvieron y el porcentaje de muestras que correspondió a cada categoría.
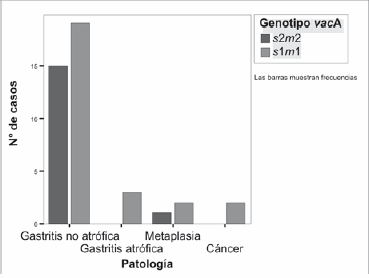
Figura 2. Regresión multinomial de la relación entre los genotipos de vacA y el desarrollo de diferentes patologías gastroduodenales. El genotipo vacA s2m1 no fue considerado debido al bajo número de pacientes en esta categoría (p > 0,05).
DISCUSIÓN
En 2003, Bravo et al. (8) analizaron los estudios histológicos de pacientes de Ibagué, Tolima, y mostraron una prevalencia de infección por H. pylori de 59,5%. En el presente estudio, realizado con pacientes residentes en Tolima, se obtuvo una prevalencia de 67,1% por medio del análisis histológico y de 58,9% mediante la detección del gen 16S ADNr, específico para la especie, de modo que esta última es más cercana a la comunicada en la publicación citada. La discrepancia encontrada entre los dos métodos usados puede deberse al azar, según lo indica el valor del índice Kappa obtenido.
Teniendo en cuenta que el gen vacA está presente en todas las cepas de H. pylori y que no se han comunicado homólogos de éste en otras especies bacterianas (6), surgió la idea de realizar la amplificación de la región señal y la región media en todas las muestras, aunque éstas fueran negativas por histopatología o por la detección del gen 16S ADNr (Figura 1). Como era de esperarse, en la mayoría de las muestras el resultado obtenido por los tres métodos fue concordante.
Los datos de la Figura 1 sugieren que la detección de H. pylori por medio del gen vacA puede ser más sensible que mediante el gen 16S ADNr. Esto también se ve reflejado en el hecho que el índice Kappa muestra una mayor concordancia entre la detección por vacA y por histología, que entre ésta última y la detección por el gen 16S ADNr. Estos resultados pueden deberse a diferencias en la sensibilidad de las parejas de los cebadores usadas para amplificar cada uno de los fragmentos blanco. La diferencia en la sensibilidad de diversas parejas de cebadores ha sido descrita por varios autores en diferentes organismos (36).
Al comparar la sensibilidad de los dos métodos moleculares usados para la detección de H. pylori (16S ADNr y vacA), se observa que ambos son sensibles. Sin embargo, al tener en cuenta el VPP, se hace evidente que existe una mayor probabilidad de que los resultados positivos obtenidos con vacA estén identificando a aquellas biopsias que resultaron ser positivas por histología, mientras que la probabilidad de identificar a la bacteria en estas muestras por medio de 16S ADNr resulta, por el contrario, baja. En términos de especificidad, los dos métodos moleculares presentan valores bastante altos y ambos presentan VPN significativos, lo que indica que existe una alta probabilidad de que por medio de estas técnicas se obtengan resultados negativos en aquellas muestras negativas para H. pylori por histología. En conjunto, estos resultados muestran que la detección por medio de vacA da resultados más comparables con los obtenidos por histología que los obtenidos usando como blanco el gen 16S ADNr.
Un estudio realizado en Alemania con 30 cepas informó que no se logró amplificar una de las muestras con los cebadores específicos para la región media. Después de analizar la región media de esta cepa, el estudio mostró una diferencia de 9,3% de nucleótidos con las cepas m2, y la denominó m3 (10). Es posible que la infección en el paciente cuya muestra resultó ser positiva para la presencia de la bacteria tanto por histología como por el 16S ADNr, pero que no produjo amplificación en ninguna de las regiones del gen vacA, esté causada por una cepa que presenta un genotipo de vacA que no puede ser amplificado por las parejas de cebadores utilizadas en este estudio. No obstante, sería necesario comprobar esta hipótesis por secuenciación.
La muestra en la que se amplificó el fragmento del gen 16S ADNr y se tipificó el gen vacA, pero en la cual no se observaron bacilos compatibles con H. pylori por medio de histología, era de un paciente con cáncer gástrico. Se ha descrito que la colonización de H. pylori en regiones atróficas del estómago es escasa y que casi nunca se da en tejidos con metaplasia (6). Además, si se tiene en cuenta que para los análisis moleculares se tomó un fragmento del antro gástrico (región colonizada preferencialmente por H. pylori) y que para los análisis histopatológicos se tomaron fragmentos del tejido en donde se observaba lesión, se puede inferir que es probable que el microorganismo se encontrara en el antro gástrico y no en la región con la lesión. La muestra en la que se tipificó el gen vacA pero que no mostró amplificación del fragmento del gen 16S ADNr o presencia de bacilos tipo H. pylori en el tejido correspondió también a un paciente con cáncer. En ésta pudo haber ocurrido lo mismo que en el caso anterior pero, teniendo en cuenta que hay indicios de que la detección del gen vacA es más sensible, es posible que se haya podido identificar este gen y no el gen 16S ADNr.
H. pylori coloniza la mucosa gástrica humana y puede permanecer allí durante décadas sin que se presente ningún síntoma (6, 10, 21-23). Por otro lado, su presencia puede causar desde una inflamación leve hasta cáncer. Se ha comunicado que este microorganismo es el agente etiológico de 70-80% de los casos de gastritis crónica y que puede ser responsable de hasta el 40% de los casos de cáncer gástrico a nivel mundial (9, 10). Esta bacteria fue clasificada como carcinógeno de clase I por la Agencia Internacional para Investigación en Cáncer en 1994 (10, 29). No obstante, en este estudio no se encontró asociación entre la presencia de la bacteria, determinada por la detección del gen 16S ADNr o por la presencia del gen vacA, y el desarrollo de gastritis no atrófica, atrófica, metaplasia o cáncer.
La frecuencia de los alelos s1, s2, m1 y m2 encontrada en este trabajo fue bastante similar entre sí y diferente, a su vez, a la encontrada en otros estudios realizados con cepas colombianas, en los que se comunica una mayor frecuencia de los alelos s1 y m1 comparados con los alelos s2, m2 (11, 29). Esto puede deberse a que en este caso el número de pacientes con enfermedades gástricas graves fue menor. Con respecto a la tipificación del gen vacA, se observó una mayor asociación entre los alelos s1 y m1 y también entre los alelos s2 y m2, lo cual concuerda con lo mostrado en otros estudios que han genotipificado cepas colombianas y de otras regiones del mundo (3, 5, 10, 11, 29).
Se determinó que una de las muestras presentaba los tipos s1, s2, m1 y m2. Es probable que este paciente tenga una infección múltiple; sin embargo, con el método usado en esta investigación (la tipificación directa del ADN extraído de la biopsia) no es posible determinar el genotipo específico de las cepas presentes en el paciente. Esta falencia puede ser superada al cultivar y aislar la bacteria. No obstante, es necesario tener en cuenta que este agente es de difícil cultivo y potencialmente peligroso (25, 30).
Al realizar el análisis estadístico para hallar una posible asociación entre la presencia de la bacteria o el genotipo de vacA con el desarrollo de patología, no se encontró una asociación significativa (Figura 2). Los valores de p > 0,05 obtenidos pueden deberse a que el número de pacientes en varias de las categorías no fue suficiente teniendo en cuenta el número de variables analizadas. Otras investigaciones realizadas con cepas colombianas sugieren que vacA no parece ser útil como marcador de virulencia, debido a que un número elevado de individuos con cepas vacA s1m1 padecen diferentes patologías gastroduodenales, incluso patologías leves como gastritis (11, 37). Esto concuerda con los hallazgos aquí referidos (Figura 2).
El presente estudio aportó datos sobre la prevalencia de H. pylori y los genotipos de vacA presentes en una muestra de individuos con trastornos gástricos. Éstos muestran que vacA sería un buen blanco para la detección de H. pylori a partir del ADN extraído de biopsias gástricas. Por otra parte, con estos resultados no fue posible demostrar una asociación entre la presencia de H. pylori o los distintos genotipos de vacA con el desarrollo de diversas patologías gástricas, como la gastritis no atrófica y atrófica, la metaplasia y el cáncer, para esta muestra de pacientes residentes en Tolima, Colombia.
Agradecimientos: a los participantes del proyecto "Tipificación del gen de la citotoxina vacuolizante de Helicobacter pylori por medio de la reacción en cadena de la polimerasa (2005- 2007)"; a los miembros del Laboratorio de Diagnóstico Molecular y Bioinformática de la Universidad de los Andes, Facultad de Ciencias, Bogotá, Colombia; a los miembros de la Universidad del Tolima y del Grupo de Citogenética, Filogenia y Evolución de Poblaciones, Ibagué, Colombia; y al Fondo de Investigaciones de la Facultad de Ciencias de la Universidad de los Andes, Bogotá, Colombia.
1. Alm RA, Ling LS, Moir DT, King BL, Brown ED, Doig PC, et al. Genomic-sequence comparison of two unrelated isolates of the human gastric pathogen Helicobacter pylori. Nature 1999, 397: 176-80. [ Links ]
2. Amieva MR, Salama NR, Tompkins LS, Falkow S. Helicobacter pylori enter and survive within multivesicular vacuoles of epithelial cells. Cell Microbiol 2002; 4: 677-90. [ Links ]
3. Atherton JC, Cao P, Peek RM Jr, Tummuru MKR, Blaser MJ, Cover TL. Mosaicism in vacuolating cytotoxin alleles of Helicobacter pylori. J Biol Chem 1995; 270: 17771-7. [ Links ]
4. Blanchard TG, Nedrud JG, Czinn SJ. Local and systemic antibody responses in humans with Helicobacter pylori infection. Can J Gastroenterol 1999; 13: 591-4. [ Links ]
5. Blaser MJ, Parsonnet J. Parasitism by the "slow" bacterium Helicobacter pylori leads to altered gastric homeostasis and neoplasia. J Clin Invest 1994; 94: 4-8. [ Links ]
6. Blaser MJ, Atherton JC. Helicobacter pylori persistence: biology and disease. J Clin Invest 2004; 113: 321-33. [ Links ]
7. Boncristiano M, Paccani SR, Barone S, Ulivieri C, Patrussi L, Ilver D, et al. The Helicobacter pylori vacuolating toxin inhibits T cell activation by two independent mechanisms. J Exp Med 2003; 198: 1887-97. [ Links ]
8. Bravo LE, Cortés A, Carrascal E, Jaramillo R, García LS. Helicobacter pylori: patología y prevalencia en biopsias gástricas en Colombia. Colombia Med 2003; 34: 124 -31. [ Links ]
9. Cassaro M, Rugge M, Gutiérrez O, Leandro G, Graham DY, Genta M. Topographic patterns of intestinal metaplasia and gastric cancer. Am J Gastroenterol 2001; 95: 1431-8. [ Links ]
10. Castillo-Rojas G, Mazari-Miriart M, López-Vidal Y. Helicobacter pylori: focus on CagA and VacA major virulence factors. Salud Pública de México 2004; 538-48. [ Links ]
11. Cittelly DM, Huertas MG, Martínez JD, Oliveros R, Posso H, Bravo MM, et al. Los genotipos de Helicobacter pylori en gastritis no atrófica difieren de los encontrados en úlcera péptica, lesiones premalignas y cáncer gástrico en Colombia. Rev Med Chil 2002; 130: 143-51. [ Links ]
12. Cover TL, Blaser MJ. Purification and characterization of the vacuolating toxin from Helicobacter pylori. J Biol Chem 1992; 267: 10570-5. [ Links ]
13. Cover TL, Krishna US, Israel DA, Peek RM Jr. Induction of gastric epithelial cell apoptosis by Helicobacter pylori vacuolating cytotoxin. Cancer Res 2003; 63: 951-7. [ Links ]
14. Czajkowsky DM, Iwamoto H, Cover TL, Shao Z. The vacuolating toxin from Helicobacter pylori forms hexameric pores in lipid bilayers at low pH. Proc Natl Acad Sci USA 1999; 96: 2001-6. [ Links ]
15. de Bernard M, Arico B, Papini E, Rizzuto R, Grandi G, Rappuoli R, et al. Helicobacter pylori toxin VacA induces vacuole formation by acting in the cell cytosol. Mol Microbiol 1997; 26: 665-74. [ Links ]
16. de Bernard M, Burroni D, Papini E, Rappuoli R, Telford J, Montecucco C. Identification of the Helicobacter pylori VacA toxin domain active in the cell cytosol. Infect Immun 1998; 66: 6014-6. [ Links ]
17. Fujikawa A, Shirasaka D, Yamamoto S, Ota H, Yahiro K, Fukada M, et al. Mice deficient in protein tyrosine phosphatase receptor type Z are resistant to gastric ulcer induction by VacA of Helicobacter pylori. Nat Genet 2003; 33: 375-81. [ Links ]
18. Galmiche A, Rassow J, Doye A, Cagnol S, Chambard JC, Contamin S, et al. The N-terminal 34 kDa fragment of Helicobacter pylori vacuolating cytotoxin targets mitochondria and induces cytochrome C release. EMBO J 2000; 19: 6361-70. [ Links ]
19. Gebert B, Fischer W, Weiss E, Hoffmann R, Haas R. Helicobacter pylori vacuolating cytotoxin inhibits T lymphocyte activation. Science 2003; 301: 1099-102. [ Links ]
20. Gebert B, Fischer W, Haas R. The Helicobacter pylori vacuolating cytotoxin: from cellular vacuolation to immunosuppressive activities. Rev Physiol Biochem Pharmacol 2004; 152: 205-20. [ Links ]
21. Hardin FJ, Wright RA. Helicobacter pylori: review and update. Hosp Physician 2002; 23-31. [ Links ]
22. Lee A, Fox J, Hazell S. Pathogenicity of Helicobacter pylori: a perspective. Infect Immun 1993; 1601-10. [ Links ]
23. Letley DP, Rhead JL, Twells RJ, Dove B, Atherton JC. Determinants of non-toxicity in the gastric pathogen Helicobacter pylori. J Biol Chem 2003; 278: 26734-41. [ Links ]
24. Li Y, Wandinger-Ness A, Goldenring JR, Cover TL. Clustering and redistribution of late endocytic compartments in response to Helicobacter pylori vacuolating toxin. Mol Biol Cell 2004; 15: 1946-59. [ Links ]
25. Logan RPH, Walker MM. Epidemiology and diagnosis of Helicobacter pylori infection. Br Med J 2001; 232: 920-2. [ Links ]
26. McNulty CAM, Wyatt JI. Helicobacter pylori. J Clin Pathol 1999; 52: 338-44. [ Links ]
27. Morales-Espinosa R, Castillo G, González-Valencia G, Ponce de León S, Cravioto A, Atherton JC, et al. Colonization of mexican patients by multiple Helicobacter pylori strains with different vacA and cagA genotypes. J Clin Microbiol 1999; 37: 3001-4. [ Links ]
28. Murillo LS, Land JA, Pleijster J, Bruggeman CA, Peña AS, Morré SA. Interleukin-1B (IL-1B) and interleukin-1 receptor antagonist (IL-1RN) gene polymorphisms are not associated with tubal pathology and Chlamydia trachomatis-related tubal factor subfertility. Hum Reprod 2003; 18: 2309-14. [ Links ]
29. Quiroga AJ, Cittelly DM, Bravo MM. Frecuencia de los genotipos babA2, oipA y cagE de Helicobacter pylori en pacientes con enfermedades gastroduodenales. Biomédica 2005; 25: 325-34. [ Links ]
30. Rivas-Traverso F, Hernández F. Helicobacter pylori: factores de virulencia, patología y diagnóstico. Rev Biomed 2000; 11: 187-205. [ Links ]
31. Rittig MG, Shaw B, Letley DP, Thomas RJ, Argent RH, Atherton JC. Helicobacter pylori-induced homotypic phagosome fusion in human monocytes is independent of the bacterial vacA and cag status. Cell Microbiol 2003; 5: 887-99. [ Links ]
32. Saiki RK, Scharf S, Faloona F, Mullis KB, Horn GT, Erlich HA, et al. Enzymatic amplification of beta-globin genomic sequences and restriction site analysis for diagnosis of sickle cell anemia. Science 1985; 230: 1350-4. [ Links ]
33. Suerbaum S, Michetti P. Helicobacter pylori Infection. N Engl J Med 2002; 347: 1175-86. [ Links ]
34. Telford JL, Covacci A, Rappuoli R, Chiara P. Immunobiology of Helicobacter pylori infection. Curr Opin Immunol 1997; 9: 498-503. [ Links ]
35. Thoreson ACE, Borre MB, Andersen LP, Elsborg S, Holck S, Conway P, et al. Development of a PCR-based technique for detection of Helicobacter pylori. Immunol Med Microbiol 1995; 10: 325-34. [ Links ]
36. Wilson IG. Inhibition and facilitation of nucleic acid amplification. Appl Environ Microbiol 1997; 63: 3741-51. [ Links ]
37. Yamaoka Y, Kodama T, Kita M, Imanishi J, Kashima K, Graham DY. Relationship of vacA genotypes of Helicobacter pylori to cagA status, cytotoxin production, and clinical outcome. Helicobacter 1998; 3: 241-53. [ Links ]
Recibido: 08/04/08
Aceptado: 18/12/08.














