Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO  uBio
uBio
Share
Revista argentina de microbiología
Print version ISSN 0325-7541On-line version ISSN 1851-7617
Rev. argent. microbiol. vol.41 no.1 Ciudad Autónoma de Buenos Aires Jan./Mar. 2009
Caracterización de aislamientos de Vibrio cholerae no-O1, no-O139 asociados a cuadros de diarrea
S. González Fraga1, 4,*, A. Villagra de Trejo2, M. Pichel1, S. Figueroa2, G. Merletti2, M. I. Caffer1, M. Cecilia de Castillo3, N. Binsztein1
1Departamento Bacteriología, INEI - ANLIS "Dr. Carlos G. Malbrán", Av. Vélez Sarsfield 563 (1281) Ciudad Autónoma de Buenos Aires;
2Hospital del Niño Jesús, Pasaje Hungría 700 (4000) San Miguel de Tucumán;
3Universidad Nacional de Tucumán (UNT), Ayacucho 491 (4000) San Miguel de Tucumán;
4Consejo Nacional de Investigaciones Científicas y Técnicas, Argentina.
*Correspondencia. E-mail: sgfraga@anlis.gov.ar
RESUMEN
La infección por Vibrio cholerae, el agente causal del cólera, se trasmite al hombre por ingestión de agua y alimentos contaminados. Aunque son los serogrupos O1 y O139 los que habitualmente se asocian al cólera epidémico, los aislamientos de otros serogrupos también son causales de gastroenteritis e infecciones extra-intestinales. Durante el período 2003-2005, se investigó la presencia de V. cholerae en la materia fecal de niños con diarrea atendidos en el Hospital del Niño Jesús, Tucumán. Se recuperaron 34 aislamientos de V. cholerae no-O1, no-O139. Se determinaron sus perfiles de virulencia por PCR, la sensibilidad a los antimicrobianos y la diversidad genética por electroforesis en campo pulsado. Se obtuvieron ocho perfiles de virulencia, aunque ningún aislamiento fue positivo para la toxina colérica ni para la toxina termoestable. Cuatro aislamientos fueron positivos para el sistema de secreción de tipo tres. El 17,6% de los aislamientos fueron resistentes o de sensibilidad intermedia a ampicilina y el 5,9% fueron resistentes a trimetoprima-sulfametoxazol. Los aislamientos resultaron muy diversos: se hallaron 27 patrones distintos en 29 aislamientos tipificables por electroforesis en campo pulsado. A pesar de su baja incidencia, V. cholerae continúa siendo un agente causal de diarrea en niños, los que se ven afectados por una amplia variedad de cepas circulantes.
Palabras clave: V. cholerae no-O1; no-O139; Factores de virulencia; PFGE.
ABSTRACT
Characterization of Vibrio cholerae non-O1 and non-O139 isolates associated with diarrhea. Vibrio cholerae, etiologic agent of cholera, is transmitted to humans by ingestion of contaminated food or water. Even though serogroups O1 and O139 are the ones usually associated to epidemic cholera, isolates from other serogroups also cause gastroenteritis and extraintestinal infections. During the period 2003-2005, presence of V. cholerae in stools was investigated in children with diarrhea that seaked assistance at the Niño Jesús Hospital in Tucumán. Thirty four isolates of V. cholerae non-O1, non-O139 were recovered. We characterized the isolates studying its virulence factors by PCR, antimicrobial susceptibility patterns and genetic diversity by pulsed-field gel electrophoresis. Eight virulence patterns were obtained although no isolate was positive for the cholera toxin or the thermostable toxin. Four isolates were positive for the type three secretion system. The 17.6% of the isolates were resistant or intermediate to ampicillin and 5.9% were resistant to trimethoprim-sulfamethoxazole. By SfiI-PFGE, all isolates were genetically very diverse, as 27 different patterns were identified in 29 typeable isolates by pulsed-field gel electrophoresis. Although it has a low incidence, V. cholerae continues to be a causative agent of diarrhea in children, who are affected by a variety of circulating strains of V. cholerae non-O1, non-O139.
Key words: V. cholerae non-O1 and non-O139 isolates; Virulence factors; PFGE.
INTRODUCCIÓN
Vibrio cholerae es el agente causal del cólera, una enfermedad infecciosa conocida desde la antigüedad. Es un habitante autóctono de los ecosistemas acuáticos, los que a su vez actúan como reservorios y vehículos de transmisión de este patógeno (8). La infección por V. cholerae es adquirida por la ingestión de agua o alimentos contaminados. Sus manifestaciones clínicas incluyen una profusa diarrea acuosa, vómitos y deshidratación, con gran pérdida de sales y agua. V. cholerae ha sido clasificado en numerosos serogrupos, de los cuales sólo el O1 y, más recientemente, el O139 (20) han causado epidemias de cólera. Sin embargo, cepas pertenecientes a los serogrupos no-O1, no-O139 han sido aisladas de pacientes con síntomas que van desde una diarrea leve hasta la deshidratación grave, semejante a la observada en el cólera (23, 26). Por otro lado, cepas de estos serogrupos se han asociado a sepsis y a cuadros extraintestinales, como también a infecciones de heridas superficiales.
En 1991 el cólera reemergió en Perú (22), luego de aproximadamente 100 años de ausencia de casos en Sudamérica. Esta epidemia fue causada por V. cholerae O1 biotipo El Tor. En Argentina ocurrieron siete brotes de cólera entre 1992 y 1998. En el país se estableció un activo programa de vigilancia que incluyó la creación de la Red de Laboratorios de Diarreas y Patógenos Bacterianos de Transmisión Alimentaria. En el marco de este programa y también a través de estudios de monitoreo en el medio ambiente, desde 1992 hasta 2007 se identificaron y caracterizaron alrededor de 3.500 aislamientos de V. cholerae O1 y no-O1 de origen humano y ambiental provenientes de distintas regiones del país (10, 17, 26). V. cholerae O1 fue aislado únicamente durante los periodos epidémicos, principalmente en el norte del país y en los meses de verano. En cambio, los aislamientos de V. cholerae no-O1, no-O139 siguieron un patrón diferente, ya que se recuperaron principalmente en primavera y verano, pero también en los meses de otoño e invierno. En general, los aislamientos clínicos estaban asociados a casos esporádicos de diarrea grave o moderada, aunque también hubo microorganismos recuperados de septicemias. En diciembre de 1999 y enero de 2000, ocurrió en Orán (provincia de Salta) un brote de diarrea que afectó a niños y adultos y que duplicó el número de coprocultivos esperados para la época del año; durante ese brote se recuperó V. cholerae no-O1 de diez pacientes y también de fuentes de agua de la zona.
Los principales factores de virulencia asociados al cólera epidémico incluyen la enterotoxina conocida como CT (11) y el factor de colonización TCP (toxin coregulated pilus) codificado en la isla de patogenicidad VPI (33), ambos habitualmente ausentes en V. cholerae no-O1, no-O139 (21). Para estos serogrupos se postula un mecanismo de patogenia diferente, en el que intervienen otros factores de virulencia. La diarrea en general es acuosa, aunque puede presentar sangre y moco (23). Por otra parte, V. cholerae no-O1, no-O139 puede producir otros productos extracelulares como la toxina termoestable NAG-ST y las citolisinas VCC/HlyA y RTX (1, 5, 21), los que pueden estar contribuyendo a la virulencia de estas cepas. También se ha descrito una neuraminidasa codificada en otra isla de patogenicidad (VPI-2), que cliva el ácido siálico de los gangliósidos presentes en las células eucariotas (19). Se cree que esta enzima aumenta la sensibilidad de las células hospedadoras a la toxina CT, ya que expone al gangliósido GM1, receptor de la toxina. La neuraminidasa está habitualmente asociada a cepas toxigénicas; sin embargo, también ha sido descrita en aislamientos de V. cholerae no toxigénicos como la variante Amazonia (15). Recientemente se ha descrito la presencia de un sistema de secreción de tipo III en cepas de V. cholerae no-O1 que puede participar en la virulencia de estas cepas (13, 32).
Diferentes estudios muestran que Vibrio cholerae es una especie de alta diversidad genética, y ello se ha observado utilizando diferentes técnicas de tipificación como multilocus enzimoelectroforesis (2), ribotipificación (14), diferentes estrategias basadas en la reacción en cadena de la polimerasa (PCR) (25, 31) y MLST (multilocus sequence typing) (16). Estas técnicas poseen diferente poder de discriminación y estudian distintas porciones del genoma. También hay estudios en los que se utilizó la electroforesis de campo pulsado (PFGE) con las enzimas de restricción NotI o SfiI, los que mostraron que esta técnica es muy discriminatoria para estudiar la diversidad genética de aislamientos de V. cholerae (4, 25, 31). En general, dichos estudios muestran un agrupamiento de los aislamientos O1 y O139 toxigénicos causales de epidemias -lo que indicaría una dispersión clonal de cepas epidémicas- y, por otra parte, una alta diversidad para los aislamientos no toxigénicos de los serogrupos O1 y no-O1.
El objetivo de este trabajo fue caracterizar los perfiles de virulencia y la diversidad genética de los aislamientos de Vibrio cholerae no-O1, no-O139 recuperados de niños con diarrea atendidos en el Hospital del Niño Jesús de San Miguel de Tucumán durante el período 2003-2005.
MATERIALES Y MÉTODOS
Muestras analizadas
Se estudiaron 14.724 coprocultivos correspondientes a niños con diarrea atendidos en el Hospital del Niño Jesús de San Miguel de Tucumán entre el 1° de enero de 2003 y el 31 de diciembre de 2005.
Aislamiento e identificación de Vibrio cholerae
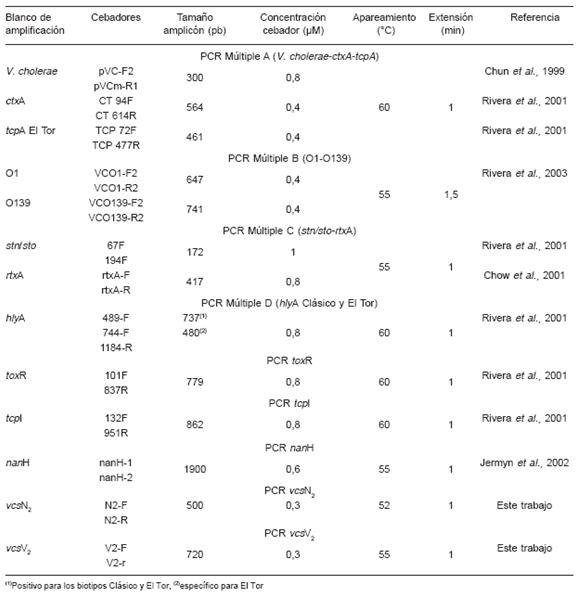
Las muestras de materia fecal se obtuvieron principalmente por hisopado anal o rectal y se conservaron en el medio de transporte Cary Blair hasta su traslado al laboratorio del hospital. Se sembraron en agar tiosulfato citrato bilis sacarosa (TCBS) (Oxoid Ltd., Basingstoke, Inglaterra), y en agar sangre preparado con sangre humana al 5% en el medio tripteína de soja (Laboratorios Britania, CABA, Argentina); estas placas se incubaron durante 24 h a 37 °C luego de un enriquecimiento de 6-8 h en agua peptonada alcalina (APA) (Oxoid Ltd.). Las colonias amarillas en TCBS (sacarosa positivas) o hemolíticas en agar sangre y oxidasa positivas fueron caracterizadas utilizando las pruebas bioquímicas para V. cholerae descritas por Dodin et al. (12). Además, se realizó la prueba de la cuerda en desoxicolato de sodio, positiva para Vibrio cholerae. Los aislamientos de V. cholerae fueron serotipificados por aglutinación en placa utilizando sueros policlonales (anti-O139, O1, Ogawa e Inaba) producidos por el Instituto Nacional de Producción de Biológicos- ANLIS "Dr. Carlos G. Malbrán". Para confirmar la identificación realizada por pruebas bioquímicas se efectuó PCR múltiple A con cebadores específicos para Vibrio cholerae (6). Los resultados de la serotipificación se confirmaron mediante PCR múltiple B con el empleo de cebadores para detectar los serogrupos O1 y O139 (28), utilizando la técnica detallada en el próximo párrafo y en la Tabla 2. Los aislamientos de V. cholerae caracterizados en este estudio se indican en la Tabla 1.
Tabla 1.
Aislamientos de Vibrio cholerae no-O1, no-O139 analizados mediante PCR
Tabla 2. Condiciones de las reacciones de PCR
Sensibilidad a los antimicrobianos
Se evaluó la sensibilidad a los antimicrobianos por el método de difusión con discos utilizando discos comerciales (Laboratorios Britania) de ampicilina (AMP, 10 μg), trimetoprima - sulfametoxazol (SXT, 1,25 / 23,75 μg), furazolidona (FUR, 100 μg), fosfomicina (FOS, 50 μg), tetraciclina (TET, 30 μg) y norfloxacina (NOR, 10 μg). Frente a la AMP, la SXT y la TET, los resultados obtenidos se interpretaron según las recomendaciones del CLSI (Clinical and Laboratory Standard Institute, USA) para V. cholerae; en el caso de los demás antimicrobianos la interpretación se basó en las recomendaciones para Enterobacteriaceae (7).
Identificación de factores de virulencia por PCR
Sobre la base de protocolos ya publicados, se realizaron diferentes reacciones de PCR para detectar genes que codifican factores de virulencia, entre ellos: ctxA (que codifica la subunidad A de CT); tcpA El Tor (que codifica la unidad estructural del factor de colonización TCP, alelo El Tor, TcpA); tcpI (que codifica una proteína asociada a la membrana interna y reguladora de la síntesis de TcpA); toxR (que codifica la proteína ToxR, reguladora de la expresión y la secreción de CT y TCP); stn/sto (que codifica la toxina termoestable NAG-ST); hlyA (que codifica la citolisina HlyA/VCC); rtxA (que codifica la toxina formadora de poros RTX); y nanH (que codifica una neuraminidasa) (5, 19, 27). Los protocolos fueron ligeramente modificados, ya que se implementó PCR múltiple A para V. cholerae, ctxA y tcpA El Tor y PCR múltiple C para stn/sto y rtxA, como se muestra en la Tabla 2. También se desarrollaron dos reacciones de PCR para los genes vcsN2 y vcsV2 que codifican la ATPasa y la proteína estructural del sistema de secreción de tipo III, recientemente encontrado en V. cholerae (13). La secuencia de los cebadores utilizados es la siguiente: N2-F: GCAGTTGAGCCAATTCCATTACAG; N2-R: CGACCAAACGAGATAATGCT; V2-F: CTATTTGGCTCACTTGATGGGGC; V2-R: GCCACATCATTGCTTGCTACC. Las condiciones de ciclado y la concentración de los cebadores (Operon Biotechnologies, Cologne, Alemania) se muestran en la Tabla 2. Para todas las reacciones de amplificación, la concentración final de MgCl2 fue de 1,5 mM. Los templados de ADN fueron preparados por el método de hervido y se utilizaron 2 μl para la amplificación por PCR (volumen final 25 μl). Como controles se utilizaron las siguientes cepas: RC138 (V. cholerae O139, ctxA +, tcpA El Tor +, hlyA El Tor, toxR +, stn/sto -, rtxA +) y RC2 (V. cholerae O1, ctxA +, tcpA El Tor -, hlyA Clásica, toxR +, stn/sto -, rtxA +), provistas por la Dra. Rita Colwell, Center of Marine Biotechnology, University of Maryland, EE.UU.; y la cepa 83-7771 (V. cholerae no-O1, no-O139, ctxA -, tcpA El Tor -, hlyA El Tor+, toxR +, stn/sto +, rtxA +), provista por el Dr. Clifford Clark, National Microbiology Laboratory, Canadá. También se utilizó una cepa autóctona BA312 como control positivo para vcsV2 y vcsN2. Como control de reactivos se agregaron 2 μl de agua en lugar del templado a la mezcla de reacción. El programa de ciclado fue el siguiente: desnaturalización inicial a 94 °C durante 5 min seguida de 30 ciclos de 1 min de desnaturalización a 94 °C, 1 min de apareamiento a temperatura variable, tiempo variable de extensión a 72 °C y una extensión final a 72 °C durante 10 min (Tabla 2).
Diversidad genética por electroforesis de campo pulsado
Se utilizó el protocolo de PFGE estandarizado de la Red PulseNet Internacional para Vibro cholerae (9). Brevemente, el ADN genómico se preparó mezclando en partes iguales una suspensión bacteriana de densidad óptica 0,9 a 610 nm en buffer Tris-EDTA 100 mM con agarosa SeaKem Gold 1%. Luego de una lisis de 1 h a 54 °C (solución para la lisis: Tris 50 mM, EDTA 50 mM, pH 8, lauril sarcosina 1%, proteinasa K 0,1 mg/ ml), se realizaron dos lavados con agua y cuatro lavados con TE 1X en baño con agitación a 50 °C. La restricción se realizó con 40 U de la enzima SfiI o con 30 U de Notl incubando toda la noche a 50 °C o 37° respectivamente. La cepa Salmonella Braenderup H9812 digerida con XbaI se utilizó como estándar. La electroforesis se realizó con agarosa SeaKem Gold 1% utilizando un equipo CHEF-DR III (Bio-Rad Laboratories, Hercules, CA, EE.UU.) en buffer TBE 0,5X con el siguiente programa: 2 a 10 seg, 13 h; 20 a 25 seg, 6 h. Para los aislamientos que presentaron una degradación completa del ADN se repitió el ensayo con el agregado de tiourea 50 μM en el buffer de corrida TBE (29). Aquellos aislamientos en los que no se resolvió la degradación fueron considerados no tipificables por PFGE. Los patrones de PFGE se analizaron por inspección visual y utilizando también el programa BioNumerics versión 4.6 (Applied Maths, Sint-Martens-Latem, Bélgica). Los dendrogramas se construyeron utilizando el coeficiente de similitud de Dice y el algoritmo UPGMA.
RESULTADOS
Aislamiento de Vibrio cholerae
La frecuencia de aislamiento de V. cholerae obtenido de los coprocultivos fue 0,23%; Shigella spp. fue el patógeno bacteriano más frecuentemente aislado de diarreas durante el período estudiado y estuvo presente en el 10,30% de los 14.724 coprocultivos estudiados. También se aisló Salmonella spp. (1,54%) y Aeromonas spp. (1,73%). No se detectaron casos de cólera ni se aisló V. cholerae de los serogrupos O1 u O139.
Se recuperaron 34 aislamientos de V. cholerae no-O1, no-O139 que se confirmaron con pruebas bioquímicas, serotipificación y PCR. En la Tabla 1 se presentan las fechas de toma de muestra, la ubicación geográfica, el sexo y la edad de los pacientes, como así también los resultados de las reacciones de PCR de los diez factores de virulencia analizados y de las que confirmaron la identificación como V. cholerae no-O1, no-O139. Los pacientes que presentaron diarrea por V. cholerae no-O1, no-O139 tenían entre 3 meses y 14 años (mediana = 1,25).
Con respecto a las manifestaciones clínicas, 19 de los 34 pacientes estudiados (56%) presentaron diarrea de tipo acuosa, otros 12 (35%) presentaron diarrea mucosa con sangre y no se contó con información de los tres pacientes restantes. En cinco de estos casos se observó asociación con otros patógenos: en cuatro muestras de diarrea acuosa se identificaron parásitos y en una de diarrea sanguinolenta se aisló Shigella sonnei. Por otro lado, 22 de los pacientes (65%) presentaron deshidratación leve, seis deshidratación moderada y uno deshidratación grave.
Sensibilidad a los antimicrobianos
La mayoría de los aislamientos (74%) fueron sensibles a todos los antimicrobianos ensayados. Cuatro aislamientos fueron resistentes a la ampicilina y dos fueron de sensibilidad intermedia. También se detectaron dos aislamientos resistentes a la trimetoprima-sulfametoxazol y un aislamiento resistente a la fosfomicina. Ningún aislamiento fue resistente a más de un antimicrobiano.
Factores de virulencia
Todos los aislamientos fueron negativos para ctxA, stn/sto y tcpA El Tor. Dos aislamientos fueron positivos para tcpI y, aunque fueron negativos para tcpA El Tor, como tcpI y tcpA pertenecen al mismo operón, es posible que posean otro alelo del factor de colonización TCP. Todos los aislamientos fueron positivos para toxR, mientras que 29 (85%) y 31 (91%) aislamientos fueron positivos para hlyA (hemolisina El Tor- ET) y rtxA, respectivamente. El gen nanH estuvo presente en 16 aislamientos (47%) y los genes vcsN2 y vcsV2 amplificaron en 4 de los 34 aislamientos (12%). Al estudiar la presencia de factores de virulencia se detectaron ocho perfiles diferentes (Figura 1 y Tabla 1), con una mayor prevalencia (47%) para aquel que poseía los genes toxR, hlyA y rtxA (perfil A), seguido del que, además de estos tres genes, también incluía nanH (perfil B), con el 20%. El resto de los perfiles de virulencia estaba representado por uno a tres aislamientos.
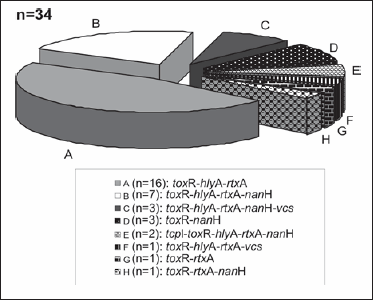
Figura 1. Distribución de los perfiles de virulencia de los aislamientos de V. cholerae no-O1, no-O139.
Diversidad genética
De los 34 aislamientos estudiados, cinco (T347, T153, T18118, T15881 y T4174) fueron no tipificables por PFGE, incluso luego de agregar tiourea en el buffer de corrida. La técnica demostró tener alto poder de discriminación, ya que se encontraron 27 patrones electroforéticos diferentes entre los 29 aislamientos comparados (Figura 2a). Solamente se identificaron dos grupos integrados por dos y tres aislamientos, cada uno con una similitud mayor al 95% al calcular el coeficiente de Dice (señalados en la Figura 2a). El primer grupo comprendió los aislamientos T1035 y T997, recuperados en enero de 2003; éstos fueron indistinguibles con las dos enzimas de restricción utilizadas (Figuras 2a y 2b) y tuvieron el mismo perfil de virulencia (perfil A). El segundo grupo estuvo integrado por los aislamientos T2508, T4582 y T987, recuperados entre enero y marzo del 2005; sus patrones de SfiI-PFGE difirieron en una sola banda y fueron indistinguibles por NotI-PFGE (Figura 2b). Estos tres aislamientos también compartían el mismo perfil de virulencia (perfil B). Los aislamientos de los dos grupos fueron recuperados en verano de pacientes que habitaban en barrios cercanos al Río Salí.
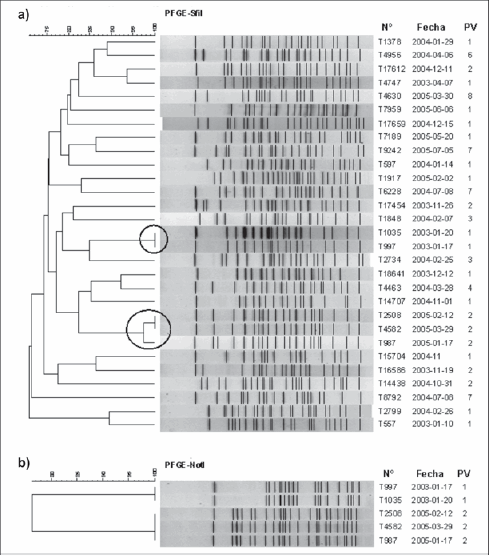
Figura 2. a) Dendrograma de SfiI- PFGE para los aislamientos de V. cholerae no-O1, no-O139. b) Dendrograma de NotI- PFGE para los aislamientos de V. cholerae no-O1, no-O139 que presentaron patrones compartidos con la enzima SfiI. En las columnas de la derecha se presenta el número que identifica al aislamiento, la fecha de aislamiento y el perfil de virulencia por PCR (PV). Los círculos señalan los grupos identificados con un coeficiente de similitud mayor del 95%.
Al analizar los aislamientos con una similitud mayor al 90% se observaron dos nuevos grupos. Estos comprendían a los aislamientos T17612 y T4747 por un lado, y a T18641 y T4463 por otro. Los aislamientos diferían en más de siete bandas al ser analizados por SfiI-PFGE y presentaban distintos perfiles de virulencia. También se identificaron grupos con mayor cantidad de aislamientos que tenían una similitud del 75% al 88%, pero que presentaban al menos diez bandas de diferencia y un máximo de cinco perfiles de virulencia diferentes.
DISCUSIÓN
A lo largo de los años 2003 al 2005 se realizó una intensiva vigilancia de diarreas por V. cholerae en el Hospital del Niño Jesús de San Miguel de Tucumán. Al confirmarse solamente 34 aislamientos de 14.724 coprocultivos, se puede afirmar que las diarreas por V. cholerae tienen baja incidencia en los pacientes pediátricos de San Miguel de Tucumán y afectan principalmente a niños de corta edad, los que en su mayoría exhiben diarrea de tipo acuosa, aunque fue llamativo el alto porcentaje de los casos (35%) que presentaron diarrea con sangre. El grado de deshidratación observado fue variable; como el tipo de diarrea predominante fue la acuosa, pensamos que la consulta temprana de las madres frente al cuadro de diarrea de sus hijos y la pronta instauración de la hidratación adecuada (oral, endovenosa, o ambas) evitó un grado de deshidratación mayor. De los 34 pacientes de los que se aisló V. cholerae, 21 (62%) vivían en zonas geográficas cercanas a ríos como el Salí, el Lules y el Canal Norte (Cuenca del Río Salí); esto se asume como un importante factor de riesgo para contraer diarreas causadas por Vibrio spp. y también por Aeromonas spp.
Aunque el tratamiento antimicrobiano de los cuadros diarreicos por Vibrio cholerae no tiene demasiada importancia en la evolución clínica, es útil para cortar la cadena epidemiológica y evitar así la diseminación del patógeno. Los antimicrobianos ensayados, sobre todo la furazolidona, la trimetoprima-sulfametoxazol y, en menor medida, la ampicilina, presentaron buena cobertura frente a estos aislamientos provenientes de una población pediátrica. Todos los aislamientos fueron sensibles a tetraciclina y norfloxacina, y aunque estos antimicrobianos no están autorizados para uso pediátrico, es importante ensayarlos para emplearlos, en caso de ser necesarios, en adultos. No se detectaron cepas multirresistentes.
Los aislamientos encontrados tuvieron diversos perfiles de virulencia; sin embargo, no se encontró correlación entre los síntomas y signos expuestos por los pacientes y los perfiles de virulencia de los aislamientos (datos no mostrados). Los aislamientos en ningún caso presentaron los genes que codifican la toxina CT (ctxA) o la toxina termoestable NAG-ST (stn/sto). En cambio, el gen que codifica el regulador ToxR (toxR) y los genes que codifican las citolosinas HlyA/VCC y RTX (hlyA y rtxA) estuvieron ampliamente distribuidos entre los aislamientos: éstos conformaron el perfil de virulencia más frecuente (perfil A, 47%). Tanto HlyA/VCC, que altera el balance osmótico de las células eucariotas (18), como RTX, que despolimeriza los filamentos de actina en células eucariotas (30), podrían explicar la patogenia de las cepas de V. cholerae CT negativas y quizás, los casos de diarrea muco-sanguinolenta. Los resultados de la caracterización de aislamientos de origen humano fueron concordantes con los encontrados para aislamientos ambientales recuperados del río Lules y de dos sitios del río Salí (17), ya que los perfiles de virulencia son muy semejantes. Esta similitud, sumada a la falta de correlación entre los perfiles de virulencia de los aislamientos de V. cholerae no-O1, no-O139 y la presentación clínica de la diarrea sugiere que, además de estos factores de virulencia del patógeno, la cantidad de microorganismos ingerida y el estado inmune del huésped tienen un papel muy importante en la evolución de la enfermedad. Por esta razón es importante la vigilancia de este patógeno, especialmente en las poblaciones más vulnerables, como los niños, los ancianos y los inmunocomprometidos.
Es interesante resaltar que se encontraron 16 aislamientos positivos para el gen que codifica la neuraminidasa (nanH), que pertenece a la isla de patogenicidad VPI-2, habitualmente asociada a cepas epidémicas portadoras de la toxina CT (19). Sin embargo, la presencia de dicho gen no implica la presencia de la isla VPI-2 completa (24). Es necesario investigar este aspecto, como también el papel de la neuraminidasa en la patogénesis de aislamientos CT negativos.
También es importante destacar la presencia del sistema de secreción de tipo III (T3SS) -hasta ahora, el único descrito para V. cholerae- en cuatro de los aislamientos estudiados, ya que este sistema puede aportar mecanismos de patogenia alternativos para las cepas de V. cholerae no toxigénicas. A su vez, se ha descrito que los genes vcs que codifican el T3SS de V. cholerae también se encuentran en la isla VPI-2 (24). En el grupo de aislamientos analizados, solamente tres aislamientos portaban el gen nanH y los genes vcs. A su vez, hubo tres aislamientos con el gen nanH y sin los genes vcs, y un aislamiento que poseía los genes vcs y no el gen nanH. Esto estaría indicando una estructura distinta de la isla VPI-2 o una localización diferente de estos genes.
Al igual que en otros estudios (4, 31), se encontró alta diversidad genética en los aislamientos de V. cholerae no-O1, no-O139 analizados por PFGE, ya que solamente se identificaron dos pequeños grupos genéticamente muy relacionados, de dos y tres aislamientos cada uno. Si bien se observaron en el dendrograma otros grupos, con una similitud de entre 75% y 95%, los aislamientos dentro de esos grupos presentaban más de siete bandas de diferencia y distintos perfiles de virulencia. Esto sugiere que estos agrupamientos incluyeron aislamientos que pertenecían probablemente a diferentes linajes.
Nuestros resultados muestran que no existe un número limitado de clones que persiste en esta región, sino que existe gran diversidad entre las cepas de V. cholerae no-O1, no-O139 capaces de causar enfermedad en el hombre, aun dentro de una región acotada, como es el caso de la cuenca del río Salí. Estos resultados coinciden con los encontrados por Bidinost et al. (3) en el estudio llevado a cabo en la provincia de Córdoba con aislamientos ambientales y humanos. Más aún, esta diversidad ha sido también demostrada por nuestro grupo en un estudio reciente aplicando MLST sobre aislamientos de V. cholerae de Argentina (16).
Finalmente, informamos que se ha detectado un aislamiento portador de los genes ctxA y tcpI, recuperado en San Miguel de Tucumán en febrero de 2007 de un paciente varón de 2 años y 6 meses, que consultó por la guardia de emergencia por diarrea con reacción inflamatoria, vómitos y mal estado general. Dicho aislamiento, caracterizado como V. cholerae no-O1, no-O139 (resultados no publicados), evidencia la transferencia horizontal de genes, ya que ctxA y tcpI están usualmente asociados a V. cholerae O1.
Es necesario destacar la importancia de reactivar la vigilancia de V. cholerae, tanto en casos clínicos como en los reservorios acuáticos, para detectar oportunamente la aparición de cepas con potencial patogénico, ya sea del serogrupo O1 o de otros serogrupos.
Agradecimientos: al Consejo Nacional de Investigaciones Científicas y Técnicas por la beca otorgada a S. González Fraga, y a M. Panagopulo por su asistencia técnica. Este estudio fue realizado con fondos del proyecto PICTR 2000-00010 otorgado por la Agencia Nacional de Promoción Científica y Tecnológica.
1. Arita M, Takeda T, Honda T, Miwatani T. Purification and characterization of Vibrio cholerae non-O1 heat-stable enterotoxin. Infect Immun 1986; 52: 45-9. [ Links ]
2. Beltrán P, Delgado G, Navarro A, Trujillo F, Selander RK, Cravioto A. Genetic diversity and population structure of Vibrio cholerae. J Clin Microbiol 1999; 37: 581-90. [ Links ]
3. Bidinost C, Saka HA, Aliendro O, Sola C, Panzetta-Duttari G, Carranza P, et al. Virulence factors of non-O1 non-O139 Vibrio cholerae isolated in Córdoba, Argentina. Rev Argent Microbiol 2004; 36:158-63. [ Links ]
4. Choudhury SR, Bhadra RK, Das J. Genome size and restriction fragment length polymorphism analysis of Vibrio cholerae strains belonging to different serovars and biotypes. FEMS Microbiol Lett 1994; 115: 329-34. [ Links ]
5. Chow IK, Yuen K, Yam W. Detection of RTX Toxin Gene in Vibrio cholerae by PCR. J Clin Microbiol 2001; 39: 2594-7. [ Links ]
6. Chun J, Huq A, Colwell R. Analysis of 16S-23S rRNA intergenic spacer regions of Vibrio cholerae and Vibrio mimicus. Appl Environ Microbiol 1999; 65: 2202-8. [ Links ]
7. Clinical and Laboratory Standards Institute. Disk diffusion. Performance standards for antimicrobial susceptibility testing; 18th Informational Supplement, 2008; M100-S18. Wayne, PA, USA. [ Links ]
8. Colwell RR. Global climate and infectious disease: the cholera paradigm. Science 1996; 274: 2025-31. [ Links ]
9. Cooper KL, Luey CK, Bird M, Terajima J, Nair GB, Kam KM, et al. Development and validation of a PulseNet standardized pulsed-field gel electrophoresis protocol for subtyping of Vibrio cholerae. Foodborne Pathog Dis 2006; 3: 51-8. [ Links ]
10. Costagliola M, López A, Malaspina A, Guerrero R, Medina D, Odizzio M, et al. Estudio de la presencia de V. cholerae en la Zona Común de Pesca Argentino-Uruguaya: período 1992-1996. Frente Marítimo 2000; 18: 53-8. [ Links ]
11. De SN. Enterotoxocity of bacteria-free culture-filtrate of Vibrio cholerae. Nature 1959; 183: 1533-4. [ Links ]
12. Dodin A, Fournier JM. Méthodes de laboratoire pour le diagnostic du vibrion cholérique et des autres vibrions. Commission des laboratoires de référence et d'expertise de l'Institut Pasteur. Paris, Institut Pasteur 1992, p. 91-148. [ Links ]
13. Dziejman M, Serruto D, Tam VC, Sturtevant D, Diraphat P, Faruque SM, et al. Genomic characterization of non-O1, non-O139 Vibrio cholerae reveals genes for a type III secretion system. Proc Natl Acad Sci USA 2005; 102: 3465-70. [ Links ]
14. Faruque SM, Chowdhury N, Kamruzzaman M, Dziejman M, Rahman MH, Sack DA, et al. Genetic diversity and virulence potential of environmental Vibrio cholerae population in a cholera-endemic area. Proc Natl Acad Sci USA 2004; 101: 2123-8. [ Links ]
15. Figueiredo SC, Neves-Borges AC, Coelho A. The neuraminidase gene is present in the non-toxigenic Vibrio cholerae Amazonia strain: a different allele in comparison to the pandemic strains. Mem Inst Oswaldo Cruz 2005; 100: 563-9. [ Links ]
16. González Fraga S, Pichel M, Binsztein N, Johnson JA, Morris JG Jr, Stine OC. Lateral gene transfer of O1 serogroup encoding genes of Vibrio cholerae. FEMS Microbiol Lett 2008; 286: 32-8. [ Links ]
17. González Fraga S, Pichel M, Costagliola M, Cecilia M, Jurquiza V, Peressutti S, et al. Environment and virulence factors of Vibrio cholerae strains isolated in Argentina. J Appl Microbiol 2007; 103: 2448-56. [ Links ]
18. Gutiérrez MG, Saka HA, Chinen I, Zoppino FC, Yoshimori T, Bocco JL, et al. Protective role of autophagy against Vibrio cholerae cytolysin, a pore-forming toxin from V. cholerae. Proc Natl Acad Sci USA 2007; 104: 1829-34. [ Links ]
19. Jermyn WS, Boyd EF. Characterization of a novel vibrio pathogenicity island (VPI-2) encoding neuraminidase (nanH) among toxigenic Vibrio cholerae isolates. Microbiology 2002; 148: 3681-93. [ Links ]
20. Kaper JB, Morris JG Jr, Levine MM. Cholera. Clin Microbiol Rev 1995; 8: 48-86. [ Links ]
21. Kurazono H, Pal A, Bag PK, Nair GB, Karasawa T, Mihara T, et al. Distribution of genes encoding cholera toxin, zonula occludens toxin, accessory cholera toxin, and El Tor hemolysin in Vibrio cholerae of diverse origins. Microb Pathog 1995; 18: 231-5. [ Links ]
22. Levine M. South America: the return of cholerae. Lancet 1991; 338: 45-6. [ Links ]
23. Morris JG. Non-O group 1 Vibrio cholerae strains not associated with epidemic disease. En: Wachsmuth IK, Blake PA, Olsvik Ø, editors. Vibrio cholerae and Cholera. Washington D.C., ASM Press, 1994, p. 103-15. [ Links ]
24. Murphy RA, Boyd EF. Three pathogenicity islands of Vibrio cholerae can excise from the chromosome and form circular intermediates. J Bacteriol 2008; 190: 636-47. [ Links ]
25. Pichel M, Rivas M, Chinen I, Martín F, Ibarra C, Binsztein N. Genetic diversity of Vibrio cholerae O1 in Argentina and emergence of a new variant. J Clin Microbiol 2003; 41: 124-34. [ Links ]
26. Rivas M, Cacace ML, Ayala LT, Baschkier A, Miliwebsky E, Caffer MI. Casos de gastroenteritis asociados a Vibrio cholerae no-01 en Orán, Salta. Rev Argent Microbiol 1996; 28: 163-9. [ Links ]
27. Rivera I, Chun J, Huq A, Sack R, Colwell RR. Genotypes associated with virulence in environmental isolates of Vibrio cholerae. Appl Environ Microbiol 2001; 67: 2421-9. [ Links ]
28. Rivera I, Lipp E, Gil A, Choopun N, Huq A, Colwell RR. Method for extraction and application of multiplex PCR to detect toxigenic V. cholerae O1 and O139 from aquatic ecosystems. Environ Microbiol 2003; 5: 599-606. [ Links ]
29. Römling U, Tümmler B. Achieving 100% typeability of Pseudomonas aeruginosa by pulsed-field gel electrophoresis. J Clin Microbiol 2000; 38: 464-5. [ Links ]
30. Sheahan KL, Cordero CL, Satchell KJ. Identification of a domain within the multifunctional Vibrio cholerae RTX toxin that covalently cross-links actin. Proc Natl Acad Sci USA 2004; 101: 9798-803. [ Links ]
31. Singh DV, Matte MH, Matte GR, Jiang S, Sabeena F, Shukla BN, et al. Molecular analysis of Vibrio cholerae O1, O139, non-O1, and non-O139 strains: clonal relationships between clinical and environmental isolates. Appl Environ Microbiol 2001; 67: 910-21. [ Links ]
32. Tam VC, Serruto D, Dziejman M, Brieher W, Mekalanos JJ. A type III secretion system in Vibrio cholerae translocates a formin/spire hybrid-like actin nucleator to promote intestinal colonization. Cell Host Microbe 2007; 1: 95-107. [ Links ]
33. Taylor RK, Miller VL, Furlong DB, Mekalanos JJ. Use of phoA gene fusions to identify a pilus colonization factor coordinately regulated with cholera toxin. Proc Natl Acad Sci USA 1987; 84: 2833-7. [ Links ]
Recibido: 26/08/08
Aceptado: 16/12/08.














