Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO  uBio
uBio
Share
Revista argentina de microbiología
Print version ISSN 0325-7541On-line version ISSN 1851-7617
Rev. argent. microbiol. vol.41 no.2 Ciudad Autónoma de Buenos Aires Apr./June 2009
Efecto de la inoculación de la cepa Sphingomonas paucimobilis 20006FA sobre la composición de un consorcio bacteriano degradador de fenantreno
L. Madueño, B. M. Coppotelli, I. S. Morelli*
Centro de Investigación y Desarrollo en Fermentaciones Industriales, CINDEFI (UNLP; CCT-La Plata, CONICET), Calle 50 y 115, La Plata, Pcia. de Buenos Aires (1900). Argentina
*Correspondencia. E-mail: lbmh@biol.unlp.edu.ar
RESUMEN
Se estudió el efecto de la inoculación con la cepa Sphingomonas paucimobilis 20006FA sobre la composición bacteriana de un consorcio degradador de fenantreno en cultivos discontinuos (batch) con 8 repiques sucesivos. El consorcio original se obtuvo a partir de un suelo prístino. A los fines del estudio, se obtuvieron y mantuvieron dos consorcios: uno inoculado (F200+I) y otro sin inocular (F200). Se estudió la diversidad bacteriana de los consorcios mediante el análisis de microorganismos cultivables (por caracterización fenotípica y genotípica) y totales (por PCR-DGGE). A lo largo de los repiques sucesivos pudo observarse en ambos consorcios una tendencia a la pérdida de la capacidad degradadora de fenantreno, acompañada por una disminución de la diversidad bacteriana. Si bien la inoculación no produjo cambios significativos en la capacidad degradadora de fenantreno de los consorcios (29,9% para F200 y 27,6% para F200+I hacia el tercer repique), sí produjo cambios en la composición bacteriana, ya que los perfiles de DGGE revelaron una dinámica estructural diferente en el consorcio inoculado. En ambos consorcios se pudo observar la presencia de una banda intensa posicionada a la misma altura que el ADN del inóculo en el gel de DGGE; sin embargo, los cultivos aislados de los consorcios que presentaban idéntica posición de banda en el perfil PCR-DGGE que la cepa S. paucimobilis 20006FA mostraron baja similitud con la cepa inoculada mediante la técnica de RAPD.
Palabras clave: Consorcios bacterianos; Fenantreno; Sphingomonas; DGGE
ABSTRACT
Effect of the inoculant strain Sphingomonas paucimobilis 20006FA on the bacterial composition of a phenanthrene-degrading consortium. The effect of the inoculant strain Sphingomonas paucimobilis 20006FA on the bacterial composition of a phenanthrene-degrading consortium obtained from a pristine soil in sequencing batch cultures was studied. Inoculated (F200+I) and non-inoculated (F200) phenanthrene-degrading consortia, were obtained. Bacterial diversity of consortia was studied at cultivable (phenotype and genotype characterization) and non-cultivable (PCR-DGGE) levels. During the successive cultures, a loss in the phenanthrene-degrading capacity and a decrease in the bacterial diversity were observed in both consortia. Although inoculation did not produce any significant changes in the consortia phenanthrene-degrading capacity (29.9% F200 and 27.6% F200+I), it did produce changes in the bacterial composition, showing a differential structural dynamics in the DGGE profiles of the inoculated consortium. In both consortia, a dominant band placed at the same position as that of the DNA of the inoculant strain in the DGGE gel could be observed. However, isolated cultures from the consortia which had an identical band position to that of S. paucimobilis 20006FA in the PCR-DGGE profile showed low similarity with respect to the inoculant strain (RAPD).
Key words: Bacterial consortium; Phenanthrene; Sphingomonas; DGGE
INTRODUCCIÓN
Los hidrocarburos policíclicos aromáticos (PAH) se producen como resultado de procesos geobiológicos naturales, aunque su principal origen son las fuentes antropogénicas. Por ello, su concentración en los suelos se ha incrementado considerablemente desde el siglo XX (12). Estos ubicuos contaminantes orgánicos exhiben propiedades tóxicas y carcinogénicas (20), por lo que la Environmental Protection Agency (EPA) y la Unión Europea (UE) han considerado a 16 de ellos contaminantes prioritarios (5).
La degradación microbiana de PAH ha recibido una gran atención como posible estrategia para la remediación de suelos contaminados con estos compuestos (16). Los PAH son típicos contaminantes para los cuales sería aplicable la estrategia de bioaumento (11), la que consiste en el aumento de las capacidades metabólicas de la comunidad microbiológica indígena por la introducción de microorganismos, a fin de incrementar la velocidad o la extensión de la degradación de un contaminante. En los últimos años se han publicado muchos experimentos relacionados con esta estrategia, algunos con resultados favorables y otros desfavorables, por lo que esta técnica aún se considera discutible tanto desde el punto de vista científico como tecnológico. Mientras algunos autores encontraron que la inoculación produce un aumento en la velocidad de biodegradación y reduce los tiempos del proceso de biorremediación de los PAH de bajo peso molecular (1, 10); otros informaron una rápida disminución de la concentración del inóculo debido a que la dinámica del suelo evoluciona hacia el reestablecimiento de la comunidad original (29). En otros casos, el establecimiento del microorganismo inoculado no produjo un aumento significativo en la actividad degradadora de la comunidad. En tal sentido, la acumulación de productos de degradación (9), la baja disponibilidad de los PAH y la competencia con la flora nativa (2, 7, 13, 17, 32) fueron mencionadas como factores que podrían afectar la actividad del inóculo.
El cultivo de consorcios microbianos es una de las técnicas utilizadas para estudiar los mecanismos que emplean los microorganismos para establecerse y permanecer en distintos ambientes. Se han realizado numerosos estudios para obtener consorcios bacterianos degradadores utilizando la técnica de cultivo batch o discontinuo (12, 33) y se ha comprobado que este sistema provee un ambiente relativamente constante para su establecimiento.
El objetivo de nuestro estudio fue determinar la influencia que ejerce sobre el establecimiento y la supervivencia de la cepa Sphingomonas paucimobilis 20006FA la competencia con la microflora autóctona del suelo. La cepa mencionada, utilizada como inoculante, fue aislada y ampliamente caracterizada en nuestro laboratorio (8). Teniendo en cuenta el objetivo planteado, se obtuvieron y mantuvieron en cultivos discontinuos con repiques sucesivos dos consorcios bacterianos degradadores de fenantreno obtenidos a partir de suelo prístino, uno de los cuales fue inoculado con la cepa S. paucimobilis 20006FA. Se estudió el efecto de la presencia de la cepa sobre la estructura y la composición microbiana y sobre la capacidad degradadora del consorcio en comparación con el consorcio obtenido a partir de la microflora autóctona solamente.
MATERIALES Y MÉTODOS
Obtención y mantenimiento de los cultivos de enriquecimiento F200 y F200+I
Se utilizaron dos sistemas de cultivo discontinuo en erlenmeyers de 250 ml con 90 ml de medio mineral líquido (MML) (30), 200 mg/l (p.p.m.) de fenantreno y 11,7 g de tierra húmeda previamente tamizada (2 mm). El cultivo F200+I se obtuvo inoculando con 200 μl de un cultivo de S. paucimobilis 20006FA con un título bacteriano de 1,4 x107 UFC/ml.
Cada 15 días se realizaron repiques de ambos sistemas en medio fresco (90 ml de MML suplementado con 200 p.p.m. de fenantreno), repicando 10 ml del cultivo anterior. Esta metodología de repiques sucesivos se repitió 8 veces hasta el sistema discontinuo N° 8. Los sistemas fueron incubados a 28 ± 2 °C con agitación (200 r.p.m.).
Preparación del inóculo de S. paucimobilis 20006FA
El inóculo se cultivó en caldo nutritivo (Oxoid Ltd., Inglaterra) diluido 1:2 con agua destilada (AD) a 32 °C durante 48 h. Las células fueron centrifugadas y el sedimento obtenido se lavó dos veces con una solución 0,85% NaCl (SF), luego se resuspendió y se ajustó a una DO de 5 a 580 nm, correspondiente a 6,3 x 109 UFC/ml. La densidad celular del inóculo fue determinada mediante diluciones seriadas y posterior siembra en placas de medio Reasoner (R2A) (24). Un volumen de 200 μl de esta suspensión se adicionó a uno de los cultivos de enriquecimiento (F200+I).
Determinación de la capacidad degradadora de fenantreno
Se inocularon 5 ml de los cultivos de enriquecimiento F200 y F200+I correspondientes a los repiques N° 3 y N° 8 en erlenmeyers que contenían 50 ml de MML y 200 p.p.m. de fenantreno como única fuente de carbono y energía. Los cultivos se incubaron a 28 °C en agitación (200 r.p.m.). Los ensayos se realizaron por duplicado.
A los 12 días de incubación se realizaron extracciones exhaustivas del fenantreno remanente en cada erlenmeyer utilizando ampollas de decantación con 10 ml de hexano como solvente de extracción y 2 ml de hexadecano como estándar interno; el tiempo de contacto de cada extracción fue de 30 min y cada medida se realizó por duplicado. La concentración de fenantreno remanente se determinó por CG-FID (31). Los valores promedio fueron comparados mediante el análisis de varianza simple a un nivel de p= 0,05. Todos los análisis estadísticos fueron realizados utilizando el software 5.0 Systat® en sistema Windows®.
Recuentos de bacterias heterótrofas cultivables (BHC)
Se tomaron 100 μl de cada cultivo de enriquecimiento y se realizaron diluciones seriadas en SF; las diluciones escogidas se sembraron en placas de Petri con medio R2A. Cada dilución se sembró por duplicado. Los resultados del recuento que se muestran son el promedio de tres réplicas y se expresan como UFC/ml.
Aislamiento e identificación de cultivos puros
A partir de las placas de R2A donde se realizaron los recuentos de BHC, se seleccionaron algunas colonias de cada consorcio. Con cada una de las colonias elegidas se realizaron aislamientos sucesivos en placas con R3A (24). Los cultivos fueron clasificados de acuerdo con la coloración de Gram, la movilidad y la morfología bacteriana, así como por el aspecto y el color de la colonia en la superficie del medio R3A. Se realizaron, además, pruebas bioquímicas convencionales (reducción de nitratos, formación de indol, utilización oxidativa-fermentativa de la glucosa según Hugh y Leifson, arginina dihidrolasa, ureasa, hidrólisis de esculina, hidrólisis de gelatina, prueba de citrato según Simmons e hidrólisis de almidón).
Se evaluó la capacidad de los cultivos aislados para degradar diferentes PAH sembrándolos sobre placas que contenían medio mineral sólido (MMS) adicionado con los siguientes PAH (6): antraceno, dibenzotiofeno, fenantreno, pireno y fluoreno. Se consideró un cultivo degradador del PAH en estudio a aquel que generó alrededor de su estría de siembra un halo translúcido o color proveniente de los productos de degradación. Las cajas se incubaron a 28 ± 2 °C durante 21 días.
Los resultados obtenidos en la caracterización de los cultivos puros (características macro y microscópicas, pruebas bioquímicas y espectro de degradación de PAH) se utilizaron como base para el cálculo de la matriz de similitud utilizando el coeficiente de similitud de apareamiento simple (Ssm). Los dendrogramas fueron calculados mediante el método del promedio aritmético no ponderado (UPGMA) (25).
Extracción de ADN
Cultivos aislados: se tomó 1 ml de cada cultivo puro crecido durante 24 h en caldo R3 (24), se centrifugó y se resuspendió en 1 ml de AD; luego se sumergieron los tubos en un baño a 100 °C durante 10 min y finalmente se realizó una centrifugación a 13.000 r.p.m. Del sobrenadante se recuperó el ADN soluble.
Cultivos de enriquecimiento: se eligieron los cultivos correspondientes a los repiques N° 3 y N° 8 para estudiar los cambios en la composición bacteriana de los consorcios establecidos.
Se tomaron aproximadamente 3 ml de cada sistema discontinuo, se realizó una centrifugación y se eliminó el sobrenadante. Para la extracción y purificación del ADN se utilizó el Kit Genomictips (20/G) de Qiagen (Qiagen Inc., Chatsworth, CA, EE.UU.).
RAPD
Se utilizaron los siguientes cebadores: cebador 9: IM-2 5'd[CTCAATGGCAGCGGCTATG] 3', cebador 6: IM-1 5'd[CGAGCTTCGCGTACCACCC] 3' y cebador 10: IM-3 5'd[GTTTCGCTCGATGCGCTACC] 3'. Las reacciones de PCR contenían 1 μl de ADN; 1 U de AmpliTaq; el buffer provisto por el fabricante de la enzima; 0,4 mM de dNTPs y 2 μM del cebador correspondiente en un volumen de reacción de 25 μl. La amplificación se realizó en un termociclador Progene FPROGO5Y (Techne, Burlington, NY, EE.UU.). El programa consistió en 4 ciclos de 5 min a 94 °C, 5 min a 40 °C y 5 min a 70 °C; 30 ciclos de 1 min a 94 °C, 1 min a 55 °C y 2 min a 70 °C; y finalmente 1 ciclo de 10 min a 70 °C. Los productos obtenidos fueron sometidos a una electroforesis a 75 V durante 90 min en gel de agarosa al 1,2% utilizando TBE (0,089 M Tris pH 8; 0,089 M ácido bórico y 0,002 M Na2EDTA) como buffer de corrida. El gel se coloreó con bromuro de etidio para evidenciar los productos de amplificación. Como marcador de peso molecular se utilizaron fragmentos de ADN doble cadena en un rango de 100 a 1000 pb en incrementos de a 100 pb (CienMarker; Biodynamics S.R.L.)
PCR- DGGE
Las muestras que se amplificaron fueron las de ADN obtenido de los cultivos puros y el proveniente de los cultivos de enriquecimiento. Se utilizaron cebadores dirigidos al gen ARNr 16S (cebador 341-FGC: CGCCCGCCGCGCCCCGCGCCGGCCCGCCGCCCCCGCCCCCTCCTACGGGAGGCAGCA; cebador 907-R: CCGTCAATTCCTTTGAGTTT) (21). Las reacciones de PCR contenían 1 μl de ADN; 1 U de AmpliTaq; el buffer previsto por el fabricante de la enzima; 200 mM de BSA; 0,4 mM de dNTPs y 2 μM de cada cebador en un volumen de reacción de 50 μl. El programa de PCR consistió en 1 ciclo de 4 min a 94 °C; 10 ciclos de 30 s a 94 °C; 45 s a 62 °C (temperatura de hidridación) y 1 min a 72 °C; 25 ciclos de 30 s a 94 °C, 45 s a 57 °C y 1 min a 72 °C; y 10 min a 72 °C. Se verificó la amplificación de cada muestra mediante electroforesis a 90 V durante 1 h en gel de agarosa 1,2% utilizando TBE como buffer de corrida. El gel se coloreó con bromuro de etidio para evidenciar los productos de la PCR.
La DGGE fue realizada en un BioRad D GENE System (BioRad, Munich, Alemania). Los productos de la PCR purificados fueron directamente aplicados en un gel de poliacrilamida al 6% (p/vol) (acrilamida-N,N'-metilenbisacrilamida, 37,5:1). El gel contenía un gradiente lineal 30%-65% desnaturalizante (100% desnaturalizante corresponde a urea 7 M y 40% de formamida vol/vol). La electroforesis fue realizada en buffer TAE (40 mM Tris pH 8,1; 20 mM ácido acético; 1 mM Na2EDTA) a una temperatura de 60 °C. La corrida electroforética se realizó a un voltaje constante de 100 V durante 16 h. Luego de la electroforesis, el gel fue teñido durante 1 h con bromuro de etidio; los geles fueron digitalizados en una cámara CCD.
RESULTADOS Y DISCUSIÓN
Obtención y mantenimiento de consorcios bacterianos degradadores de fenantreno
Utilizando la técnica de enriquecimiento en cultivos discontinuos con repiques sucesivos se logró obtener y mantener dos consorcios bacterianos degradadores de fenantreno, a los que se denominó F200 y F200+I. Los consorcios se mantuvieron 120 días mediante 8 repiques sucesivos durante los cuales conservaron su capacidad degradadora, evidenciada por la aparición de un color amarillo que resulta de la acumulación de productos de degradación (27). En ambos consorcios se observó también la capacidad de formación de biopelículas sobre la superficie del vidrio, aunque ésta fue perdiéndose durante los repiques sucesivos (datos no mostrados).
Fue posible diferenciar a los dos consorcios por sus características macroscópicas: el cultivo de enriquecimiento F200+I, que había sido inoculado con la cepa S. paucimobilis 20006FA, presentó inicialmente y hasta el cuarto repique una mayor intensidad de color; además conservó la capacidad de formar biopelículas durante un mayor número de repiques que el cultivo F200.
En cuanto al recuento de bacterias heterótrofas cultivables en el medio R2A, ambos consorcios mantuvieron un número relativamente estable e independiente del tiempo de incubación (7,29 x 109 ± 1,34 x 108 para F200 y 1,61 x 109 ± 4,62 x 108 para F200+I) luego del segundo repique, lo que sugiere que los sistemas habían alcanzado un estado pseudoestacionario. Para el estudio de la estructura, la composición y la actividad degradadora del consorcio, se seleccionaron los repiques número 3 y 8; esto es, al inicio del estado pseudoestacionario y al finalizar el experimento.
Estudio de la capacidad degradadora de fenantreno de los consorcios
La Tabla 1 muestra los porcentajes de eliminación de fenantreno luego de 12 días de incubación en los consorcios 3F200, 8F200, 3F200+I y 8F200+I. En todos los casos los porcentajes de eliminación obtenidos fueron significativamente mayores que la pérdida abiótica (0,85% ± 0,10%), lo que da cuenta de la existencia de procesos biológicos. Se observó que la inoculación con la cepa S. paucimobilis 20006FA no produjo un cambio significativo en los porcentajes de eliminación de fenantreno alcanzados por los consorcios.
Tabla 1. Porcentaje de eliminación de fenantreno de los consorcios 3F200, 8F200, 3F200+I y 8F200+I luego de 12 días de incubación en MML suplementado con 200 mg/l de fenantreno.

Tampoco se observaron diferencias significativas entre los porcentajes de eliminación de fenantreno de los consorcios a través de los repiques sucesivos.
Estudio de la estructura y composición bacteriana
A partir de las placas en las cuales se obtuvieron entre 30 y 300 colonias en los recuentos de BHC y de acuerdo con la diversidad de morfotipos presentes, se realizaron entre 10 y 20 aislamientos por consorcio. Estos cultivos aislados se tomaron como representativos de las poblaciones cultivables mayoritarias.
Se pudo observar una disminución en la diversidad de fenotipos morfológicos de las colonias causada por la inoculación con la cepa S. paucimobilis 20006FA y por los repiques sucesivos (datos no mostrados). En concordancia con lo observado macroscópicamente, las observaciones microscópicas mostraron una mayor diversidad en los consorcios 3F200 y 3F200+I, con presencia de poblaciones tanto gram-positivas como gram-negativas. A través de los repiques sucesivos, los consorcios fueron perdiendo diversidad de morfologías microscópicas, y las poblaciones aisladas en 8F200 y 8F200+I fueron casi exclusivamente gram-negativas.
Es importante destacar que luego de 8 repiques sucesivos en medios con fenantreno como única fuente de carbono y energía, aproximadamente el 80% de las poblaciones presentes en los consorcios no mostraron evidencias de ser capaces de degradar PAH en cultivos puros (mediante la aparición de un halo claro por la solubilización del PAH ensayado en medio sólido o por la aparición de productos coloreados). La persistencia de estos microorganismos a través de los repiques sucesivos parece sugerir la existencia de relaciones comensales y/o sintróficas con aquellas poblaciones capaces de realizar la fisión inicial de los PAH. Precisamente, la existencia de este tipo de relaciones funcionales son las que definen a los consorcios bacterianos, circunstancia que convalida la técnica de cultivo usada como método de obtención y mantenimiento de consorcios.
La Figura 1 muestra el dendrograma obtenido con los porcentajes de similitud fenotípica entre los cultivos aislados de los cuatro consorcios. Para el cálculo de los porcentajes de similitud fenotípica fueron consideradas las características morfológicas y bioquímicas, como así también el espectro de degradación de PAH (antraceno, fenantreno, pireno, dibenzotiofeno y fluoreno). En el análisis se agregó la cepa S. paucimobilis 20006FA de manera de poder compararla con los cultivos aislados. Se observó que ni la inoculación ni los repiques sucesivos determinaron una marcada diferenciación y separación de clusters (Figura 1). En el dendrograma se observan dos grandes clusters, el A y el B, que se asemejan entre sí en un 62,2%, lo que muestra la gran similitud encontrada entre los cultivos de los cuatro consorcios estudiados. Ambos clusters incluyen poblaciones degradadoras de PAH. Dentro del cluster A se puede observar la presencia de un subcluster indicado mediante la línea punteada. Este subcluster está integrado por cuatro cultivos degradadores de fenantreno, de los cuales uno corresponde a la cepa S. paucimobilis 20006FA y los restantes provienen de tres (3F200, 8F200 y 8F200+I) de los cuatro consorcios analizados. La agrupación de estos cultivos aislados junto con la cepa inoculada en un subcluster con una similitud del 71,7% estaría indicando la gran semejanza existente entre ellos. Curiosamente, no se aislaron cultivos fenotípicamente semejantes a la cepa inoculada en el consorcio 3F200+I.
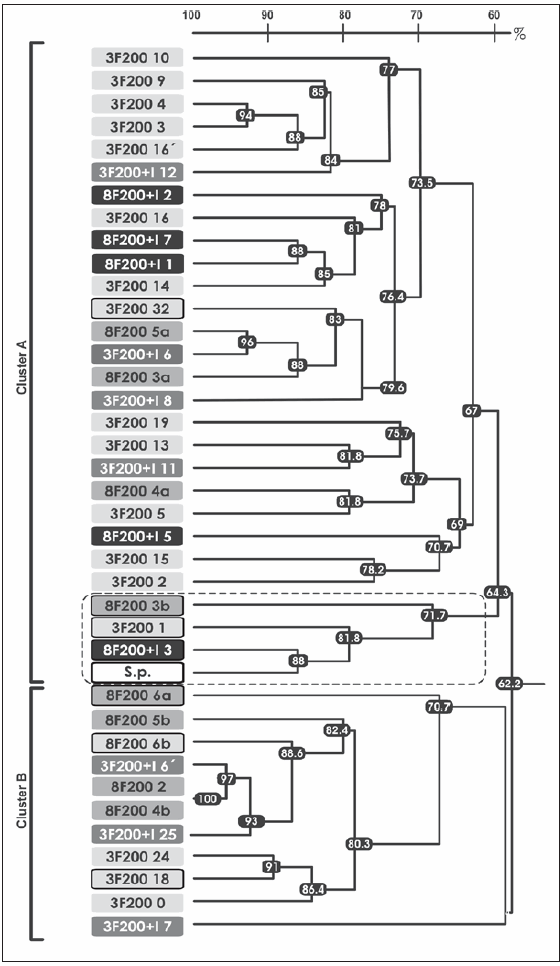
Figura 1. Dendrograma derivado del agrupamiento por ligamiento promedio no ponderado (UPGMA) de los coeficientes de similitud obtenidos mediante la comparación de los 38 cultivos aislados a partir de los consorcios 3F200, 8F200, 3F200+I y 8F200+I entre sí y con la cepa S. paucimobilis 20006FA; las distancias se expresan en porcentaje de similitud. Los cultivos aislados de los consorcios inoculados están en letra blanca (3F200+I y 8F200+I) y los aislados de los consorcios sin inocular en letra negra (3F200 y 8F200). Los aislados degradadores se encuentran recuadrados en un marco negro. Sp: S. paucimobilis 20006FA. El recuadro con línea punteada muestra el grupo donde se encuentra la cepa inoculada.
PCR-DGGE
La Figura 2a muestra los perfiles de bandas del ADN total que se obtuvieron mediante la técnica de PCR-DGGE de los cuatro consorcios estudiados. Para realizar comparaciones se incluyó el ADN de la cepa S. paucimobilis 20006FA.
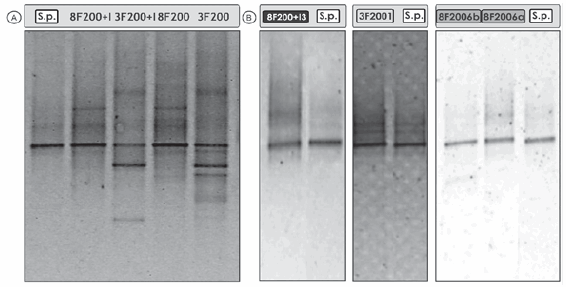
Figura 2. a) Perfiles de PCR-DGGE de los consorcios 3F200, 8F200, 3F200+I y 8F200+I. Sp: S. paucimobilis 20006FA. b) Perfiles de PCR-DGGE de los cultivos degradadores de fenantreno 3F2001, 8F2006a, 8F2006b y 8F200+I3 aislados de los consorcios 3F200, 8F200 y 8F200+I. Sp: S. paucimobilis 20006FA.
Al comparar los perfiles obtenidos a partir de los consorcios 3F200 y 3F200+I, observamos que la inoculación produjo la desaparición de algunas bandas y la aparición de otras que no se detectaron en el consorcio sin inocular (3F200), lo que podría relacionarse en el primer caso con la disminución de la diversidad bacteriana, y en el segundo caso con una dinámica estructural diferente en el consorcio 3F200+I debido a la inoculación. En tal sentido, se ha indicado que un inoculante puede producir cambios en la estructura de la comunidad microbiana, establecerse dentro de ella y desplazar a alguno de los integrantes de la comunidad microbiológica indígena (34).
También fue posible observar que los repiques sucesivos causaron una drástica disminución en el número de bandas de los perfiles obtenidos por PCR-DGGE, lo que se evidenció al comparar el consorcio 3F200 con el 8F200 y el consorcio 3F200+I con el 8F200+I. Stoffels et al. (26) estudiaron la dinámica de comunidades degradadoras de PAH utilizando cultivos discontinuos y sistemas de biofiltros, y encontraron que los primeros generaban una pérdida de la diversidad bacteriana que podía conducir a la obtención de cultivos prácticamente puros, mientras que el sistema de biofiltros seleccionaba un mayor número de cultivos bacterianos con presencia de grupos filogenéticamente poco relacionados (26). La pérdida de la diversidad encontrada en el cultivo discontinuo podría deberse a que, a diferencia de lo que ocurre en el sistema de biofiltros, ese sistema no genera ambientes con nichos ecológicos variados como para que bacterias poco relacionadas filogenéticamente puedan crecer y ejercer una función dentro del consorcio.
Otra de las causas posibles de la pérdida de la diversidad con los repiques sucesivos es el incremento en la concentración de metabolitos intermediarios, lo cual se manifiesta por el aumento de color observado en los repiques sucesivos (datos no mostrados). Estos metabolitos han demostrado ser inhibidores del crecimiento y de la actividad de las bacterias degradadoras de PAH (18). La acumulación de metabolitos intermediarios podría explicar también la limitada degradación de fenantreno (Tabla 1) y la baja recuperación de cultivos aislados capaces de metabolizar PAH en los cuatro consorcios.
En la Figura 2a también se puede visualizar que los cuatro consorcios mantuvieron la presencia de una banda intensa que se encuentra posicionada a la misma altura en el gradiente desnaturalizante que el ADN de la cepa inoculada. Por otra parte, el ADN de cuatro de las poblaciones degradadoras de PAH aisladas de los consorcios (3F200 1, 8F200+I 3, 8F200 6a y 8F200 6b) coincidió con la posición de la banda obtenida para la cepa S. paucimobilis 20006FA (Figura 2b). Los aislados 3F200 1 y 8F200+I 3 mostraron, además, una alta similitud fenotípica con la cepa de S. paucimobilis 20006FA y entre sí, por lo que se ubicaron junto a aquella en un cluster con 81,8% de similitud (Figura 1). Los miembros del género Sphingomonas parecen estar especializados en la degradación de compuestos aromáticos (35). Las Sphingomonas son el mayor grupo de bacterias degradadoras de PAH presentes en muchos sitios contaminados (4, 23) y han demostrado poseer habilidades específicas para degradar esos contaminantes. Son consideradas como importantes biocatalizadores para la remediación del suelo debido a su amplia distribución y a la diversidad de sus capacidades metabólicas frente a contaminantes orgánicos recalcitrantes (19).
Sin embargo, el análisis por RAPD mostró diferencias genotípicas entre los aislados 3F200 1 y 8F200+I 3 y la cepa S. paucimobilis 20006FA (Figura 3). Algunos estudios recientes basados en la técnica de RAPD y de otras que implican la amplificación o detección de un fragmento del gen 16S del ARNr concluyeron que, gracias a su alta sensibilidad, la técnica de RAPD es útil para diferenciar subespecies dentro de la misma especie (14, 22, 28). Esta capacidad de distinguir subespecies se fundamenta en el hecho de que el RAPD diferencia variaciones de secuencia existentes en el genoma completo de microorganismos de la misma especie.
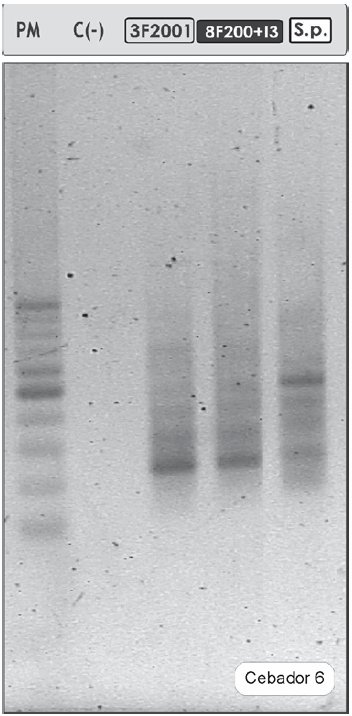
Figura 3. Perfiles de bandas obtenidos por RAPD-PCR de los cultivos 3F2001 y 8F200+I3 y de la cepa S. paucimobilis 20006FA. Se muestran los resultados obtenidos con el cebador 6: IM-1.
Por otro lado, ninguno de los ADN de los cultivos aislados del consorcio 3F200+I coincidió con la posición de la banda obtenida para la cepa S. paucimobilis 20006FA, lo que nuevamente sugiere que la cepa inoculada no pudo ser aislada de este consorcio.
Estudios previos de inoculación de la cepa S. paucimobilis 20006FA en microcosmos de suelo contaminado con fenantreno demostraron que su establecimiento produjo un fuerte impacto sobre la diversidad de la comunidad bacteriana del suelo, pero sin lograr un efecto significativo sobre la degradación del fenantreno (9). En el presente trabajo, el estudio de los consorcios bacterianos degradadores de fenantreno permitió simplificar el sistema de evaluación, con independencia de factores como la biodisponibilidad de nutrientes y del contaminante, limitación de oxígeno, etc., de modo de tratar de determinar específicamente si la inoculación con cepa S. paucimobilis 20006FA logra desplazar o acomodar las poblaciones degradadoras autóctonas.
Utilizando un esquema polifásico fue posible caracterizar desde el punto de vista de su composición bacteriana dos consorcios bacterianos degradadores de hidrocarburos. En concordancia con los resultados encontrados por Coppotelli et al. (9), se determinó que el consorcio inoculado mostró una menor diversidad bacteriana, evidenciada tanto al analizar el número de microorganismos cultivables como los patrones de bandas obtenidos por PCR-DGGE. Sin embargo, no fue posible determinar fehacientemente el establecimiento de la cepa S. paucimobilis 20006FA en el consorcio inoculado, a pesar de que el tamaño del inóculo fue equivalente al utilizado en los experimentos de Coppotelli et al. (9). Si bien los perfiles de DGGE de todos los consorcios mostraron la presencia de una banda posicionada a la misma altura que el ADN de la cepa inoculada (Figura 2), las limitaciones inherentes a esta técnica hacen que la presencia de bandas con posiciones similares en los geles de DGGE no confirme la presencia de la misma secuencia o especie bacteriana en las muestras (15). Por otra parte, ninguno de los aislados proveniente del consorcio inoculado mostró una alta similitud genotípica con la cepa inoculada.
Si bien la inoculación de la cepa S. paucimobilis 20006FA logró un impacto sobre la flora bacteriana autóctona (demostrado en el hecho de que los consorcios inoculados y sin inocular exhibieron claras diferencias en su composición), luego de 3 repiques sucesivos en medio con fenantreno no fue posible encontrar evidencias claras del establecimiento de la cepa. Esto podría deberse a que, a diferencia de lo que ocurre en el suelo, donde hay numerosos factores que mitigan la competencia (multiplicidad de recursos, heterogeneidad espacial, etc.) (3), en las condiciones establecidas para los cultivos discontinuos y a la tasa de dilución utilizada, la cepa no sería capaz de competir y mantenerse dentro del consorcio.
Estudios futuros en sistemas continuos o con el fenantreno absorbido a soportes que simulen las condiciones del suelo permitirán dilucidar no sólo el efecto de la inoculación con la cepa S. paucimobilis 20006FA sobre las poblaciones degradadoras autóctonas, sino además establecer diferencias respecto de la acumulación de productos de degradación del fenantreno y de la posible cadena trófica de degradación del mismo.
Agradecimientos: Este trabajado ha sido parcialmente financiado por la Agencia Nacional de Promoción Científica y Tecnológica (PICT2006-884). Madueño L. tiene una beca de postgrado cofinanciada por CONICET y OIL M&S. Coppotelli B. es becaria postdoctoral del CONICET.
1. Abbondanzi F, Bruzzi L, Campisi T, Frezzati A, Guerra R, Iacodini A. Biotreatability of polycyclic aromatic hydrocarbons in brackish sediments: preliminary studies of an integrated monitoring. Int Biodeterior Biodegradation 2006; 57: 214-21. [ Links ]
2. Allard AS, Remberger M, Neilson AH. The negative impact of aging on the loss of PAH components in a creosotecontaminated soil. Int Biodeterior Biodegradation 2000; 46: 43-9. [ Links ]
3. Atlas RM, Bartha R. Ecología Microbiana y Microbiología Ambiental. Addison Wesley, Madrid, 2000, p. 185. [ Links ]
4. Bastiaens L, Springael D, Wattiau P, Harms H, deWachter R, Verachtert H, et al. Isolation of adherent polycyclic aromatic hydrocarbon (PAH)-degrading bacteria using PAHsorbing carriers. Appl Environ Microbiol 2000; 66: 1834-43. [ Links ]
5. Berthe-Corti L, Del Panno MT, Hulsch R, Morelli IS. Bioremediation and bioaugmentation of soils contaminated with polyaromatic hydrocarbons. Curr Trends Microbiol 2007; 3: 1-30. [ Links ]
6. Bogart A, Hemmingsen B. Enumeration of phenanthrene degrading bacteria by an overlay technique and its use in evaluation of petroleum contamined sites. Appl Environ Microbiol 1992; 58: 2579-82. [ Links ]
7. Cavalca L, Colombo M, Larcher S, Gigliotti C, Collina E, Andreoni V. Survival and naphthalene-degrading activity of Rhodococcus sp. strain 1BN in soil microcosm. J Appl Microbiol 2002; 92: 1058-65. [ Links ]
8. Coppotelli BM. Obtención de inoculantes bacterianos y evaluación de su aplicación en procesos de biorremediación de suelos contaminados con PAH. Trabajo de Tesis. Facultad de Ciencias Exactas. UNLP, 2008. [ Links ]
9. Coppotelli BM, Ibarrolaza A, Del Panno MT, Morelli IS. Effects of the inoculant strain Sphingomonas paucimobilis 20006FA on soil bacterial community and biodegradation in phenanthrene-contaminated soil. Microb Ecol 2008; 55: 173-83. [ Links ]
10. Cunliffe M, Kertesz MA. Effect of Sphingobium yanoikuyae B1 inoculation on bacterial community dynamics and polycyclic aromatic hydrocarbon degradation in aged a freshly PAH-contaminated soils. Environ Pollut 2006; 144: 228-37. [ Links ]
11. El Fantroussi S, Agathos SN. Is bioaugmentation a feasible strategy for pollutant removal and site remediation? Curr Opin Microbiol 2005; 8: 268-75. [ Links ]
12. Giordano A, Stante L, Pirozzi F, Raffaele C, Bortone G. Sequencing batch reactor performance treating PAH contamined lagoon sediments. J Hazard Mater 2005; B119: 159-66. [ Links ]
13. Gomes N, Kosheleva I, Abraham W, Smalla K. Effects of the inoculant strain Pseudomonas putida KT2442 (pNF142) and naphthalene contamination on the soil bacterial community. FEMS Microbiol Ecol 2005; 54: 21-33. [ Links ]
14. Høl L, Dalsgaard A, Larsen JL, Warner JM, Oliver JD. Comparison of ribotyping and randomly amplified polymorphic DNA PCR for characterization of Vibrio vulnificus. Appl Environ Microbiol 1997; 63: 1674-8. [ Links ]
15. Jackson CR, Roden EE, Churchill PF. Denaturing gradient gel electrophoresis can fail to separate 16S rDNA fragments with multiple base differences. Mol Biol Today 2000; 1: 49-51. [ Links ]
16. Johnsen AR, Winding A, Karlson U, Roslev P. Linking of microorganisms to phenanthrene metabolism in soil by analysis of (13)C-labeled cell lipids. Appl Environ Microbiol 2002; 68: 6106-13. [ Links ]
17. Johnson DL, Maguire KL, Anderson DR, McGrath SP. Enhanced dissipation of chrysene in planted soil: the impact of a rhizobial inoculum. Soil Biol Biochem 2004; 36: 33-8. [ Links ]
18. Kanzuga C, Aitken M. Products from the incomplete metabolism of pyrene by polycyclic aromatic hydrocarbon degrading bacteria. Appl Environ Microbiol 2000; 66: 1917-22. [ Links ]
19. Leys NM, Ryngaert A, Bastiaens L, Verstraete W, Top EM, Springael D. Occurrence and phylogenetic diversity of Sphingomonas strains in soils contaminated with polycyclic aromatic hydrocarbons. Appl Environ Microbiol 2004; 70: 1944-55. [ Links ]
20. Maliszewska-Kordybach B, Smreczak B. Habitual function of agricultural soils as affected by heavy metals and polycyclic aromatic hydrocarbons contamination. Environ Int 2003; 28: 719-28. [ Links ]
21. Muyzer G, Brinkhoff T, Nübel U, Santegoedes C, Schäfer H, Wawer C. Denaturing gradient electrophoresis (DGGE) in microbial ecology. En: Akkermanns ADL, van Elsas JD, de Brujin F, editors. Molecular Microbial Ecology Manual. Dordrecht, Kluwer Academic Press, 1998, p.1-27. [ Links ]
22. Pérez Pulido R, Nabil Ben O, Hikmate A, López RL, Martínez Camañero M, Gálvez A. Microbiological study of lactic acid fermentation of caper berries by molecular and culture-dependent methods. Appl Environ Microbiol 2005; 71: 7872-9. [ Links ]
23. Pinyakong O, Habe H, Supaka N, Pinpanichkarn P, Juntongjin K, Yoshida T, et al. Identification of novel metabolites in the degradation of phenanthrene by Sphingomonas sp. strain P2. FEMS Microbiol Lett 2000; 191: 115-21. [ Links ]
24. Reasoner D, Geldreich E. A new medium for the enumeration and subculture of bacteria from potable water. Appl Environ Microbiol 1985; 49: 1-7. [ Links ]
25. Sokal RR, Michener CD. A statistical method for evaluating systematic relationships. Univ Kansas Sci Bull 1958; 38: 1409-38. [ Links ]
26. Stoffels M, Amann R, Ludwig W, Hekmat D, Scheifer KH. Bacterial community dynamics during star-up of a tricklebed biorector degrading aromatic compounds. Appl Environ Microbiol 1998; 64: 930-9. [ Links ]
27. Story SP, Parker SH, Kline JD, Tzeng TRJ, Mueller JG, Kline EL. Identification of four structural genes and two putative promoters necessary for utilization of naphtalene, phenanthrene, and fluoranthrene by Sphingomonas paucimobilis var EPA 505. Gene 2000; 260: 155-69. [ Links ]
28. Urso R, Rantsiou K, Dolci P, Rolle L, Comi G, Cocolin L. Yeast biodiversity and dynamics during sweet wine production as determined by molecular methods. FEMS Yeast Res 2008; 8: 1053-62. [ Links ]
29. van Veen J, van Overbeek L, van Elsas J. Fate and activity of microorganisms introduced into soil. Microbiol Mol Biol Rev 1997; 61: 121-35. [ Links ]
30. Vecchioli GI, Del Panno MT, Painceira, MT. Use of selected authochtonous soil bacteria to enhance degradation of hydrocarbons in soil. Environ Pollut 1990; 67: 249-58. [ Links ]
31. Vecchioli G, Constanza O, Giorgieri S, Remmler M. Extend of cleaning achievable for bioremediation of soil contaminated with petrochemical sludges. J Chem Tech Biotechnol 1997; 70: 331-6. [ Links ]
32. Venosa AD, Suidan MT, Wrenn BA, Strohmeier KL, Haines JR, Loye Eberhart B, et al. Bioremediation of an experimental oil spill on the shoreline of Delaware Bay. Environ Sci Technol 1996; 30: 1764-75. [ Links ]
33. Ventaka Mohan S, Shailaja S, Krishna MR, Reddy KB, Sarma PN. Bioslurry phase degradation of di-ethyl phthalate (DEP) contaminated soil in periodic discontinuous mode operation: influence of bioaugmentation and substrate partition. Process Biochem 2006, 41: 644-52. [ Links ]
34. Viñas M, Sabate J, Espuny MJ, Solanas AM. Bacterial community dynamics and polycyclic aromatic hydrocarbon degradation during bioremediation of heavily creosote-contaminated soil. Appl Environ Microbiol 2005; 71: 7008-18 [ Links ]
35. Wattiau P, Springael D, Agathos SN, Wuertz S. Use of the pAL5000 replicon in PAH-degrading mycobacteria: application for strain labelling and promoter probing. Appl Microbiol Biotechnol 2002; 59: 700-5. [ Links ]
Recibido: 09/09/08
Aceptado: 08/04/09














