Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Revista argentina de microbiología
versión impresa ISSN 0325-7541versión On-line ISSN 1851-7617
Rev. argent. microbiol. v.41 n.3 Ciudad Autónoma de Buenos Aires jul./sep. 2009
Serovariedades de Salmonella enterica subespecie enterica en porcinos de faena y su resistencia a los antimicrobianos
M. P. Ibar1*, G. Vigo2, P. Piñeyro3, M. I. Caffer4, P. Quiroga5, C. Perfumo3, D. Centrón5, G. Giacoboni1
1Laboratorio de Diagnóstico e Investigaciones Bacteriológicas,
2Cátedra de Microbiología,
3Cátedra de Patología Especial. Facultad de Ciencias Veterinarias, Universidad Nacional de La Plata. Calle 60 y 118 (1900) La Plata;
4Servicio de Enterobacterias, INEI-ANLIS Dr. Carlos G. Malbrán, Ciudad Autónoma de Buenos Aires;
5Departamento de Microbiología, Parasitología e Inmunología, Facultad de Medicina, UBA. Ciudad Autónoma de Buenos Aires, Argentina.
*Correspondencia. E-mail: mariela.ibar@fcv.unlp.edu.ar
RESUMEN
Se realizó un estudio para determinar la prevalencia de Salmonella y sus serovariedades en cerdos de faena, para evaluar sus perfiles de resistencia a los antimicrobianos y para conocer la presencia de integrones de clase 1 como posibles reservorios de resistencia. A partir de un total de 386 muestras de porcinos provenientes de cuatro frigoríficos de las provincias de Buenos Aires y de Santa Fe (Argentina), se identificaron 93 (24,1%) cepas de Salmonella enterica subespecie enterica, 52 (55,9%) de contenido cecal y 41 (44,1%) de nódulo linfático ileocecal. Se hallaron 13 serovariedades de S. enterica, las más prevalentes fueron S. Schwarzengrund, S. Heidelberg, S. subespecie I 6,8:e,h:-, S. Derby y S. Bredeney. Se probaron 15 antimicrobianos por el método de dilución en agar: amikacina, gentamicina, ciprofloxacina, cefalotina, cefotaxima, enrofloxacina, fosfomicina, polimixina-B, tetraciclina, cloranfenicol, estreptomicina, trimetoprima-sulfametoxazol, ampicilina, nitrofurantoína y ácido nalidíxico. Según se estableció mediante la determinación de la CIM, el 73% de las cepas de S. enterica subespecie enterica fueron sensibles a todos los antimicrobianos probados. Se observó resistencia a tetraciclina en 24 (25,8%) de las 93 cepas, a cloranfenicol en 22 (23,7%), a estreptomicina en 22 (23,7%) a trimetoprima-sulfametoxazol en 20 (21,5%), a ampicilina en 18 (19,4%), a nitrofurantoína en 3 (3,2%) y a ácido nalidíxico en 3 (3,2%). Algunos aislamientos de S. Typhimurium, S. Heildelberg, S. Derby y S. Orion presentaron multirresistencia y portaban el gen de la integrasa clase 1. Los mayores porcentajes de resistencia correspondieron a los antimicrobianos habitualmente utilizados en veterinaria y en las explotaciones porcinas.
Palabras clave: Cerdos; Salmonella; Multirresistencia a los antimicrobianos; Integrones de clase 1; Salud pública
ABSTRACT
Serovars of Salmonella enterica subspecies enterica and its antimicrobial resistance in slaughterhouse pigs. A study was carried out in order to determine the prevalence of Salmonella and its serovars among porcine slaughterhouses, to evaluate the antimicrobial resistance profiles and to know the presence of class 1 integrons as possible reservoir of resistance. From a total of 386 samples from four porcine slaughterhouses of Buenos Aires and Santa Fe Provinces (Argentina), 93 (24,1%) Salmonella enterica subspecies enterica strains were identified, 52 (55,9%) from cecal contents and 41 (44,1%) from ileocecal lymph nodes. Thirteen serovars of S. enterica were found, the most prevalent were: S. Schwarzengrund, S. Heidelberg, S. subspecie I 6,8:e,h:-, S. Derby and S. Bredeney. Fifteen antimicrobials by the agar dilution method were tested: amikacin, gentamicin, ciprofloxacin, cephalotin, cefotaxime, enrofloxacin, fosfomycin, polimixin-B, tetracycline, chloramphenicol, streptomycin, trimethoprim-sulfamethoxazole, ampicillin, nitrofurantoin, and nalidixic acid. According to the CIM determination, 73% Salmonella enterica subspecies enterica strains were sensible to all the antimicrobials tested. Antimicrobial resistance was observed to tetracycline in 24 (25,8%) of 93 strains, to chloramphenicol in 22 (23,7%), to streptomycin in 22 (23,7%), to trimethoprim-sulfamethoxazole in 20 (21,5%), to ampicillin in 18 (19,4%), to nitrofurantoin in 3 (3,2%) and to nalidixic acid in 3 (3,2%). Some isolates of S. Typhimurium, S. Heidelberg, S. Derby, S. Orion showed multidrug resistance and carried the class 1 integrase gene. The highest percentage of resistance corresponded to the antimicrobials currently used in veterinary and porcine farms.
Key words: Pigs; Salmonella; Antimicrobial multidrug resistance; Class 1 integrons; Public health
INTRODUCCIÓN
Las bacterias pertenecientes al género Salmonella incluyen un gran número de serovariedades que habitan el tracto intestinal de diversas especies animales, domésticas y silvestres (4, 7). Algunas de ellas tienen predilección por un hospedador particular y causan enfermedades bien definidas en el hombre y los animales, mientras que otras se encuentran en un gran número de reservo-rios y de allí se diseminan al medio ambiente. En los porcinos, la salmonelosis con presentación clínica está asociada a S. Choleraesuis, aunque también pueden presentarse cuadros relacionados con S. Typhimurium y excepcionalmente con S. Typhisuis. En el intestino de esta especie animal, se pueden encontrar serovariedades potencialmente patógenas para el ser humano y, por consiguiente, los cerdos pueden constituir una fuente de infección a través del consumo de sus subproductos y un factor de riesgo para la salud pública (10).
En sistemas de cría intensiva de cerdos, la utilización de antimicrobianos es una práctica frecuente. De esta manera, se favorece la selección de bacterias que poseen mecanismos de resistencia a los antibióticos administrados y aumenta el número de animales portadores de bacterias resistentes. Muchos de los genes que codifican para mecanismos de resistencia a los antibióticos se localizan en plásmidos, transposones e integrones, estructuras típicamente sujetas a la transferencia horizontal de genes. Varios estudios han demostrado que la presencia del gen de la integrasa de clase 1 está asociada a un perfil de multirresistencia antibiótica en la familia Enterobacteriaceae, independientemente de la especie y del origen de la muestra (2, 13, 20).
La información disponible referida a las serovariedades de Salmonella aisladas de cerdos en nuestro país, y la resistencia antibiótica que estas acarrean, se remite a hallazgos clínicos aislados y a escasos estudios planificados (17, 23). La presencia de integrones de clase 1 en Salmonella spp. de origen porcino no ha sido estudiada en nuestro país.
El objetivo de este trabajo fue conocer la prevalencia de Salmonella enterica subespecie enterica y de sus diferentes serovariedades en los cerdos presentes en las plantas de faena, estudiar sus perfiles de resistencia a los antimicrobianos y conocer la presencia de integrones de clase 1 como posibles reservorios de resistencia anti-microbiana.
MATERIALES Y MÉTODOS
Origen de las muestras
Entre los meses de agosto y octubre del año 2007 se recolectaron muestras en las plantas de faena de 4 frigoríficos, 3 ubicados en la provincia de Buenos Aires y uno en la de Santa Fe, identificados con las letras A, B, C y D. El número de animales muestreados en cada frigorífico fue de 80, 106, 40 y 160, respectivamente. En total se tomaron 386 muestras: 193 de contenido cecal y 193 de nódulo linfático ileocecal correspondiente a un mismo animal. Las muestras fueron rotuladas, refrigeradas y enviadas en envases plásticos estériles al laboratorio para su posterior procesamiento.
Procesamiento de las muestras
La marcha bacteriológica se realizó inmediatamente después de la llegada de las muestras al laboratorio, de acuerdo con la norma FDA/BAM (8).
De cada animal se tomaron 10-25 g de contenido cecal y el nódulo linfático ileocecal. El nódulo linfático fue sumergido en alcohol y flameado a la llama para descontaminar. Ambas muestras fueron diluidas 1/10 en agua peptonada tamponada (Laboratorios Britania, CABA, Argentina) e incubadas a 37 °C durante 24 h (preenriquecimiento). Posteriormente, 1 ml del caldo de preenriquecimiento fue inoculado en 10 ml de caldo tetrationato (Laboratorios Britania) e incubado durante 24 h a 37 °C (enriquecimiento). Una ansada de este caldo fue sembrada en agar entérico Hektoen (Laboratorios Britania) que contenía 10 µg/ml de novobiocina (Sigma Chemical Co, St. Louis, EE.UU.); luego se incubó a 37 °C durante 48 h.
Aislamiento, identificación y serotipificación de Salmonella spp.
Dos colonias con características compatibles con Salmonella spp. fueron sembradas en agar tripticasa de soja (Laboratorios Britania) e incubadas durante 48 h a 37 °C. La identidad de los cultivos fue confirmada por pruebas bioquímicas convencionales. Para la identificación de S. enterica subespecie enterica se utilizaron las siguientes pruebas: fermentación de dulcitol, sorbitol, salicina, lactosa, mucato; utilización del malonato, ONPG, tartrato de Jordan y gelatina de Kohn (12).
La serotipificación de los aislamientos identificados como S. enterica fue realizada de acuerdo con el esquema de Kauffman-White (11), utilizando antisueros polivalentes, monovalentes y factores somáticos y flagelares producidos en el Servicio de Antígenos y Antisueros del Instituto Nacional de Producción de Reactivos y Biológicos-ANLIS Dr. Carlos G. Malbrán.
Pruebas para determinar la sensibilidad a los antimicrobianos
Se determinó la concentración inhibitoria mínima(CIM) por el método de dilución en agar según las normas del Clinical Laboratory Standards Institute (CLSI) (6).
Se ensayaron los siguientes antimicrobianos: ampicilina (AMP) (ICN Biomedicals Inc., Ohio, EE.UU.), cloranfenicol (CMP) (ParafarmR, Buenos Aires, Argentina), tetraciclina (TET) (Vetanco S.A., Buenos Aires, Argentina), estreptomicina (S) (Waco Pure Chemical Industries, Ltd, Osaka, Japón), gentamicina (GEN) (Sigma Chemical Co, St. Louis, EE.UU.), amikacina (AKN) (Sigma Chemical Co), cefalotina (CEF) (ICN Biomedicals Inc.), ácido nalidíxico (NAL) (Sigma Chemical Co), trimetoprimasulfa-metoxazol (TMS) (Vetanco S.A.), cefotaxima (CTX) (ICN Bio-medicals Inc.), enrofloxacina (ENR) (Vetanco S.A.), ciprofloxacina (CIP) (ICN Biomedicals Inc.), polimixina B (POL) (ICN Biomedicals Inc.), fosfomicina (FOS) (Cevasa S.A., Buenos Aires, Argentina) y nitrofurantoína (NIT) (Sigma Chemical Co). La cepa de referencia utilizada fue Escherichia coli ATCC 25922.
Determinación de la presencia del gen de la integrasa 1 (IntI 1)
Los aislamientos de S. enterica subespecie enterica se sembraron en placas de agar tripticasa de soja, se incubaron 24 h a 37 °C y una colonia se subcultivó en caldo tripticasa de soja y se incubó a 37 °C durante 12 h. La extracción de ADN se realizó mediante los reactivos del kit WizardR Genomic, (Promega Corporation, Madison, WI, EE.UU.) para bacterias gram-negativas. La concentración de ADN se midió con espectrofotómetro y se diluyó con agua destilada estéril para obtener una concentración final de 5 µg/µl.
La reacción de PCR se llevó a cabo en un volumen final de 25 µl que contenía 5 µl de ADN diluido y los siguientes reactivos: agua destilada estéril 16,5 µl; buffer 2,5 µl; MgCl2 1,5 mM; cebadores (Invitrogen, Buenos Aires, Argentina) 0,2 µM cada uno (0,5 µl de cada uno); mezcla de dNTPs (Promega Corporation, Madison, WI, EE.UU.), 25 mM de cada uno (0,25 µl) y 2,5 U de Taq polimerasa en 0,25 µl (GoTaq, Promega Corporation, Madison, EE.UU.).
Para la determinación del gen de la integrasa de clase 1 se utilizaron los siguientes cebadores: Inti1r 5- cga ggc ata gac tgt ac-3 e Inti1f¨ 5-ttc gaa tgt cgt aac cgc-3 (17). Se inició el programa de la PCR con una desnaturalización a 94 °C durante 1 min. Luego se realizaron 35 ciclos, constando cada uno de las siguientes etapas: iniciación 95 °C durante 5 min, hibridación a 52 °C durante 20 seg y extensión a 72 °C durante 20 seg. Como control positivo se utilizó la cepa E 705 (18) y como control negativo agua destilada estéril. Posteriormente se corrieron 12 µl en gel de agarosa al 1% a 107 V durante 40 min, utilizando un marcador de 1000 pb (MilMarker, Biodynamics, Buenos Aires, Argentina). El gel se visualizó con un transilu-minador de luz UV.
RESULTADOS
Aislamiento, identificación y serotipificación de Salmonella spp.
Se obtuvieron 93 cepas de Salmonella enterica subespecie enterica de las 386 muestras procesadas (24,1%), de las cuales 52 (55,9%) fueron de contenido cecal y 41 (44,1%) de nódulo linfático ileocecal. Las cepas aisladas se distribuyeron en 13 serovariedades de Salmonella; la proporción correspondiente a cada una se expresa en la Figura 1. La distribución de las sero-variedades según el frigorífico y el tipo de muestra de la que provinieron se describen en la Tabla 1.
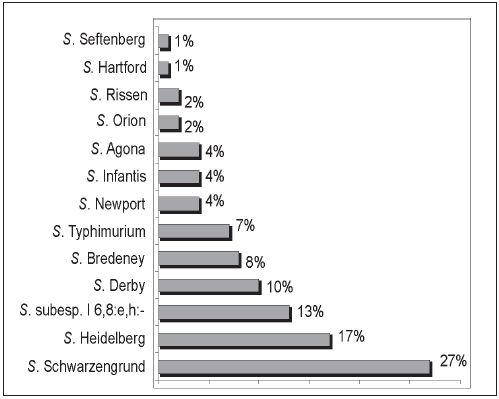
Figura 1. Serovariedades de Salmonella enterica subesp. enterica aisladas en cerdos de faena.
Tabla 1. Distribución de serovariedades de Salmonella subespecie enterica aisladas de cerdos en cuatro frigoríficos argentinos
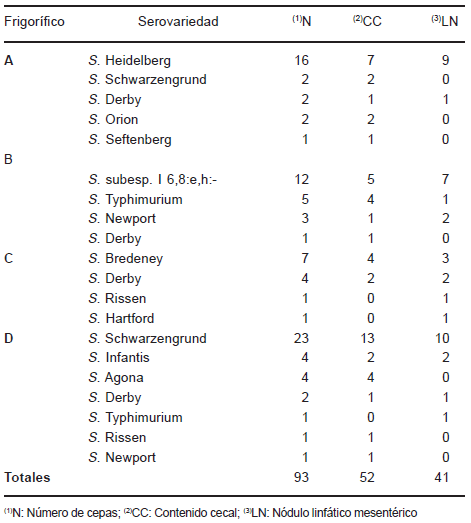
En 20 animales se aisló S. enterica, tanto de contenido cecal como de nódulo linfático ileocecal. Las combinaciones de serovariedades halladas fueron respectivamente, S. Bredeney/S. Bredeney (1 animal); S. Derby/S. Bredeney (1 animal); S. Newport/S. Newport (1 animal); S. subesp. I 6,8:e,h:-/ S. subesp. I 6,8:e,h:- (4 animales); S. Typhimurium/ S. subesp. I 6,8:e,h:- (1 animal); S. Schwarzengrund/S. Heidelberg (2 animales); S. Heidelberg/ S. Heidelberg (2 animales); S. Schwarzen-grund/ S. Schwarzengrund (4 animales); S. Infantis/S. Schwarzengrund (1 animal); S. Schwarzengrund/S. Infantis (2 animales); S. Newport/ S. Schwarzengrund (1 animal).
Concentración inhibitoria mínima (CIM)
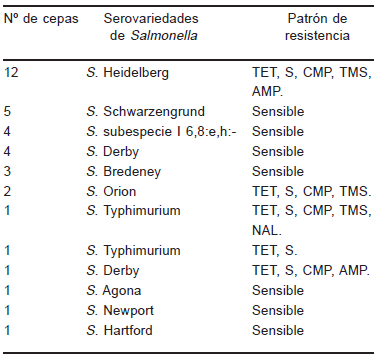
La prueba de sensibilidad a los antimicrobianos por el método de dilución (CIM) mostró que 68 (73%) de las 93 cepas fueron sensibles a todos los antimicrobianos, mientras que 25 (27%) de las 93 cepas fueron resistentes a uno o más de los siguientes antimicrobianos: AMP, TET, CMP, S, TMS, NIT y NAL. Los resultados del ensayo de la CIM se detallan en la Tabla 2. Se observaron resistencias a 1, 2, 4, 5 y 6 antimicrobianos. Los patrones de resistencia se detallan en la Figura 2.
Tabla 2. Sensibilidad a los antimicrobianos de los aislamientos de Salmonella enterica subesp. enterica basada en la determinación de la CIM
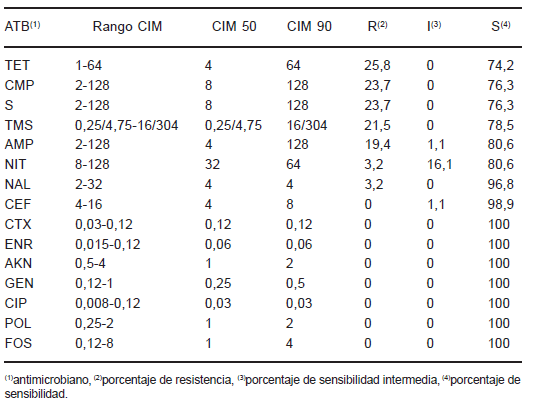
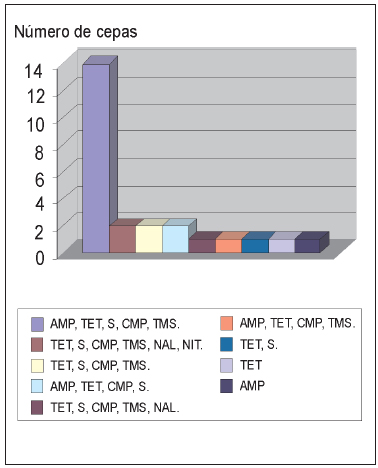
Figura 2. Frecuencias relativas de los patrones de resistencia a los antimicrobianos hallados en los aislamientos de Salmonella enterica subesp. enterica según la CIM
Determinación del gen intI 1
La expresión del gen de la integrasa 1 se observó en 36 (38,7%) de los 93 aislamientos de S. enterica identificados. Diecisiete de las cepas portadoras del gen presentaron multirresistencia a 5, 4 y 2 antimicrobianos (TET, S, CMP, TMS, AMP y NAL), mientras que las 19 restantes fueron sensibles a todos los antibióticos probados. La mayor prevalencia de integrones de clase 1 se encontró en el frigorífico A (44%) y la menor en el frigorífico D (16%). Los frigoríficos B y C presentaron prevalencias de 22% y 19%, respectivamente.
Los patrones de resistencia y las serovariedades de S. enterica subespecie enterica en las que se pudo determinar la presencia de integrones de clase 1 se muestran en la Tabla 3.
Tabla 3. Serovariedades de Salmonella enterica subesp. enterica que expresaron el gen de la integrasa 1 y sus patrones de resistencia a los antimicrobianos
DISCUSIÓN
En las muestras procesadas de contenido cecal y de nódulo linfático ileocecal, las 5 serovariedades de Sal-monella entérica más prevalentes fueron S. Schwarzen-grund, S. Heidelberg, S. subesp. I 6,8:e,h:-, S. Derby y S. Bredeney. Estas serovariedades difieren de las encontradas por Vigo et al. (22) en la provincia de Buenos Aires, ya que en dicho estudio predominaron las serova-riedades S. Typhimurium, S. Ohio y S. Rissen.
En concordancia con datos de otros países, en este estudio detectamos aislamientos de S. Typhimurium, que es una de las serovariedades usualmente prevalentes y que representa un riesgo para la salud pública (18).
Los aislamientos de Salmonella enterica subesp. enterica fueron más frecuentes en el contenido cecal que en el nódulo linfático ileocecal (55,9% y 44,1%, respectivamente). A su vez, en el contenido cecal se hallaron cuatro serovariedades que no se encontraron en el nódulo linfático ileocecal (S. Agona, S. Rissen, S. Newport, S. Orion); las 3 primeras pertenecieron al mismo frigorífico (D), donde se registró la mayor diversidad de serova-riedades. La serovariedad S. Hartford sólo se encontró en un animal del frigorífico C a partir de nódulo linfático, aunque no de contenido cecal. Los resultados obtenidos demuestran que la siembra simultánea del nódulo linfático ileocecal y del contenido cecal de un mismo animal aumenta la probabilidad de obtener más aislamientos y mayor diversidad de serovariedades.
La diversidad de serovariedades halladas y su localización se podrían atribuir a que los animales se contaminan en el camión de transporte al frigorífico, en los corrales de espera (15) y en la etapa de evisceración, donde se produce la contaminación al cortar los intestinos y diseminar la materia fecal a la carcasa (3). Según Loynachan et al. (14), Salmonella spp. pueden producir infecciones en numerosos órganos tales como tonsilas, colon, ciego, nódulo linfático y timo a partir de las 3 horas posinoculación, tiempo que se considera compatible con el de transporte de los animales a su destino final.
Si bien otros autores encontraron mayor proporción en el nódulo linfático ileocecal (18,8%) que en el contenido del ileon (13,9%), ambas fracciones representan una importante fuente de contaminación en los frigoríficos (21).
Los antibióticos frente a los cuales Salmonella enterica subesp. enterica mostró resistencia fueron: TET (25,8%), CMP (23,7%), S (23,7%), TMS (21,5%), AMP (19.4%), NIT (3,2%) y NAL (3,2%). Resultados similares hallaron Castagna et al. (5) en Brasil, quienes también aislaron Salmonella spp. de nódulo linfático y contenido cecal, y observaron cepas resistentes a TET, AMP, S y NAL, aunque para CMP tuvieron menor proporción de cepas resistentes (16,6%). Los hallazgos de este trabajo también coincidieron con los de los autores citados respecto de los bajos niveles de resistencia a las quinolonas y del porcentaje de cepas con multirresistencia (27%), que en nuestro caso se asoció a las serovariedades S. Typhi-murium, S. Heildelberg, S. Derby y S. Orion.
Otros trabajos coinciden en los valores de la resistencia a TET y S, pero presentan mayor proporción de multirresistencia, sobre todo en S. Typhimurium con el patrón ampicilina-cloranfenicol-estreptomicina y tetraci-clina (R-ACSSuT) relacionado con el fagotipo DT 104 (1, 9), el cual no ha sido identificado en este estudio. En nuestro país hay registros de S. enterica spp. aisladas de cerdos, con multirresistencia a seis, cinco y cuatro antimicrobianos(AKN-S-TET-NAL-COL-CMP; AMP-GEN-S-NAL-CMP y S-NAL-CMP-NIT) en diferentes patrones, correspondientes a S. Typhimurium y S. Ohio (22).
Los mayores porcentajes de resistencia coinciden con los antibióticos corrientemente utilizados en medicina veterinaria y en la explotación porcina. En estudios anteriores, donde se analizó la resistencia a antimicrobianos de aislamientos de Escherichia coli provenientes de materia fecal de cerdos, también se observó que los mayores niveles de resistencia correspondieron a los antibióticos AMP, TET y S, con el mismo perfil de multirresistencia (16).
No existen datos sobre estudios clínicos referidos a proporción de cepas de Salmonella enterica subesp. enterica sensibles a antibióticos y portadoras del gen intI1. Sin embargo, la alta frecuencia aquí detectada, que llega al 38%, sugiere que el medio ambiente podría jugar un papel muy importante como reservorio de la mul-tirresistencia a antibióticos. Los antecedentes de la presencia de integrones de clase 1 en aislamientos de Salmonella spp. de origen clínico en la Argentina corresponden a cepas que exhiben el patrón de resistencia a los antimicrobianos ampicilina, cefotaxima, ceftazidima, amikacina y gentamicina (19). En nuestro estudio no se registraron cepas resistentes a gentamicina. Diecisiete de las 36 cepas que resultaron positivas para el gen de la integrasa 1 presentaron resistencia a 2 o más antimi-crobianos, en tanto que 19 fueron sensibles. Las cepas multirresistentes portadoras del gen intI1 tuvieron en común la resistencia a TET, S, CMP y TMS, excepto S. Derby que no presentó resistencia a TMS.
La gran cantidad y diversidad de serovariedades de S. enterica aisladas en este estudio de cerdos de frigoríficos, así como el considerable porcentaje de cepas portadoras de integrones de clase 1, entre las que se incluyeron varias multirresistentes, demuestran que sería importante tomar medidas higiénico-sanitarias en los corrales de espera y principalmente, en la planta de faena del frigorífico, para evitar la contaminación de las carcasas y su diseminación, ya que eso implica un gran riesgo para la salud pública.
Agradecimientos: nuestro agradecimiento por la provisión de los antisueros al Servicio de Antígenos y Antisueros. ANLIS Dr. Carlos G. Malbrán. Este trabajo fue subsidiado por FONCyT- ANPCyT, PICT2005-33987.
1. Agustin AI, Carramiñana JJ, Rota C, Herrera A. Antimicrobial resistance of Salmonella spp. from pigs at slaughter in Spain in 1993 and 2001. Lett Appl Microbiol 2005; 41: 39-44. [ Links ]
2. Álvarez-Fernández M, Rodríguez-Souza T, Brey-Fernández E, López- Meléndez C, Piñeiro L. Asociación entre integrones de clase 1 con resistencia a múltiples antimicrobianos y plásmidos conjugativos en Enterobacteriaceae. Rev Esp Quimioterap 2003; 16: 394-7. [ Links ]
3. Berends BR, Van Knapen F, Snijders JMA, Mossel DAA. Identification and quantification of risk factors regarding Salmonella spp. on pork carcasses. Int J Food Microbiol 1997; 36: 199-206. [ Links ]
4. Cabello C, Cabello F. Zoonosis con reservorios silvestres: amenazas a la Salud Pública y a la economía. Rev Med Chil 2008; 136: 385-93. [ Links ]
5. Castagna SMF, Bessa MC, Carvalho M, Cardoso M, Costa M. Antimicrobial resistent patterns of Salmonella spp. isolated from slaughtered pigs in the state of Rio Grande do Sul Brasil. Arq Fac Vet UFRGS 2001; 29: 44-9. [ Links ]
6. Clinical and Laboratory Standards Institute. Disk diffusion. Performance standards for antimicrobial susceptibility testing; 15th informational supplement, 2005; M100-S15. Wayne, Pa, USA. [ Links ]
7. Davies Ph, Dalziel R, Gibberns JC, Wilesmith JW, Ryan JM, Evans SJ, et al. National survey for Salmonella in pigs, cattle and sheep at slaughter in Great Britain (1999-2000). J Appl Microbiol 2005; 96: 750-60. [ Links ]
8. Food and Drug Administration. Bacteriological Analytical Manual. 8th Edition USA. 1995; Chapter 5, Salmonella. [ Links ]
9. Gebreyes WA, Altier C. Molecular characterization of multidrug-resistant Salmonella enterica subesp. enterica serovar Typhimurium isolates from swine. J Clin Microbiol 2002; 40: 2813-22. [ Links ]
10. Griffith RW, Schwartz KJ, Meyerholdz DK. Salmonella. En: Straw BE, Zimmerman JJ, D´Allaire S, Taylor D, editors. Diseases of Swine, 9th edition. Blackwell Publishing, 2006, p. 739-54. [ Links ]
11. Grimont PAD, Weill FX. Antigenic formulae of the Salmonella serovars. WHO Collaborating Centre for Reference and Research on Salmonella, 9th edition. Institut Pasteur, Paris, France, 2007. [ Links ]
12. Koneman EW, Allen SD, Janda WM, Schreckenberg PC, Winn WC. Enterobacteraceae. En: Koneman EW, editor. Diagnóstico Microbiológico. Buenos Aires (BA), Editorial Médica Panamericana S.A. Springer, 1999, p. 171-250. [ Links ]
13. Leverstein-van Hall MA, Blok HEM, Donders ART, Paauw A, Fluit AC, Verhoef J. Multidrug resistance among Enterobacteriaceae is strongly associated with the presence of integrons and is independent of species or isolate origin. J Infect Dis 2003; 187: 251-9. [ Links ]
14. Loynachan AT, Nugen JM, Erdman MM, Harris DL. Acute infection of swine by various Salmonella serovars. J Food Prot 2004; 67: 1484-8. [ Links ]
15. Matthew M, Erdman BS, Sthephanie D, Wedel BS, Harris DL. Genotypic and phenotypic comparison of swine Salmonella isolates from farm and abattoir. J Swine Health Prod 2003; 11: 169-72. [ Links ]
16. Moredo FA, Vigo GB, Cappuccio JA, Piñeyro P, Perfumo CJ, Giacoboni GI. Resistencia a los antimicrobianos de aislamientos de Escherichia coli obtenidos de cerdos de la República Argentina. Rev Argent Microbiol 2007; 39: 227-9. [ Links ]
17. Noseda RP, Bigalli MC, Andrich M, Cordiviola JM, Bardón JC, Martínez AH, et al. Aislamiento de Salmonella en muestras clínicas humanas, animales y alimentos durante 1966 y 2001.Vet Argent 2002; 19: 752-9. [ Links ]
18. Oliveira CJB, Oliveira Silva Carvalho LF, Fernández AS, Tavechio AT, Camacho Pereira Menezes C, Domingues FJ. Antimicrobial resistance of Salmonella serotypes isolated from slaughter-age pigs and environmental samples. Microb Drug Resist 2002; 8: 407-11. [ Links ]
19. Orman BE, Piñeiro SA, Arduino S, Galas M, Melano R, Caffer MI, et al. Evolution of multirresistance in nontyphoid Salmonella serovars from 1984 to 1998 in Argentina. Antimicrob Agents Chemother 2002; 46: 3963-70. [ Links ]
20. Quiroga MP, Andrés P, Petroni A, Soler-Bistué A, Guerriero L, Jordá Vargas L, et al. Complex class 1 integrons with diverse variable regions including aac(6)-Ib-cr and a novel allele qnrB10 associated to ISCR1 in clinical enterobacteria from Argentina. Antimicrob Agents Chemother 2007; 51: 4466-70. [ Links ]
21. Viera Pinto M, Temudo P, Martins C. Ocurrence of Salmonella in the ileum, ileocolic lymph nodes, tonsils, mandibular lymph nodes and carcasses of pigs slaughtered for consumption. J Vet Med B 2005; 52: 476-81. [ Links ]
22. Vigo G, Moredo F, Capuccio J, Piñeyro P, Caffer M, Perfumo C. Frecuencia, serovariedades y sensibilidad antimi-crobiana de Salmonella spp. aisladas de cerdos faenados provenientes de una granja con presentación clínica de la infección. V Congreso de producción porcina del MERCOSUR, 2006, p. 218. Córdoba, Argentina. [ Links ]
23. Vigo GB, Capuccio JA, Piñeyro PE, Machuca MA, Quiroga MA, Moredo FA, et al. Bacteriological and serological longitudinal study of Salmonella enterica in a positive three site farrow-to-finish farm. XIII Abraves, 2007. Florianópolis, SC, Brasil. [ Links ]
Recibido: 21/04/09
Aceptado: 08/07/09














