Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO  uBio
uBio
Compartilhar
Revista argentina de microbiología
versão impressa ISSN 0325-7541versão On-line ISSN 1851-7617
Rev. argent. microbiol. v.42 n.1 Ciudad Autónoma de Buenos Aires jan./abr. 2010
ARTICULOS ESPECIALES
Integrones: los coleccionistas de genes
J. A. Di Conza1, 2*, G. O. Gutkind1
1Laboratorio de Resistencia Microbiana, Cátedra de Microbiología, Facultad de Farmacia y Bioquímica, UBA. Junín 956 (1113) Ciudad Autónoma de Buenos Aires;
2Cátedra de Microbiología General, Facultad de Bioquímica y Ciencias Biológicas, UNL. Argentina.
*Correspondencia. E-mail: jdiconza@ffyb.uba.ar
RESUMEN
Los integrones son estructuras genéticas que han despertado gran interés, debido a que algunos de ellos vehiculizan genes de resistencia a los antimicrobianos. Están formados por un fragmento que codifica una integrasa (intI) y, a continuación, una secuencia attI a la que se unen los genes en casetes que codifican diferentes mecanismos de resistencia. Dentro de intI, en su extremo 3´, hay una secuencia promotora Pc a partir de la cual se transcriben los casetes de resistencia integrados, ya que estos genes carecen de promotor. Sin embargo, estos casetes presentan una secuencia específica denominada attC, la cual es reconocida por la integrasa que se une, por recombinación, a la secuencia attI del integrón en la orientación adecuada para su expresión. Los integrones se han clasificado según la secuencia de su integrasa, pero en la actualidad se prefiere clasificarlos según su localización. Se habla, en general, de "integrones móviles" para referirse a aquellos asociados a secuencias de inserción, transposones y/o plásmidos conjugativos, los que en su mayoría median mecanismos de resistencia, y de "superintegrones", de localización cromosómica y con grandes arreglos de genes en casetes. Los integrones móviles de clase 1 son los más abundantes en aislamientos clínicos y suelen estar asociados a transposones del subgrupo Tn21, seguidos por los de clase 2, derivados principalmente de Tn7. Estos elementos no son móviles por sí mismos, pero su asociación con elementos que sí lo son facilita su transferencia horizontal, lo que explica su amplia difusión entre las bacterias. Esta revisión intenta recopilar la información disponible acerca de los integrones móviles descritos en Argentina hasta la fecha.
Palabras clave: Integrasa; Gen en casete; Resistencia a antibióticos
ABSTRACT
Integrons: gene collectors. Integrons gained great interest due to their participation in resistance gene recruitment and expression. Their basic structure includes a fragment that encodes an integrase (intI) followed by a recognition sequence (attI) into which they may incorporate gene cassettes (encoding resistance mechanisms). A promoter (Pc) embedded within the integrase gene controls the transcription of integrated resistance markers, as these genes do not have their own promoters. When in cassettes, resistance genes are flanked by specific sequences (attC), which are recognized by the integrase that, by site specific recombination, incorporates them after attI in proper orientation for their expression. In the past, integrons were classified according to their sequence homology; currently they are classified according to their location. In general, they are divided into "mobile" integrons (those associated with insertion sequences, transposons and/or plasmids, being most of them associated with resistance mechanisms), and chromosomally-located "super" integrons with large arrangements of cassette genes. "Mobile" class 1 integrons are the most abundant in clinical isolates and are generally associated with Tn21 subgroup transposons, followed by class 2, derived primarily from Tn7. These elements are not mobile themselves, but their association with mobile platforms that facilitate horizontal transfer, explains their wide distribution among bacteria. This review also attempts to describe the mobile integrons described so far in Argentina.
Key words: Integrase; Gene cassette; Antibiotic resistance
INTRODUCCIÓN
Más de medio siglo ha transcurrido desde el reconocimiento de la participación de las primeras estructuras de transferencia de los genes de resistencia adquiridos por las bacterias para evadir la acción de los antibióticos hasta reconocer la participación de sistemas de movilización que contribuyen a la inclusión de nuevos mecanismos, lo que amplía así el espectro de resistencia.
Las metodologías disponibles tiempo atrás hicieron que inicialmente se evaluaran los marcadores de resistencia cotransferidos por conjugación de diferentes plásmidos R. Más tarde fueron analizadas las estructuras de estos plásmidos, lo que llevó al reconocimiento de los diversos procesos que movilizan estos marcadores entre distintas estructuras génicas. Sólo tras la optimización de los métodos de secuenciación y el advenimiento del análisis computacional fue posible reconocer la importancia de nuevas plataformas genéticas, a las que hoy llamamos integrones.
Desde los comienzos de su descripción y hasta la actualidad, se continúa investigando y tratando de comprender en profundidad el comportamiento molecular y evolutivo de los integrones, los cuales fueron rápidamente asociados al reclutamiento y la pérdida de genes. Los integrones no son elementos genéticos raros, ya que luego de analizar más de 600 genomas secuenciados en forma parcial o completa, se encontró que el 9% de ellos poseen integrones (12). En este trabajo se realizó una revisión sobre estas estructuras genéticas movilizables que parecieran estar favoreciendo distintos eventos de diversificación genética dentro de los microorganismos procariotas, dado que si bien no son esenciales para las bacterias, aportan genes adicionales que les permiten una mejor adaptación a ambientes hostiles. Aunque estos elementos fueron encontrados en bacterias recuperadas de ambientes naturales (Vibrio, Pseudomonas, Xantomonas, Shewanella), se prestó especial atención a la participación y el impacto de estos elementos sobre los microorganismos frecuentemente encontrados en contextos clínicos, donde despliegan una marcada variedad de mecanismos para adaptarse a esos entornos y resistir la acción de los agentes antibióticos.
MESA DE OFERTAS ADAPTATIVAS: DEFINICIÓN
En la era de la Biología Molecular, contamos con numerosos equipos comerciales o kits que nos facilitan el clonado de genes, incluso disponemos de sistemas para la expresión in vivo e in vitro de esos genes. En forma análoga, se podría pensar a los integrones como verdaderos "equipos naturales" de clonado y expresión de genes, que brindan la maquinaria completa para una eficiente adquisición (y escisión) de genes a través de eventos de recombinación específica de sitio (9).
La definición más divulgada de integrón es aquella formulada por Hall y Collis (40), como "un elemento dinámico que contiene los determinantes genéticos de los componentes de un sistema de recombinación específica de sitio que reconoce y captura genes en casete móviles". La estructura mínima de un integrón incluye: 1- el gen para la integrasa (intI), 2- un sitio adyacente de recombinación (attI), y 3- al menos un promotor (Pc, P2), orientado para la expresión de los genes capturados. El gen intI contiene su propio promotor y, en general, se expresa en forma divergente a los genes capturados (Figura 1). La integrasa, que pertenece a la familia de las tirosina-recombinasas, es la que cataliza la recombinación entre las secuencias específicas. Es por ello que las bacterias que poseen integrones tienen la posibilidad de incorporar genes que se encuentran en el citoplasma bacteriano bajo la forma de genes en casete. Estos genes en casete no son parte necesaria de los integrones, pero forman parte de ellos una vez que han sido integrados. De esta manera, los integrones pueden adquirir nuevos determinantes, por ejemplo, de virulencia, de resistencia a antibióticos, nuevas funciones metabólicas, etc., lo cual brinda a la bacteria hospedadora una amplia versatilidad para adaptarse a nuevas condiciones de supervivencia (12, 45).

Figura 1. Estructuras básicas de un integrón. Se observan los tres componentes fundamentales que definen a estas plataformas genéticas, necesarios para la adquisición de genes en casete (mediante recombinación específica de sitio) y para su expresión: 1- el gen que codifica la integrasa (intI), 2- el sitio adyacente de recombinación (attI) y 3- los posibles promotores presentes, orientados adecuadamente para la expresión de los casetes insertados (Pc y P2). El gen de la integrasa contiene su propio promotor Pint.
Si bien estas plataformas genéticas fueron descritas por primera vez vinculadas a aislamientos clínicos con algunos pocos arreglos de genes en casete que codificaban resistencia a antibióticos (105), se reconoce que los integrones se hallan ampliamente distribuidos, ya que se han descrito en diferentes nichos ecológicos (12), los cuales pueden actuar como potenciales reservorios.
ASUNTOS DE FAMILIA: CLASIFICACIÓN
Los primeros integrones descritos fueron agrupados en clases (1, 2 y 3) de acuerdo con la secuencia nucleotídica del gen de la integrasa (intI) que los caracterizaba. Sin embargo, este sistema de clases se ha ido desdibujando debido a la gran divergencia de integrasas descritas en los más diversos microorganismos, lo que ha llevado a una clasificación práctica basada en una variedad de criterios, en dos grandes grupos: I- el de los llamados integrones de resistencia a antibióticos (a veces denominados "integrones móviles"), que será el foco de interés en esta revisión dada su relevancia clínica y la cantidad de literatura disponible, y II- el de los integrones presentes en el cromosoma bacteriano (también referidos como "superintegrones"), sólo esporádicamente asociados a determinantes de resistencia (12, 30, 41, 57).
Tanto la descripción de los integrones móviles como la de los superintegrones estuvo basada en ciertas características extraídas a partir de un número de secuencias limitado; actualmente el cuadro de diversidad es más complejo y las características que definen a estos dos grupos tienden a solaparse, lo cual lleva a debatir sobre la conveniencia de esta agrupación práctica (12, 57).
Los integrones móviles presentan, en general, casetes de resistencia a antibióticos agrupados en arreglos relativamente pequeños, y es aquí donde se incluirían los integrones clase 1, 2 y 3 antes descritos (57, 98).
Los integrones de clase 1 son los más prevalentes en aislamientos clínicos y se hallan altamente asociados con bacilos gram negativos multirresistentes. En la actualidad han cobrado una gran importancia, no sólo por el amplio espectro de bacterias en las que han sido comunicados, sino también por su frecuente nexo con casetes que codifican, por ejemplo, distintas metalocarbapenemasas (30, 119). A pesar de que el contenido de casetes suele ser pequeño en este grupo de integrones, a la fecha se encuentran informados más de 100 arreglos diferentes dentro de esta clase, lo cual está directamente relacionado con la variedad de mecanismos de resistencia a casi todas las familias de antibióticos ya encontrados en casetes.
El análisis y la comparación de la secuencia del transposón Tn7 y su familia muestra una organización similar a la de los integrones e indica la presencia de un posible gen codificante de una integrasa de clase 2 (intI2) localizado en el extremo 5' del primer casete presente en el Tn7. Así, los integrones clase 2 que forman parte del Tn7 (y otros relacionados) portan genes en casete en su estructura y han sido documentados en aislamientos clínicos de bacilos gram negativos, aunque en menor frecuencia que los anteriores. A menudo el gen intI2 es interrumpido prematuramente por un codón de terminación. Esto lo convierte en un pseudogen, lo que explicaría los pocos arreglos que se encuentran descritos en las bases de datos, en comparación con el número de arreglos de los integrones clases 1 (22, 28). La secuencia de aminoácidos de IntI2 presenta una identidad del 40% con IntI1 (87, 91). Sin embrago, recientemente se han descrito nuevos integrones clase 2 -en Providencia stuartii (8) y en Escherichia coli (54)- en los que se observa una sustitución en la base que originaba el codón de terminación temprano dentro de intI2, lo cual demuestra la funcionalidad de la integrasa.
Los integrones clase 3 presentan en su estructura el gen intI3, que codifica la integrasa de clase 3. La secuencia de aminoácidos deducida para esta integrasa muestra una identidad del 60,9 % con la integrasa IntI1 (3, 91). Dichos integrones también portan casetes de resistencia a antibióticos. Sin embargo, son muy pocos los informes encontrados y la mayoría se limitan al continente asiático. En el primer caso descrito, este gen se localizó en el extremo 5' del casete blaIMP-1 (que codifica una carbapenemasa) presente en un aislamiento clínico de Serratia marcescens (3), más tarde hallado por el mismo grupo de trabajo también en Pseudomonas putida (102). En esa misma región geográfica se ha detectado en forma reciente esta clase de integrones en el cromosoma de aislamientos ambientales de Delftia spp. (120). Fuera de Asia, su presencia se describió en Portugal, en un aislamiento urinario de Klebsiella pneumoniae cuya región variable presentaba el casete blaGES-1 (21).
Un determinado gen en casete no parece ser exclusivo de una clase de integrón estipulada; a modo de ejemplo, el casete blaIMP-1, originalmente encontrado en un integrón de clase 3, se halla actualmente descrito como parte de integrones de clase 1 (3, 48, 59, 76).
LOS TÍPICOS Y LOS ATÍPICOS: ESTRUCTURA
Como se indicó anteriormente, los integrones clase 1 son los de mayor prevalencia en aislamientos clínicos. Contienen dos segmentos conservados que flanquean una región central (región variable), donde se insertan los casetes que codifican la resistencia a antibióticos. El segmento 5' conservado (5'CS) incluye al gen de la integrasa (intI1) y el sitio adyacente de recombinación reconocido por la integrasa IntI1 (attI1). Dentro de esta estructura se encuentran uno o más promotores que aseguran la expresión de los casetes. El segmento 3' conservado (3'CS) en los integrones clase 1 es menos definido. En general, este segmento incluye un gen que codifica resistencia a compuestos de amonio cuaternario (qacE![]() 1), otro de resistencia a sulfonamidas (sul1), y un marco de lectura abierto (orf5) de función desconocida (Figura 2a).
1), otro de resistencia a sulfonamidas (sul1), y un marco de lectura abierto (orf5) de función desconocida (Figura 2a).

Figura 2. Diferentes clases de integrones. Se señalan en color las regiones conservadas y en gris las regiones variables. a- Integrón de clase 1, en verde se marca la región 5´CS y en naranja la región 3´CS. La región variable quedó constituida por tres genes en casete seleccionados al azar. b- Integrón de clase 2, en rojo se marca la región 5´CS (el asterisco en intI2* indica que es un pseudogen) y en azul la región 3´CS. Se respetó la región variable presente en el Tn7. c- Integrón de integrasa invertida, en violeta se marca la región 5´CS (la orientación del gen intI es opuesta a la observada en los diferentes integrones). Diferentes orf como genes en casete estarían presentes downstream del gen de la integrasa.
Al analizar detalladamente el segmento 3'CS se observa que qacE![]() 1 es una forma truncada del gen qacE fusionado con un segmento no relacionado que codifica 21 aminoácidos. QacE
1 es una forma truncada del gen qacE fusionado con un segmento no relacionado que codifica 21 aminoácidos. QacE![]() 1 pertenece a una familia de proteínas que puede exportar diferentes drogas e incluye a las proteínas QacC, QacE, y Ebr. En general, son pequeñas proteínas con sólo cuatro segmentos transmembrana. Aun con el truncado, QacE
1 pertenece a una familia de proteínas que puede exportar diferentes drogas e incluye a las proteínas QacC, QacE, y Ebr. En general, son pequeñas proteínas con sólo cuatro segmentos transmembrana. Aun con el truncado, QacE![]() 1 parece funcionar parcialmente como proteína exportadora de múltiples drogas, ya que aislamientos que contienen qacE
1 parece funcionar parcialmente como proteína exportadora de múltiples drogas, ya que aislamientos que contienen qacE![]() 1 son resistentes a bajos niveles de colorantes y a sales de amonio cuaternario, pero no se observa eflujo de bromuro de etidio (75).
1 son resistentes a bajos niveles de colorantes y a sales de amonio cuaternario, pero no se observa eflujo de bromuro de etidio (75).
La presencia ubicua del gen sul1 (que codifica una dihidropterato sintetasa) en estos elementos puede resultar sorpresiva, aunque las sulfonamidas (antimicrobianos sintéticos) junto con las sales de mercurio han sido empleadas para el tratamiento de las enfermedades infecciosas desde mediados de la década del 30 (24).
Sin embargo, la región 3´CS puede ser diferente en algunos integrones (37, 75), incluso hay autores que han descrito la ausencia del gen sul1 en el 10% de los integrones clase 1 estudiados (49). La analogía del transposón Tn402 -carente de una estructura 3´CS típica- con estos últimos ha dado lugar al modelo que explica el origen y la subsecuente diseminación de integrones clase 1 entre distintas especies de Proteobacteria (34).
Los genes en casete son integrados en lo que se denomina comúnmente región variable (40). El número de genes en casete en esta región es precisamente variable: se describieron desde integrones con región variable nula, como en In0 (11), hasta algunos con más de siete genes en casete (48, 51, 61, 78); sin embargo, la presencia de dos o tres casetes suele ser lo más habitual en las secuencias depositadas en las bases de datos. En la actualidad, se encuentran descritas numerosas combinaciones de genes en casete, incluso varias copias de un mismo casete en un mismo integrón, tal como las dos copias del casete blaOXA-2 en el integrón In1 (104).
Los integrones clase 2, tal como ya se señaló, se encuentran con menor frecuencia en los aislamientos clínicos comparados con los integrones clase 1. Tienen una región 5´CS típica con las particularidades correspondientes a esta clase (intI2, attI2). En la mayoría de los integrones de clase 2, la zona 3´CS está compuesta por 5 genes (tnsA, tnsB, tnsC, tnsD y tnsE) involucrados en la transposición del Tn7 y derivados (Figura 2b) (43, 44). En general, los arreglos de casetes dentro de la región variable son más constantes (como ya se ha fundamentado); lo más frecuente es encontrar el arreglo detallado en el integrón presente en Tn7 en diferentes enterobacterias y en otros aislamientos clínicos. Ese integrón posee el siguiente orden: dfrA1 (que confiere resistencia a trimetoprima), sat2 (que otorga resistencia a estreptotricina, un antibiótico muy empleado en la industria de alimentos y en veterinaria, pero no en la clínica humana), aadA1 (que codifica una enzima que otorga resistencia a espectinomicina y a estreptomicina) y, finalmente, un marco de lectura abierto, orfX, de función desconocida (28, 33, 79).
En el año 2000 fue descrito el primer híbrido entre dos clases de integrones (en un aislamiento de Acinetobacter baumannii), constituido por una integrasa de clase 2, el sitio attI2, los tres primeros casetes presentes en Tn7, mientras que el orfX y los 5 genes tnsA-E fueron reemplazados por la región 3´CS, característica de los integrones clase 1. Esta estructura quimérica podría ser el resultado de la recombinación homóloga entre ambos integrones coexistentes en el mismo microorganismo (77).
Por último, cabe señalar que son pocas las publicaciones referidas a casos clínicos de integrones clase 3 en los que éstos hayan sido caracterizados en detalle. Se sabe que poseen una localización plasmídica y no se conoce con precisión si comparten una región 3´ conservada (3, 21).
Normalmente el gen de la integrasa (intI) está en orientación opuesta respecto de los genes codificados en los casetes; sin embargo, algunos integrones muestran una integrasa en la misma orientación que los genes de los arreglos en casetes asociados. Estos han sido denominados integrones de integrasa invertida (Figura 2c) y forman un grupo monofilético de integrones bien definido que fueron hallados en una gran variedad de grupos taxonómicos bacterianos ambientales (12).
EL MÁS GRANDE DE LA FAMILIA: EL "SUPERINTEGRÓN"
Desviaremos temporariamente la atención sobre los integrones de resistencia para referirnos a otras clases de integrones que fueron confinadas al cromosoma bacteriano y que están asociadas a grandes arreglos de genes en casete. El género Vibrio, donde se describieron los primeros ejemplos, presenta desde 50 hasta más de 200 casetes. Esta gran estructura ha conducido a su peculiar denominación, la de "superintegrón" (SI). Las siguientes características se han usado frecuentemente para describir un SI: 1- una localización cromosómica, 2- muchos genes en casete asociados, 3- un alto grado de identidad entre las secuencias attC de estos casetes, y 4- una descendencia principalmente lineal entre un grupo determinado de microorganismos (57). El descubrimiento de SI en diferentes géneros de proteobacterias ha impactado en la comprensión de la evolución del genoma bacteriano. Estos elementos se han descrito en patógenos para el hombre, los animales y las plantas, así como también en bacterias no patógenas. Es muy probable que la mayoría de los genes en casete que forman parte de un SI no sean expresados; así, estos persisten en ausencia de presión selectiva y proveen las bases genéticas para la evolución y la subsecuente retención de nuevas funciones para la adaptación (12, 57).
Por ejemplo, estas estructuras de SI se describieron por primera vez en Vibrio cholerae en una región de aproximadamente 100 kb (en un principio denominados integrones clase 4 y 5), y desde su descripción se destaca que se observa sólo una asociación marginal con genes de resistencia (17, 53, 58, 92). Una porción del genoma de V. cholerae parece tener un arreglo de genes únicos separados por cortas secuencias repetidas e invertidas de 123-126 pb, conocidas como VCR (V. cholerae repeated sequences); estas VCR comparten una identidad del 95-97%. La secuencia consenso de las VCR está significativamente relacionada con la secuencia consenso de los attC. Esta estructura contiene más de 170 casetes, la mayoría no definidos (orf), y ocupan aproximadamente el 4% del genoma. Por esto, puede interpretarse a esta fracción del genoma de V. cholerae como un gran arreglo de genes en casete. Los casetes encontrados en V. cholerae no se expresan a partir de un sólo promotor, lo que es claramente evidente, ya que no todos se encuentran orientados en la misma dirección. Como se dijo anteriormente, estos "superintegrones" se encuentran en muchos géneros bacterianos e inclusive en otras especies del género Vibrio.
La mayoría de los orf identificados en integrones cromosómicos están más relacionados con funciones de virulencia u otras funciones que poco tienen que ver con la resistencia a antibióticos, aunque se han descrito unos pocos casetes de resistencia, como por ejemplo blaCARB-7 y blaCARB-9 (100). Por otro lado, también se ha visto que los attC de los casetes blaP3 y dfrVI y los recientemente descritos qnrVC1 y qnrVC2 son idénticos a los VCR. Estos hallazgos sugieren que existe un grupo de casetes disponibles o compartidos entre integrones de resistencia y superintegrones (32, 97, 98).
ERES EL ÚNICO CULPABLE: EL GEN EN CASETE
La estructura de un gen en casete está compuesta por dos componentes funcionales: una secuencia codificante, generalmente carente de promotor propio, flanqueada por secuencias palindrómicas no perfectas que participan activamente de la recombinación específica de sitio. En los casetes de resistencia a antibióticos, esta estructura es conocida como elemento de 59-bases (59-be) o, según su nomenclatura más moderna, como secuencia attC. El tamaño de los casetes es considerablemente variado y depende de la proteína que codifique (9, 31, 40, 91). Los más de 100 casetes descritos confieren resistencia a casi todas las familias de antibióticos (ß-lactámicos, aminoglucósidos, trimetoprima, sulfamidas, cloranfenicol, rifampicina, lincosaminas, macrólidos, quinolonas) y además, a desinfectantes y antisépticos. Las proteínas codificadas por estos casetes confieren resistencia por distintos mecanismos, por ejemplo, enzimas que inactivan al antibiótico (aminoglucósidos, cloranfenicol y b-lactámicos), desvío metabólico (trimetoprima) y eflujo (cloranfenicol). Además, se han identificado varios marcos de lectura abiertos con función desconocida (30, 40). Un comentario destacado merecen los genes de resistencia a quinolonas transferibles. El primer casete identificado (2006), al que se llamó aac(6´)- Ib-cr, es una variante del gen aac(6´)-Ib que codifica una aminoglucósido acetiltransferasa. Las sustituciones en este gen llevan a la posibilidad de acetilación de un amino presente en algunas quinolonas (85, 94); de esta manera se identificó el primer mecanismo de inactivación enzimática de dos familias no relacionadas estructuralmente. Otros genes en casetes que confieren resistencia únicamente a quinolonas (denominados qnrVC1 y qnrVC2), a través de un mecanismo que protege el sitio diana del antibiótico, fueron descritos en el 2008. Con anterioridad, este mecanismo había sido ampliamente descrito en integrones clase 1 inusuales o atípicos (los que se describen más adelante), pero los genes asociados carecen de sitios attC típicos. Estos dos nuevos casetes presentan sitios attC característicos de superintegrones y fueron asociados a integrones clase 1 típicos presentes en V. cholerae (32).
Un examen exhaustivo de las secuencias publicadas muestra que en algunos casos existen casetes fusionados, donde dos potenciales regiones codificantes se encuentran dentro de un único casete. Se cree que estos casetes surgieron por fusión de otros dos, cada uno con un único gen (91); por ejemplo, la fusión de casete aadA1- oxa9 (112, 113) o los encontrados en el integrón In53, aacA1-orfG y oxa10-aadA1 (61). Se ha descubierto, incluso, la fusión de un intrón del grupo II (16).
Las regiones attC difieren considerablemente en su secuencia y su tamaño, desde unos 51 pb hasta 141 pb, y si bien son heterogéneas entre ellas, muestran características en común (91). Comparten una región de aproximadamente 25 pb a cada extremo que conforman las secuencias consenso (19, 106), las que están repetidas e invertidas en forma imperfecta una respecto de otra, y cada una de ellas comprende un par de dominios de unión a la integrasa (106). Las secuencias más conservadas de los attC son heptámeros denominados sitios core, los cuales se muestran como secuencias palindrómicas. Uno de ellos, cuya secuencia consenso es GTTAGGC o GTTRRRY (R = purina, Y = pirimidina), está localizado upstream del gen de un casete integrado, y el sitio core inverso, que se lo identifica con la secuencia consenso GCCTAAC o RYYYAAC, queda ubicado downstream del gen (19, 91). Las diferencias entre ambas regiones aseguran que los casetes se inserten en la orientación que permite su expresión (18, 106).
Cuando se considera todo el pool de casetes conocidos (provenientes de integrones móviles y de superintegrones), los que codifican resistencia a antibióticos corresponden a menos del 10%. La mayoría de los casetes no poseen función conocida: al 65% no se les conoce homólogos y el 13% presenta homología con proteínas de función desconocida. El 22% restante codificaría para un amplio rango de funciones (la mayoría deducida por predicción con proteínas análogas, y muy pocas con funciones determinadas experimentalmente), siendo las más prevalentes factores de virulencia, modificaciones del ADN, funciones relacionadas con fagos, sistemas toxina-antitoxina (que suelen prevenir, en general, la pérdida de ADN plasmídico), acetiltransferasas, sistemas de metilación o restricción, lipasas, enzimas que participan en la biosíntesis de polisacáridos, etc. En general, estos casetes forman parte de integrones cromosómicos o superintegrones descritos en Vibrio spp. y en otras especies ambientales (12, 30).
LA INTEGRASA ¿CÓMO TE UBICO?
El análisis de las distintas integrasas permitió establecer que el origen de los integrones móviles que portan casetes de resistencia es claramente diferente, lo cual no sólo es avalado por los análisis filogenéticos sino también al considerar el origen ambiental de muchos de ellos (lo que incluso sugiere una evolución producida antes de la era antibiótica). Esto indica que muchos integrones encontrados en ambientes naturales han tenido la capacidad de adquirir casetes de resistencia y así aumentar enormemente su potencial de adaptación ante la presión ejercida por el hombre (12, 98).
La integrasa IntI1 pertenece a la familia de las l integrasas (tirosina-recombinasas) y cataliza no sólo la integración de genes en casete, sino también su escisión. Si bien los miembros de esta familia presentan baja homología en su secuencia de aminoácidos, se han identificado dos regiones (designadas box I y box II) relativamente conservadas. Dentro del box I cabe destacar, entre otros, el motivo ALER, característico de las integrasas (lo que ha permitido identificar nuevas variantes en diversos géneros bacterianos), mientras que en el box II lo característico es la tirosina ubicada en el extremo carboxilo (Y312) terminal (35, 60).
La IntI1 reconoce dos tipos diferentes de sitios de recombinación: el sitio attI, que forma parte de la región 5´CS de los integrones, y los miembros de la familia attC, presentes en los genes en casete (93).
La arquitectura de los sitios tipo attI1 es distinta de la descrita para sitios tipo attC. Al contrario de lo que ocurre con los sitios attC, los sitios attI de las diferentes clases de integrones no están relacionados entre sí y sus secuencias no muestran muchas características en común (38, 93). El sitio attI1 es uno de los más examinados y estudiados en detalle (42, 73, 93), e incluye una única estructura de sitio simple que contiene dos regiones de unión a IntI1 orientadas en forma invertida (sitios core 1 y 2), además de presentar sitios adicionales de unión de la integrasa localizados upstream del sitio simple (38, 73).
La integrasa IntI1 puede catalizar al menos dos posibles reacciones, a saber: entre el sitio attI1 y un attC y entre dos attC. Si el integrón receptor no contiene casetes, la reacción de recombinación durante la integración de un casete es necesariamente attI1 x attC. En cambio, si contiene uno o más casetes, la reacción de recombinación durante una nueva integración tiene dos posibilidades: attI1 x attC o attC x attC.
La diferencia entre la arquitectura del sitio attI1 y la de los sitios attC parece jugar un papel importante, al asegurar que los casetes se integren preferencialmente en forma adyacente al sitio attI1 del integrón y en la misma orientación (20). Esta localización es la óptima para la expresión del gen contenido en el casete entrante (18). Se ha demostrado que los eventos de recombinación específica de sitio mediada por IntI1 son más eficientes cuando involucran un sitio attI1 con un attC que cuando ocurren entre dos attC (20, 42, 73, 93, 106). Sin embargo, la pérdida de casetes (reacción de escisión) suele ocurrir por la recombinación preferencial de dos sitios attC (52). En la Figura 3 se resume el modelo de movilización simplificado de genes en casete dentro de un integrón; las particularidades del mecanismo de integración fueron propuestas por Bouvier et al. (13), mientras que las del proceso de escisión fueron delineadas por Mac Donald et al. (52). En parte de estos eventos se requieren estructuras de ADN de hebra simple, motivo por el cual se sostiene que ocurren ya sea durante la replicación del ADN o durante la conjugación de elementos móviles (13, 52).

Figura 3. Modelo de integración y escisión de casetes. Proceso por el cual los genes en casete circulares de resistencia a antibióticos se insertan preferencialmente en el sitio específico attI1 en un integrón clase 1. Se seleccionó blaVIM-2 (que codifica resistencia a carbapenemes) como ejemplo de gen que se va a incorporar. Dentro de la región variable contamos, además, con genes casete que codifican resistencia a aminoglucósidos (aacA4) y a cloranfenicol (cmlA5). Además, se muestra la estructura del gen en casete circular blaVIM-2, donde se destaca el sitio de reconocimiento de la integrasa 59-be o attC como parte de su estructura. intI1, gen que codifica la integrasa; IntI1: integrasa clase 1. Las flechas de los casetes indican la dirección de la transcripción.
Si bien los genes en casete se encuentran integrados y forman parte de un integrón o se ubican en alguna otra localización no específica, pueden existir libres como moléculas de ADN circular cerradas de modo covalente. En la naturaleza, los casetes circularizados presumiblemente se generan por escisión, y es probable que sean los intermediarios más importante en la diseminación de casetes cuando se encuentran en esta conformación circular (19).
Finalmente, es interesante destacar que la especificidad de las secuencias attI asociadas a una determinada integrasa no impide que sean reconocidas por otras integrasas y que ocurra así la recombinación. Por ejemplo, la IntI1 también reconoce los sitios attI2 y attI3 de los integrones clase 2 y 3, aunque la eficiencia de recombinación entre attI2 y attI3 con un attC es mucho más baja que la reacción attI1 x attC (38). Queda claro entonces que la integrasa IntI1 podría participar en la movilización de casetes presentes en otras clases de integrones.
LA DIFERENCIA ENTRE SÓLO ESTAR Y SER: AHORA, A EXPRESAR LO INCORPORADO
Los genes en casete generalmente no incluyen una secuencia promotora. La expresión de estos genes de resistencia a antibióticos está bajo el control transcripcional del promotor Pc (inicialmente llamado P1 o Pant) presente en la región 5'CS del integrón, ya que todos los casetes están integrados en la misma orientación. Se ha estudiado en detalle el promotor Pc de los integrones clase 1 (en In2, localizado entre las bases 254 a 225 upstream del punto de recombinación -N° de acceso: AF071413-) y se han encontrado 4 variantes de este promotor en integrones presentes en aislamientos clínicos (31, 39). Cada una de estas variantes posee diferente fuerza promotora (Tabla 1), lo que confiere diferentes niveles de resistencia ante la presencia del mismo gen codificante (18, 50). En un subgrupo de integrones clase 1 se ha originado un segundo promotor, P2, por inserción de tres residuos de G entre los motivos preexistentes -10 y -35 (en In2, localizado entre las bases 136 a 107 upstream del punto de recombinación -Nº de acceso: AF071413-). Así se extiende el espacio intermotivos requerido para que este promotor sea activo, de 14 pb a 17 pb (Tabla 1). Cuando P2 está presente y es activo, su fuerza es mayor que la de todas las versiones de Pc, excepto Pc (fuerte) (18, 50). Frecuentemente, P2 activo está asociado con Pc (débil) y, en este caso, P2 es el principal responsable de la expresión de los genes de resistencia en integrones clase 1 en diferentes microorganismos (39, 47, 55). El análisis de más de 100 secuencias depositadas en bases de datos muestra que existe un número equitativo de cada variante de promotor y que, en general, no existen asociaciones preferenciales entre las distintas variantes de promotores y los genes en casete insertados en attI. Aunque esta información no puede ser tomada como un dato epidemiológico verdadero, ambas observaciones sugieren que no habría ventaja en la selección de integrones con capacidad promotora superior (25).
Tabla 1. Regiones promotoras descritas en los integrones clase 1

Todos los casetes de un integrón se transcriben a partir del Pc (o Pc y P2). Sin embargo, cuando hay más de un casete, sólo se detectan bajos niveles del transcripto de tamaño completo, con predominio de moléculas de ARNm más cortas (18). Estos transcriptos más cortos tienen tamaños discretos y diferentes, y todos se originan a partir de Pc. El hecho de que los transcriptos terminen prematuramente en puntos discretos parece corresponderse con el extremo del casete. De estas observaciones surge la posibilidad de que los sitios attC (que incluyen secuencias repetidas e invertidas), también actúen como terminadores de transcripción, como atenuadores o como señales para el clivaje endonucleolítico de los transcriptos (18, 91). Esto lleva a una mayor abundancia de los transcriptos del gen del primer casete adyacente a Pc (al compararlo con el mismo casete cuando está localizado más distal) y, eventualmente, a más niveles de resistencia (18).
En conclusión, la expresión de los genes de resistencia (sin promotor propio) está marcadamente influenciada por su posición en el arreglo de casetes (25).
Se ha encontrado que un pequeño número de casetes presentan su propio promotor; el primer caso descubierto fue el casete cmlA, que codifica una proteína de eflujo en la membrana interna (resistencia a cloranfenicol) (10). Otros ejemplos son los casetes qacE y qacG, que también codifican una proteína de eflujo, o el casete fusionado oxa-10-aadA1. Estos casetes se transcriben, además, desde el promotor Pc (25, 31, 61).
ÉRAMOS POCOS: INTEGRONES CLASE 1 COMPLEJOS O INUSUALES
Algunos integrones no sólo participan en la adquisición o la expulsión de genes en casetes y su expresión, sino que además se han asociado a otros elementos que participan en el reclutamiento (y a veces en la expresión) de genes de resistencia que no se hallan en forma de casete.
Estos integrones con características inusuales contienen una copia completa del segmento conservado 5'CS, pero presentan dos copias completas o parciales del segmento 3'CS (39). Entre las dos copias de 3'CS hay una región de 2,1 kb, idéntica en todos los casos, seguida por una región variable que contiene genes de resistencia (Figura 4). En la región idéntica se encuentra un marco de lectura abierto denominado orf513 (anteriormente orf341), que podría codificar una recombinasa que reconocería otro sitio de recombinación donde se insertarían los genes de resistencia carentes de attC (111, 114).
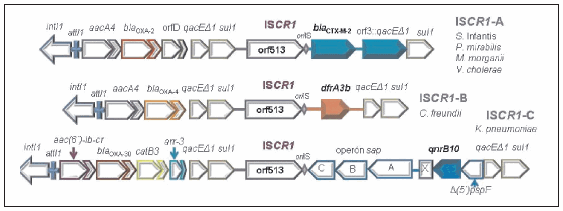
Figura 4. Integrones clase 1 inusuales (complejos): Estructura descritas en Argentina. En cada esquema se destaca en letra violeta el elemento ISCR1 (región conservada de 2,1 kb, incluye el orf513) que se halla estrechamente asociado a estos integrones clase 1 (110, 111). ISCR1-A, -B y -C corresponden a distintos ejemplos de ISCR1 descritos en Argentina, los que se han asociado con alelos que confieren resistencia a cefotaxima (6, 27, 84, 103), a trimetoprima (5) y a ciprofloxacina (85), respectivamente. El integrón esquematizado en ISCR1-C, además del determinante de resistencia a quinolonas asociado qnrB10, posee el gen en casete aac(6´)-Ib-cr, que codifica una nueva variante de la enzima modificadora de aminoglucósidos y que también modifica a la ciprofloxacina, con lo que reduce su actividad. En el gráfico se indican las especies bacterianas donde fueron descritos. Aunque en muchos aislamientos se continuó con la caracterización de la estructura o su entorno, por razones de simplicidad se esquematiza aquí sólo hasta el gen sul1 de la segunda copia del segmento 3´CS.
La secuencia contiene los componentes típicos de un elemento CR (región común). Algunos estudios recientes sugieren que los elementos CR serían un subgrupo dentro de una familia de secuencias de inserción (IS) inusuales denominada IS91, por lo que se ha propuesto renombrarlos como secuencias de inserción CR (ISCR) (110). Estos nuevos elementos de captura denominados ISCR han sido clasificados teniendo en cuenta diversas características, y a aquellos asociados a determinados genes de resistencia (no como genes en casete) que forman parte de integrones clase 1 inusuales se los denomina ISCR1 (111).
La característica distintiva de replicación por transposición en rolling circle de ISCR1 propuesta por Toleman et al. (110) podría explicar los cambios estructurales observados en los integrones inusuales. Los análisis realizados sobre estos elementos han concluido que contienen en su secuencia posibles regiones promotoras -10 y -35 que permitirían o reforzarían la expresión de genes de resistencia adyacentes (95).
Los primeros integrones inusuales comunicados fueron In6, In7 y el integrón presente en pSAL-1 (69, 107, 116). Los genes de resistencia asociados a ISCR1 en estos integrones fueron el gen de resistencia a cloranfenicol catA2 para In6, el gen de resistencia a trimetoprima dfrA10 para In7; y pSAL-1, por su parte, contiene el gen de resistencia a b-lactámicos ampC y su regulador ampR, provenientes del cromosoma de Morganella morganii (39, 69, 107, 116).
Además, estos integrones comparten la característica de presentar una deleción del extremo 5' en la segunda copia del 3'CS; así, In6, In7 y pSAL-1 muestran deleciones de 248 pb, 76 bp y 179 pb, respectivamente (69, 107, 116).
Algunos nuevos ejemplos de ISCR1 son los asociados con otros genes que codifican resistencia a trimetoprima (dfrA23, dfrA3b, dfrA19), a aminoglucósidos (armA), a quinolonas (qnr) o a diferentes antibióticos a-lactámicos [b-lactamasas clase A (blaCTX-M-2, blaCTX-M-9, blaPER-3) y b-lactamasas clase C (blaCMY-8, blaCMY-9, blaMOX-1)] .
¿PERO SI YO NO ME ESCAPÉ?: LOCALIZACIÓN
Los integrones de resistencia móviles no son elementos que presenten la maquinaria necesaria para movilizarse. Asociarse a secuencias de inserción, a transposones y a plásmidos conjugativos fueron los eventos o accidentes que permitieron su movilización y diseminación entre microorganismos de diferentes ambientes. Muchos de estos aislamientos, de genotipo no relacionado, poseen integrones cuya región variable es idéntica, lo que sugiere una transferencia de genes inter e intraespecie. A su vez, la predominante presencia de uno o más casetes en un mismo orden y en diferentes cepas sugiere que la movilización de la estructura completa del integrón es más probable que la movilización individual de los genes en casete (55).
Una parte de los integrones clase 1 están inmersos en transposones defectivos o presentan restos de transposones. El Tn402 es, probablemente, uno de los pocos transposones activos que lleva un integrón (14) que contiene el gen intI1, el sitio attI1, genes en casetes, qacE y un módulo de transposición tni completo, todo flanqueado por las secuencias invertidas repetidas IRi e IRt. Como se ha mencionado anteriormente, se considera que el Tn402, tras participar en varios eventos de inserciones/deleciones y de adquisición/escisión, sería uno de los eslabones hacia la evolución de los integrones clase 1 presentes en los aislamientos clínicos (14, 34).
Los transposones Tn1404, Tn1696 y Tn21 son ejemplos claros de asociación a integrones clase 1 (70, 71, 74, 118), mientras que la familia de los Tn7 está asociada a integrones clase 2 (87, 108). Estos transposones se han localizado tanto en el cromosoma como en plásmidos. En este caso son, en su mayoría, plásmidos conjugativos de elevado peso molecular (> 100 kb) y pertenecen a distintos grupos de incompatibilidad, como por ejemplo IncFI (118), IncFII (36) e IncL/M (117).
La asociación con diferentes secuencias de inserción (IS) ha facilitado también la dispersión de los integrones entre especies bacterianas. Se han descrito varias IS entre las que se cuentan IS26, IS1353 e IS1326, como así también IS1999 e IS2000, entre otras. En algunos casos, no se encuentran situados por fuera de la estructura del integrón, sino que usurpan su propia estructura (62, 72, 115). Cabe recordar que las secuencias ISCR1 descritas anteriormente forman parte de los integrones clase 1 complejos o inusuales.
Resumiendo, las integrasas y las transposasas son todas recombinasas; las primeras recombinan material dentro de su estructura, las segundas recombinan las estructuras que las contienen en otros materiales genéticos (recombinación hacia adentro versus recombinación hacia afuera).
Y NOS TILDARON DE UBICUOS Y PROMISCUOS, ¿SERÁ ASÍ?
En un principio estos elementos fueron asociados a aislamientos clínicos y se los vinculó insistentemente con la resistencia a antibióticos. Así, distintos estudios en diferentes partes del mundo concluyeron que los integrones de clase 1 son los más frecuentes en aislamientos clínicos. Un ejemplo de esta situación epidemiológica fue descrita en el centro y oeste de Europa (55, 56), donde se estudiaron bacilos gram negativos de diferentes hospitales de nueve países europeos, y el 43% (70/163) resultó ser portador de integrones. Es importante notar en estos y en otros tantos trabajos que, estadísticamente, los aislamientos portadores de integrones se encuentran asociados a la multirresistencia a antibióticos, por ejemplo, a aminoglucósidos, quinolonas y compuestos b- lactámicos (incluyendo cefalosporinas de tercera generación y monobactamas), aun si dichos determinantes no siempre están formando parte de éstos (56, 82). También son varios los trabajos en los cuales se describe que, además de la elevada prevalencia de integrones, múltiples integrones de una misma clase pueden encontrarse en un mismo aislamiento, como la coexistencia de integrones clase 1 y clase 2 en una misma bacteria (4, 16, 28, 55, 79). En un principio, los integrones móviles se hallaron solamente en bacterias gram negativas, fundamentalmente en enterobacterias y algunos otros microorganismos como P. aeruginosa, P. putida, Achromobacter sp., Stenotrophomonas maltophilia, complejo Burkholderia cepacia, Vibrio sp., A. baumannii y mucho otros más (30). Desde unos años atrás se sabe que la presencia de integrones no está restringida a este grupo de microorganismos, ya que se ha detectado el gen qacE![]() 1 en enterococos y estafilococos (46), un integrón truncado en Mycobacterium fortuitum (98) y un integrón clase 1 completo y funcional en Corynebacterium glutamicum (65). Hasta el momento, la presencia de integrones en bacterias gram positivas se considera un evento inusual; sin embargo, recientemente se ha comunicado la existencia de integrones clase 1 en aislamientos clínicos de Staphylococcus aureus, Staphylococcus epidermidis, Staphylococcus haemolyticus, Streptococcus sp. y Enterococcus faecalis (todos estos relevamientos fueron realizados en China) (101, 121, 122).
1 en enterococos y estafilococos (46), un integrón truncado en Mycobacterium fortuitum (98) y un integrón clase 1 completo y funcional en Corynebacterium glutamicum (65). Hasta el momento, la presencia de integrones en bacterias gram positivas se considera un evento inusual; sin embargo, recientemente se ha comunicado la existencia de integrones clase 1 en aislamientos clínicos de Staphylococcus aureus, Staphylococcus epidermidis, Staphylococcus haemolyticus, Streptococcus sp. y Enterococcus faecalis (todos estos relevamientos fueron realizados en China) (101, 121, 122).
Asimismo, se han hallado diferentes integrones en bacilos gram negativos provenientes de animales como primates (31), cerdos y peces (64), como así también de otros animales de zoológico que podrían actuar como reservorios de integrones clase 1 y 2 (1). Estos datos, junto con la descripción de estos elementos en miembros del género Salmonella aislados de animales, humanos y agua de río, entre otras fuentes (64), alertan sobre la diseminación de la resistencia a antibióticos y el problema en la salud pública. Algunos autores postulan a las bacterias gram positivas como los principales reservorios de integrones clase 1 en granjas de gallinas (63).
Existen numerosos estudios y publicaciones que documentan el hallazgo de integrones provenientes de aislamientos obtenidos en ambientes naturales como ríos, lagos, suelos, mares y otros lugares sin conocida exposición a antibióticos. Un trabajo realizado en un hábitat natural y en ausencia de presión selectiva continua (entorno acuático) mostró que la gran mayoría del bajo porcentaje de bacterias gram negativas con integrones (3,6%) aparecían sin casetes de resistencia integrados (96). La amplia detección de estos elementos y de nuevas integrasas en diferentes poblaciones apoya la hipótesis de que estas plataformas genéticas participan en la emergencia de los integrones encontrados con tanta frecuencia en aislamientos multirresistentes, en los que actúan, además, como reservorio de genes en casete (12, 66, 99).
LOS TUYOS, LOS MÍOS Y LOS NUESTROS: INTEGRONES DE RESISTENCIA LOCALES
Poco es lo que se ha estudiado sobre una base epidemiológica cierta sobre integrones de resistencia en nuestro país; sin embargo, se dispone de muchas publicaciones y presentaciones en las que se han descrito y caracterizado los integrones presentes en los microorganismos aislados, con características peculiares.
En el ámbito clínico, se han efectuado algunos estudios de relevancia de estas plataformas genéticas en bacterias gram negativas. En particular, en un hospital de la ciudad de Santa Fe, se han estudiado todos los bacilos gram negativos recuperados en un determinado período de tiempo, y se observó: 1- una mayor frecuencia de integrones clase 1 que de clase 2 (4X), 2- la presencia simultánea de integrones clase 1 y 2 en un mismo aislamiento, 3- la ausencia de integrones clase 3, y 4- una muy significativa asociación entre la presencia de estos elementos y la multirresistencia a antibióticos (79, 81, 82).
Tras realizar una recopilación de los datos disponibles, podemos enunciar que se han descrito en nuestro país un gran número de integrones clase 1 con una extensa variedad de arreglos de genes en casete, incluso nuevas variantes alélicas de genes en casete, como por ejemplo el casete aac(6')-Iq en una cepa multirresistente de K. pneumoniae aislada en Buenos Aires (15) y el casete blaOXA-101 en diferentes enterobacterias aisladas en Santa Fe (80). Los integrones clase 1 fueron descritos en muchas especies de enterobacterias y otros bacilos gram negativos; los arreglos observados se resumen en la Tabla 2, donde además se destacan las especies portadoras y la ciudad de donde proceden los aislamientos. Incluso, se han comunicado algunos integrones clase 1 que carecen de 3´CS (29).
Tabla 2. Arreglos de genes en casete que codifican mecanismos de resistencia descritos en integrones clase 1 presentes en aislamientos clínicos de Argentina

Asimismo, se describieron integrones clase 1 inusuales o complejos y se caracterizó su estructura (Figura 4) como así también la parte del transposón o plásmido donde se encontraban localizados. El más estudiado fue aquel asociado a la enzima CTX-M-2 (dado que es la ßlactamasa de espectro extendido prevalente en Argentina), razón por la cual se lo describió en diferente enterobacterias (6, 27, 84), en V. cholerae (103) e incluso en Pseudomonas (99a). Se describieron elementos relacionados con el Tn1696 y otros relacionados con tranposones tipo Tn1000 upstream y downstream del integrón inusual asociado a blaCTX-M-2, respectivamente (83, 103). En un aislamiento de E. coli recuperado en España, se halló un integrón similar que contiene blaCTX-M-2 en su estructura; contiene además elementos relacionados con Tn21 y carece de secuencias tipo Tn1000 downstream del integrón (115). Asimismo, se detectaron otros integrones inusuales (Figura 4) asociados con resistencia a trimetoprima (5) y a quinolonas (85).
Finalmente, varios arreglos de casetes fueron descritos en integrones clase 2 (Figura 5). Como era de esperar, la mayoría de las especies estudiadas presentan el arreglo correspondiente al transposón Tn7. Estos fueron descritos en diferentes enterobacterias, en algunos bacilos gram negativos no fermentadores de glucosa y en Helicobacter pylori (22, 79, 88). Dos nuevos arreglos en integrones clase 2 han sido descritos en Argentina, uno muy particular en A. baumannii, con 5 genes en casete y el gen catB2 asociado a un sitio attI2 (pseudocasete) como parte del arreglo (89), y otro con un único gen en casete (sat2) en B. cenocepacia (90). Además de los ya caracterizados, es de nuestro conocimiento que la mayor variabilidad de nuevos arreglos fue descrita en A. baumannii (sat2 solo, dfrA1 solo, dfrA1-sat2, sat2-aadA1-orfX:IS26); incluso un casete infrecuente que codifica resistencia a b-lactámicos ha aparecido como parte de un nuevo gran arreglo (88).

Figura 5. Arreglos en integrones clase 2 descritos en Argentina. Estructura del transposon Tn7 (44, 109) y de distintos arreglos publicados o presentados en Argentina (22, 79, 88-90). Las líneas de puntos en los genes tns implican que no fueron investigados o detectados en todos los casos. En el gráfico se mencionan las especies bacterianas donde fueron descritos.
PARA DESTACAR
El grado del conocimiento sobre los integrones ha cambiado enormemente en los últimos años, ya que se ha pasado de una etapa descriptiva a una funcional. Si bien la información disponible en la actualidad es muy amplia, todavía quedan puntos importantes por entender e investigar. A pesar de ello, se pueden establecer algunos conceptos generales que aluden a diferentes aristas de este tema.
Cuando se evalúan diferentes variables de estos elementos (integrones y superintegrones), como el grado de movilidad y de distribución filogenética, su frecuencia en un grupo taxonómico dado, la cantidad de regiones codificantes en casetes, la diversidad y el tamaño del pool de genes en casete, el número de casetes asociados, la relación de adquisición o pérdida de casetes, su capacidad para reconocer una alta variabilidad de secuencias dianas de recombinación y el grado de paralogía en los arreglos observados; se explica desde una óptica evolutiva cómo muchos microorganismos portadores de integrones pueden cambiar y adaptarse rápidamente a nichos ambientales diferentes e impredecibles. Esta flexibilidad no sólo contribuye a reclutar genes extraños que pueden, en última instancia, dotar de una mayor aptitud a la bacteria, sino que también le permite descartar genes que no le proveen una función importante.
Si el análisis se circunscribe al ámbito clínico, se pueden subrayar los siguientes ítems: 1- existe una alta frecuencia de aislamientos clínicos con integrones asociados a resistencia a antibióticos, 2- el número de casetes de resistencia presentes en un integrón es variable (de uno a más de cinco), 3- existe una real versatilidad de arreglos de casetes que codifican resistencia a diferentes familias de antibióticos, 4- están emergiendo nuevos genes en casete, sobre todo de algunos implicados en mecanismos de resistencia contra nuevos b-lactámicos o fluoroquinolonas, y 5- existen diferentes variantes involucradas en la expresión de estos mecanismos. Estos aspectos no deben desestimarse y particularmente deben alertar y comprometer a los profesionales e investigadores que se desarrollan en el área de la salud, para minimizar los posibles fracasos terapéuticos que puedan sobrevenir.
Por otro lado, la frecuente movilización en bloque de estos elementos asociados a resistencia (ya sea por transposición o conjugación) y su estable permanencia en varios linajes microbianos remarca no sólo su valor adaptativo, sino también la posible selección y diseminación de bacterias multirresistentes al administrar un agente antimicrobiano (o al hacerlo en forma inadecuada).
Se debe destacar que estas plataformas genéticas no quedan restringidas a aislamientos provenientes de diferentes nosocomios, ya que también se han detectado en aislamientos derivados de pacientes de la comunidad procedentes de diferentes nichos ambientales, e incluso de animales de granja, zoológicos, mascotas, etc., y que todos ellos pueden actuar como potenciales reservorios para su diseminación.
Finalmente, ¿qué cabe esperar? Que los integrones se tornen cada vez más eficientes en el reclutamiento de genes y que a su vez se asocien con estructuras cada vez más promiscuas, lo que llevará a la rápida emergencia y diseminación de microorganismos multirresistentes.
Agradecimientos: estamos especialmente agradecidos con la Lic. Ayelén Porto por la lectura crítica del manuscrito y por los detalles de discusión aportados. También queremos agradecer a la Dra. Marcela Radice y al Dr. Pablo Power por los trabajos realizados en conjunto y la gentileza en la información suministrada. Este trabajo fue financiado con subsidios ANPCyT y UBA otorgados a GG y con fondos ANPCyT y UNL otorgados a JDC. Ambos autores son miembros de la carrera de investigador científico de CONICET.
1. Ahmed AM, Motoi Y, Sato M, Maruyama A, Watanabe H, Fukumoto Y, et al. Zoo animals as reservoirs of gram-negative bacteria harboring integrons and antimicrobial resistance genes. Appl Environ Microbiol 2007; 73: 6686-90. [ Links ]
2. Almuzara M, Radice M, de Garate N, Kossman A, Cuirolo A, Santella G, et al. VIM-2-producing Pseudomonas putida, Buenos Aires. Emerg Infect Dis 2007; 13: 668-9. [ Links ]
3. Arakawa Y, Murakami M, Suzuki K, Ito H, Wacharotayankun R, Ohsuka S, et al. A novel integron-like element carrying the metallo-b-lactamase gene blaIMP. Antimicrob Agents Chemother 1995; 39: 1612-5. [ Links ]
4. Arcangioli M, Leroy-Sétrin S, Martel J, Chaslus-Dancla. E. A new chloramphenicol and florfenicol resistance gene flanked by two integron structures in Salmonella Typhimurium DT104. FEMS Microbiol Let 1999; 174: 327-32. [ Links ]
5. Arduino SM, Catalano M, Orman BE, Roy PH, Centrón. D. Molecular epidemiology of orf513-bearing class 1 integrons in multiresistant clinical isolates from Argentinean hospitals. Antimicrob Agents Chemother 2003; 47: 3945-9. [ Links ]
6. Arduino SM, Roy PH, Jacoby GA, Orman BE, Pineiro SA, Centrón. D. blaCTX-M-2 is located in an unusual class 1 integron (In35) wich includes orf513. Antimicrob Agents Chemother 2002; 46: 2303-6. [ Links ]
7. Barbolla R, Catalano M, Orman BE, Famiglietti A, Vay C, Smayevsky J, et al. Class 1 integrons increase trimethoprimsulfamethoxazole MICs against epidemiologically unrelated Stenotrophomonas maltophilia isolates. Antimicrob Agents Chemother 2004; 48: 666-9. [ Links ]
8. Barlow RS, Gobius KS. Diverse class 2 integrons in bacteria from beef cattle sources. J Antimicrob Chemother 2006; 58: 1133-8. [ Links ]
9. Bennett PM. Integrons and gene cassettes: a genetic construction kit for bacteria. J Antimicrob Chemother 1999; 43: 1-4. [ Links ]
10. Bissonnette L, Champetier S, Buisson JP, Roy PH. Characterization of the nonenzymatic chloramphenicol resistance (cmlA) gene of the In4 integron of Tn1696: similarity of the product to transmembrane transport proteins. J Bacteriol 1991; 173: 4493-502. [ Links ]
11. Bissonnette L, Roy PH. Characterization of In0 of Pseudomonas aeruginosa plasmid pVS1, an ancestor of integrons of multiresistance plasmids and transposons of gram-negative bacteria. J Bacteriol 1992; 174: 1248-57. [ Links ]
12. Boucher Y, Labbate M, Koenig JE, Stokes HW. Integrons: mobilizable platforms that promote genetic diversity in bacteria. Trends Microbiol 2007; 15: 301-9. [ Links ]
13. Bouvier M, Demarre G, Mazel D. Integron cassette insertion: a recombination process involving a folded single strand substrate. EMBO J 2005; 24: 4356-67. [ Links ]
14. Brown HJ, Stokes HW, Hall RM. The integrations In0, In2, and In5 are defective transposon derivates. J Bacteriol 1996; 178: 4429-37. [ Links ]
15. Centron D, Roy PH. Characterization of the 6'-Naminoglycoside acetyltransferase gene aac(6')-Iq from the integron of a natural multiresistance plasmid. Antimicrob Agents Chemother 1998; 42: 1506-8. [ Links ]
16. Centron D, Roy PH. Presence of a group II intron in a multiresistant Serratia marcescens strain that harbors three integrons and a novel gene fusion. Antimicrob Agents Chemother 2002; 46: 1402-9. [ Links ]
17. Clark CA, Purins L, Kaewrakon P, Focareta T, Manning PA. The Vibrio cholerae O1 chromosomal integron. Microbiology 2000; 146: 2605-12. [ Links ]
18. Collis CM, Hall RM. Expression of antibiotic resistance genes in the integrated cassettes of integrons. Antimicrob Agents Chemother 1995; 39: 155-62. [ Links ]
19. Collis CM, Hall. RM. Genes cassettes from the insert region of integrons are excised as covalently closed circles. Mol Microbiol 1992; 6: 2875-85. [ Links ]
20. Collis CM, Recchia GD, Kim MJ, Stokes HW, Hall RM. Efficiency of recombination reactions catalyzad by class 1 integron integrase IntI1. J Bacteriol 2001; 183: 2535-42. [ Links ]
21. Correia M, Boavida F, Grosso F, Salgado MJ, Lito LM, Cristino JM, et al. Molecular characterization of a new class 3 integron in Klebsiella pneumoniae. Antimicrob Agents Chemother 2003; 47: 2838-43. [ Links ]
22. Crespo O, Catalano M, Pineiro S, Matteo M, Leanza A, Centron D. Tn7 distribution in Helicobacter pylori: a selective paradox. Int J Antimicrob Agents 2005; 25: 341-4. [ Links ]
23. Cuirolo A, Almuzara M, Radice M, Gutkind G. Molecular characterization of IMP-13 producing Pseudomonas aeruginosa. 46th Interscience Conference on Antimicrobial Agents and Chemotherapy, 2006, p. 109, San Francisco, EE.UU. [ Links ]
24. Davies. J. Inactivation of antibiotics and the dissemination of resistance genes. Science 1994; 264: 375-82. [ Links ]
25. Depardieu F, Podglajen I, Leclercq R, Collatz E, Courvalin P. Modes and modulations of antibiotic resistance gene expression. Clin Microbiol Rev 2007; 20: 79-114. [ Links ]
26. Di Conza J, Ayala J, Porto A, Mollerach M, Gutkind G. Molecular characterization of InJR06, a class 1 integron located in a conjugative plasmid of Salmonella enterica serotype Typhimurium. Int Microbiol 2005; 8: 287-90. [ Links ]
27. Di Conza J, Ayala J, Power P, Mollerach M, Gutkind G. Novel Class 1 integron (InS21) carrying the blaCTX-M-2 gene in Salmonella enterica serovar Infantis. Antimicrob Agents Chemother 2002; 46: 2257-61. [ Links ]
28. Dubois V, Parizano MP, Arpin C, Coulange L, Bezian MC, Quentin C. High genetic stability of integrons in clinical isolates of Shigella spp. of worldwide origin. Antimicrob Agents Chemother 2007; 51: 1333-40. [ Links ]
29. Faccone D, Barreiro L, Pasterán F, Melano R, Rapoport M, Galas M, et al. Genetic support of blaGES-1: novel location in Tn402-like transposons of Pseudomonas aeruginosa (Pae) from Argentina. 48th Interscience Conference on Antimicrobial Agents and Chemotherapy, 2008, Resumen C1- 1062, p. 120, Washington, DC, EE.UU. [ Links ]
30. Fluit AC, Schmitz FJ. Resistance integrons and superintegrons. Clin Microbiol Infect 2004; 10: 272-88. [ Links ]
31. Fluit AC, Schmitz. FJ. Class 1 integrons, gene cassettes, mobility, and epidemiology. Eur J Clin Microbiol Infect Dis 1999; 18: 761-70. [ Links ]
32. Fonseca EL, Dos Santos Freitas F, Vieira VV, Vicente AC. New qnr gene cassettes associated with superintegron repeats in Vibrio cholerae O1. Emerg Infect Dis 2008; 14: 1129-31. [ Links ]
33. Gassama-Sow A, Diallo MH, Boye CS, Garin B, Sire JM, Sow AI, et al. Class 2 integron-associated antibiotic resistance in Shigella sonnei isolates in Dakar, Senegal. Int J Antimicrob Agents 2006; 27: 267-70. [ Links ]
34. Gillings M, Boucher Y, Labbate M, Holmes A, Krishnan S, Holley M, et al. The evolution of class 1 integrons and the rise of antibiotic resistance. J Bacteriol 2008; 190: 5095-100. [ Links ]
35. Gravel A, Messier N, Roy PH. Point mutations in the integron integrase IntI1 that affect recombination and/or substrate recognition. J Bacteriol 1998; 180: 5437-42. [ Links ]
36. Guerra B, Soto S, Helmuth R, Mendoza MC. Characterization of a self-transferable plasmid from Salmonella enterica serotype Typhimurium clinical isolates carrying two integronborne gene cassettes together with virulence and drug resistance genes. Antimicrob Agents Chemother 2002; 46: 2977-81. [ Links ]
37. Hall RM, Brown HJ, Brookes DE, Stokes HW. Integrons found in different locations have identical 5' ends but variable 3' ends. J Bacteriol 1994; 176: 6286-94. [ Links ]
38. Hall RM, Collis CM, Kim MJ, Partridge SR, Recchia GD, Stokes HW. Mobile gene cassettes in evolution. Ann N Y Acad Sci 1999; 87: 68-80. [ Links ]
39. Hall RM, Collis CM. Antibiotic resistance in gram-negative bacteria: the role of gene cassettes and integrons. Drug Resist Updat 1998; 1: 109-19. [ Links ]
40. Hall RM, Collis CM. Mobile gene cassettes and integrons: capture and spread of genes by site-specific recombination. Mol Microbiol 1995; 15: 593-600. [ Links ]
41. Hall RM, Stokes HW. Integrons or super integrons? Microbiology 2004; 150: 3-4. [ Links ]
42. Hansson K, Skold O, Sundstrom L. Non-palindromic attI sites of integrons are capable of site-specific recombination with one another and with secondary targets. Mol Microbiol 1997; 26: 441-53. [ Links ]
43. Hansson K, Sundstrom L, Pelletier A, Roy PH. IntI2 integron integrase in Tn7. J Bacteriol 2002; 184: 1712-21. [ Links ]
44. Heikkila E, Sundstrom L, Skurnik M, Huovinen P. Analysis of genetic localization of the type I trimethoprim resistance gene from Escherichia coli isolated in Finland. Antimicrob Agents Chemother 1991; 35: 1562-9. [ Links ]
45. Holmes AJ, Gillings MR, Nield BS, Mabbutt BC, Nevalainen KM, Stokes HW. The gene cassette metagenome is a basic resource for bacterial genome evolution. Environ Microbiol 2003; 5: 383-94. [ Links ]
46. Kazama H, Hamashima H, Sasatsu M, Arai T. Distribution of the antiseptic-resistance gene qacE![]() 1 in gram-positive bacteria. FEMS Microbiol Lett 1998; 165: 295-9. [ Links ]
1 in gram-positive bacteria. FEMS Microbiol Lett 1998; 165: 295-9. [ Links ]
47. Kim TE, Kwon HJ, Cho SH, Kim S, Lee BK, Yoo HS, et al. Molecular differentiation of common promoters in Salmonella class 1 integrons. J Microbiol Methods 2007; 68: 453-7. [ Links ]
48. Laraki N, Galleni M, Thamm I, Riccio ML, Amicosante G, Frere JM, et al. Structure of In31, a blaIMP-containing Pseudomonas aeruginosa integron phyletically related to In5, which carries an unusual array of gene cassettes. Antimicrob Agents Chemother 1999; 43: 890-901. [ Links ]
49. Leverstein-Van Hall MA, Paauw A, Box AT, Blok HE, Verhoef J, Fluit AC. Presence of integron-associated resistance in the community is widespread and contributes to multidrug resistance in the hospital. J Clin Microbiol 2002; 40: 3038-40. [ Links ]
50. Levesque C, Brassard S, Lapointe J, Roy PH. Diversity and relative strength of tandem promoters for the antibiotic-resistance genes of several integrons. Gene 1994; 142: 49-64. [ Links ]
51. Levesque C, Piché L, Larose C, Roy PH. PCR mapping of integron reveals several novel combinations of resistance genes. Antimicrob Agents Chemother 1995; 39: 185-91. [ Links ]
52. MacDonald D, Demarre G, Bouvier M, Mazel D, Gopaul DN. Structural basis for broad DNA-specificity in integron recombination. Nature 2006; 440: 1157-62. [ Links ]
53. Manning PA, Clark CA, Focareta T. Gene capture in Vibrio cholerae. Trends Microbiol 1999; 7: 93-95. [ Links ]
54. Marquez C, Labbate M, Ingold AJ, Chowdhury PR, Ramirez MS, Centron D, et al. Recovery of a functional class 2 integron from an Escherichia coli strain mediating a urinary tract infection. Antimicrob Agents Chemother 2008; 52: 4153-4. [ Links ]
55. Martinez-Freijo P, Fluit A, Schmitz F, Verhoef J, Jones M. Many class I integrons comprise distintc stable structures ocuurring in diferent species of Enterobacteriaceae isolated from widespread geographic regions in Europe. Antimicrob Agents Chemother 1999; 43: 686-9. [ Links ]
56. Martinez-Freijo P, Fluit AC, Schmitz FJ, Grek VSC, Verhoef J, Jones ME. Class 1 integrons in gram-negative isolates from different European hospitals and association with decreased susceptibility to multiple antibiotic compounds. J Antimicrob Chemother 1998; 42: 689-96. [ Links ]
57. Mazel D. Integrons: agents of bacterial evolution. Nat Rev Microbiol 2006; 4: 608-20. [ Links ]
58. Mazel D, Dychinco B, Webb VA, Davies J. A distinctive class of integron in the Vibrio cholerae genome. Science 1998; 280: 605-8. [ Links ]
59. Mendes RE, Castanheira M, Toleman MA, Sader HS, Jones RN, Walsh TR. Characterization of an integron carrying blaIMP-1 and a new aminoglycoside resistance gene, aac(6')- 31, and its dissemination among genetically unrelated clinical isolates in a Brazilian hospital. Antimicrob Agents Chemother 2007; 51: 2611-4. [ Links ]
60. Messier N, Roy PH. Integron integrases possess a unique additional domain necessary for activity. J Bacteriol 2001; 183: 6699-706. [ Links ]
61. Naas T, Mikami Y, Imai T, Poirel L, Nordmann P. Characterization of In53, a class 1 plasmid- and composite transposon-located integron of Escherichia coli which carries an unusual arrays of gene cassettes. J Bacteriol 2001; 183: 235-49. [ Links ]
62. Naas T, Poirel L, Karim A, Nordmann P. Molecular characterization of In50, a class 1 integron encoding the gene for the extended-spectrum beta-lactamase VEB-1 in Pseudomonas aeruginosa. FEMS Microbiol Lett 1999; 176: 411-9. [ Links ]
63. Nandi S, Maurer JJ, Hofacre C, Summers AO. Gram-positive bacteria are a major reservoir of class 1 antibiotic resistance integrons in poultry litter. Proc Natl Acad Sci U S A 2004; 101: 7118-22. [ Links ]
64. Nastasi A, Mammina C. Presence of class 1 integrons in multigrug-resistant, low-prevalence Salmonella serotypes, Italy. Emer Infect Dis 2001; 7: 455-8. [ Links ]
65. Nesvera J, Hochmannova J, Patek M. An integron of class I is present on the plasmid pCG4 from gram-positive bacterium Corynebacterium glutamicum. FEMS Microbiol Lett 1998; 169: 391-5. [ Links ]
66. Nield BS, Holmes AJ, Gillings MR, Recchia GD, Mabbutt BC, Helena-Nevalainen KM, et al. Recovery of new integron classes from environmental DNA. FEMS Microbiol Lett 2001; 195: 59-65. [ Links ]
67. Orman BE, Piñeiro SA, Arduino S, Galas M, Melano R, Caffer MI, et al. Evolution of multiresistance in nontyphoid Salmonella serovars from 1984 to 1998 in Argentina. Antimicrob Agents Chemother 2002; 46: 3963-70. [ Links ]
68. Pagniez G, Radice M, Cuirolo A, Rodriguez O, Rodriguez H, Vay C, et al. Prevalence of metallo-beta-lactamase in carbapenem resistant Pseudomonas aeruginosa at a university hospital of Buenos Aires City. Rev Argent Microbiol 2006; 38: 33-7. [ Links ]
69. Parsons Y, Hall RM, Stokes HW. A new trimethoprim resistance gene, dhfrX, in the In7 integron of plasmid pDGO100. Antimicrob Agents Chemother 1991; 35: 2436-9. [ Links ]
70. Partridge SR, Brown HJ, Stokes HW, Hall RM. Transposons Tn1696 and Tn21 and their integrons In4 and In2 have independent origins. Antimicrob Agents Chemother 2001; 45: 1263-70. [ Links ]
71. Partridge SR, Collis CM, Hall RM. Class 1 integron containing a new gene cassette, aadA10, associated with Tn1404 from R151. Antimicrob Agents Chemother 2002; 46: 2400-8. [ Links ]
72. Partridge SR, Hall RM. In34, a complex In5 family class 1 integron containing orf513 and dfrA10. Antimicrob Agents Chemother 2003; 47: 342-9. [ Links ]
73. Partridge SR, Recchia GD, Scaramuzzi C, Collis CM, Stokes HW, Hall RM. Definition of the attI1 site of class 1 integrons. Microbiology 2000; 146: 2855-64. [ Links ]
74. Partridge SR, Recchia GD, Stokes HW, Hall RM. Family of class 1 integrons related to In4 from Tn1696. Antimicrob Agents Chemother 2001; 45: 3014-20. [ Links ]
75. Paulsen IT, Littlejohn TG, Radstrom P, Sundstrom L, Skold O, Swedberg G, et al. The 3' conserved segment of integrons contains a gene associated with multidrug resistance to antiseptics and disinfectants. Antimicrob Agents Chemother 1993; 37: 761-8. [ Links ]
76. Penteado AP, Castanheira M, Pignatari AC, Guimaraes T, Mamizuka EM, Gales AC. Dissemination of blaIMP-1-carrying integron In86 among Klebsiella pneumoniae isolates harboring a new trimethoprim resistance gene dfr23. Diagn Microbiol Infect Dis 2009; 63: 87-91. [ Links ]
77. Ploy MC, Denis F, Courvalin P, Lambert T. Molecular characterization of integrons in Acinetobacter baumannii: description of a hybrid class 2 integron. Antimicrob Agents Chemother 2000; 44: 2684-8. [ Links ]
78. Poirel L, Lambert T, Turkoglu S, Ronco E, Gaillard JL, Nordmann P. Characterization of class 1 integrons from Pseudomonas aeruginosa that ontain the blaVIM-2 carbapenem- hydrolyzing b-lactamase gene and of two novel aminoglycoside resistance gene cassettes. Antimicrob Agents Chemother 2001; 45: 546-52. [ Links ]
79. Porto A. Mecanismos de adquisición de genes de resistencia a antibióticos. Integrones presentes en Enterobacteriaceae [Tesina de Grado]. Santa Fe: Universidad Nacional del Litoral, 2006. [ Links ]
80. Porto A, Ayala J, Gutkind G, Di Conza J. A novel OXA-10- like b-lactamase is present in different Enterobacteriaceae. Diagn Microbiol Infect Dis 2010; 66: 228-9. [ Links ]
81. Porto A, Rico M, Méndez E, Di Conza J. Las enterobacterias resistentes a múltiples drogas están fuertemente asociadas a la presencia de integrones. Congreso SADEBAC- IV Actividad Científica Anual de la Sociedad Argentina de Bacteriología Clínica, 2006, Resumen 15514, edición electrónica, Ciudad Autónoma de Buenos Aires, Argentina. [ Links ]
82. Porto A, Vaccari MC, Méndez E, Di Conza J. Integrones clase 1 y 2 presentes en aislamientos hospitalarios de enterobacterias. Revista FABICIB 2005; 9: 141-7. [ Links ]
83. Power P, Di Conza J, Gutkind G. blaCTX-M-2 gene present in InS21 is associated with Tn1000 and other truncated transposition- related genes. 106th General Meeting, 2006, Abstract A-007, p. 2, Orlando, Florida, EE.UU. [ Links ]
84. Power P, Galleni M, Di Conza J, Ayala JA, Gutkind G. Description of In116, the first blaCTX-M-2-containing complex class 1 integron found in Morganella morganii isolates from Buenos Aires, Argentina. J Antimicrob Chemother 2005; 55: 461-5. [ Links ]
85. Quiroga MP, Andres P, Petroni A, Soler Bistue AJ, Guerriero L, Vargas LJ, et al. Complex class 1 integrons with diverse variable regions, including aac(6')-Ib-cr, and a novel allele, qnrB10, associated with ISCR1 in clinical enterobacterial isolates from Argentina. Antimicrob Agents Chemother 2007; 51: 4466-70. [ Links ]
86. Radice M, Stortz M, Rodríguez H, Vay C, Famiglietti A, Gutkind G. Caracterización de BLEE en Pseudomonas aeruginosa aisladas en un hospital de Buenos Aires. VII Congreso Argentino de la Sociedad Argentina de Infectología, 2007, p. 20, Mar del Plata, Argentina. [ Links ]
87. Radstrom P, Skold O, Swedberg G, Flensburg J, Roy PH, Sundstrom L. Transposon Tn5090 of plasmid R751, which carries an integron, is related to Tn7, Mu, and the retroelements. J Bacteriol 1994; 176: 3257-68. [ Links ]
88. Ramirez MS. Caracterización molecular y funcional de integrones de clase 2 en aislamientos clínicos y ambientales. [Tesis Doctoral]. Buenos Aires: Universidad de Buenos Aires, 2008. [ Links ]
89. Ramirez MS, Quiroga C, Centron D. Novel rearrangement of a class 2 integron in two non-epidemiologically related isolates of Acinetobacter baumannii. Antimicrob Agents Chemother 2005; 49: 5179-81. [ Links ]
90. Ramirez MS, Vargas LJ, Cagnoni V, Tokumoto M, Centron D. Class 2 integron with a novel cassette array in a Burkholderia cenocepacia isolate. Antimicrob Agents Chemother 2005; 49: 4418-20. [ Links ]
91. Recchia GD, Hall RM. Gene cassettes: a new class of mobile element. Microbiology 1995; 141: 3015-27. [ Links ]
92. Recchia GD, Hall RM. Origins of the mobile gene cassettes found in integrons. Trends Microbiol 1997; 5: 389-94. [ Links ]
93. Recchia GD, Stokes HW, Hall RM. Characterisation of specific and secondary recombinations sites recognised by the integron DNA integrase. Nucleic Acids Res 1994; 22: 2071-8. [ Links ]
94. Robicsek A, Strahilevitz J, Jacoby GA, Macielag M, Abbanat D, Park CH, et al. Fluoroquinolone-modifying enzyme: a new adaptation of a common aminoglycoside acetyltransferase. Nat Med 2006; 12: 83-8. [ Links ]
95. Rodriguez-Martinez JM, Poirel L, Canton R, Nordmann P. Common region CR1 for expression of antibiotic resistance genes. Antimicrob Agents Chemother 2006; 50: 2544-6. [ Links ]
96. Rosser SJ, Young HK. Identification and characterization of class 1 integrons in bacteria from an aquatic environment. J Antimicrob Chemother 1999; 44: 11-8. [ Links ]
97. Rowe-Magnus DA, Guérout AM, Mazel D. Super-integrons. Res Microbiol 1999; 150: 641-51. [ Links ]
98. Rowe-Magnus DA, Mazel D. Resistance gene capture. Curr Opin Microbiol 1999; 2: 483-8. [ Links ]
99. Rowe-Magnus DA, Mazel D. Integrons: natural tools for bacterial genoma evolution. Curr Opin Microbiol 2001; 4: 565-9. [ Links ]
99 a.Ruggiero M, Di Conza J, Famiglietti A, Vay C, Gutkind G. Genetic environment characterization of blacTX-M-2 in Pseudomonas aeruginosa in Argentina. 49th ICAAC, 2009, Resumen C2-633, p. 100, San Francisco, CA, EE.UU. [ Links ]
100. Saka HA, Sola C, Pasterán F, Galas M, Culasso C, Petroni A, et al. Ampicillin resistance in Vibrio cholerae non-O1, non-O139 recovered in Córdoba, Argentina, is largely mediated by CARB-like b-lactamases. XI Congreso Argentino de Microbiología, Resumen 34. Rev Argent Microbiol 2007; 39 Supl 1: 29.
101. Shi L, Zheng M, Xiao Z, Asakura M, Su J, Li L, et al. Unnoticed spread of class 1 integrons in gram-positive clinical strains isolated in Guangzhou, China. Microbiol Immunol 2006; 50: 463-7. [ Links ]
102. Shibata N, Doi Y, Yamane K, Yagi T, Kurokawa H, Shibayama K, et al. PCR typing of genetic determinants for metallo-b-lactamases and integrases carried by gram-negative bacteria isolated in Japan, with focus on the class 3 integron. J Clin Microbiol 2003; 41: 5407-13.
103. Soler Bistue AJ, Martin FA, Petroni A, Faccone D, Galas M, Tolmasky ME, et al. Vibrio cholerae InV117, a class 1 integron harboring aac(6')-Ib and blaCTX-M-2, is linked to transposition genes. Antimicrob Agents Chemother 2006; 50: 1903-7. [ Links ]
104. Stokes HW, Hall RM. The integron In1 in plasmid R46 includes two copies of the oxa-2 gene cassette. Plasmid 1992; 28: 225-34. [ Links ]
105. Stokes HW, Hall RM. A novel family of potentially mobile DNA elements encoding site-specific gene-integration functions: integrons. Mol Microbiol 1989; 3: 1669-83. [ Links ]
106. Stokes HW, O'Gorman DB, Recchia GD, Parsekhian M, Hall RM. Structure and function of 59-base element recombination sites associated with mobile gene cassettes. Mol Microbiol 1997; 26: 731-45. [ Links ]
107. Stokes HW, Tomaras C, Parsons Y, Hall RM. The partial 3'-conserved segment duplications in the integrons In6 from pSa and In7 from pDGO100 have a common origin. Plasmid 1993; 30: 39-50. [ Links ]
108. Sundstrom L, Roy PH, Skold O. Site-specific insertion of three structural gene cassettes in transposon Tn7. J Bacteriol 1991; 173: 3025-8. [ Links ]
109. Tietze E, Brevet J, Tschape H. Relationships among the streptothricin resistance transposons Tn1825 and Tn1826 and the trimethoprim resistance transposon Tn7. Plasmid 1987; 18: 246-9. [ Links ]
110. Toleman MA, Bennett PM, Walsh TR. Common regions e.g. orf513 and antibiotic resistance: IS91-like elements evolving complex class 1 integrons. J Antimicrob Chemother 2006; 58: 1-6. [ Links ]
111. Toleman MA, Bennett PM, Walsh TR. ISCR elements: novel gene-capturing systems of the 21st century? Microbiol Mol Biol Rev 2006; 70: 296-316. [ Links ]
112. Tolmasky ME, Crosa JH. Genetic Organization of antibiotic resistance genes aac(6')-Ib, aadA, and oxa-9 in the multirresistance transposon Tn1331. Plasmid 1993; 29: 31-40. [ Links ]
113. Tolmasky ME. Bacterial resistance to aminoglycosides and b-lactamse the Tn1331 transposon paradigm. Front Biosci 1999; 5: d20-9.
114. Valentine CR, Heinrich MJ, Chissoe SL, Roe BA. DNA sequence of direct repeats of the sulI gene of plasmid pSa. Plasmid 1994; 32: 222-7. [ Links ]
115. Valverde A, Canton R, Galan JC, Nordmann P, Baquero F, Coque TM. In117, an unusual In0-like class 1 integron containing CR1 and blaCTX-M-2 and associated with a Tn21-like element. Antimicrob Agents Chemother 2006; 50: 799-802. [ Links ]
116. Verdet C, Arlet G, Barnaud G, Lagrange PH, Philippon A. A novel integron in Salmonella enterica serovar Enteritidis, carrying the blaDHA-1 gene and its regulator gene ampR originated from Morganella morganii. Antimicrob Agents Chemother 2000; 44: 222-5. [ Links ]
117. Villa L, Pezella C, Tosini F, Visca P, Petrucca A, Carattoli A. Multiple-antibiotic resistance mediated by structurally related IncL/M plasmid carrying an extended-spectrum blactamase gene and a class 1 integron. Antimicrob Agents Chemother 2000; 44: 2911-4. [ Links ]
118. Villa L, Visca P, Tosini F, Pezzella C, Carattoli A. Composite integron array generated by insertion of an orf341-type integron within a Tn21-like element. Microb Drug Resist 2002; 8: 1-8.
119. Walsh TR, Toleman MA, Poirel L, Nordmann P. Metallo-ß-lactamases: the quiet before the storm? Clin Microbiol Rev 2005; 18: 306-25. [ Links ]
120. Xu H, Davies J, Miao V. Molecular characterization of class 3 integrons from Delftia spp. J Bacteriol 2007; 189: 6276-83. [ Links ]
121. Xu Z, Shi L, Alam MJ, Li L, Yamasaki S. Integron-bearing methicillin- resistant coagulase-negative staphylococci in South China, 2001-2004. FEMS Microbiol Lett 2008; 278: 223-30. [ Links ]
122. Xu Z, Shi L, Zhang C, Zhang L, Li X, Cao Y, et al. Nosocomial infection caused by class 1 integron-carrying Staphylococcus aureus in a hospital in South China. Clin Microbiol Infect 2007; 13: 980-4. [ Links ]
Recibido: 21/04/09
Aceptado: 27/11/09














