Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Revista argentina de microbiología
versión impresa ISSN 0325-7541versión On-line ISSN 1851-7617
Rev. argent. microbiol. v.42 n.2 Ciudad Autónoma de Buenos Aires abr./jun. 2010
MICROBIOLOGÍA BÁSICA
Amplificación del gen CAP59 en Cryptococcus neoformans y Cryptococcus gattii directamente a partir de una suspensión de levaduras
N. Siachoque, V.M. Jewtuchowicz, C. Iovannitti, M.T. Mujica*
Departamento de Microbiología, Parasitología e Inmunología de la Facultad de Medicina de la Universidad de Buenos Aires. Paraguay 2155 piso 11 (1121) Buenos Aires, Argentina.
*Correspondencia. E-mail: mtmujica@gmail.com
RESUMEN
Cryptococcus es una levadura capsulada de la clase Basidiomycetes y es el agente etiológico de la criptococosis. La criptococosis es una de las infecciones oportunistas más comunes en pacientes inmunosuprimidos, aunque puede afectar a individuos inmunocompetentes. En los últimos años, la identificación de especies fúngicas de importancia médica se ha logrado gracias a la amplificación de determinadas regiones del ADN o de genes fúngicos mediante la reacción en cadena de la polimerasa (PCR). El gen CAP59 está implicado en la síntesis de la cápsula en Cryptococcus neoformans y Cryptococcus gattii, y es de utilidad en la identificación molecular de sus serotipos. En este trabajo utilizamos aislamientos de distintos serotipos provenientes de una colección de cultivos y de C. neoformans recuperados de pacientes con criptococosis. La amplificación del gen CAP59 se realizó a partir de suspensiones estandarizadas de levaduras de los diferentes aislamientos de Cryptococcus, las que actuaron como molde. Este procedimiento fue rápido, sencillo y económico y no requirió ningún paso previo a la PCR, lo que puede facilitar en forma considerable la realización de estudios taxonómicos en los laboratorios que disponen de herramientas de biología molecular.
Palabras clave: Gen CAP59; Cryptococcus neoformans; Criptococosis; Cryptococcus gattii
ABSTRACT
CAP59 gene amplification in Cryptococcus neoformans and Cryptococcus gattii directly from a yeast suspension. Cryptococcus is an encapsulated yeast of class Basidiomycetes, etiologic agent of cryptococcosis. Cryptococcosis is one of the most common opportunistic infections in immunosuppressed patients, although it can affect immunocompetent individuals. In recent years, the identification of medically important fungal species has been achieved through the amplification of specific regions or genes of fungal DNA by polymerase chain reaction (PCR). The CAP59 gene is involved in the synthesis of the capsule in Cryptococcus neoformans and Cryptococcus gattii, and is useful in the molecular identification of serotypes. In this research, we use yeasts of different serotypes from collection strains and C. neoformans isolates recovered from patients with cryptococcosis. A standardized yeast suspension from different Cryptococcus isolates as template allowed CAP59 gene amplification. This procedure was quick, simple, and inexpensive and required no PCR steps. This is important for taxonomic studies in laboratories with implemented molecular biology tools.
Keys word: CAP59 gene; Cryptococcus neoformans; Cryptococcosis; Cryptococcus gattii
La levadura capsulada Cryptococcus pertenece a la clase Basidiomycetes y es el agente etiológico de la criptococosis, una micosis que en las últimas tres décadas se ha convertido en una de las infecciones oportunistas más comunes en pacientes inmunosuprimidos, aunque también puede afectar a individuos inmunocompetentes (10). C. neoformans es un microorganismo con distribución universal y es encontrado, sobre todo, en individuos inmunocomprometidos, particularmente en pacientes infectados con el virus de la inmunodeficiencia humana. En contraste, Cryptococcus gattii puede infectar a individuos inmunocompetentes y se lo aísla generalmente de áreas subtropicales o tropicales, aunque se ha recuperado en regiones más templadas (6, 7, 11, 14).
Durante varias décadas, la estructura del polisacárido capsular permitió agrupar a las cepas de C. neoformans en dos variedades que incluían a los 5 serotipos: C. neoformans variedad neoformans para las cepas de los serotipos A, D y AD, y C. neoformans variedad gattii para las cepas con los serotipos B y C (10). Con base en estudios genómicos, se propuso que el serotipo A fuera reclasificado como C. neoformans var. grubii, mientras que el serotipo D continuó perteneciendo a la especie neoformans y a la variedad neoformans (4). Una especie nueva e independiente, C. gattii, ha sido recientemente propuesta para lo que antes se conocía como C. neoformans var. gattii (8). Estos cambios nos llevan a considerar como válidos a C. neoformans var. grubii (serotipo A), C. neoformans var. neoformans (serotipo D), C. gattii (serotipos B y C) y el híbrido de C. neoformans perteneciente al serotipo AD. El significado de los serotipos de Cryptococcus puede extenderse más allá de lo taxonómico a lo epidemiológico (1, 15), por la posibilidad de que, según sea el serotipo, Cryptococcus afecte pacientes con diferentes estados inmunes, presenten diferentes cursos clínicos y tengan distinta respuesta a los antifúngicos (7, 11, 14). En los últimos años se han desarrollado numerosos métodos basados en la amplificación de determinados genes o regiones del ADN mediante la reacción en cadena de la polimerasa (PCR) para la identificación de especies fúngicas y serotipos de C. neoformans (3, 5). Así, el análisis de la secuencia nucleotídica del gen URA5 y su posterior análisis filogenético permitió separar los serotipos A y D en dos grupos bien definidos (5). El gen que codifica la fosfolipasa B1 (PLB1) resultó útil en la diferenciación de las variedades y serotipos a través de PCR y un análisis posterior de polimorfismos en la longitud de los fragmentos de restricción (RFLP) (9). Los genes CAP10, CAP59, CAP60 y CAP64 se consideran esenciales para la formación de la cápsula en C. neoformans (12) y están relacionados con los serotipos A, B, C, D y el híbrido AD (3).
El aislamiento y la purificación del ADN es un paso clave en los estudios de biología molecular. Los métodos de extracción de ADN fúngico involucran la ruptura de la pared celular mediante la lisis enzimática o mecánica y, en general, requieren pasos adicionales para la obtención de ADN puro.
En este trabajo evaluamos la utilidad de amplificar el gen CAP59 a partir de suspensiones estandarizadas de levaduras de Cryptococcus de los distintos serotipos, las que actuaron como molde en la PCR, sin ningún paso previo.
Aislamientos en estudio. Se usaron las cepas de referencia C. neoformans var. grubii serotipo A (ATCC 6352), C. gattii serotipo B (ATCC 32269), C. gattii serotipo C (ATCC 32608), C. neoformans var. neoformans serotipo D (ATCC 28957) y 20 aislamientos de Cryptococcus provenientes de pacientes con criptococosis, de los cuales 14 se obtuvieron de líquido cefalorraquídeo (LCR) y 6 de hemocultivos. La identificación de los aislamientos se realizó mediante criterios morfológicos y bioquímicos: producción de melanina en medio Staib, crecimiento a 37 ºC, producción de ureasa en medio de Christensen, estudio de asimilación de fuentes de carbono utilizando el sistema API ID 32 C (bioMérieux, Marcy-L'Étoile, Francia) y utilización de la glicina-canavanina-azul de bromotimol (GCB) (7, 8,10).
Estudios moleculares. Los diferentes aislamientos se cultivaron en agar Sabouraud y se incubaron a 37 °C durante 48 h. Para la amplificación del gen CAP59 se realizaron suspensiones de las levaduras en agua destilada estéril con opacidades correspondientes a 0,5, 1, 2 y 3 de la escala de Mc Farland; una alícuota de 10 μl de cada suspensión se usó como molde para la PCR.
Para los aislamientos de C. neoformans provenientes de cultivos de pacientes con criptococosis se realizaron suspensiones en agua destilada con opacidad correspondiente a 2 de la escala de Mc Farland.
PCR. La reacción de PCR se realizó según el siguiente protocolo: tampón PCR 1 x 5 μl, 200 μM de cada de-soxirribonucleósido trifosfato (dATP, dGTP, dCTP, dTTP), 0,3 mM de MgCl2, 0,4 μM de cada iniciador (CAP59F: 5'-CCTTGCCGAAGTTCGAAACG-'3 y CAP59R: 5'-AATCGGTGGTTGGATTCAGTGT-'3) (3), 10 μl de molde, 1,25 U/μl Taq DNA polimerasa (Invitrogen, Argentina) y suficiente agua destilada para que el volumen final de reacción fuera de 50 μl. La amplificación del gen CAP59 fue optimizada en nuestro laboratorio con los siguientes ciclos: 1 min a 94 °C seguido de 30 ciclos de 1 min a 94 °C, 30 seg a 65 °C y 1 min a 72 °C, con una extensión final de 15 min a 72 °C. Se empleó un termociclador MiniCycler (TM MJ Research Inc, NY, EE.UU.); en cada experimento se incluyeron controles negativos.
Detección del producto de PCR. Los fragmentos amplificados se separaron por electroforesis en gel de agarosa al 1,5% en tampón TBE (0,09 M Tris, 0,09 M ácido bórico y 2 mM de EDTA, pH 8,3) durante 90 minutos a 90 voltios y se visualizaron con bromuro de etidio bajo irradiación con luz UV.
Determinación de serotipos. La identificación de los serotipos de Cryptococcus provenientes de pacientes con criptococosis se realizó a partir de la amplificación del gen CAP59 (como ya se detalló) y posterior RFLP con las enzimas BsmFI (BioLabs Inc®), HpaII (Fermentas®) y BshTI (Fermentas®), de acuerdo con las indicaciones dadas por sus fabricantes (3). Los fragmentos digeridos se separaron por electroforesis en gel de agarosa en tampón TBE a 90 voltios durante 2 horas y se visualizaron con bromuro de etidio bajo irradiación con luz UV.
Este estudio demostró que las diluciones correspondientes a una opacidad igual al tubo 1, 2 ó 3 de la escala de Mc Farland de las diferentes cepas de referencia de C. neoformans var. grubii, C. neoformans var. neoformans y C. gattii serotipos B y C con los cebadores correspondientes, permitieron la amplificación del gen CAP59 directamente a partir del cultivo, y como se esperaba, se obtuvo un producto de amplificación de un tamaño aproximado de 370 pb (3) (Figura 1). La PCR a partir de los aislamientos clínicos de C. neoformans resultó positiva para el gen CAP59 en el 100% de los casos. En la Figura 2 se observan los productos amplificados mediante PCR del gen CAP59 en 9 de los 20 aislamientos estudiados. En la Figura 3 se observa el patrón de RFLP que se obtuvo con la enzima BsmFI; se aprecia un sitio de restricción de aproximadamente 200 pb en 8 de los 19 aislamientos que se identificaron como C. neoformans var. grubii (serotipo A). El aislamiento que no presentó sitio de restricción (Figura 3) fue posteriormente identificado como C. gattii serotipo B, al presentar un corte con la enzima BshTI (dato no mostrado).
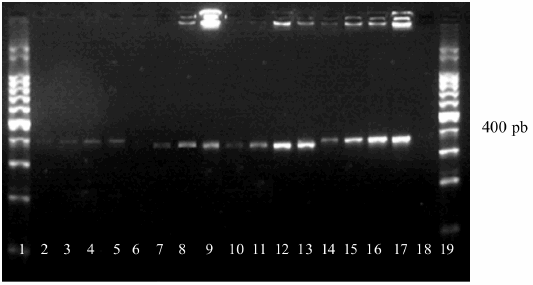
Figura 1. Productos de la amplificación del gen CAP59 en C. neoformans y C. gattii. Calles 2, 3, 4 y 5: C. neoformans var. grubii; calles 6, 7, 8 y 9: C. gattii serotipo B; calles 10, 11, 12 y 13: C. gattii serotipo C; calles 14, 15, 16 y 17: C. neoformans var. neoformans. En todos los casos se partió de suspensiones celulares con opacidad correspondiente al tubo 0,5; 1; 2 y 3 de la escala de Mc Farland, respectivamente. Calle 18: control negativo; calles 1 y 19: marcador de peso molecular de 100 pb.
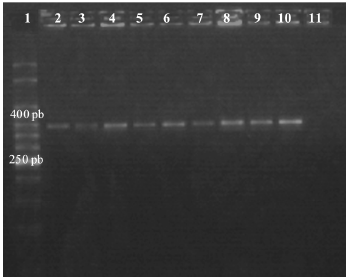
Figura 2. Productos de la amplificación del gen CAP59 en aislamientos de C. neoformans provenientes de pacientes con diagnóstico de criptococosis. La amplificación se realizó a partir de suspensiones celulares con opacidad igual al tubo 2 de la escala Mc Farland. Calle 1: marcador de peso molecular de 50 pb; calles 2-7: aislamientos de Cryptococcus obtenidos de líquido cefalorraquídeo; calles 8-10: aislamientos de Cryptococcus obtenidos de hemocultivos; calle 11: control negativo.
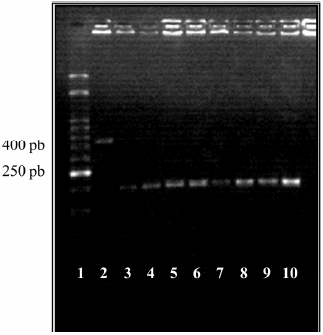
Figura 3. Amplificación del gen CAP59 a partir de suspensiones celulares con opacidad igual al tubo 2 de la escala Mc Farland de cultivos de Cryptococcus recuperados de pacientes, y posterior RFLP con la enzima BsmFI. Calle 1: marcador de peso molecular de 50 pb; calle 2: no existió para ese aislamiento, proveniente de líquido cefalorraquídeo (LCR), sitio de restricción de la enzima; calles 3-10: fragmentos de la restricción enzimática de 200 bp obtenidos a partir de aislamientos recuperados de hemocultivos y de LCR correspondientes al serotipo A de C. neoformans var. grubii.
La extracción de ADN es un paso esencial para el análisis molecular de especies fúngicas. Está ampliamente aceptado que la extracción de ADN puro y de elevado peso molecular a partir de la célula fúngica es dificultosa debido a la característica de su pared. En Cryptococcus, además de la pared celular, la presencia de la cápsula podría dificultar aún mucho más la extracción del material genético. Sin embargo, nosotros desarrollamos y estandarizamos este método sensible, rápido y simple para amplificar ADN a partir de C. neoformans y C. gattii, en donde las células de levaduras no requieren un precultivo en medio líquido, no es necesario usar solventes orgánicos potencialmente tóxicos (como el fenol, el alcohol isoamílico o el cloroformo) ni tampoco procedimientos de ruptura mecánica o enzimática de la pared (13), ya que el ADN se obtiene a partir de una suspensión de levaduras en el mismo proceso de amplificación de la PCR; en este caso, para el gen CAP59. Este método también nos permitió la amplificación de la región rADN a partir de una suspensión de la levadura de Cryptococcus con opacidad igual al tubo 0,5 de la escala de Mc Farland (resultados no mostrados). La omisión de los procedimientos de extracción y purificación del ADN disminuyen significativamente el tiempo necesario para hacer una identificación mediante PCR. Como limitación a esta metodología podemos mencionar la imposibilidad de disponer de stock de ADN para estudios posteriores.
La amplificación del gen CAP59 y el posterior análisis mediante la técnica de RFLP con diferentes enzimas de restricción permitió la determinación molecular de los serotipos de C. neoformans y C. gattii (3). Esto representa una alternativa de análisis molecular importante debido a que, si bien la serotipificación de los aislamientos de C. neoformans por métodos inmunológicos fue el método de diferenciación más difundido y aceptado por los laboratorios de microbiología, actualmente no se realiza, ya que depende de los reactivos comerciales (que no están fácilmente disponibles), de la presencia de la cápsula en la levadura y de la estabilidad del polisacárido capsular (2). Así, para la definición de los serotipos de C. neoformans y C. gattii, las técnicas moleculares mediante el análisis de ácidos nucleicos son más adecuadas, pues usan como blanco el ADN en lugar de un carácter expresado (producción de cápsula). En este contexto, los métodos que permitan una extracción sencilla, fácil y económica resultan de utilidad para posteriores estudios taxonómicos en laboratorios que tengan implementadas herramientas de biología molecular.
Agradecimientos: este trabajo fue realizado con el subsidio M - 426 de la Secretaría de Ciencia y Técnica de la Universidad de Buenos Aires.
1. Bennett JE, Kwon-Chung KJ, Howard D. Epidemiologic differences among serotypes of Cryptococcus neoformans. Am J Epidemiol 1984; 105: 582-6. [ Links ]
2. Cherniak R, Morris LC, Belay T, Spitzer ED, Casadevall A. Variation in the structure of glucuronoxylomannan in isolates from patients with recurrent cryptococcal meningitis. Infect Immun 1995; 63: 1899-905. [ Links ]
3. Enache-Angoulvant A, Chandenier J, Symoens F, Lacub P, Douchet C, Poirot J, et al. Molecular identification of Cryptococcus neoformans serotypes. J Clin Microbiol 2007; 45: 1261-5. [ Links ]
4. Franzot SP, Salkin IF, Casadevall A. Cryptococcus neoformans var. grubii: separate varietal status for Cryptococcus neoformans serotype A isolates. J Clin Microbiol 1999; 37: 838-40. [ Links ]
5. Iwen PC, Hinrichs SH, Rupp ME. Utilization of the internal transcribed spacer regions as molecular targets to detect and identify human fungal pathogens. Med Mycol 2002; 40: 87-109. [ Links ]
6. Kidd S, Hagen F, Tscharke R, Huynh M, Bartlett K, Fyfe M, et al. A rare genotype of Cryptococcus gattii caused the cryptococcosis outbreak on Vancouver Island (British Columbia, Canada). Proc Natl Acad Sci 2004; 101: 17258-63. [ Links ]
7. Kwon-Chung KJ. Cryptococcosis. En: Kwon-Chung KJ, Bennett JE, editors. Medical Mycology. Lea and Febiger, Philadelphia, PA. 1992, p. 397-446. [ Links ]
8. Kwon-Chung KJ, Varma A. Do major species concepts support one, two or more species within Cryptococcus neoformans? Fems Yeast Res 2006; 6: 574-87. [ Links ]
9. Latouche GN, Huynh M, Sorrell TC, Meyer W. PCR-restriction fragment lenght polymorphism analysis of the phospholipase B (PLB1) gene for subtyping of Cryptococcus neoformans isolates. Appl Environ Microbiol 2003; 69: 2080-6. [ Links ]
10. Mitchell TG, Perfect JR. Cryptococcosis in the era of AIDS- 100 years after the discovery of Cryptococcus neoformans. Clin Microbiol Rev 1995; 8: 515-48. [ Links ]
11. Mitchell DH, Sorrell TC, Allworth AM, Heath CH, McGregor AR, Papanaoum K, et al. Cryptococcal disease of the CNS in immunocompetent hosts: influence of cryptococcal variety on clinical manifestations and outcome. Clin Inf Dis 1995; 20: 611-6. [ Links ]
12. Okabayashi K, Hasegawa A. Watanabe T. Microreview: capsule-associated genes of Cryptococcus neoformans. Mycopathologia; 2007; 163: 1-8. [ Links ]
13. Scherer S, Stevens, DA. Application of DNA typing methods to epidemiology and taxonomy of Candida species. J Clin Microbiol 1987; 25: 675-9. [ Links ]
14. Sorrell TC. Cryptococcus neoformans variety gattii. Med Mycol 2001; 39: 155-68. [ Links ]
15. Viviani MA, Cogliati M, Esposto MC, Lemmer K, Tintelnot K, Colom Valiente MF, et al. Molecular analysis of 311 Cryptococcus neoformans isolates from 30 month ECMM survey of cryptococcosis in Europe. Fems Yeast Res 2006; 6: 614-9. [ Links ]
Recibido: 08/09/09
Aceptado: 12/01/10














