Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO  uBio
uBio
Compartilhar
Revista argentina de microbiología
versão impressa ISSN 0325-7541versão On-line ISSN 1851-7617
Rev. argent. microbiol. v.42 n.2 Ciudad Autónoma de Buenos Aires abr./jun. 2010
MICROBIOLOGÍA CLÍNICA Y ENFERMEDADES INFECCIOSAS
Optimización del procesamiento y la conservación de muestras clínicas destinadas al diagnóstico molecular de coqueluche
L. Pianciola1 *, M. Mazzeo1, D. Flores2, D. Hozbor2
1Laboratorio Central, Subsecretaría de Salud de Neuquén. Gregorio Martínez 65, Neuquén;
2Instituto de Biotecnología y Biología Molecular, Facultad de Ciencias Exactas, Universidad Nacional de La Plata. CCT La Plata CONICET. Calles 49 y 115 (1900) La Plata. Argentina.
*Correspondencia: luispianciola@yahoo.com.ar
RESUMEN
Coqueluche o pertussis es una infección respiratoria aguda, muy contagiosa y particularmente grave en menores de un año. En la enfermedad clásica, el diagnóstico clínico puede realizarse sin dificultad. En otros casos se requiere la confirmación por el laboratorio. Los métodos usados son el cultivo, la serología y la PCR. Para esta última, la muestra de elección es el aspirado nasofaríngeo y la metodología más sencilla de procesamiento utiliza proteinasa K. Aunque por lo general los resultados son satisfactorios, frecuentemente se presentan dificultades relacionadas con la naturaleza mucosa de los especímenes. Por otra parte, existen incertidumbres respecto de las condiciones óptimas de conservación de las muestras. En el presente estudio se evaluaron distintas técnicas de procesamiento y diversas condiciones de conservación de los materiales. Los resultados obtenidos permitieron seleccionar un método que optimiza el procesamiento de estas muestras, con un desempeño comparable al de los métodos comerciales y costos muy inferiores. Asimismo, las experiencias diseñadas para evaluar la conservación de las muestras nos permitieron elaborar una serie de recomendaciones, que pueden resultar muy valiosas cuando se debe guiar la derivación desde los lugares donde se atienden los pacientes hasta los centros donde se realizan las determinaciones basadas en metodologías moleculares.
Palabras clave: Coqueluche; Extracción de ADN; PCR
ABSTRACT
Optimization of processing and storage of clinical samples to be used for the molecular diagnosis of pertussis. Pertussis or whooping cough is an acute, highly contagious respiratory infection, which is particularly severe in infants under one year old. In classic disease, clinical diagnosis may present no difficulties. In other cases, it requires laboratory confirmation. Generally used methods are: culture, serology and PCR. For the latter, the sample of choice is a nasopharyngeal aspirate, and the simplest method for processing these samples uses proteinase K. Although results are generally satisfactory, difficulties often arise regarding the mucosal nature of the specimens. Moreover, uncertainties exist regarding the optimal conditions for sample storage. This study evaluated various technologies for processing and storing samples. Results enabled us to select a method for optimizing sample processing, with performance comparable to commercial methods and far lower costs. The experiments designed to assess the conservation of samples enabled us to obtain valuable information to guide the referral of samples from patient care centres to laboratories where such samples are processed by molecular methods.
Key words: Pertussis; DNA extraction; PCR
Coqueluche o pertussis es el nombre que recibe una infección respiratoria aguda, muy contagiosa y particularmente grave en menores de un año, que luego de más de 60 años de vacunación aún produce en el mundo 30 millones de casos por año, con más de 300 000 muertes (3, 12). Bordetella pertussis es el agente etiológico principal de la patología. Bordetella parapertussis también causa la enfermedad, aunque con menor frecuencia y, generalmente, produce cuadros más leves (1). La enfermedad continúa siendo un problema para la salud pública, a pesar de décadas de coberturas vacunales adecuadas (8). En los últimos 20 años, junto con un aumento del número de casos, se produjo un corrimiento de éstos hacia el grupo etario de los adolescentes y los adultos, quienes son considerados el reservorio actual de la enfermedad (11).
En la enfermedad clásica, el diagnóstico clínico puede realizarse sin dificultad frente a la presencia de tos paroxística seguida de vómitos, estridor inspiratorio, marcada linfocitosis y ausencia de fiebre. En cambio, en aquellos casos en los que la clínica dista de la presentación clásica, como ocurre en pacientes adultos, en lactantes o en aquellos individuos con historia vacunal previa, se requiere de confirmación mediante técnicas de laboratorio (14). El análisis en el laboratorio también es requerido para el diagnóstico etiológico diferencial entre B. pertussis y B. parapertussis (1) y para identificar otros agentes infecciosos que pueden causar síntomas similares (15). La técnica microbiológica que implica el aislamiento del agente causal es una metodología altamente específica y se considera el "estándar de oro"; sin embargo, su sensibilidad es baja. La serología es más sensible, pero el formato que se emplea en la actualidad en nuestro país requiere de dos muestras de suero, una de las cuales corresponde a la fase convaleciente de la enfermedad, lo que hace que esta metodología redunde en un diagnóstico tardío y, por consiguiente, de escasa utilidad clínica (10).
En relación con los métodos moleculares, se recomienda la PCR en tiempo real, ya que es más sensible y específica. Ésta presenta el inconveniente de su mayor costo respecto del formato convencional de PCR, que es el usado en nuestro medio. Si se opta por esta última metodología, es importante guiarse por el algoritmo que exige confirmación de los resultados positivos mediante ensayos independientes. El uso de las técnicas de amplificación, en cualquiera de los formatos antes descritos, ha permitido disminuir los tiempos de obtención de resultados de días a horas y ha incrementado la sensibilidad del método considerado el estándar de oro (cultivo) en casi un 40%, con una especificidad estimada del 97 al 98% respecto del cultivo. Estas ventajas han motivado su uso masivo.
Tanto para las metodologías moleculares como para el diagnóstico microbiológico, la muestra de elección es el aspirado nasofaríngeo y, como una alternativa, especialmente en adolescentes y adultos, el hisopado nasofaríngeo. El procedimiento más sencillo propuesto para la extracción de ADN a partir de estas muestras es su tratamiento con proteinasa K (7). Si bien los resultados obtenidos con esta metodología son satisfactorios, a menudo se presentan dificultades relacionadas con la naturaleza mucosa de las muestras. Esto trae aparejado la necesidad de realizar repeticiones del análisis (que en nuestra experiencia suele comprender el 30% del total de muestras recibidas), para poder interpretar adecuadamente el resultado. Estos inconvenientes deslucen las ventajas de la metodología, además de aumentar los costos.
El objetivo de este trabajo fue optimizar el procesamiento de las muestras destinadas al diagnóstico molecular de coqueluche y evaluar las condiciones óptimas de conservación y derivación de estos materiales, aspectos que son fundamentales en una región como la nuestra, en la que las distancias a los centros de complejidad, donde son procesadas, son grandes.
Para la primera etapa del trabajo, que tuvo por finalidad lograr una mejor calidad de la muestra utilizada para el diagnóstico molecular de coqueluche, se seleccionaron de la bibliografía y analizaron distintos métodos de preparación de muestras para especímenes respiratorios (Tabla 1). Para la segunda parte se determinaron las temperaturas más adecuadas para conservar las muestras y el período máximo en el que no se observa deterioro del material. También se determinó si el procesamiento de las muestras antes de su derivación permitiría prolongar el período máximo de conservación.
Tabla 1. Descripción de los métodos de procesamiento de muestras nasofaríngeas destinadas al diagnóstico molecular de coqueluche
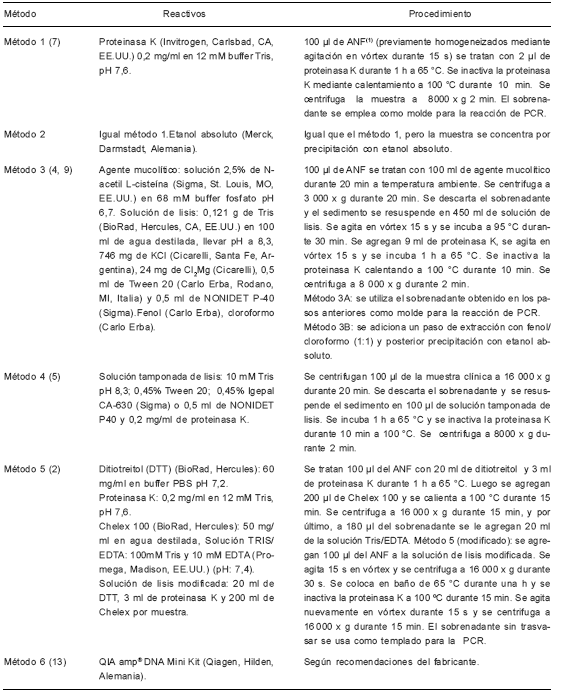
(1) ANF: aspirado nasofaríngeo.
En relación con el primer objetivo, se analizaron comparativamente las metodologías de obtención de ADN descritas en la Tabla 1 y algunas de sus variantes. Como material de partida se empleó un pool de 10 muestras de aspirado nasofaríngeo con resultado negativo por la metodología convencional y que habían sido conservadas en congelador a - 80 °C hasta su utilización. Dicho pool fue contaminado con una suspensión de B. pertussis cepa Tohama (CIP 8132), cedida por el Laboratorio Nacional de Referencia. La suspensión de B. pertussis se preparó a partir del desarrollo bacteriano obtenido en placas de agar Bordet Gengou (Difco, Sparks, MD, EE. UU.) suplementado con sangre de carnero al 15%, luego de 72 horas de incubación a 37 °C y de un repique posterior de 24 h en el mismo medio y condiciones de incubación.
La suspensión bacteriana se preparó de forma de obtener una concentración final de B. pertussis de 103 UFC/ml en el pool de aspirados nasofaríngeos. Sobre las muestras así preparadas se aplicaron las distintas metodologías para la extracción del ADN (Tabla 1). El ADN molde obtenido en cada caso fue utilizado en la reacción de PCR convencional, que emplea los cebadores que hibridan con la secuencia promotora de la toxina pertussis y que genera como producto de la reacción una secuencia de 191 pb (8). Como control positivo se usó una suspensión de B. pertussis cepa Tohama calentada 10 min en baño a 100 °C y centrifugada a 8 000 x g durante 5 minutos. Para evaluar los distintos métodos, se analizaron y compararon los límites de detección de cada uno de ellos. Como puede observarse en la Figura 1, los métodos 1, 2 y 4, que no utilizan agentes mucolíticos, no permitieron una buena resolución de bandas. Recién al diluir las muestras 1:10, fue posible vislumbrar la banda esperada. También puede observarse que el método 2 resulta menos sensible que el método 1. Los métodos 3A (calles 5 y 6), 3B (calles 7 y 8) y 5 (calles 12 y 13) arrojaron un resultado similar al método 6 (comercial), utilizado como método de referencia (calles 14 y 15).
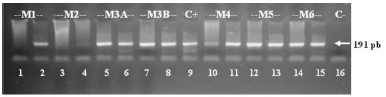
Fig. 1. Electroforesis en gel de agarosa 2% (p/v) de los productos de PCR obtenidos a partir de una muestra clínica de naturaleza mucosa contaminada con una suspensión bacteriana de B. pertussis (B. pertussis Tohama 105 UFC/ml).
Calle 1: método 1 sin diluir la muestra, calle 2: método 1 con muestra diluida 1/10, calle 3: método 2 sin diluir la muestra, calle 4: método 2 con muestra diluida 1/10, calle 5: método 3A con muestra sin diluir, calle 6: método 3A con muestra diluida 1/10, calle 7: método 3B con muestra sin diluir, calle 8: método 3B con muestra diluida 1/10, calle 10: método 4 con muestra sin diluir, calle 11: método 4 con muestra diluida 1/10, calle 12: método 5 con muestra sin diluir, calle 13: método 5 con muestra diluida 1/10, calle 14: método 6 con muestra sin diluir, calle 15: método 6 con muestra diluida 1/10. Como control de la reacción se empleó el material obtenido de la lisis de una suspensión bacteriana de B. pertussis (calle 9) y como control negativo agua (calle 16). Para la visualización se empleó bromuro de etidio a 0,5 mg/ml.
Para investigar si la extracción con fenol/cloroformo y posterior precipitación con alcohol incrementaba la sensibilidad, se compararon los métodos 3A y 3B entre sí. Los dos métodos ensayados brindaron resultados similares, ya que dieron un resultado positivo hasta una concentración de bacterias en la muestra de 1,6 x 102 UFC/ml. Dado que el método 3B es mucho más laborioso, más caro y presenta mayores posibilidades de introducir contaminaciones por incluir más pasos, se descartó su utilización.
Al comparar los métodos 3 y 5 se observó que el método 5, que introduce la utilización de CHELEX®, permitió detectar hasta una dilución más de la muestra (1,6 x 101 UFC/ml), lo que indica mayor sensibilidad en la determinación. El método 5 finalmente fue comparado con el empleo de un equipo comercial de extracción y purificación de ADN (método 6), considerado un método de alto rendimiento. Ambos métodos mostraron un comportamiento similar, ya que permitieron detectar señal hasta una dilución 10-5 de la muestra (0,42 UFC/reacción). Este dato es de gran importancia, ya que el método propuesto representa un costo inferior (un quinto del valor del método comercial), lo que lo hace muy aplicable al diagnóstico de rutina, especialmente cuando se procesa un número considerable de muestras. Esta metodología (método 5) plantea, sin embargo, la necesidad de realizar varias etapas, lo que potencialmente podría introducir contaminaciones en el proceso. Por esa razón, se adecuó el método para realizarlo en un solo paso (método 5 modificado). Los resultados alcanzados fueron similares a los obtenidos originalmente, por lo que se recomienda realizar la metodología en un solo paso. Este método de procesamiento está siendo validado en nuestro laboratorio frente a muestras clínicas. Durante el año 2009 hemos procesado simultáneamente por el método convencional y por este método un total de 425 muestras. Los resultados preliminares muestran un desempeño muy satisfactorio, que disminuye la necesidad de repeticiones, desde un promedio de 30% de las muestras, a menos de 5%.
Para evaluar las condiciones de conservación de las muestras se ensayaron las distintas posibilidades al alcance de los laboratorios clínicos: temperatura ambiente, heladera o congelador. Se trabajó con un pool de 10 muestras clínicas adicionado con una suspensión de B. pertussis cepa Tohama (CIP 8132) con un título de 1,6 x 105 UFC/ml, determinado por recuento en placa (por triplicado). Una parte de ese pool fue procesada empleando el método 5 modificado ya descrito, y el material resultante fue dividido en 15 alícuotas de 10 ml cada una, las que fueron rotuladas con la letra "P" (muestras procesadas). Otra porción de la muestra original sin procesar fue dividida en 15 alícuotas de 100 ml cada una y fueron rotuladas "NP" (muestras no procesadas). Cinco tubos rotulados como P y 5 como NP fueron conservados a temperatura ambiente, otros 5 P y 5 NP se colocaron en heladera, y finalmente, un número igual de tubos de P y NP fueron colocados en congelador. Las temperaturas oscilaron durante los 30 días de la experiencia entre los 18 y los 24 °C a temperatura ambiente, entre los 4 y los 6 °C en la heladera, y entre -18,5 y -21,5 °C en el congelador. De cada una de las condiciones analizadas se tomaron muestras a las 24 y 72 horas, y a los 7, 14 y 30 días. Las muestras P fueron utilizadas directamente en las PCR y las NP fueron procesadas por el método 5 modificado, antes de incluirlas en los ensayos moleculares. El producto final del procesamiento de los tubos NP y los tubos P fue diluido en forma seriada abarcando concentraciones entre 1,6 x 104 y 1,6 x 100 UFC/ml. Se emplearon alícuotas de cada una de las diluciones como templados en el procedimiento de PCR estándar de nuestro laboratorio. La experiencia completa se realizó por triplicado en tres momentos sucesivos, no superpuestos. Los resultados obtenidos se muestran en la Tabla 2. Luego de 24 h de conservación, en todas las muestras se observó el mismo resultado, es decir, presencia de amplicón hasta la dilución 10-2 (equivalente a 0,5 UFC/tubo de reacción). La calidad de las muestras no se vio alterada por haber sido conservadas 24 h bajo distintas condiciones. Tampoco parece haber influido en el resultado la preparación previa del material. A las 72 h, en todas las condiciones ensayadas se observó el mismo resultado: presencia de amplicón hasta la dilución de 0,5 UFC/tubo de reacción. Sin embargo, es posible advertir un leve deterioro en las muestras que fueron conservadas sin procesamiento previo. Ya a los 7 días se hace evidente el deterioro de las muestras conservadas sin preparación previa, especialmente las conservadas a temperatura ambiente y en congelador. En estas condiciones de conservación, el límite de detección disminuyó en relación con las otras condiciones, puesto que se observó amplicón sólo hasta diluciones 10-1 de las muestras (5 UFC/tubo de reacción). Es de destacar los resultados obtenidos a partir de las muestras procesadas: si las muestras se conservan procesadas por el método 5 modificado, los resultados que se obtienen son similares a los de las 24 h, con un límite de detección de 0,5 UFC/tubo de reacción. Asimismo, las muestras no procesadas parecen no continuar deteriorándose más allá de los 7 días, ya que a los 14 días se observan los mismos resultados que los alcanzados luego de 7 días de conservación. Respecto de las muestras procesadas, puede observarse un deterioro en las muestras conservadas a temperatura ambiente, en las cuales la positividad desciende a diluciones de 10-1 (5 UFC/tubo de reacción). Las otras condiciones de conservación siguen rindiendo un límite de detección de 0,5 UFC/tubo de reacción. Finalmente, a los 30 días de conservación, el deterioro fue evidente en las muestras conservadas a temperatura ambiente, tanto en aquellas no procesadas como en las que habían sido procesadas con anterioridad. Las muestras conservadas en heladera se mantuvieron adecuadamente, tanto las que fueron procesadas como las que no lo fueron. Las muestras conservadas en congelador durante 30 días no mostraron variación respecto de lo observado a los 14 días, aunque fue evidente que las muestras se conservaron mejor en heladera, tanto cuando fueron procesadas como cuando no lo fueron.
Tabla 2. Evaluación de la estabilidad de las muestras clínicas empleadas para PCR frente a distintas condiciones ambientales de conservación
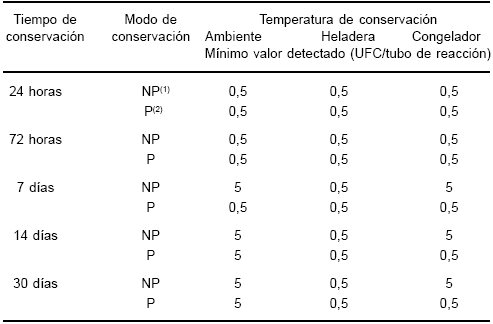
(1)NP: muestras clínicas conservadas sin ningún tratamiento previo; (2)P: muestras clínicas conservadas luego de ser procesadas por el método 5 modificado.
En resumen, las muestras no procesadas se deterioran en mayor medida que las procesadas cuando la conservación se hace a temperatura ambiente o en congelador. La conservación en heladera se muestra como la condición más adecuada, tanto para las muestras procesadas como para las no procesadas, aun durante períodos prolongados de conservación.
Teniendo en cuenta estos resultados, y que el procesamiento previo de las muestras en los laboratorios de baja complejidad puede significar una sobrecarga de difícil manejo, recomendamos que las muestras clínicas para el diagnóstico molecular de coqueluche sean refrigeradas en heladera desde el momento de su obtención, y que se deriven refrigeradas. No es necesario ningún procesamiento previo a la derivación. Si por cualquier razón de fuerza mayor fuera necesaria una conservación prolongada de la muestra, ésta deberá realizarse en heladera. Sin embargo, para el diagnóstico oportuno, se recomienda no sobrepasar las 72 horas desde la obtención de la muestra hasta el arribo de ésta al laboratorio de referencia.
1. Bergfors E, Trollfors B, Taranger J, Lagergärd T, Sundh B, Zackrisson G. Parapertussis and pertussis: differences and similarities in incidence, clinical course, and antibody responses. Int J Infect Dis 1999; 3: 140-6. [ Links ]
2. Birkebaek NH, Kristiansen M, Seefeldt T, Degn J, Moller A, Heron I, et al. Bordetella pertussis and chronic cough in adults. Clin Infect Dis 1999; 29: 1239-42. [ Links ]
3. Bonacorsi S, Farnoux C, Bidet P, Caro V, Aizenfisz S, Benhayoun M, et al. Treatment failure of nosocomial pertussis infection in a very-low-birth-weight neonate. J Clin Microbiol 2006; 44: 3830-2. [ Links ]
4. Broccolo F, Scarpellini P, Locatelli G, Zingale A, Brambilla AM, Cichero P, et al. Rapid diagnosis of mycobacterial infections and quantitation of Mycobacterium tuberculosis load by two real-time calibrated PCR assays. J Clin Microbiol 2003; 41: 4565-72. [ Links ]
5. Cadario ME. Comunicación personal. [ Links ]
6. Crowcroft NS, Pebody RG. Recent developments in pertussis. Lancet 2006; 367: 1926-36. [ Links ]
7. Glare EM, Paton JC, Premier RR, Lawrence AJ, Nisbet LT. Analysis of a repetitive DNA sequence from Bordetella pertussis and its application to the diagnosis of pertussis using the polymerase chain reaction. J Clin Microbiol 1990; 28: 1982-7. [ Links ]
8. Grimprel E, Bégué P, Anjak L, Betsou F, Guiso N. Comparison of polymerase chain reaction, culture, and Western Immunoblot serology for diagnosis of Bordetella pertussis infection. J Clin Microbiol 1993; 31: 2745-50. [ Links ]
9. Jensen JS, Borre MB, Dohn B. Detection of Mycoplasma genitalium by PCR amplification of the 16 rRNA gen. J Clin Microbiol 2003; 41: 261-6. [ Links ]
10. Lingappa JR, Lawrence W, West-Keefe S, Gautom R, Cookson BT. Diagnosis of community acquired pertussis infection. Comparison of both culture and fluorescent-antibody assays with PCR detection using electrophoresis or dot blot hybridization. J Clin Microbiol 2002; 40: 2908-12. [ Links ]
11. Muñoz FM. Pertussis in infant, children, and adolescents: diagnosis, treatment, and prevention. Semin Pediatr Infect Dis 2006; 17: 14-9. [ Links ]
12. Plotkin S. Aims, scope and findings of the global pertussis initiative. Pediatr Infect Dis J 2005; 24 Suppl 5: S5-6. [ Links ]
13. Riffelmann M, Wirsing von König CH, Caro V, Guiso N for the Pertussis PCR Consensus Group. Nucleic acid amplification test for diagnosis of Bordetella infections. J Clin Microbiol 2005; 43: 4925-9. [ Links ]
14. Walsh PF, Kimmel L, Feola M, Tran T, Lim C, De Salvia L, et al. Prevalence of Bordetella pertussis and Bordetella parapertussis in infants presenting to the emergency department with bronchiolitis. J Emerg Med 2008; doi: 10.1016/j.emermed.2008.04.048. [ Links ]
15. van Kruijssen AM, Templeton KE, van der Plas RN, van Doorn HR, Claas ECJ, Sukhai RN, et al. Detection of respiratory pathogens by real-time PCR in children with clinical suspicion of pertussis. Eur J Pediatr 2007; 166: 1189-91. [ Links ]
Recibido: 6/10/09
Aceptado: 3/3/10














