Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO  uBio
uBio
Share
Revista argentina de microbiología
Print version ISSN 0325-7541
Rev. argent. microbiol. vol.43 no.1 Ciudad Autónoma de Buenos Aires Jan./Mar. 2011
MICROBIOLOGÍA CLÍNICA Y DE ALIMENTOS
ß-lactamasas AmpC plasmídicas tipo CMY-2 emergentes en Tucumán, Argentina
María A. Jure1 *, Constanza Presti1, Norma M. Cudmani2, Lidia M. Grellet3, Carolina López4, Eduardo H. Musa4, Olga C. Aulet1, Carlos Nieto5, Lucila Saavedra6, Marta Cecilia de Castillo1
1Instituto de Microbiología "Dr. Luis C. Verna", Cátedra de Bacteriología, Facultad de Bioquímica, Química, Farmacia y Biotecnología, Universidad Nacional de Tucumán. Ayacucho 491 2° piso, San Miguel de Tucumán;
2Laboratorio de Bacteriología, Hospital Avellaneda. San Miguel de Tucumán;
3Laboratorio de Bacteriología, Hospital de Concepción. Concepción, Pcia. de Tucumán;
4Laboratorio de Bacteriología, Centro de Microbiología Médica. San Miguel de Tucumán;
5Instituto de Microbiología "Dr. Luis C. Verna", Cátedra de Microbiologia General, Facultad de Bioquímica, Química, Farmacia y Biotecnología, Universidad Nacional de Tucumán. San Miguel de Tucumán;
6Laboratorio de Biología Molecular, Centro de Referencia para Lactobacilos (CERELA). San Miguel de Tucumán, Argentina
*Correspondencia. E-mail: majure@fbqf.unt.edu.ar, magejure@gmail.com
RESUMEN
En los últimos años, algunas enterobacterias como Klebsiella pneumoniae, Proteus mirabilis y Escherichia coli adquirieron resistencia a las cefalosporinas de tercera generación (C3G) por la adquisición de ß-lactamasas de tipo AmpC plasmídicas, mecanismo que se está comunicando cada vez con mayor frecuencia en el mundo. El objetivo de esta investigación fue detectar la emergencia de ß-lactamasas AmpC plasmídicas en Tucumán e identificar sus tipos predominantes. Se estudió la sensibilidad a diferentes antimicrobianos de 733 aislamientos clínicos consecutivos de enterobacterias obtenidos en el período marzo-julio de 2009 en tres centros asistenciales de nuestra provincia. Se realizaron pruebas de sensibilidad por difusión y se seleccionaron tres aislamientos de E. coli y uno de P. mirabilis resistentes a C3G y a cefoxitina. En estos aislamientos se determinó la CIM por E-test y se evaluó el mecanismo enzimático de resistencia mediante sinergia con discos de ácido aminofenilborónico y ensayo microbiológico de Masuda. Se realizó PCR usando cebadores dirigidos al grupo CIT de las AmpC plasmídicas. Se obtuvo un amplicón de 462 pb que fue secuenciado; presentó un 100% de identidad con blaCMY-2 en los cuatro aislamientos. En conclusión, las ß-lactamasas AmpC tipo CMY-2 emergieron en nuestro medio, de modo que es importante implementar una vigilancia sistemática de estas resistencias para evitar potenciales consecuencias clínicas y epidemiológicas.
Palabras clave: Enterobacteriaceae; ß-lactamasa AmpC plasmídica; Tipo CMY-2
ABSTRACT
CMY-2-type plasmid-mediated AmpC ß-Lactamases emerging in Tucumán, Argentina. In the last years, Enterobacteriaceae such as Klebsiella pneumoniae, Proteus mirabilis and Escherichia coli, have acquired resistance to third-generation cephalosporins (C3G) because of the presence of plasmid-mediated AmpC ß-lactamases. The aim of this work was to detect plasmid AmpC enzymes and to investigate the predominant types in our region. Between March and July 2009, 733 consecutive isolates of Enterobacteriaceae derived from hospitals and outpatient centers were studied. Susceptibility testing was performed by disk diffusion; one P. mirabilis and three E. coli strains showed resistance to cephamycins (cefoxitin) and C3G. An E-test to determine MIC and a synergy test by aminophenylboronic disks were performed. Enzymatic activity against cefoxitin was confirmed by a microbiological assay. A polymerase chain reaction (PCR) for the detection of plasmid-mediated ampC genes of different groups was performed and a 462-bp amplicon was obtained when using primers directed against the CIT group; the obtained sequences were compared to blaCMY-2 sequences, showing 100% identity. The emergence of CMY-2-type plasmid-mediated AmpC ß-lactamases indicated the importance of implementing systematic monitoring of these resistances to avoid potential clinical and epidemiological consequences.
Key words: Enterobacteriaceae; Plasmid-mediated AmpC ß-lactamases; CMY-2 type
En nuestro medio, la presencia de enterobacterias resistentes a las cefalosporinas de tercera generación (C3G) es una realidad; detectar precozmente esta resistencia en el laboratorio conduce a tratamientos exitosos.
Géneros como Enterobacter spp., Citrobacter spp. y Serratia spp. pueden hiperproducir ß-lactamasa de tipo AmpC en forma cromosómica constitutiva, lo que les confiere resistencia a las C3G. En los últimos años, otras enterobacterias como Klebsiella pneumoniae y Proteus mirabilis, que naturalmente carecen de estas enzimas, o Escherichia coli que la produce en niveles basales, adquirieron también resistencia asociada a la producción de AmpC codificada en plásmidos (en adelante, AmpC plasmídicas).
En Argentina se documentaron dos tipos de AmpC plasmídicas: tipo FOX en K. pneumoniae (1994) y tipo CMY-2 en Citrobacter koseri, en K. pneumoniae (2007) y en Shigella flexneri (2008) (6, 10, 11).
El objetivo de este trabajo fue detectar la emergencia de AmpC plasmídicas y caracterizar sus tipos predominantes en nuestra región.
Se estudió el comportamiento antimicrobiano de 733 aislamientos clínicos de E. coli y otras enterobacterias que naturalmente no poseen el gen cromosomal ampC. Los aislamientos correspondieron a E. coli (66%), Klebsiella spp. (21%) y Proteus spp. (13%), y se obtuvieron en tres centros asistenciales de Tucumán (Hospital Nicolás Avellaneda, Hospital Regional de Concepción y Centro de Microbiología Médica de Tucumán) en el período marzo-julio de 2009.
La prueba de sensibilidad se realizó por difusión con discos siguiendo las normativas del Clinical and Laboratory Standards Institute (CLSI) (3). Los antibióticos ensayados fueron ampicilina, amoxicilina/clavulánico, cefalotina, cefoxitina, cefotaxima, ceftacidima, imipenem, cefotaxima/clavulánico, ceftacidima/clavulánico, piperacilina/tazobactama, trimetoprima/sulfametoxazol, ciprofloxacina, gentamicina y amicacina.
Los criterios para seleccionar cepas probablemente productoras de AmpC plasmídica fueron sensibilidad disminuida o resistencia a C3G y resistencia a cefoxitina. En los aislamientos seleccionados se determinó la concentración inhibitoria mínima (CIM) por E-test de los siguientes antibióticos: cefoxitina, ceftacidima, cefotaxima, ceftriaxona y cefepima. No se incluyó en este estudio ningún aislamiento productor de ß-lactamasa de espectro extendido (BLEE ), por su sensibilidad a cefoxitina.
Las enzimas AmpC plasmídicas son inhibidas por el ácido 3-aminofenilborónico (AFB). Para investigar su presencia se llevó a cabo una prueba de sinergia colocando discos con AFB entre los de cefoxitina y/o ceftacidima/cefotaxima (13), y luego se practicó el ensayo microbiológico de Masuda, que consistió en evaluar diferentes concentraciones del extracto enzimático obtenido a partir de cada aislamiento frente a la cefoxitina (8).
Una vez detectada la presencia de la enzima en términos fenotípicos, se realizó la caracterización genética por PCR. Teniendo en cuenta la prevalencia de enzimas AmpC plasmídicas en nuestro país, se escogieron los cebadores descritos por Pérez-Pérez y Hanson (9), que amplifican genes codificadores de enzimas del grupo CIT (LAT-1 a LAT-4, CMY-2 a CMY-7 y BIL 1). Se utilizó como templado el ADN total; como control positivo se empleó un aislamiento de S. flexneri productor de AmpC tipo CMY-2 cedido por el Servicio de Antimicrobianos del Instituto Malbrán.
El producto de PCR fue enviado a secuenciar con un analizador ABI3130XL (Applied Biosystems, EE .UU .) al Instituto de Biotecnología, Unidad de Genómica, Centro de Investigaciones en Ciencias Veterinarias y Agronómicas (INTA Castelar).
Con el objeto de evaluar si el fragmento obtenido se localizaba en un plásmido, se procedió a la extracción del ADN plasmídico de los aislamientos clínicos seleccionados como probables productores de AmpC plasmídica. Esto se realizó por dos metodologías: mediante la técnica de Birnboim y Dolly (2) y con un equipo comercial (Axy Prep Plasmid Miniprep Kit, Axygen Biosciences, Basilea, Suiza), luego se transformaron células competentes de E. coli DH5α. Los transformantes se seleccionaron en placas que contenían agar Luria-Bertani adicionado con 100 μg/ml de cefoxitina.
De los 733 aislamientos clínicos de enterobacterias estudiados, se seleccionaron cuatro aislamientos provenientes de orina: tres de ellos correspondieron a E. coli (aislamientos C20 y A964, de pacientes ambulatorios, y aislamiento C44, de un paciente hospitalizado) y uno correspondió a P. mirabilis (aislamiento A2, de un paciente ambulatorio). Todos ellos fueron resistentes a las aminopenicilinas, las cefalosporinas de espectro restringido y la cefoxitina, y presentaron sensibilidad disminuida o resistencia a las cefalosporinas de espectro extendido, mientras que fueron sensibles a cefepima, piperacilina/tazobactama, meropenem e imipenem.
La prueba de sinergia con AFB y el ensayo de Masuda confirmaron el mecanismo enzimático en los 4 aislamientos (Figuras 1 y 2).

Figura 1. Prueba de sinergia con discos de ácido 3-aminofenil borónico (AFB), ceftacidima (CAZ) y cefotaxima (CTX).

Figura 2. Ensayo microbiológico de Masuda. Placa de agar Mueller Hinton hisopada con E. coli ATCC 25923; ee: discos de papel de filtro impregnados con 5 μl (ee1), 10 μl (ee2) o 15 μl (ee3) de extracto enzimático; bf: discos de papel de filtro impregnados con buffer PBS (15 μl); FOX: cefoxitina (30 μg).
Por PCR se obtuvo un amplicón de 462 pb. Las secuencias de los productos de PCR de los aislamientos seleccionados presentaron un 100% de identidad con K. pneumoniae blaCMY-2 (n° de acceso a GenBank; X 91840).
El ADN plasmídico obtenido a partir de los aislamientos clínicos de E. coli fue transferido por transformación a cepas receptoras de E. coli DH5α.
Los transformantes presentaron valores de CIM de cefoxitina > a 256 μg/ml (Tabla 1), sinergia con los discos de AFB y el ensayo microbiológico de Masuda positivos, con lo cual se confirmó en términos fenotípicos que se trataba de una resistencia enzimática. Además, se confirmó la presencia del gen blaCMY-2 en las cepas transformantes mediante PCR de colonia con los cebadores específicos antes descritos, con los que se obtuvo un fragmento del tamaño esperado (462 pb).
Tabla 1. Perfiles de sensibilidad a los antimicrobianos en los aislamientos clínicos de E. coli y P. mirabilis seleccionados y en sus respectivos transformantes
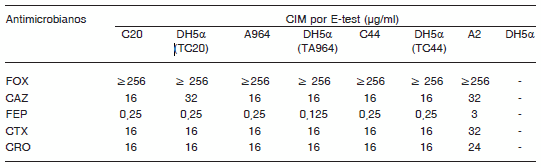
FOX: cefoxitina; CAZ: ceftacidima; FEP: cefepima; CTX: cefotaxima; CRO: ceftriaxona. C20: E. coli, Hospital de Concepción; A964: E. coli, Hospital Avellaneda; C44: E. coli, Hospital de Concepción; A2: P. mirabilis, Hospital Avellaneda; DH5α (TC20): E. coli transformante C20; DH5α (TA964): E. coli transformante A964; DH5α (TC44): E. coli transformante C44.
En P. mirabilis no se pudieron obtener plásmidos por los métodos empleados, por lo tanto no se confirmó su localización plasmídica.
La búsqueda y detección de las enzimas AmpC en los laboratorios se ve limitada debido a que no existen métodos fenotípicos estandarizados; por otra parte, la resistencia a cefoxitina como una herramienta de detección de AmpC plasmídica es una metodología muy inespecífica, ya que la reducción en la permeabilidad de la membrana externa puede originar un fenotipo de resistencia similar (4, 12).
En este trabajo, de los 733 aislamientos clínicos de enterobacterias obtenidos en un período de 5 meses, sólo 4 presentaron el perfil de resistencia enunciado en el criterio de selección y fueron positivos en la prueba de sinergia con los discos con AFB.
En un período de 6 meses y sobre un total de 25 000 aislamientos clínicos de enterobacterias, Rapoport et al. (11) seleccionaron 15 con un perfil de resistencia inusual a ß-lactámicos y que resultaron positivos en la prueba de sinergia con discos con AFB. La presencia de AmpC plasmídica perteneciente al grupo CIT se confirmó por PCR en la totalidad de esos casos. Doy et al. y Rapoport et al. sugirieron implementar el uso de discos de AFB para detectar esta enzima (5, 11); en el presente estudio, dicha metodología nos permitió detectar este mecanismo inusual en cepas resistentes a cefoxitina.
Se han documentado valores variables de incidencia de AmpC plasmídica en Enterobacteriaceae en distintas áreas geográficas. Desde el primer informe en Corea del Sur en 1989 (1), se ha detectado AmpC plasmídica en China y en EE .UU ., con incidencias de 2,8% y 1,2%, respectivamente (7, 4).En Argentina, Rapoport et al. informaron en 2008 un porcentaje menor de 0,1% de enterobacterias productoras de enzimas del grupo CIT (11); en nuestro trabajo este porcentaje fue del 0,55%.
La ß-lactamasa CMY-2 fue descrita por primera vez en 1990 y se la ha detectado en E. coli, Klebsiella spp., Proteus spp., Salmonella spp. y Shigella spp. Es la enzima de mayor prevalencia y la más ampliamente distribuida en el mundo (1).
En Argentina, la ß-lactamasa CMY-2 fue detectada en varias especies bacterianas como E. coli, P. mirabilis, C. koseri, K. pneumoniae y S. flexneri (10, 11, 14, 15). En Tucumán se detectó por primera vez en E. coli y P. mirabilis.
En conclusión, hemos demostrado que las ß-lactamasas AmpC plasmídicas tipo CMY-2 emergieron en nuestro medio. Consideramos que es muy importante implementar su búsqueda rutinaria en todos los centros asistenciales, para evitar su propagación a otros microorganismos.
Agradecimientos: los autores agradecen a las Dras. Clara Ruiz y Cristina Allori y a las Bioqcas. Rosa Cangemi y Norma Porcel por su colaboración en la recepción y conservación de las cepas estudiadas, y al Dr. Fernando Pasterán por proveer la cepa de S. flexneri utilizada como control positivo. Este trabajo fue financiado por la Secretaría de Ciencia y Técnica de la Universidad Nacional de Tucumán. Programa CIUNT 26/D417.
1. Bauernfeind A, Stemplinger I, Junwirth R, Giamarellou H. Characterization of the plasmidic ß-lactamase CMY-2, wich is responsible for cephamycin resistance. Antimicrob Agents Chemother 1996; 40: 221-4. [ Links ]
2. Birnboim H, Dolly J. A rapid alkaline extraction procedure for screening recombinant plasmid DNA. Nucleic Acids Res 1979; 7: 1513-23. [ Links ]
3. Clinical and Laboratory Standards Institute. Performance Standards for Antimicrobial Susceptibility Testing; 17th Informational Supplement, 2007; M100-S17. Wayne, PA, EE .UU . [ Links ]
4. Coudron P, Moland E, Thomson K. Occurrence and detection of AmpC ß-lactamases among Escherichia coli, Klebsiella pneumoniae, and Proteus mirabilis isolates at a Veterans Medical Center. J Clin Microbiol 2000; 38: 1791-6. [ Links ]
5. Doi Y, Paterson DL. Detection of plasmid-mediated class C ß-lactamases. Int J Infect Dis 2007;11: 191-7. [ Links ]
6. González M, Pérez-Díaz J, Ayala J, Casellas JM, Martínez-Beltrán J, Bush K, Baquero F. Gene sequence and biochemical characterization of FOX-1 from Klebsiella pneumoniae, a new AmpC type plasmid mediated ß-lactamase with two molecular variants. Antimicrob Agents Chemother 1994; 38: 2150-7. [ Links ]
7. Li Y, Li Q, Du Y, Juang X, Tang J, Wang J, Guilan L, Jiang Y. Prevalence of plasmid-mediated AmpC ß-lactamases in a Chinese University Hospital from 2003-2005: first report of CMY-2 -type AmpC ß-lactamase resistance in China. J Clin Microbiol 2008; 46: 1317-21. [ Links ]
8. Masuda G, Tomioka S, Hasegawa M. Detection of ß-lactamase production by gram-negative bacteria. J Antibiotics1976; 29: 662-4. [ Links ]
9. Pérez-Pérez FJ, Hanson ND. Detection of plasmid-mediated AmpC ß-lactamase genes in clinical isolates by using multiplex PCR. J Clin Microbiol 2002; 40: 2153-62. [ Links ]
10. Radice M, Cittadini R, Stortz M, Ruggiero M, Gutkind G, Vay C. Emergence of plasmid-mediated AmpC ß-lactamases in ESBL producing enterobacteria in Buenos Aires, Argentina. 47th Interscience Conference on Antimicrobial Agents and Chemotherapy (ICAAC), 2007. Resumen C-2 1512, Chicago, EE .UU. [ Links ]
11. Rapoport M, Monzani V, Pasteran F, Morvay L, Faccone D, Petroni A, Galas M. CMY-2-type plasmid-mediated AmpC ß-lactamase finally emerging in Argentina. Int J Antimicrob Agents 2008; 31: 385-7. [ Links ]
12. Tan T, Yong L, He J, Koh T, Yang Hsu L. Evaluation of screening methods to detect plasmid-mediated AmpC in Escherichia coli, Klebsiella pneumoniae, and Proteus mirabilis. Antimicrob Agents Chemother 2009; 53: 146-9. [ Links ]
13. Yagi T, Wachino J, Kurokawa H, Suzuki S, Yamane K, Doi Y, Shibata N, Uato H, Shibayama K, Arakawa Y. Practical methods using boronic acid compounds for identification of class C ß-lactamase-producing Klebsiella pneumoniae and Escherichia coli. J Clin Microbiol 2005; 43: 2551-8. [ Links ]
14. Cejas D, Radice M, Almuzara M, Quinteros M, Famiglietti A, Vay C, Gutkind G. Detección y caracterización de enzimas de tipo AmpC de localización plasmídica en Escherichia coli. XIII Jornadas Argentinas de Microbiología, 2008. Resumen p. 389, Rosario, Santa Fe, Argentina. [ Links ]
15. Cejas D, Almuzara M, Giovanakis M, Fernández Canigia L, Vay C, Quinteros M, Pagnila G, Lascialandare S, Mutti D, Gutkind G, Radice M. Análisis de prevalencia de enzimas de tipo AmpC de codificación plasmídica (PAMPC) en Enterobacteriaceae. X Congreso de la Sociedad Argentina de Infectología, 2010. Resumen 26906, p. 75, Mar del Plata, Argentina. [ Links ]
Recibido: 29/12/09
Aceptado: 15/12/10














