Introducción
El arroz constituye la fuente de hidratos de carbono más importante para la tercera parte de la población mundial; sin embargo, obtener una óptima producción implica la aplicación de fertilizantes, así como el control de plagas y enfermedades1. Burkholderia glumae y Burkholderia gladioli son considerados los agentes etiológicos del añublo bacterial de la panícula de arroz (ABPA)26,34. Esta enfermedad se manifiesta en la etapa de floración y provoca infertilidad, con decoloración y manchado de la gluma en desarrollo debido a la producción de toxoflavina36. La enfermedad ha sido reportada por primera vez en Perú en 201 324.
El agricultor arrocero no posee un método de control efectivo para el tratamiento del ABPA26: a pesar del empleo intensivo de pesticidas, el 50% de la producción puede perderse debido a esta enfermedad42. Hasta el momento el único agente químico capaz de controlar la enfermedad es el ácido oxolínico26; sin embargo, dicho compuesto está prohibido por las autoridades sanitarias porque constituye un riesgo debido a la generación de resistencia y a la presencia de cepas naturalmente resistentes a este agente químico28. A la fecha las estrategias de prevención son las más empleadas.
Los microorganismos asociados al arroz han coevolucionado con su hospedero en virtud de su capacidad para reducir enfermedades en el nicho ecológico compartido empleando diferentes estrategias26. Bajo esa perspectiva, el control biológico se ha mostrado prometedor6,11. Estrategias basadas en mecanismos biológicos como el quorum quenching20, la resistencia sistémica inducida12 y la producción de metabolitos antimicrobianos50 constituyen las más prometedoras. Shrestha et al.49 reportaron que cepas de Bacillus amyloliquefaciens fueron capaces de inhibir el crecimiento de B. glumae 336 g-1. La búsqueda de microorganismos que comparten el hábitat con B. glumae durante el proceso de desarrollo de la enfermedad se considera de interés, ya que estos podrían servir para controlar el patógeno.
Contar con un cepario de microorganismos con potencial como antagonistas del citado patógeno permitirá disponer de opciones para desarrollar productos biopesticidas, capaces de reducir el uso de agroquímicos perjudiciales para el medioambiente, sobre todo, de antibióticos. Cuando se emplean de manera indiscriminada estos últimos generan resistencia y se transforman en un problema de salud pública. Por otro lado, el uso de biopesticidas contribuiría a reducir los costos de producción, con lo que se elevaría el nivel de competitividad del agricultor.
El objetivo del presente trabajo fue aislar, caracterizar, seleccionar e identificar bacterias endofíticas (BE) de cultivos locales de arroz con actividad antimicrobiana frente a B. glumae. Asimismo, se quiso aislar cepas de B. glumae y B. gladioli en áreas de cultivo de arroz de la zona norte del Perú y estudiar su diversidad genética y los factores de virulencia presentes. La hipótesis planteada es que los microorganismos endofíticos aislados de cultivos locales de arroz han coevolucionado con las cepas fitopatógenas de B. glumae que predominan en dicha región y, por ende, podrían presentar actividad antimicrobiana frente a estas.
Material y métodos
Colecta del material vegetal
La colecta de las plantas de arroz sin síntomas de enfermedad se realizó siguiendo la metodología descrita por Baldani et al.5 y las de plantas con sintomatología de ABPA de acuerdo con Nandakumar et al.43. Se realizaron muestreos en campos de los departamentos de Tumbes, Piura, Amazonas y San Martín entre junio y agosto de 2018 (tabla 1 del material suplementario). Los campos fueron georreferenciados y se colectaron 3 plantas sanas por parcela. Cada planta fue seccionada en raíz, tallo, hojas, espigas y grano; este material fue transportado a 4 °C hasta el laboratorio. Para obtener aislamientos de agentes causales de ABPA los granos con signos de dicha enfermedad fueron colocados en sobres de papel y transportados al laboratorio a temperatura ambiente.
Aislamiento y conservación de bacteriasSe empleó la metodología descrita por Sun et al.51 para obtener BE de raíz, hojas y tallos. La confirmación del proceso de desinfección superficial se realizó sembrando una alícuota del último enjuague en TSA (Himedia, India), con incubación a 30 °C por 3 días. Solo las muestras que evidenciaron ausencia de crecimiento bacteriano en dicho medio fueron seleccionadas para el proceso de aislamiento a partir de tejido macerado empleando mortero y pilón estériles. Para aislar bacterias esporuladas y Burkholderia se siguió la metodología sugerida porCottyn et al.13. Para el aislamiento de B. glumae y B. gladioli una alícuota de tejido macerado fue sembrada en el medio selectivo CCNT modificado33. La composición de dicho medio fue la siguiente: extracto de levadura 2g/l, peptona 1 g/l, meso-inositol 4g/l, cetrimide 10mg/l, cloranfenicol 10mg/l, novobiocina 10mg/l, agar-agar 18 g/l en agua destilada. El pH del medio se ajustó a 4,8 y las placas fueron incubadas a 41 °C durante 4 días. La presencia de un pigmento amarillo fluorescente indicaba la producción de toxoflavina por B. glumae y/o B. gladioli. Todos los aislamientos fueron conservados a corto y mediano plazo en cuña de agar y en crioviales con glicerol al 30%, respectivamente, con almacenamiento a -30 °C en el último caso.
Actividad inhibitoria frente a B. glumaeLa metodología de selección se realizó de acuerdo a lo descripto por Lima et al.39, empleando discos de papel de filtro estériles (6mm), y se llevó a cabo en 2 etapas: 1) selección primaria; y 2) selección secundaria. La primera consistió en la evaluación cualitativa (resultado positivo o negativo) de la actividad inhibitoria del conjunto de BE obtenidas frente a B. glumae THT (10 aislamientos BE/placa). B. glumae THT fue sembrada en 2 ml de medio Luria Bertani (LB) e incubada a 180rpm a 30°C (24h), para ser luego sembrada (100^l) en superficie en placas de Petri con medio King B. Las BE fueron sembradas en 2 ml de TSB e incubadas bajo las mismas condiciones descritas anteriormente. Con alícuotas de 20 ^l de cada BE se impregnaron los discos de papel y estos se distribuyeron sobre una placa de Petri previamente sembrada con B. glumae THT. Este material se incubó a 30 °C (72 h). Los aislamientos que generaron un halo de inhibición de al menos 2 mm2 pasaron a la etapa de selección secundaria, en la que se midió el área de inhibición de B. glumae THT (en mm2), sembrando una sola cepa de BE/placa. Para la selección secundaria, se empleó la misma metodología de siembra e incubación, tanto para las BE seleccionadas como para B. glumae THT. B. glumae THT fue usada como cepa patrón; esta cepa virulenta fue aislada de granos de arroz de la variedad «La Esperanza», con síntomas del ABPA, obtenidos en la región San Martín.
Amplificación y secuenciación del gen 16S ARNr en BE con capacidad inhibitoria frente a B. glumae THTSe obtuvieron cultivos líquidos en TSB (24 h, 30 °C, 120 rpm) hasta alcanzar la fase final de crecimiento logarítmico (DÜ60o ^0,6). Posteriormente, las células fueron recogidas mediante centrifugación (13.680 x g, 3 min), a temperatura ambiente. Para la obtención de ADN genómico se utilizó el kit comercial GeneJET Genomic DNA Purification, siguiendo las instrucciones del fabricante (Thermo Fisher Scientific, EE. UU.). La integridad del ADN extraído fue evaluada luego de ser sometido a electroforesis en gel de agarosa al 1% en tampón Tris borato y tinción con GelRed (Biotium Inc.); la visualización se hizo en un transiluminador UV. Se utilizó como marcador de peso molecular ADN del fago Lambda digerido con la enzima HindIII. La cuantificación del ADN se llevó a cabo utilizando un espectrofotómetro NanoDrop, modelo ND1000 (Thermo Fisher Scientific, EE. UU.). El ADN extraído fue almacenado a -20 °C. La identificación de las bacterias aisladas se realizó mediante el análisis de la secuencia del gen ribosomal 16S. El gen fue amplificado mediante PCR usando los cebadores fD1 y rD154. La especificidad de la amplificación fue verificada visualizando los productos de PCR en un transiluminador UV, luego de separarlos por electroforesis en geles de agarosa al 1% y tenñirlos con GelRed (Biotium Inc.). Los productos de PCR fueron secuenciados con la tecnología Sanger como servicio externo en la companñía Macrogen (Seúl, Corea del Sur). Las secuencias obtenidas fueron analizadas en el servidor EzBioCloud para determinar su afiliación taxonómica.
Producción de sideróforosLas cepas de BE seleccionadas fueron sembradas en caldo LB e incubadas a 37 °C, 200 rpm, durante 24 h. Las células fueron cosechadas en fase estacionaria y, empleando medio succinato normal (SM) y medio succinato desferrado (SM-Fe), fueron lavadas y resuspendidas. La concentración de células fue estandarizada (DO600 = 1,0) y con alícuotas de 100 ^l de estas suspensiones se inocularon Erlenmeyers (125 ml) que contenían 9ml de medio SM o medio SM-Fe (0,1% v/v). Tras 30 horas de incubación a 30 °C y 170 rpm se obtuvo el sobrenadante por centrifugación (9.500 x g, 15 min), que se empleó para la cuantificación de sideróforos. La estimación cuantitativa de la producción de sideróforos se realizó a través de la metodología CAS-Shuttle Assay48. Se midió a 630 nm la absorbancia de una mezcla de 0,5 ml de sobrenadante y 0,5 ml de reactivo CAS, tomando como absorbancia de referencia aquella correspondiente a 0,5 ml de caldo (SM o SM-Fe) no inoculado + 0,5 ml de reactivo CAS. El porcentaje de unidades de sideróforos fue calculado como ([Ar-As] /Ar)*100, donde Ar fue la absorbancia de la referencia y As la de la muestra48.
Resistencia a la toxoflavina
La resistencia a la toxoflavina se evaluó de acuerdo con lo descrito por Choi et al.11. Las BE fueron sembradas en 2 ml de caldo LB e incubadas en agitación a 200 rpm y 37 °C (24 h). Se preparó una solución de toxoflavina (SIGMA-Aldrich) en metanol (25 mg/ml), la cual se almacenó en oscuridad a 4 °C. Se prepararon tubos con 2 ml de caldo LB suplementados con 20 ^g de toxoflavina/ml, que fueron inoculados a razón de 1% (v/v), además de un tubo control. Todos ellos se incubaron a 37°C y 180 rpm durante 24 horas. Un incremento en la DO60o nm =0,2 en presencia de toxoflavina fue considerado resultado positivo. De la misma manera, se evaluaron 2 concentraciones de toxoflavina mayores: 30 y 40 ^g/ml.
Estimación de la diversidad genética de cepas de B. glumaeLas condiciones de crecimiento y extracción de ADN genó-mico fueron similares a las empleadas con las BE. Para estimar la biodiversidad se generaron perfiles genómicos empleando el cebador BOXA1R, según describen Versalo-vic et al.52. Los productos de PCR se separaron en geles de agarosa al 1,2% preparados en tampón Tris borato. La elec-troforesis fue realizada empleando una corriente continua de 60 V y los geles fueron fotografiados bajo luz UV. Como marcador de tamaño molecular se utilizó una escalera de fragmentos de ADN de 100 pb a 10 kb. El análisis comparativo de los perfiles electroforéticos obtenidos mediante BOX-PCR fue realizado empleando el paquete informático InfoQuest FP (versión 4.5) (Bio Rad), que contiene el algoritmo UPGMA basado en el coeficiente de correlación de Pearson. Todos los aislamientos con un nivel de similitud igual o superior al 90% en sus perfiles genómicos fueron considerados clones de una misma cepa, y se eligió un solo representante para la identificación taxonómica. Se calculó el índice de diversidad a nivel de cepa como el cociente entre el número de cepas y el número total de aislamientos42.
Amplificación y secuenciación parcial del gen 16S ARNr de cepas representantes de B. glumaeLa caracterización taxonómica se realizó mediante el análisis de la secuencia del gen ribosomal 16S, tal como ya se describió para las BE con capacidad inhibitoria frente a B. glumae THT.
Evaluación de la virulencia de cepas de B. glumae y B. gladioliSe siguió la metodología sugerida por Karki et al.32, empleando B. glumae cepas THT, A y B3HT como controles positivos y Rhizobium tropici CIAT 899 como control negativo. Dichas cepas fueron reactivadas y purificadas en TSA e incubadas a 30°C (48 h). Las cepas de B. glumae, de B. gladioli y las empleadas como controles fueron sembradas en 2ml de caldo TSB, e incubadas a 30 °C y 180 rpm (48 h). Las células fueron lavadas y resuspendidas en una solución de MgCl2 10 mM estéril mediante 2 centrifugaciones sucesivas (9.500 x g, 5min) y colocadas en microtubos estériles.
Se ajustó la DO de cada tratamiento a 0,1 (5 x 105 ufc/ml). Para la prueba de maceración de catáfila de cebolla se colocaron 4 catáfilas de la variedad «Criolla» en placas de Petri limpias y la cara interna de estas se inoculó con 5 ^l de la suspensión bacteriana, con la punta de una micropipeta. Estas placas se incubaron a 30 °C durante 48 horas, tras lo cual se midió el área de maceración (en mm2) como indicador del potencial fitopatogénico y, por ende, de la virulencia de las cepas. Se realizaron 4 repeticiones por tratamiento, con un diseño experimental completamente aleatorio (DCA).
Producción de toxoflavinaSe realizó una valoración cualitativa de la producción de toxoflavina por cepas de B. glumae y B. gladioli observando el desarrollo de color amarillo por estrías sucesivas en agar King B, con incubación a 37 °C durante 48 horas. Para la valoración se empleó la siguiente escala: -: sin producción de toxoflavina; +: producción baja; ++: producción intermedia; y+++: producción elevada32.
Producción de pigmentosSe empleó la metodología sugerida por Karki et al.32. Las cepas purificadas fueron sembradas por estría en el medio CPG (casaminoácidos, 1,0 g/l; peptona 10,0 g/l; glucosa 10,0g/l, agaragar 18,0g/l) e incubadas a 30°C durante 14 días.
Producción de enzimas extracelularesPara estimar la producción de proteasas se empleó la metodología sugerida por Huber et al.30 para la de celulasas y poligalacturonasas (pectinasas), la sugerida por Degrassi et al.14. Para evaluar la actividad proteasa se empleó agar nutritivo suplementado al momento del servido con 1% de leche descremada, bajo condiciones estériles. Para evaluar la actividad celulasa y poligalacturonasa se empleó agar carboxilmetilcelulosa (CMC) y agar pectina, respectivamente. Las cepas fueron sembradas en LB e incubadas a 170 rpm a 30 °C hasta alcanzar la fase estacionaria (24 h). El pellet fue recuperado por centrifugación (9.500 x g, 5min)yresus-pendido en LB, ajustando la concentración celular a una DO600 = 1,0. Se inocularon las placas de cada medio con 5 ^l de suspensión bacteriana, y se incubó a 37 °C por 48 horas. Estas actividades se determinaron midiendo el halo de hidrólisis formado alrededor de las colonias, expresado como área de hidrólisis (mm2).
Sensibilidad a agentes bactericidasSe empleó la metodología sugerida por Kim et al.34 para evaluar la sensibilidad al peróxido de hidrógeno (H2O2), y la de Maeda et al.40 para determinar la sensibilidad al ácido oxolínico (OA). Las cepas fueron sembradas en caldo LB e incubadas en agitación (200rpm) a 37 °C durante 24 horas, y luego sembradas en superficie (100 ^l) en placas de agar TSA. Se embebieron discos de papel de filtro estériles (6 mm) en solución de H2O2 (30%) (Himedia®, India) y se dispusieron sobre las placas de Petri, que se incubaron a 37°C por
Control biológico de Burkholderia glumae en Perú
24 horas. Se midió el área de inhibición expresado en mm2. Para el ensayo de sensibilidad al OA se emplearon discos embebidos con 0, 10, 50, 100, 250 y 500 ^g/ml de OA, que se obtuvieron partiendo de una solución de OA (20% p/v) (Oxilobac®, Colombia). Los discos fueron colocados sobre placas de Petri que contenían agar Müller-Hinton (Merck®, Alemania) previamente sembrado en superficie con las cepas de B. glumae y B. gladioli en estudio.
Ensayos de motilidadSe empleó la metodología sugerida por Kim et al.34. Las cepas de B. glumae fueron estriadas en agar King B para verificar la pureza del cultivo. Cada cepa fue cosechada y resuspendida en caldo LB a una DO60o = 1,0. El medio LB fue suplementado al 0,3% de agar para evaluar la capacidad de swarming, y al 0,7% agar para evaluar la capacidad swim-ming en cada una de las cepas en estudio. Ambas pruebas fueron valoradas empleando la siguiente escala: -: sin motilidad; +, ++ o +++, según el grado de capacidad swimming y swarming.
Análisis estadísticoLos resultados de las pruebas de actividad inhibitoria, producción de sideróforos y virulencia fueron evaluados mediante el test de normalidad Anderson-Darling y verificación de la homocedasticidad. Cumpliendo ambos requisitos se procedió a realizar el ANOVA paramétrico, usando el test de Tukey al 95% como test de comparaciones múltiples, con el objetivo de ver diferencias significativas entre tratamientos, empleando el paquete estadístico InfoStat 201815.
Resultados y discusión
Colecta, recuperación y aislamiento de BELas BE brindan una serie de ventajas a la planta; se citan entre las principales el biocontrol de fitopatógenos y la producción de factores de promoción del crecimiento2,29. El arroz constituye el modelo de interacción planta-bacteria endófita más estudiado, y se han reportado endófitos en la raíz12, las hojas, los tallos y las semillas9. En el presente estudio se obtuvieron 189 aislamientos de BE (tabla 1), con mayor recuperación de endófitos de raíz y tallo, tal como reportaron Mbai et al.41. Las BE provienen del suelo7, pasando transitoriamente como endofíticas17 y, finalmente, solo algunas seleccionadas son transmitidas por el grano25.
Selección de BE con capacidad inhibitoria contra B. glumaeEn la selección primaria de las 189 BE obtenidas solo 34 (18,0%) fueron capaces de inhibir el crecimiento de B. glumae THT. La inhibición de crecimiento fue variable, con áreas de inhibición desde 2 hasta 451 mm2 (datos no mostrados).
El control de B. glumae ofrece alternativas químicas, culturales y biológicas26; este último es un componente importante en el manejo integrado de la enfermedad49. Actualmente destacan el uso de agentes de biocontrol como Paenibacilluspolymyxa JH235, Bacillusamyloliquefaciens49, Bacillus oryzicola11, B. gladioli47 y Streptomyces50, así como la búsqueda metagenómica de enzimas degradadoras de toxoflavina11.
En la selección secundaria solo 9 aislamientos (26,5% de los 34 reunidos en la selección primaria y 4,8% sobre los aislamientos totales) fueron confirmados con actividad inhibitoria mayor de 150mm2 (tabla 2, figs. 1 A y B). De esos 9 aislamientos 5 fueron endófitos radiculares, 2 caulinares y 2 foliares. No se encontraron BE con capacidad inhibitoria del crecimiento de B. glumae THT procedentes de espiga ni de grano.
Caracterización genética de las BE seleccionadasLas BE con actividad inhibitoria del crecimiento de B. glumae THT correspondieron a los géneros Burkholderia y Bacillus, de acuerdo con el análisis filogenético de la secuencia del gen 16S ARNr (tabla 2).
Las cepas TUR04-01 y TUR04-03 presentaron un 99,93% y 99,72% de identidad, respectivamente, con Burkholderia vietnamiensis LMG 10929t. Por su versatilidad ecológica y habilidad para usar una gran variedad de compuestos como única fuente de carbono y producir metabolitos secundarios, no sorprende que el género Burkholderia constituya una alternativa de biocontrol para otros miembros del mismo género10,47.
Las cepas SMBH14-02, TUT05-02 y TUR07-04 presentaron un 99,79%, 100% y 99,93% de identidad, respectivamente, con Bacillus megaterium NBRC 15308t. Las cepas AMH12-02 y SMBR13-04 presentaron un 99,93% y 100% de identidad, respectivamente, con Bacillus aryabhattai B8W22T. La cepa SMNC17-02 presentó una identidad del 99,76% con Bacillus subtilis subsp. stecoris D7XPN1T y, finalmente, la cepa TUR07-02 fue un 99,78% idéntica a la cepa Bacillus vele-zensis CR-502t (tabla 2). El género Bacillus tiene un uso potencial en el biocontrol de bacterias fitopatógenas8,16,45, debido a la producción de una serie de lipopéptidos antimicrobianos como la iturina y la surfactina, así como la jinggangmicina46.
Producción de sideróforos por BE seleccionadasLa oxidación del hierro (Fe2+ a Fe3+) se da en presencia de oxígeno y a pH neutro, y la forma oxidada es poco soluble y no disponible para las bacterias; es por ello que la producción de sideróforos constituye una estrategia directa para la nutrición bacteriana en condiciones limitantes de hierro53. El 44,4% de las BE antagonistas produjeron sideróforos en medio de cultivo con trazas de Fe3+, mientras que el 66,7% produjeron sideróforos en medio desferrado. Todas las cepas incrementaron la producción de sideróforos cuando en el medio líquido hubo privación del hierro (tabla 2).
Las cepas de B. vietnamiensis TUR04-01 y TUR04-03 produjeron la mayor concentración de sideróforos solubles, tanto en presencia de hierro como en medio desferrado (tabla 2). Se ha reportado que el salicilato, la ornibactina y la maleobactina constituyen los sideróforos más comunes producidos por el género Burkholderia19,23. El género Bacillus, a diferencia del género Burkholderia, produce side-róforos del tipo catecoles (bacilibactinas), que favorecen el crecimiento de la planta y protegen contra patógenos4.

Tabla 1: Distribución de los aislamientos de BE de arroz de acuerdo con el origen y órgano seleccionado

Figura 1: Actividad inhibitoria de B. vietnamiensis TUR04-03 (A) y B. aryabhattai AMH12-02 (B) contra B. glumae THT en medio TSA incubado a 30°C por 72 horas.
La producción de sideróforos se ha asociado al control de fitopatógenos por limitación del Fe en el medio de cultivo. En nuestro estudio la actividad inhibitoria de las BE contra B. glumae THT estuvo correlacionada de manera positiva con la producción de sideróforos en condiciones normales de Fe (r = 0,58) así como en ausencia de Fe (r = 0,74).
Resistencia a la toxoflavina por BE seleccionadasLa toxoflavina es una fitotoxina con características antimicrobianas, antifúngicas e incluso herbicidas, que genera peróxido de hidrógeno (H2O2) en presencia de luz y oxígeno, interrumpiendo la cadena respiratoria del arroz44. Se han reportado cepas bacterianas capaces de tolerar hasta 40 ^g/ml de toxoflavina, y es suficiente > 3 ^g/ml para inhibir el crecimiento de Escherichia coli35.
Se ha informado de que la cepa JH2 de P. polymyxa, aislada de semillas de arroz sanas, presenta el gen tflA, que expresa una enzima degradadora de esta fitotoxina35. Un estudio de metagenómica de Choi et al.11 da cuenta del gen txeA, que codifica una metaloenzima con actividad degradadora de toxoflavina. A la concentración de 20 ^g/ml, 8 cepas portadoras de dicho gen fueron capaces de tolerar (DO600 <0,5) toxoflavina, mientras que B. aryabhattai SMBR13-04 fue sensible. Asimismo, B. subtilis subsp. stecoris SMNCT17-02 fue sensible a 30 ^g/ml. Solo el 55,6% fue tolerante a 40 ^g/ml de toxoflavina (tabla 2). Sería interesante determinar la posible presencia de los genes tflA o txeA en el genoma bacteriano de las cepas endofíticas tolerantes a toxoflavina.
Aislamiento, diversidad genética e identificación de cepas nativas de B. glumae y B. gladioliSe obtuvieron 17 aislamientos en medio CCNT modificado, 16 de grano y uno de tallo. Las 17 cepas fueron agrupadas en 10 perfiles genómicos BOX diferentes (A, B, C, D1, D2, D3, E1, E2, E3, E4) (tabla 3). Se eligió una cepa representante de cada perfil genómico para posteriores estudios de confirmación filogenética y caracterización de los principales factores de virulencia. La técnica de huella genómica BOX-PCR es ampliamente usada debido a su destacado poder de resolución intercepa, atendiendo a sus diferencias genómicas13,31.
En nuestro estudio se observó una diferenciación genó-mica entre cepas de B. glumae acorde con su distribución geográfica. Lee et al.36 demostraron que cepas de B. glu-mae de diferentes orígenes geográficos presentan rearreglos genómicos irregulares y esporádicos, sobre todo reordenamientos de elementos genéticos por inversión a gran escala, lo cual permite su adaptación a nuevos ambientes.
La amplificación y secuenciación parcial del gen 16S ARNr de las 10 cepas representantes de cada perfil genómico BOX-PCR detectó similitud entre 9 de ellas (perfiles B, C, D1, D2, D3, E1, E2, E3 y E4) y B. glumae LMG2196T; la cepa restante (perfil A) la presentó con B. gladioli NBRC13700T. En nuestro estudio solo un aislamiento fue recuperado de tallo e identificado como B. glumae, probablemente debido al estilo de vida endofítico38.

Tabla 2: Caracterización de bacterias endófitas de arroz procedentes de la zona norte del Perú preseleccionadas por su capacidad inhibitoria frente a Burkholderia glumae cepa THT

Tabla 3: Caracterización de aislamientos de B. glumae y B. gladioli obtenidos de muestras de grano y tallo de arroz cultivado en cuatro departamentos de la zona norte del Perú
La producción de toxoflavina fue superior en la cepa B. glumae TUT05-07 (tabla 3). La toxoflavina es producida tanto por B. glumae como por B. gladioli bajo el control del regulador transcripcional ToxR del operón toxABCDE22, y constituye el principal factor de virulencia, aunque se ha reportado que la virulencia es menor en B. gladioli31,36.
Los pigmentos desempeñan un rol importante en la adaptación ecológica de los microorganismos, asociados a la protección frente a las radiaciones o a la exacerbación de los factores de virulencia32. Dos cepas nativas y 2 cepas patrón de B. glumae no produjeron pigmentos visibles ni en colonias ni difusibles al medio. Se detectó producción de un pigmento azul en 4 cepas nativas y en la cepa patrón B. glumae THT. Tres cepas nativas y una cepa patrón produjeron pigmento azul, pero los bordes fueron marrones (tabla 3). Karki et al.31 reportaron que cepas de B. glu-mae produjeron pigmentos con propiedades antimicrobianas y que, además, incrementaron la tolerancia a luz UV, y se postula que dichos pigmentos pueden ser utilizados para sobrevivir en condiciones oligotróficas o de estrés27,32.
El ensayo de virulencia en catáfila de cebolla mostró que la cepa B. glumae SMNCG18-05 produjo la mayor área de maceración, mientras que la menor la produjo B. glumae AMG12-06 (tabla 3). Karki et al.31 informaron de que existe una correlación directa entre el ensayo de maceración de catáfila de cebolla y la prueba de infectividad en panículas de arroz31, por lo que la primera constituye una prueba rápida y de menor costo para determinar la virulencia de cepas de B. glumae. Nosotros no observamos correlación entre los niveles de maceración de catáfila de cebolla y la producción de toxoflavina (r = -0,05). Aunque la producción de toxoflavina en B. glumae está a menudo asociada a un fenotipo virulento, no es condición suficiente para provocar enfermedad, según postulan Karki et al.32.
En relación con las enzimas extracelulares que pueden ayudar en los procesos de colonización e infección se encuentran proteasas, celulasas y poligalacturonasas (pec-tinasas). El 80% de las cepas presentaron actividad proteasa un día después de la siembra; sin embargo, todas las cepas mostraron actividad proteasa, en mayor o menor grado, después de 3 días de incubación (tabla 3). La producción de proteasas extracelulares en el género Burkholderia es muy común56. Esta actividad puede cobrar importancia en etapas avanzadas de la enfermedad, cuando se requieren polipépti-dos, oligopéptidos y aminoácidos, que pueden ser liberados a partir de la hidrólisis de las proteínas del tejido vegetal3. Lelis et al.37 comunicaron que la metaloproteasa codificada por el gen prtA constituye un factor de virulencia de B. glumae.
El 53,3% de las cepas demostró actividad celulolítica; la máxima actividad se detectó en la cepa B. glumae SMNCG18-05 (tabla 3). En otros patógenos las celulasas hidrolizan la pared celular de la planta huésped y desempenñan un papel importante y sinérgico en la patogénesis, junto con otras enzimas hidrolíticas. Hasta el momento no se ha reportado actividad celulolítica en B. glumae y B. gladioli; sin embargo, se infiere de estos hallazgos que las celulasas producidas podrían tener un rol sinérgico en la virulencia53.
Prueba de ello es la correlación obtenida entre la mace-ración de catáfila y la producción de celulasas (r = 0,61).
Finalmente, se hallaron 5 cepas de B. glumae con actividad poligalacturonasa evaluada cualitativamente (tabla 3). Se ha propuesto que la actividad poligalacturonasa en bacterias fitopatógenas ayudaría a obtener nutrientes de la pared celular cuando la enfermedad se encuentra en un estado avanzado14. B. glumae expresa 2 isoformas de endopoli-galacturonasas (pehA y pehB)49 y la actividad enzimática depende principalmente de la densidad celular, la fuente de carbono y los productos de descomposición de los polímeros de pectina26. No se ha informado actividad poligalacturo-nasa en B. gladioli18. Por otro lado, el 58,3% no presentó actividad, probablemente debido a la carencia de los citados genes, o a una actividad débil debido a la expresión del gen pehB solamente bajo condiciones in vitro. No se encontró una correlación entre la virulencia y la actividad poligalac-turonasa. Degrassi et al.14 reportaron que cepas mutantes de B. glumae (pehA- y pehB-) presentaron similar virulencia que las cepas originales en panículas de arroz.
La cepa B. glumae SMNCG18-05 presentó la mayor sensibilidad al H2O2, mientras que la cepa B. glumae TUT05-07 fue la más resistente (tabla 4). La tolerancia al H2O2 se asocia con la presencia y efectividad de las enzimas cata-lasa y peroxidasa para superar el estrés oxidativo por H2O2. La catalasa permite la detoxificación del H2O2 evitando el dano oxidativo en las células microbianas. Curiosamente, la correlación entre actividad celulolítica y sensibilidad al peróxido de hidrógeno fue elevada (r = 0,80), es decir, una mayor actividad endoglucanasa (endo-1,4-p-glucanasas), celobiohidrolasa (exo-1,4-p-glucanasas) y p-glucosidasa46, por alguna circunstancia, está relacionada con una menor actividad catalasa y/o peroxidasa en las cepas de B. glumae.
La hipótesis de una actividad celulolítica superior dependiente de una menor resistencia al H2O2 podría explicar esta elevada correlación. Forsberg et al.21 informaron de la presencia de monooxigenasas líticas de polisacáridos dependientes de cobre en actinobacterias, que requieren H2O2 para clivar polisacáridos como la celulosa. Si bien no ha sido reportada en patógenos de plantas, se recomienda la búsqueda de esta actividad enzimática específica para comprobar dicha hipótesis.
Al menos 7 cepas (46,7%) presentaron resistencia a 10 ^g/ml de OA; la sensibilidad a este antibiótico fue variable y mayor en B. glumae AMG12-06. Al aumentar la concentración, la mayoría de las cepas se mostraron más o menos sensibles. Solo B. gladioli TUG05-07 puede ser considerada como altamente resistente (tabla 4).
La resistencia intrínseca a los antibióticos es un indicativo de adaptación ecológica al ambiente, y en la mayoría de los casos refleja uso inadecuado de antibióticos30. Se ha reportado una fácil generación de resistencia en aislamientos de B. glumae, asociada a las sustituciones Ser83Arg y Ser83Ile en GyrA40. En Perú han existido reportes de uso indebido de OA mediante aplicaciones clandestinas desde 2013, las cuales podrían influir en la generación de cepas resistentes.
La motilidad tipo swimming y swarming desempena un papel fundamental en el comportamiento social y virulento de B. glumae26. El movimiento a través de un líquido empleando flagelos se denomina swimming, y cuando el movimiento ocurre sobre una superficie, como una biopelícula, se denomina swarming34. Las cepas con mayor capacidad de swimming correspondieron a B. glumae TUG07-11, SMNCG18-06 y THT; por el contrario, la actividad estuvo ausente en B. gladioli TUG05-07, probablemente porque carece de flagelos y, por ende, tiene menor capacidad invasiva (figs. 2 A y B).
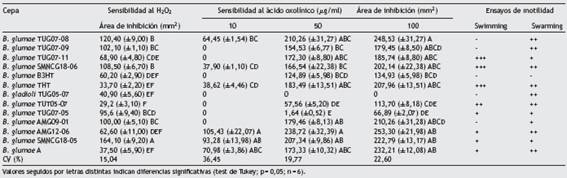
Tabla 4: Factores de virulencia en cepas de B. glumae y B. gladioli obtenidas de grano y tallo de arroz cultivado en 4 departamentos de la zona Norte del Perú

Figura 2: Diferentes niveles de swimming en B. glumae TUG07-09 (A) y B. glumae TUG07-11 (B) creciendo en LB suplementado con 0,5% agar.
Se observó una correlación débil positiva (r = 0,20) y negativa (r = -0,22) entre la virulencia, el swimming y el swarming, respectivamente. La motilidad es uno de los factores de virulencia cruciales para B. glumae34.
En conclusión, encontramos que cepas nativas de 3 especies de Bacillus, además de B. vietnamiensis, son endófitos con actividad inhibitoria contra B. glumae THT bajo condiciones in vitro, con características relacionadas con el biocontrol. Asimismo, este constituye el primer reporte sobre la diversidad genética y el estudio de los principales factores de virulencia de cepas de B. glumae y B. gladioli aisladas en Perú. La gran variabilidad genética encontrada entre las cepas de B. glumae y B. gladioli y la diversa expresión de factores de virulencia nos demuestra la elevada adaptabilidad de estos patógenos al medio ambiente. Es preciso lograr una mejor comprensión de la enfermedad para poder diseñar estrategias de control de estas enfermedades empleando agentes microbianos.
Financiación
El presente trabajo ha sido financiado por la Universidad Nacional de San Martín-Tarapoto-Perú, con resolución n.° 296-2018-UNSM/CU-R/NLU; Red Agromicrobios-Universidad Nacional Agraria La Molina-Perú.
Conflicto de intereses
Los autores han declarado que no presentan ningún conflicto de intereses existentes.
Agradecimientos
A los bachilleres Luz E. Valdiviezo Saavedra, Jean C. Rodríguez Peralta y Rony Castro Tuanama por el apoyo en los trabajos de caracterización genética.
Anexo. Material adicionalSe puede consultar material adicional a este artículo en su versión electrónica disponible en doi:10.1016/j.ram. 2019.12.002












 uBio
uBio 

