Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Mastozoología neotropical
versión impresa ISSN 0327-9383versión On-line ISSN 1666-0536
Mastozool. neotrop. vol.25 no.2 Mendoza dic. 2018
ARTÍCULO
Variabilidad genética de visones americanos Neovison vison asilvestrados en la provincia de Santa Cruz: ¿se cumple la paradoja genética de las especies invasoras?
Martina S. Malerba1, 3, Laura Fasola2, 3, Ignacio Roesler1, 3, 4, María Inés Pereda3, Andrés de Miguel3, Lucía Martín3 y Bettina Mahler1
1 Laboratorio de Ecología y Comportamiento Animal, Instituto de Ecología, Genética y Evolución de Buenos Aires, Consejo Nacional de Investigaciones Científicas y Técnicas – Universidad de Buenos Aires, Facultad de Ciencias Exactas y Naturales, Ciudad Autónoma de Buenos Aires, Argentina.[Correspondencia: Martina Malerba <ushmartu@gmail.com>]
2 Consejo Nacional de Investigaciones Científicas y Técnicas (CONICET)-Delegación Técnica Patagonia, Administración de Parques Nacionales. San Carlos de Bariloche, Río Negro, Argentina.
3 Proyecto Macá Tobiano – Aves Argentinas/Asociación Ornitológica del Plata y Asociación Ambiente Sur.
4 EDGE affiliated. Zoological Society of London.
RESUMEN
Las especies invasoras constituyen una amenaza para la biodiversidad nativa. El éxito de las especies invasoras pese a su baja variabilidad genética se conoce como "paradoja genética”. El visón americano (Neovison vison) fue introducido en varias regiones del planeta como Europa, Asia y América del Sur con fines peleteros. En Argentina fue introducido en la década de 1930 y, como resultado de la liberación de individuos en la naturaleza, lograron establecerse poblaciones silvestres en la región Patagónica. El objetivo del presente trabajo es estudiar la diversidad genética de una población asilvestrada de la provincia de Santa Cruz y compararla con la de una población en cautiverio. Analizamos un fragmento de 359 pb de la región control de ADN mitocondrial y encontramos índices de variabilidad similares entre la población silvestre y la población cautiva. Al igual que en poblaciones asilvestradas de otras regiones del planeta, no se observa una reducción en la variabilidad genética en la población silvestre de Santa Cruz, probablemente como consecuencia de liberaciones y escapes recurrentes desde diversas fuentes de cautiverio.
ABSTRACT
Genetic variation of wild American mink (Neovison vison) in the Province of Santa Cruz. Is the genetic paradox of invasive species met?
Alien species represent a threat to native biodiversity. The success of invasive species despite their reduced genetic diversity is known as "the genetic paradox”. The American mink (Neovison vison) was introduced in several regions of the world, like Europe, Asia and South America. In Argentina, they were introduced in the 1930’s by the fur industry and wild populations were established in Patagonia due to liberations and escapes. In this study, we analyzed the genetic diversity of a wild American mink population of northern Santa Cruz province and compared it with a captive population. We sequenced a 359-bp fragment of the mtDNA control region and found that genetic variability was similar between both groups. Like in other wild invasive populations, genetic variability was not reduced in the population of Santa Cruz, probably as a consequence of frequent releases and escapes from different sources of captive minks.
Palabras clave: Biodiversidad; Diversidad haplotípica; Diversidad nucleotídica; Genética poblacional; Patagonia.
Key words: Biodiversity; Haplotype diversity; Nucleotide diversity; Patagonia; Population genetics.
Recibido 6 abril 2018.
Aceptado 3 agosto 2018.
Editor asociado: I. Tomasco
INTRODUCCIÓN
Las especies invasoras son reconocidas como la segunda causa de pérdida de biodiversidad a nivel mundial, después de la pérdida de hábitat (Schmitz & Simberloff 1997; Simberloff 2001; Genovesi 2009). Se trata de aquellas especies introducidas por el ser humano que se han dispersado y establecido fuera de su área de distribución natural y constituyen una amenaza para la biodiversidad nativa (Convenio sobre la Diversidad Biológica [CDB] 1992). El proceso de invasión involucra cambios espacio-temporales del tamaño poblacional que frecuentemente llevan a una reducción sucesiva de la diversidad genética durante la introducción, el establecimiento y la propagación de aquellos individuos introducidos (Schrieber & Lachmuth 2016). En una población originada a partir de unos pocos eventos de introducción y unos pocos individuos invasores se espera una baja variabilidad genética, ya que constituye una sub-representación de los genotipos y fenotipos de la población original (i.e. efecto fundador). El éxito de las invasiones biológicas a pesar de la baja variabilidad genética constituye una "paradoja genética” (Schrieber & Lachmuth 2016) porque muestra un patrón contrario a la relación positiva encontrada entre la variabilidad genética y el "fitness” (éxito reproductivo) (Markert et al. 2010; Reed & Frankham 2003). Estudios recientes encontraron evidencias contrapuestas a este patrón y sugieren que las poblaciones con baja diversidad genética podrían ver aumentado su fitness a causa del cuello de botella que atraviesan en el proceso de invasión, que tendría un efecto beneficioso en caracteres particulares y purgaría mutaciones deletéreas, entre otros efectos (Parisod et al. 2005; Facon et al. 2011; Estoup et al. 2016). La reducción de la variabilidad genética estaría compensada a su vez por factores ecológicos que favorecen el establecimiento y permanencia de las poblaciones introducidas, tales como la ausencia de depredadores, la disponibilidad de nichos ecológicos, la susceptibilidad del ecosistema receptor a la invasión (invasibilidad), la presión de propágulos y las características intrínsecas de la especie invasora (invasividad) (Mack et al. 2000).
El visón americano (Neovison vison) es uno de los mamíferos invasores con mayor distribución en el planeta (Global Invasive Species Database [GISD] 2018). Se encuentra en estado silvestre en numerosos países de Europa, Asia y Sudamérica donde fue introducido en la naturaleza principalmente como consecuencia de la industria peletera, a través de escapes accidentales o intencionales, y en ocasiones como forma de enriquecer la fauna local (Jaksic et al. 2002; Zalewski et al. 2011; Korablev et al. 2017). Representa una amenaza a la biodiversidad nativa generando impactos negativos en aves, anfibios y mamíferos (MacDonald & Harrington 2003; Bonesi & Palazon 2007; Zalewski et al. 2009). La interacción que se establece entre el visón y la fauna nativa se da principalmente por depredación de este último sobre especies autóctonas y por competencia con especies ecológicamente equivalentes (Jaksic et al. 2002; Fasola et al. 2009; Ibarra et al. 2009; Peris et al. 2009; Fasola & Valenzuela 2014). Incluso se presume que es un eslabón clave en la transmisión de enfermedades desde el ámbito doméstico al silvestre (Sepúlveda et al. 2014).
A principios de la década del 30, el visón fue introducido en la Patagonia argentina y chilena, con el fin de iniciar el comercio de pieles en la región (Jaksic et al. 2002). Los primeros criaderos de visón que se instalaron en la Patagonia argentina se encontraban en las provincias de Chubut y Tierra del Fuego, y operaron incluso hasta fines de la década de 1970 (Fasola et al. 2011). En 1960 se detectaron por primera vez poblaciones silvestres en el Parque Nacional Los Alerces ubicado en la provincia de Chubut, originadas como consecuencia de escapes y liberaciones desde criaderos de la zona (Chehébar et al. 1986; Fasola et al. 2011). Actualmente el visón se encuentra en estado silvestre al norte de la provincia de Neuquén, en Río Negro, Chubut y oeste de Santa Cruz (Fasola et al. 2011; Guichón et al. 2016; Valenzuela et al. 2016; Fasola & Roesler 2018). Proveniente de otro foco más austral, la población de la Isla Grande de Tierra del Fuego (Fasola & Valenzuela 2014) se ha expandido a otras partes del archipiélago fueguino (Rozzi & Sherriffs 2003; Valenzuela et al. 2013).
En la provincia de Santa Cruz, la especie fue declarada perjudicial (Ley Provincial 3353/14) como consecuencia de los efectos negativos sobre algunas especies endémicas de la región (Roesler et al. 2012; Barnett et al. 2014). Uno de los mayores impactos negativos sobre la biodiversidad nativa se registró con la reducción del 3% de la población global de macá tobiano (Podiceps gallardoi), un ave acuática críticamente amenazada y endémica de Santa Cruz, por un único visón americano en la meseta del lago Buenos Aires (Roesler et al. 2012). Este caso particular llevó a implementar en 2013 un "Programa de Control del Visón Americano” en el marco del Proyecto Macá Tobiano (coordinado por Aves Argentinas-Asociación Ambiente Sur; Fasola & Roesler 2016). En los programas de control, tradicionalmente se monitorea el tamaño poblacional en el tiempo para evaluar los efectos de la remoción de individuos sobre la población controlada (Melero et al. 2010; Porteus et al. 2012). Sin embargo, la inclusión de estudios genéticos como una herramienta de monitoreo puede brindar información sobre el tamaño efectivo poblacional, la tasa de inmigración y la estructuración genética de la especie invasora (Schwartz et al. 2007, Zalewski et al. 2016). Además, ayuda a establecer la conectividad y la direccionalidad del flujo de individuos, permitiendo plantear hipótesis sobre barreras al movimiento que pueden ser incluidas en las estrategias de control (Bryce et al. 2011).
El objetivo de este trabajo es analizar la variabilidad genética de una población silvestre de visón americano del noroeste de la provincia de Santa Cruz y comparar sus niveles de diversidad genética con los de una población cautiva de la provincia de Buenos Aires. Los criaderos mantienen una alta variabilidad genética de sus stocks renovando periódicamente parte de los individuos reproductores (Gosálvez & Díaz 1992). De esta forma, evitan que la endogamia dé origen a problemas genéticos, de salud, infertilidad y baja viabilidad. Este es un patrón general para diferentes poblaciones cautivas, en las que se observan altos niveles de diversidad genética (Zalewski et al. 2011; Korablev et al. 2017). Considerando esta característica compartida por las poblaciones cautivas, se utilizó un criadero actualmente activo en la provincia de Buenos Aires como referencia de la variabilidad genética de los criaderos que dieron origen a los individuos asilvestrados en Santa Cruz. Estos no pudieron ser muestreados dado que dejaron de funcionar hace al menos cinco décadas. Se espera encontrar una menor variabilidad genética en la población asilvestrada, producto de un efecto fundador de unos pocos individuos liberados. Además, se analizará la distribución espacial de la variabilidad genética de la población silvestre, que podría indicar una estructuración geográfica en los movimientos del visón americano en la provincia de Santa Cruz.
MATERIALES Y MÉTODOS
Toma de muestras
Se trabajó con 42 muestras de tejido de individuos de visón americano provenientes de dos poblaciones, una población silvestre (n=19) y otra de criadero (n=23). Los animales silvestres fueron capturados en la provincia de Santa Cruz en el marco del "Programa de Control de Visón Americano” del Proyecto Macá Tobiano (Fasola & Roesler 2016), en el período comprendido entre diciembre 2013 y mayo 2014. Dieciocho muestras fueron extraídas de individuos del área de la meseta del lago Buenos Aires y solamente una de un individuo proveniente de la meseta La Siberia localizada 250 km al sur del lago Buenos Aires, donde la llegada de visones parece tener otro origen (Fig. 1A). El área de la meseta del lago Buenos Aires incluye el Parque Nacional Patagonia. Se pudo determinar el sexo en dieciocho de los diecinueve individuos, siendo siete hembras y once machos. Las muestras de la población en cautiverio (n=23) se obtuvieron en junio de 2014 durante una faena del establecimiento "Coliñir” de Pedro Massen, Batán, provincia de Buenos Aires (Fig. 1A). De estas muestras, diez correspondieron a machos y trece a hembras. De cada individuo se tomó una muestra de tejido muscular y se almacenó en 1 ml de buffer DMSO (20% v/v DMSO, 250 mM EDTA, NaCl).

Fig. 1. A) Presencia actual e histórica de visones americanos. Círculos naranjas: principales criaderos que funcionaron en Patagonia (Fasola & Valenzuela 2014); círculo azul (I): criadero en funcionamiento, Batán, Buenos Aires; cuadrados rojos: sitios de recolección de muestras correspondientes a poblaciones silvestres, meseta del lago Buenos Aires (II) y meseta de La Siberia (III). Se capturo un único individuo en la meseta La Siberia. B) Detalle de la meseta del lago Buenos Aires, donde fue aplicado el "Programa de Control de Visón Americano”. Los puntos amarillos indican los sitios de ubicación de las trampas donde se capturaron 17 individuos: río Telken (n=4); laguna El Cervecero, río Ecker y río Pinturas (n=10); río Columna (n=3). Los gráficos de torta indican la distribución de haplotipos en las tres subcuencas: Violeta: Hap 1; Rojo: Hap 4; Naranja: Hap 5; Azul: Hap 6; Verde: Hap 9. Los límites del Parque Nacional Patagonia se muestran en gris oscuro.
Análisis genéticos
La extracción de ADN se realizó siguiendo el protocolo modificado de deshidratación con NaCl y etanol (Miller et al. 1988) para 40 muestras de un total de 42. No se logró obtener de forma exitosa ADN de las dos muestras restantes con el protocolo de extracción por deshidratación, por lo que en estos casos la extracción se realizó con el kit DNeasy® Blood & Tissue (Qiagen).
Se amplificó un fragmento de 359 pares de bases perteneciente a la región control mitocondrial utilizando los primers LGL283 y ISM015 (Hundertmark et al. 2002). Esta región es ampliamente usada en estudios de variabilidad genética en mamíferos porque acumula mutaciones en forma relativamente rápida, presenta herencia exclusivamente materna y no tiene recombinación (Korablev et al. 2017). La amplificación por PCR fue realizada en un volumen de reacción de 10 μl con las siguientes concentraciones: 50-100 ng ADN, 2.5 mM MgCl2, 1 mM dNTPs, 0.5 μM primers sentido y antisentido y 0.25 U Taq polimerasa (Invitrogen). Las muestras se amplificaron implementando un protocolo que consta de un ciclo de desnaturalización a 95°C durante 15 min., 30 ciclos a 95°C durante 30 seg., 52°C durante 60 seg. y 72°C durante 60 seg., seguido por una extensión final a 72°C durante 30 minutos. Los productos de amplificación fueron purificados con un protocolo Exo/SAP antes de ser enviados a secuenciar al Servicio de Secuenciación, INTA Castelar.
Los haplotipos obtenidos se compararon con 31 fragmentos de ADNmt publicados en un trabajo previo y depositados en GenBank (JF430903–JF430933; Zalewski et al. 2011). Las nuevas secuencias fueron depositadas en GenBank (MH429412-MH429420).
Análisis de variabilidad genética
Las secuencias se alinearon con el programa Bioedit 7.2.6 (Hall 1999) y se editaron manualmente. Los índices de variabilidad genética para cada población se calcularon con el programa Arlequin 3.5 (Excoffier & Lischer 2010): número de haplotipos, diversidad haplotípica (h) y diversidad nucleotídica (π). La diversidad génica, h (Nei 1973), también llamada diversidad haplotípica cuando se utilizan datos de haplotipos como en el caso del ADNmt, tiene como ventaja ser menos sensible al efecto del tamaño muestral (Freeland 2006). Por otro lado, se estimó la diversidad nucleotídica (π), que cuantifica la divergencia media entre secuencias tomadas de a pares y suele ser más informativa en genomas de evolución relativamente rápida como es el caso de ADNmt en especies animales.
Se construyó una red de haplotipos utilizando el algoritmo Median Joining del paquete PEGAS perteneciente al programa R versión 3.5.1 para evaluar la distribución de haplotipos entre poblaciones. Además, se analizó la distribución de haplotipos en el área de manejo de la meseta del lago Buenos Aires.
Por último, se analizaron las relaciones entre los haplotipos del presente trabajo y los descriptos por García et al. (2017) para la distribución original de la especie. Sobre la base de las secuencias homólogas de 321 pares de bases obtenidas de GenBank, editadas con el programa Bioedit 7.2.6 (Hall 1999), y extrayendo los haplotipos únicos con DnaSP v6.11.01 (Rozas et al. 2017), se construyó un árbol bayesiano utilizando el programa MrBayes (Ronquist y Huelsenbeck 2003). Se usaron 6x105 pasos (MCMC) con muestreo cada 100 iteraciones, dos corridas en simultáneo de 4 cadenas. El soporte de los nodos del árbol obtenido corresponde a la probabilidad posterior. Se consideró alcanzada la convergencia de ambas cadenas cuando el valor de "split frequency” fue de 0.01. Además se evaluó la verosimilitud de este parámetro y el alcance del estado estacionario con el programa TRACER v.1.5 (Rambaut & Drummond 2007). Como grupo externo se utilizó una secuencia recuperada con BLAST de GenBank correspondiente a Mustela frenata (código GenBank: HM106321.1).
RESULTADOS
Se amplificó con éxito un fragmento de 359 pb de la región control de ADN mitocondrial en un total de 39 de los 42 individuos muestreados, 21 provenientes de cautiverio y 18 provenientes de Santa Cruz. El fragmento amplificado mostró 18 sitios variables que conformaron nueve haplotipos (Tabla 1, GenBank: MH429412-MH429420). Seis de los nueve haplotipos encontrados (Hap 1-Hap 6) fueron descritos previamente (Zalewski et al. 2011).
Tabla 1
Detalle de los haplotipos y sitios variables tomando como referencia las posiciones del haplotipo GenBank JF30910 (Zalewski et al. 2011). (--) indica indels.
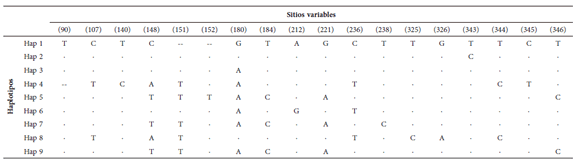
Se encontró un único haplotipo compartido entre la población cautiva y la silvestre (Hap 1), el cual está presente en el 36% de los individuos totales (Tabla 2). De los ocho haplotipos restantes, cuatro se encontraron exclusivamente en los animales cautivos y los otros cuatro exclusivamente en los individuos de la población silvestre de Santa Cruz (Tabla 2). La muestra proveniente de la meseta de La Siberia presentó el haplotipo Hap 4, que también se encontró en 4 individuos capturados en la meseta del lago Buenos Aires.
Tabla 2
Distribución de haplotipos en las poblaciones de estudio (Silvestre n=18 y Cautiverio n=21) y para el total de individuos (n=39).

No se observaron diferencias en los índices de variabilidad genética entre la población cautiva y la población silvestre (Tabla 3). La red de haplotipos muestra que los haplotipos encontrados en la población silvestre son de origen múltiple (Fig. 2).
Tabla 3
Índices de diversidad genética en ambos grupos Silvestre y Cautiverio, con sus respectivos desvíos estándar.
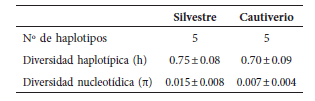

Fig. 2. Red de haplotipos que muestra la relación entre los 9 haplotipos hallados en las poblaciones silvestre (noroeste de la provincia de Santa Cruz) y cautiva (criadero de la provincia de Buenos Aires) de visón americano. El tamaño del círculo es relativo al número de individuos que portan ese haplotipo y el largo de las ramas proporcional al número de cambios entre haplotipos. El color rojo representa la proporción de individuos encontrados en la población cautiva y el color celeste la proporción hallada en la población silvestre.
A pesar de que el apoyo estadístico de los nodos del árbol de la Fig. 3 no es alto y no permite conocer las relaciones filogenéticas entre los haplotipos, muestra que los haplotipos hallados en Patagonia y en el criadero de la provincia de Buenos Aires se agrupan con dos linajes diferentes del área de distribución original de la especie en Norteamérica.

Fig. 3. Árbol construido a partir de un análisis bayesiano que muestra la relación entre los haplotipos pertenecientes a los grupos silvestre (S) y cautiverio (C), y los descriptos por un trabajo previo provenientes de la distribución nativa (DO) de la especie (García et al. 2017). Los números ubicados sobre los nodos indican la probabilidad posterior obtenida para ese grupo. Para Hap1_DO-Hap10_DO se detallan sus respectivos códigos de acceso GenBank. El árbol se enraizó con una secuencia perteneciente a Mustela frenata (OutG; código GenBank: HM106321.1).
El análisis de distribución de haplotipos entre las vías de acceso a la meseta del lago Buenos Aires mostró que los individuos capturados en los ríos Telken (n=3) y Columna (n=4) presentan los mismos haplotipos (Hap 4 y Hap6), mientras que la diversidad de haplotipos es mayor en la subcuenca que involucra a la laguna El Cervecero, el río Ecker y el río Pinturas (n=10; Hap 4, Hap 6, Hap 1, Hap 5 y Hap 9; Fig. 1B).
DISCUSIÓN
Los resultados que se presentan en este trabajo constituyen los primeros datos de diversidad genética para una población de visón americano asilvestrada en Sudamérica. Contrariamente a lo esperado para un efecto fundador en la población silvestre (i.e. baja variabilidad genética) se encontraron niveles similares de diversidad genética entre los grupos de visones provenientes de la población silvestre y la de cautiverio. Niveles de diversidad genética similares a los de poblaciones cautivas han sido encontrados en poblaciones asilvestradas de visones americanos en otras regiones del mundo. En estudios realizados sobre poblaciones silvestres de Polonia (Zalewski et al. 2011), la Península Ibérica (García et al. 2017) y Rusia (Korablev et al. 2017) se encontraron valores de diversidad genética elevados tal como ocurre en la población de Santa Cruz.
La alta variabilidad genética de las poblaciones invasoras del visón americano puede ser producto de los múltiples eventos de liberación o escape a partir de diversas poblaciones cautivas con variantes genéticas diferentes, dependiendo del origen de su stock. En el trabajo realizado por García et al. (2017), se encontró que los dos grandes linajes de visón americano presentes en su área de distribución nativa (América del Norte) se hallan representados en las poblaciones asilvestradas de la Península Ibérica. Se encontró un patrón similar para los individuos muestreados en este trabajo, que se asocian con dos linajes de la distribución original, indicando que los individuos invasores provienen de stocks diferentes. En ese mismo trabajo se postula que esta diversidad de variantes genéticas podría potencialmente conducir a un aumento del fitness y así aumentar la viabilidad de las poblaciones invasoras y la invasividad de la especie. La zona de la meseta del lago Buenos Aires, en la que se muestreó la población de este estudio, se encuentra equidistante de dos localidades donde existieron establecimientos peleteros de visón americano, que durante un tiempo prolongado pudieron haber sido fuente de individuos a través de escapes accidentales o intencionales. Se sabe que establecimientos peleteros en Chubut liberaron gran parte de sus animales a la naturaleza antes de cerrar (Jaksic et al. 2002) y su presencia en el noroeste de la provincia de Santa Cruz fue confirmada a principios de los 80 (Roesler et al. 2012). El origen de esta población asilvestrada posiblemente tenga dos fuentes. Una primera fuente pudo haber sido un antiguo criadero que funcionó en la localidad de Sarmiento, en el centro sur de la provincia de Chubut (Fasola et al. 2011). La segunda potencial fuente la constituyeron criaderos que funcionaron en la localidad de Coyhaique en Chile, que además trasladaron parte de los individuos al criadero de la localidad de Sarmiento (Jaksic et al. 2002). En base a la ecología de la especie y su dependencia de los sistemas acuáticos, aquellos movimientos a través de una red hidrográfica serán más probables. En otras áreas donde el visón es invasor se ha probado en base a técnicas moleculares que ciertas características del paisaje, como elevaciones, actúan como barreras mientras que los valles son facilitadores para el avance de individuos (Fraser et al. 2013). Dado que las posibles rutas para la llegada de visones al área de la población silvestre estudiada desde las fuentes propuestas no implican conexión directa a través de una red hidrográfica, ni el cruce de elevaciones o el avance por sistemas de valles o bajos, es adecuado pensar que el origen pudo ser mixto y que la llegada desde cada uno haya ocurrido a una velocidad diferente, pero desconocida. De esta forma, introducciones múltiples, una expansión poblacional rápida y la presencia de individuos de diferentes linajes podrían explicar el patrón genético observado. En general, las poblaciones invasoras del visón americano no se ajustan a la "paradoja genética” y su éxito podría explicarse en parte por la alta diversidad genética de los grupos asilvestrados.
Si bien los resultados de este trabajo son preliminares, pudimos observar una diferencia en la distribución de haplotipos entre las vías de acceso a la meseta, con una mayor diversidad de haplotipos en el sistema medio (laguna Cervecero, río Ecker y río Pinturas). Debido a que el número de individuos analizados por cada ruta de acceso es bajo, no fue posible comparar cuantitativamente la distribución de haplotipos entre las microcuencas y solo se realizó una comparación cualitativa.
El conocimiento de la diversidad genética de las poblaciones silvestres de visón americano en la región patagónica y el análisis de la distribución espacial de haplotipos proporcionan información sobre el origen de las poblaciones así como del movimiento de animales entre diferentes áreas, revelando barreras o desaceleradores de la dispersión. Actualmente, los programas de control de especies exóticas en general, y de esta especie en particular (Zalewski et al. 2009; Oliver et al. 2016), utilizan esta información para direccionar de manera más eficiente los esfuerzos de remoción en distintas cuencas hidrológicas. Con la obtención de un mayor número de muestras y una ampliación del tipo de marcadores genéticos estudiados se buscará profundizar el estudio de la población bajo control en la provincia de Santa Cruz, analizando la capacidad dispersiva y el movimiento de individuos entre subcuencas. Esto permitirá ajustar la estrategia de remoción dentro del "Programa de control del visón americano” en la meseta del lago Buenos Aires y replicar la experiencia en otras áreas de la región.
AGRADECIMIENTOS
Agradecemos al equipo y voluntarios del Proyecto Macá Tobiano involucrado en las tareas de campo (H. Casañas, P. Hernández, M. Figuera Tomas, D. Friedrich, L. Rosset, C. Ferreyra, J. Klavins, L. Pagano, P. Buchanan, N. Cossa, J.S. Verón, R. Lapido, entre otros). Personal de la Administración de Parques Nacionales, de la Fundación Flora y Fauna, del Consejo Agrario Provincial de Santa Cruz también asistieron en las tareas de campo. Agradecemos a las estancias del área que permitieron el acceso a los ríos: Sres. Pedro, Juan y Norma Garitaonandia de "La Vizcaína” y "Estancia San Carlos”, Ing. María Rosa Couto e hijos de "El Rincón”, Dr. Puricelli de "La Paloma”, Sr. M. García de "El Unco” y Sr. Cesar Castillo de "La Lucha”, Flia. Czetanovic de "El Correntoso”, Sr. Ericksen de "Manantiales”, Sr. Santiago de "La Madrugada” y el Sr. Sayhueque de "La Buitrera”. Esta es la publicación científica #15 del Proyecto Macá Tobiano.
LITERATURA CITADA
1. Barnett, J. M., S. Imberti, & I. Roesler. 2014. Distribution and habitat use of the Austral Rail Rallus antarcticus and perspectives on its conservation. Bird Conservation International 24:114-125. [ Links ]
2. Bonesi, L., & S. Palazon. 2007. The American mink in Europe: status, impacts, and control. Biological Conservation 134:470-483. [ Links ]
3. Bryce, R., M. K. Oliver, L. Davies, H. Gray, J. Urquhart, & X. Lambin. 2011. Turning back the tide of American mink invasion at an unprecedented scale through community participation and adaptive management. Biological Conservation 144:575-583. [ Links ]
4. Chehébar, C. E., A. Gallur, G. Giannico, M. D. Gottelli, & P. Yorio. 1986. A survey of the Southern River otter Lutra provocax in Lanin, Puelo and Los Alerces National Parks, Argentina, and evaluation of its conservation status. Biological Conservation 38:293-304. [ Links ]
5. Convenio Sobre Diversidad Biologica. 1992. <https://www.cbd.int/doc/legal/cbd-es.pdf> [ Links ]
6. Estoup, A., V. Ravigné, R. Hufbauer, R. Vitalis, M.Gautier, & B. Facon. 2016. Is there a genetic paradox of biological invasion? Annual Review of Ecology, Evolution, and Systematics 47:51-72. [ Links ]
7. Excoffier, L., & H. E. L. Lischer. 2010. Arlequin suite ver 3.5: A new series of programs to perform population genetics analyses under Linux and Windows. Molecular Ecology Resources 10:564-567. [ Links ]
8. Facon, B. et al. 2011. Inbreeding depression is purged in the invasive insect Harmonia axyridis. Current Biology 21:424-427. [ Links ]
9. Fasola, L., C. Chehébar, D. W. Macdonald, G. Porro, & M. H. Cassini. 2009. Do alien North American mink compete for resources with native South American river otter in Argentinean Patagonia? Journal of Zoology 277:187-195. [ Links ]
10. Fasola, L., J. Muzio, C. Chehébar, M. Cassini, & D.W. Macdonald. 2011. Range expansion and prey use of American mink in Argentinean Patagonia: dilemmas for conservation. European Journal of Wildlife Research 57:283-294. [ Links ]
11. Fasola, L., & I. Roesler. 2016. Invasive predator control program in Austral Patagonia for endangered bird conservation. European Journal of Wildlife Research 62:601-608. [ Links ]
12. Fasola, L., & I. Roesler. 2018. A familiar face with a novel behavior raises challenges for conservation: American mink in arid Patagonia and a critically endangered bird. Biological Conservation 218:217-222. [ Links ]
13. Fasola, L., & A. Valenzuela. 2014. Invasive carnivores in Patagonia: defining priorities for their management using the American mink (Neovison vison) as a case study. Ecología Austral 24:173-182. [ Links ]
14. Fraser, E. J., D. W. Macdonald, M. K. Oliver, S. Piertney, & X. Lambin. 2013. Using population genetic structure of an invasive mammal to target control efforts - An example of the American mink in Scotland. Biological Conservation 167:35-42. [ Links ]
15. Freeland, J. R. 2006. Molecular Ecology. P. 388, in: eLS. John Wiley & Sons. [ Links ]
16. García, K., Y. Melero, S. Palazón, J. Gosálbez, & J.Castresana. 2017. Spatial mixing of mitochondrial lineages and greater genetic diversity in some invasive populations of the American mink (Neovison vison) compared to native populations. Biological Invasions 19:2663-2673. [ Links ]
17. Genovesi, P. 2009. Invasive alien species in a changing world. Biodiversity 10:3-4. [ Links ]
18. Global Invasive Species Database. 2018. Species profile: Neovison vison. <http://www.iucngisd.org/gisd/speciesname/Neovison+vison on 18-07-2018. [ Links ]>
19. Gosálvez-Lara, L. F., & P. Díaz-Jiménez. 1992. Producción de visones. Hojas divulgadoras (España. Ministerio de Agricultura, Pesca y Alimentación). Instituto Nacional de Reforma y Desarrollo Agrario. n. 11/91. Madrid, España. 31pp. [ Links ]
20. Guichón, M. L., M. Monteverde, L. Piudo, J.Sanguinetti, & D. Martino. 2016. Mamíferos introducidos en la provincia de Neuquén: estado actual y prioridades de manejo. Mastozoología Neotropical 23:255-255. [ Links ]
21. Hall, T. 1999. BioEdit: a user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT. Nucleic Acids Symposium Series 41:95-98. [ Links ]
22. Hundertmark, K. J., G. F. Shields, I. G. Udina, R. T. Bowyer, A. A. Danilkin, & C. C. Schwartz. 2002. Mitochondrial phylogeography of moose (Alces alces): Late Pleistocene divergence and population expansion. Molecular Phylogenetics and Evolution 22:375-387. [ Links ]
23. Ibarra, J. T., L. Fasola, D. W. Macdonald, R. Rozzi, & C. Bonacic. 2009. Invasive american mink mustela vison in wetlands of the cape horn biosphere reserve, southern Chile: What are they eating? Oryx 43:87-90. [ Links ]
24. Jaksic, F. M., J. A. Iriarte, J. E. Jiménez, & D. R. Martínez. 2002. Invaders without frontiers: Cross-border invasions of exotic mammals. Biological Invasions 4:157-173. [ Links ]
25. Korablev, N. P., M. P. Korablev, P. N. Korablev, & N.O. Volkov. 2017. Polymorphism of the American mink (Neovison vison (Schreber, 1777)) populations inhabiting the Caspian-Baltic watershed inferred by means of mtDNA D-loop. Russian Journal of Biological Invasions 8:45-54. [ Links ]
26. Macdonald, D. W., & L. A. Harrington. 2003. The american mink: the triumph and tragedy of adaptation out of context. New Zealand Journal of Zoology 30:421-441. [ Links ]
27. Mack, R. N., D. Simberloff, W. M. Lonsdale, H. Evans, M. Clout, & F. A. Bazzaz. 2000. Biotic invasions: Causes, epidemiology, global consequences, and control. Ecological Applications 10:689-710. [ Links ]
28. Markert, J. A. et al. 2010. Population genetic diversity and fitness in multiple environments. BMC Evolutionary Biology 10:205. [ Links ]
29. Melero, Y., S. Palazón, L. Bonesi, & J. Gosàlbez. 2010. Relative abundance of culled and not culled American mink populations in northeast Spain and their potential distribution: Are culling campaigns effective? Biological Invasions 12:3877-3885. [ Links ]
30. Miller, S. A., D. D. Dykes, & H. F. Polesky. 1988. A simple salting out procedure for extracting DNA from human nucleated cells. Nucleic Acids Research 16:1215. [ Links ]
31. Nei, M. 1973. Analysis of gene diversity in subdivided populations. Proceedings of the National Academy of Sciences USA 70:3321-3323. [ Links ]
32. Oliver, M. K., S. B. Piertney, A. Zalewski, Y. Melero, & X. Lambin. 2016. The compensatory potential of increased immigration following intensive American mink population control is diluted by male-biased dispersal. Biological Invasions 18:3047-3061. [ Links ]
33. Parisod, C., C. Trippi, & N. Galland. 2005. Genetic variability and founder effect in the pitcher plant Sarracenia purpurea (Sarraceniaceae) in populations introduced into Switzerland: from inbreeding to invasion. Annals of Botany 95:277-286. [ Links ]
34. Peris, S. J., J. Sanguinetti, & M. Pescador. 2009. Have Patagonian waterfowl been affected by the introduction of the American mink Mustela vison? Oryx 43:648-654. [ Links ]
35. Porteus, T., M. Short, S. Richardson, & J. Reynolds. 2012. Empirical development of strategy for the control of invasive American mink by trapping. European Journal of Wildlife Research 58:403-413. [ Links ]
36. Rambaut, A., & A. J. Drummond. 2007. Tracer v. 1.4. <http://beast.bio.ed.ac.uk/Tracer. [ Links ]>
37. Reed, D. H., & R. Frankham. 2003. Correlation between fitness and genetic diversity. Conservation Biology 17:230-237. [ Links ]
38. Roesler, I., S. Imberti, H. Casañas, & N. Volpe. 2012. A new threat for the globally endangered hooded grebe Podiceps gallardoi: the American mink Neovison vison. Bird Conservation International 22:383-388. [ Links ]
39. Ronquist, F., & J. P. Huelsenbeck. 2003. MrBayes 3: Bayesian phylogenetic inference under mixed models. Bioinformatics Applications Note 19:1572-1574. [ Links ]
40. Rozas, J. et al. 2017. DnaSP 6: DNA sequence polymorphism analysis of large data sets. Molecular Biology and Evolution 34:3299-3302. [ Links ]
41. Rozzi, R., & M. Sherriffs. 2003. El visón (Mustela vison Schreber, Carnivora: Mustelidae), un nuevo mamífero exótico para la Isla Navarino. Anales del Instituto de la Patagonia 31:97-104. [ Links ]
42. Schmitz, D. C., & D. Simberloff. 1997. Biological invasions: A growing threat. Issues in Science and Technology 13:33-40. [ Links ]
43. Schrieber, K., & S. Lachmuth. 2016. The Genetic Paradox of Invasions revisited: The potential role of inbreeding × environment interactions in invasion success. Biological Reviews 92:939-952. [ Links ]
44. Schwartz, M. K., G. Luikart, & R. S. Waples. 2007. Genetic monitoring as a promising tool for conservation and management. Trends in Ecology and Evolution 22:25-33. [ Links ]
45. Sepúlveda, M. A., R. S. Singer, E. A. Silva-Rodríguez, A. Eguren, P. Stowhas, & K. Pelican. 2014. Invasive American mink: Linking pathogen risk between domestic and endangered carnivores. EcoHealth 11:409-419. [ Links ]
46. Simberloff, D. 2001. Eradication of island invasives: Practical actions and results achieved. Trends in Ecology and Evolution 16:273-274. [ Links ]
47. Valenzuela, A. E. J, A. Raya Rey, L. Fasola, R. A. Sáenz Samaniego, & A. Schiavini. 2013. Trophic ecology of a top predator colonizing the southern extreme of South America: feeding habits of invasive American mink (Neovison vison) in Tierra del Fuego. Mammalian Biology 78:104-110. [ Links ]
48. Valenzuela, A. E. J, M. A. Sepúlveda, & J. L. Cabello. 2016. Mamíferos exóticos invasores el visón americano en Patagonia: un análisis histórico y socioecológico de la investigación y el manejo. Mastozoología Neotropical 23:289-304. [ Links ]
49. Zalewski, A., A. Michalska-Parda, M. Ratkiewicz, M.Kozakiewicz, M. Bartoszewicz, & M.Brzeziński. 2011. High mitochondrial DNA diversity of an introduced alien carnivore: comparison of feral and ranch American mink Neovison vison in Poland. Diversity and Distributions 17:757-768. [ Links ]
50. Zalewski, A., S. B. Piertney, H. Zalewska, & X. Lambin. 2009. Landscape barriers reduce gene flow in an invasive carnivore: geographical and local genetic structure of American mink in Scotland. Molecular Ecology 18:1601-1615. [ Links ]
51. Zalewski, A., H. Zalewska, S. G. Lunneryd, C. Andre, & G. Mikusinski. 2016. Reduced genetic diversity and increased structure in American mink on the Swedish coast following invasive species control. PLOS ONE 11:e0157972. Public Library of Science. [ Links ]














