Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Revista de la Sociedad Entomológica Argentina
versión impresa ISSN 0373-5680
Rev. Soc. Entomol. Argent. vol.73 no.1-2 La Plata jun. 2014
FORO
Avances tecnológicos y sus aplicaciones en la cibertaxonomía
Technological advances and its applications in cybertaxonomy
Cigliano, María Marta, Martina E. Pocco & Hernán L. Pereira
Centro de Estudios Parasitológicos y de Vectores (CEPAVE), CCT La Plata, CONICET División Entomología, Museo de La Plata, Universidad Nacional de La Plata, Argentina. E-mail: cigliano@fcnym.unlp.edu.ar
RESUMEN. Los rápidos avances en la ciber-infraestructura, la bioinformática, la fotografía digital y las tecnologías de la información contribuyen a acceder fácilmente al conocimiento taxonómico y permiten generar información a una escala acorde con las necesidades de la crisis de la biodiversidad. El conocimiento taxonómico básico, como literatura taxonómica, catálogos de nombres científicos, registros de ejemplares, localización de ejemplares tipo en colecciones biológicas, y claves interactivas de varios grupos de organismos ya están disponibles a través de Internet. En este artículo, se analiza cómo la información taxonómica, principalmente la entomológica, es presentada a la comunidad en Internet; se discuten las diferentes formas en que la taxonomía ya ha hecho uso de este medio o podrá hacerlo en un futuro cercano, y cómo los actuales medios de compartir datos, especialmente a través de Internet, están siendo utilizados para hacer más eficientes las tareas taxonómicas, realzar las publicaciones y mejorar los vínculos con los usuarios.
PALABRAS CLAVE: Portales de biodiversidad; Cibertaxonomía; Colecciones entomológicas.
ABSTRACT. The rapid advances in cyber-infrastructure, bioinformatics, digital imaging and related information technologies facilitate the access to taxonomic data and allow generating new information on a scale appropriate to meet the decline in biodiversity. Basic taxonomic information, such as taxonomic literature, catalogues, specimen-level data, location of types, and interactive keys are already available, at least for some groups, on the Web. In this article we explore the modern means of disseminating taxonomic information, mostly entomological information, in the Internet; we review some of the different ways taxonomists have so far made use of the Web or will be able to do so in the near future, and how modern means of sharing data, especially via the Internet, are being used to streamline taxonomic work and improve links with end users.
KEY WORDS: Biodiversity portals; Cybertaxonomy; Insect collections.
INTRODUCCIÓN
La disponibilidad y fácil accesibilidad a las modernas tecnologías sumada a la necesidad de conocer y describir la biodiversidad que nos rodea, hacen que este sea un momento propicio para revitalizar los estudios de morfología comparada y de taxonomía. Los rápidos avances en la ciber-infraestructura, la bioinformática, la fotografía digital y las tecnologías de la información contribuyen a acceder fácilmente al conocimiento taxonómico y permiten generar información a una escala acorde con las necesidades de la crisis de la biodiversidad (Wilson, 1992; Raven, 1997; Wheeler et al., 2012a).
El conocimiento taxonómico básico, como literatura taxonómica digitalizada, catálogos de nombres científicos, registros de ejemplares, localización de ejemplares tipo en colecciones biológicas, y claves interactivas de varios grupos de organismos ya están disponibles a través de Internet (Godfray et al., 2007). La digitalización y disponibilidad de la información de biodiversidad a través de varias iniciativas globales como "Species2000/Catalogue of Life" (CoL), "Encyclopedia of Life" (EOL), "Global Biodiversity Information Facility" (GBIF), "Biodiversity Heritage Library" (BHL), "Biodiversity Heritage Library-Europe" (BHL-Europe) cobra cada vez mayor importancia y volumen, existiendo en la actualidad más de 680 proyectos de bases de datos de biodiversidad disponibles en Internet (http://www.tdwg.org/biodiv-projects/). Asimismo, la taxonomía está llamada a jugar un rol fundamental en la "Nueva Biología" ("New Biology") (Kelling et al., 2009; NRCNA, 2009). En esta concepción, la biología se incorpora al conjunto de disciplinas de las ciencias que se basan en el análisis de un gran volumen de datos ("data-intensive sciences") (Newman et al., 2003; Michener & Jones, 2012). Las disciplinas de datos masivos permiten el análisis de sistemas complejos de manera eficiente y se complementan con el método científico de observación, generación de hipótesis y experimentación para mejorar la comprensión de la naturaleza (Kelling et al., 2009). Esta nueva visión de la biología, implica la obtención, revisión y análisis de datos masivos a partir de diversas fuentes, que incluyen tanto técnicas moleculares, tecnologías ambientales y de la digitalización a gran escala, así como también las observaciones y/o registros de datos originales (Kelling et al., 2009). También existe una presión cada vez mayor para que los datos científicos estén disponibles y de libre acceso en Internet (Penev et al., 2010). Por lo tanto, los servicios de Web que hacen uso de nombres científicos para organizar y catalogar la información sobre los organismos, serán una pieza esencial en la "Nueva Biología" (Patterson et al., 2010; Patterson, 2014).
La cibertaxonomía o "etaxonomy", también llamada "Web-based taxonomy", es una manera de integrar herramientas electrónicas estandarizadas, ciber-infraestructura, informática e ingeniería informática a la taxonomía tradicional (Wheeler, 2007, 2008, 2010; Page, 2008). La forma en que los conocimientos taxonómicos son documentados, consultados y utilizados está cambiando con la cibertaxonomía (Schram, 2004; Padial et al., 2010). Más aún, es probable que en el futuro exista una taxonomía interactiva que permita la descripción y publicación de nuevos taxones en Internet, facilitando la actualización dinámica de los conocimientos taxonómicos, de libre acceso desde cualquier lugar (Godfray, 2002, 2005; Godfray et al., 2007).
En este artículo exploramos cómo la información taxonómica, principalmente la entomológica, es presentada a la comunidad en Internet; luego revisamos las diferentes formas en que la taxonomía ya ha hecho uso de este medio o podrá hacerlo en un futuro cercano, y cómo las modernas tecnologías para compartir datos están siendo utilizadas para hacer más eficientes las tareas taxonómicas y mejorar los vínculos con los usuarios.
Información taxonómica disponible en Internet (Web)
Información de ejemplares
Las colecciones biológicas contienen muestras de la biodiversidad del mundo y representan una fuente de información fundamental para la investigación básica y para las ciencias aplicadas del medio ambiente (Smith & Blagoderov, 2012). Los ejemplares de colección y sus datos asociados (metadatos) representan un vasto depósito de información sobre la biodiversidad, los ecosistemas y los recursos naturales. El universo de datos de las colecciones biológicas ha sido estimado entre 1,5x109 y 3x109 ejemplares (Ariño, 2010; Smith & Blagoderov, 2012). La digitalización de esa información es una actividad que los museos e instituciones académicas reconocen como el mejor medio para "sacar de la oscuridad" las colecciones biológicas y ponerlas a disposición de un público más amplio (Smith & Blagoderov, 2012). El valor de la información digitalizada de los ejemplares de colección depende, principalmente, de la calidad de la identificación taxonómica y de la cantidad y calidad de datos asociados a ellos. Generalmente, la fecha, la localidad y el colector son registrados junto con la información taxonómica del ejemplar, siendo más valioso aún si los datos de procedencia han sido georreferenciados. Numerosos museos y colecciones tienen proyectos que contemplan la digitalización de sus ejemplares tipo y de los materiales de colección, o bien presentan catálogos de tipos electrónicos disponibles en Internet (Godfray et al., 2007).
La informatización de colecciones biológicas responde muchas veces a emprendimientos gubernamentales, como por ejemplo el Sistema Nacional de Datos Biológicos de la Argentina (SNDB), que es una iniciativa del Ministerio de Ciencia, Tecnología e Innovación Productiva (MINCyT) conjuntamente con el Consejo Interinstitucional de Ciencia y Tecnología (CICyT). El SNDB tiene como misión conformar una base de datos unificada de información biológica, a partir de datos taxonómicos, ecológicos, cartográficos, bibliográficos, y de catálogos sobre recursos naturales. Las colecciones entomológicas de las principales instituciones académicas argentinas contribuyen con el SNDB y sus datos se hacen públicos a través del portal del sistema, que a su vez presenta la misma plataforma que GBIF y contribuye con sus datos biológicos al mismo. La informatización de colecciones del SNDB se implementa siguiendo la técnica tradicional de digitalización de colecciones de insectos, que consiste en ingresar manualmente la información de las etiquetas de los ejemplares en una base de datos y asignarle un identificador global único ("Globally unique identifier"). Los datos de las colecciones son provistos a SNDB siguiendo el estándar Darwin Core (ver glosario).
Varias colecciones ponen a disposición en Internet imágenes digitales de alta resolución de los ejemplares tipo que alojan. Este servicio presenta una gran ventaja ya que puede obviar la necesidad de enviar materiales en préstamo, evitando los riesgos asociados a la movilización de los mismos (Hauser et al., 2005; Lampe et al., 2005).
Un enfoque diferente, pero que también permite el acceso a imágenes digitales de ejemplares tipo es a través del uso de la telemicroscopía ("telemicroscopy"). La telemicroscopía permite examinar, manipular y fotografiar ejemplares desde un acceso remoto a través de una conexión de red. Tres prototipos de estos microscopios digitales, ROBOT ("Remotely Operable Bench-marker Of Types"), operados a distancia son utilizados en las grandes colecciones entomológicas del "Smithsonian Institution", Washington, DC, "Natural History Museum", Londres y el "Museum National d'Histoire Naturelle", Paris, con muy buenos resultados (Wheeler et al., 2012b).
Otros proyectos como "Sepsidnet" (http://sepsidnet-rmbr.nus.edu.sg/) desarrollan colecciones digitales de referencia de un grupo determinado de insectos, en este caso los Sepsidae (Diptera) mundiales. Esta base de datos en línea contiene imágenes de alta calidad de caracteres diagnósticos que han sido obtenidas a partir de ejemplares tipo depositados en diferentes colecciones globales (Ang et al., 2013) mientras que otros emprendimientos ponen a disposición en Internet imágenes de insectos preservados en portaobjetos. Utilizando cámaras fotográficas adosadas a microscopios confocales, equipados con platinas motorizadas y mediante programas especializados, se componen mosaicos de fotos de alta resolución y aumento tomadas en sucesivos planos focales. Las imágenes así compuestas permiten la generación de una imagen virtual en tres dimensiones de los ejemplares ("ciber-ejemplares" o "ejemplares virtuales") (Favret, 2013; Ang et al., 2013). Otros sistemas ("Natural-colour") permiten generar modelos en 3D respetando el color natural de los insectos (entre 3 mm a 30 mm de longitud) fotografiados desde todos los ángulos posibles (Nguyen et al., 2014).
La digitalización de ejemplares presenta ciertas desventajas relacionadas con el insumo de tiempo de registro de los datos y con el peligro que representa, para una buena conservación, la manipulación de los materiales. Para solucionar estas desventajas, varios proyectos han aprovechado los nuevos desarrollos en el campo de la digitalización y fotografías digitales que aceleran estos procesos y permiten la digitalización de colecciones completas de insectos preservados en alfiler (Smith & Blago-derov, 2012). Tales emprendimientos emplean un enfoque "industrial", que automatizan las tareas, reduciendo el registro de metadatos e incorpora la "digitalización masiva" (Blagoderov et al., 2012). La "digitalización masiva" consiste en escanear directamente los cajones, cajas o bandejas de colección que contienen los ejemplares. Uno de los primeros proyectos que utilizó este tipo de enfoque toma fotos panorámicas, de altísima resolución ("gigapixel"), de los cajones de colecciones entomológicas que pueden contener cientos de ejemplares. Este proyecto utiliza el GigaPan (www.gigapan.com), que fue desarrollado inicialmente a partir de la colaboración entre "Carnegie Mellon University" y el grupo Ames de Robótica Inteligente de la NASA para uso en los Rovers de la NASA en Marte, y está siendo utilizado en la colección entomológica de "North Carolina State University Insect Museum" (Bertone et al., 2012). El sistema GigaPan obtiene imágenes de cientos de megapíxeles a giga píxeles (miles de millones de píxeles) que son utilizadas para generar fotos panorámicas de gran navegabilidad. Se trata de un robot equipado con una cámara digital montada sobre un trípode. El robot posiciona la cámara para capturar imágenes individuales del área de interés y utiliza un "dedo" robótico que se acopla a la cámara y va capturando múltiples imágenes. El software GigaPan se utiliza luego para unir las fotos parciales en una gran panorámica que tiene una resolución semejante a la resolución de cada imagen individual pero cubriendo un área mayor (Bertone et al., 2012). Más recientemente SatScan, desarrollado en asociación con el "Natural History Museum", Londres (Blagoderov et al., 2012), y actualmente en uso en la "Australian National Insect Collection" (Mantle et al., 2012), ha permitido obtener estas fotos panorámicas ("panoramas") con una distorsión mínima. SatScan presenta un software asociado que permite seleccionar y registrar imágenes de especímenes individuales. Esta técnica de escaneo de cajón ("tray scanning system") se ha incorporado como parte del programa de digitalización del proyecto Invert-Net de EEUU (Dietrich et al., 2012) y ha dado lugar a un nuevo instrumento, de menor costo, DScan (Schmidt et al., 2012). InvertNet es parte del programa "Advancing Digitization of Biological Collections (ADBC)" de la "U.S. National Science Foundation" cuyo fin es proveer el acceso digital a unos 60 millones de ejemplares depositados en 22 colecciones de artrópodos distribuidas en el centro-oeste de EEUU.
Estas nuevas implementaciones también tienen ciertas desventajas, principalmente relacionadas con los errores que pueden acarrear una mala identificación de los ejemplares, más allá de los costos de los "scanners". Por lo tanto, no son pocos los museos e instituciones que consideran que solo aquellas partes bien curadas de las colecciones son las que deben ser digitalizadas (Haston et al., 2012; Mantle et al., 2012; Tulig et al., 2012; Ang et al., 2013).
Catálogos taxonómicos
Elaborar un catálogo de nombres científicos es una de las tareas más relevantes que un sistemático puede llevar a cabo para facilitar la investigación futura del grupo en estudio. Los catálogos y las "checklists" suelen ser fácilmente adaptables a la Web y por lo tanto existen numerosos proyectos que brindan tales recursos en Internet. Los catálogos varían de acuerdo a sus contenidos, algunos son solo listas de nombres, mientras que otros presentan información completa sobre las sinonimias, citas bibliográficas, ubicación de ejemplares tipo, e incluso información acerca de la distribución, ecología de las especies y claves interactivas. También difieren en el alcance geográfico, algunos son regionales (Cuadro 1), mientras que otros son mundiales.
Cuadro 1. Ejemplo de catálogos regionales
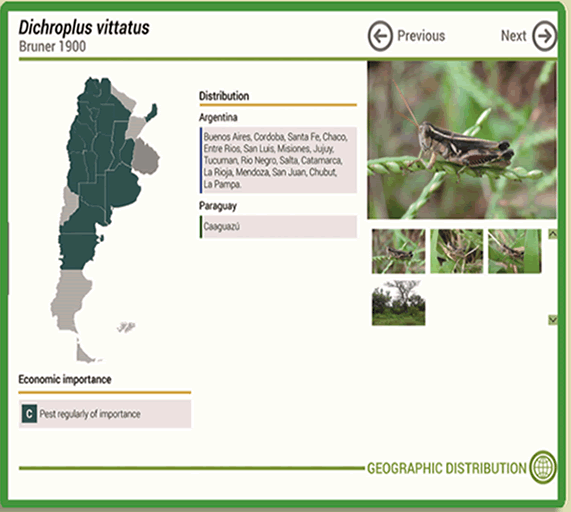
"Especies de Acridomorfos (Orthoptera) de Argentina y Uruguay/Acridomorph (Orthoptera) species of Argentina and Uruguay" de Carbonell et al. (2006), es una lista de referencia bilingüe (castellano/inglés) que contiene la clasificación, nombres de géneros, especies y subespecies. Para cada especie se brindan las sinonimias, la distribución geográfica, las principales plantas hospedadoras, su importancia económica, observaciones de interés y las citas bibliográficas más relevantes (Fig. 1). Las especies se ilustran con fotos de los ejemplares macho, hembra y del hábitat, si fueron tomadas en el campo (Fig. 2).Presenta diferentes posibilidades de búsquedas (geográfica, clasificación, alfabética). Este catálogo ha sido publicado en formato de CD-ROM. En la actualidad y aprovechando tecnologías actuales se está realizando una nueva versión, actualizada y con un nuevo diseño, que se accederá desde Internet o podrá ser utilizada en una tableta directamente en el campo. Las ilustraciones corresponden a la nueva versión.
Catálogos globales
"Species2000" es una federación autónoma de curadores de bases de datos taxonómica cuyo objetivo es cotejar y validar una "checklist" de todas las especies mundiales. Esto se logra a partir de la organización de bases de datos de especies globales que cubren cada uno de los mayores grupos de organismos (por ejemplo OSF (Orthoptera Species File, ver más abajo). La clasificación resultante es utilizada por "Global Biodiversity Information Facility" (GBIF) y por la "Encyclopedia of Life" (EoL), entre otros "portales agregadores" de datos de biodiversidad.
"Integrated Taxonomic Information System" (ITIS) es una asociación de agencias federales y otras organizaciones de los Estados Unidos, Canadá y México. La base de datos ITIS es una referencia automatizada de nombres científicos y comunes principalmente de la biota de América del Norte. ITIS contiene más de 762.000 nombres científicos y comunes de todos los reinos.
"Catalogue of Life" se crea en 2001 como resultado de la colaboración entre las organizaciones "Species2000" e ITIS. "Catalogue of Life" es el catálogo mundial de especies más completo que existe en la actualidad. Está constituido por una lista única de especies y su clasificación. "Catalogue of Life" contiene información esencial sobre los nombres, las relaciones y la distribución de más de 1,5 millones de especies, cubriendo el 70% de la diversidad mundial. Esta cifra aumenta a medida que la información va siendo compilada por las diversas instituciones académicas, organizaciones y portales proveedores de datos que contribuyen con más de 130 bases de datos taxonómicas y/o catálogos de autoridad, por ejemplo, OSF.
"Encyclopedia of Life" (EOL) tiene por objetivo poner a disposición del público el acceso global al conocimiento de la vida en la Tierra (Wilson, 2003). La "Encyclopedia of Life" (EOL) es un compendio en línea de acceso libre cuyo fin es documentar las más de 1.9 millones de especies conocidas para la ciencia. Se compila a partir de bases de datos existentes y de la contribución de expertos profesionales y amateurs. Su objetivo es construir una página por especie que contenga la información disponible de la misma, incluyendo fotos, videos, grabaciones, texto, gráficos, etc. Asimismo, EOL incorpora automáticamente enlaces a páginas de la literatura contenida en la "Biodiversity Heritage Library" donde aparecen los nombres científicos, a través de las herramientas de inteligencia taxonómica que localizan, verifican y registran estos nombres (EOL, 2014).
"Biodiversity Heritage Library" (BHL). Un consorcio de bibliotecas de los mayores museos de ciencias naturales e instituciones de investigación, principalmente de los EEUU, se constituyó para establecer la BHL. Las instituciones participantes tienen más de dos millones de volúmenes de bibliografía sobre biodiversidad, reunida durante más de 200 años de existencia. En colaboración con "Internet Archive" y a través del propio esfuerzo, la BHL ha digitalizado millones de páginas de literatura taxonómica, representando decenas de miles de títulos en más de 134.000 volúmenes. La bibliografía sobre la diversidad biológica tiene una distribución global limitada, debido a que la mayoría se encuentra disponible solo en algunas bibliotecas del mundo (Agosti & Johnson, 2002; Agosti, 2006). Estas colecciones son de un valor excepcional debido a que la taxonomía biológica depende, en gran medida, de la bibliografía histórica. La BHL trabaja con la finalidad de que la literatura sobre biodiversidad expanda su disponibilidad al mundo entero.
"Biodiversity Heritage Library for Europe" (BHL-Europe). Este emprendimiento está constituido por 28 museos de historia natural, jardines botánicos e instituciones de iniciativas de biodiversidad de Europa. El mismo tiene entre otros objetivos, poner a disposición del público la información europea sobre diversidad biológica que contienen dichas instituciones. La información está disponible a través de su propio portal como así también se encuentra disponible a través de la Biblioteca Digital Europea - EUROPEANA (www.europeanaconnect.eu).
"Zoobank" y el registro de nombres zoológicos. Antes de que existiera Internet, diversas instituciones proveían resúmenes de los trabajos taxonómicos publicados, incluyendo y registrando las descripciones de especies y de taxones. "Zoological Record" es la base de datos mundial de biología animal más antigua. Está considerada como la principal referencia taxonómica mundial, con una cobertura que se remonta a 1864. Esta base de datos ha actuado como el registro no oficial de los nombres de animales. En la actualidad se sustenta en la plataforma "Thomson Reuters Web of Science" y continua siendo un servicio comercial por suscripción al que se accede a través de la Web. La posibilidad de hacer obligatorio el registro de nombres de nuevos taxones ha sido reiteradamente discutida, pero hasta el presente no existe unanimidad de criterios al respecto dentro de la comunidad zoológica. Sin embargo, Zoobank ha sido propuesto como portal administrativo para el registro no obligatorio de nombres zoológicos y actos nomenclaturales, de trabajos publicados y de registro de autores (Polaszek et al., 2005; Acosta, 2007; Pyle & Michel, 2008). Recientemente, la "International Commission on Zoological Nomenclature (ICZN)" ha extendido y detallado los métodos de publicación permitidos por el Código, particularmente aquellos relacionados a la publicación electrónica, estableciendo a Zoobank como Registro Oficial de Nomenclatura Zoológica (ICZN, 2012). Esta nueva enmienda permite la publicación de actos nomenclaturales en revistas en formato electrónico siempre y cuando los trabajos se registren previamente en Zoobank.
Catálogos de autoridad entomológicos
Existen numerosos catálogos entomológicos disponibles en Internet. Varios de ellos responden, en cierta forma, al concepto de "unitary taxonomy" (Godfray 2002, 2007) que consiste en reunir todos los recursos relevantes a la sistemática de un grupo en particular en un único sitio Web que sirva como referente para la comunidad de especialistas del grupo. Los distintos portales ofrecen diferentes tipos de información según las necesidades y características de los taxones.
"Antweb" es una base de datos mundial que contiene la clasificación, imágenes, registros de ejemplares, mapas, e información de la historia natural de las 15.680 especies y subespecies conocidas de hormigas. El servidor está localizado en "The California Academy of Sciences" y financiado por donaciones privadas y proyectos de la "US National Science Foundation" (Antweb, 2014).
"Mosquito Taxonomic Inventory (MTI)" es una base de datos que es actualizada con nueva información en forma continua y curada por especialistas, abarca la diversidad global de la familia Culicidae (más de 3.500 especies) con énfasis en la taxonomía y sistemática. Este sitio presenta la clasificación, bibliografía, información filogenética y un glosario de la anatomía del grupo (Harbach, 2013). Este sitio se sustenta en Scratchpads (http://scratchpads.eu/), que es un sistema de programas de publicación de datos que se utiliza para construir, compartir y administrar información sobre la diversidad biológica (Smith et al., 2009).
"Catalogue of the Craneflies of the World" (CCW). Este catálogo, cuyo autor es Pjotr Oosterbroek ("Zoological Museum of the University of Amsterdam") abarca géneros y especies de las familias Pediciidae, Limoniidae, Cylindrotomidae y Tipulidae (Insecta, Diptera, Tipuloidea). Además de brindar la información taxonómica, datos biológicos y bibliografía, esta base de datos incluye información actualizada sobre la distribución de las especies.
"Orthoptera Species File" (OSF) brinda un espacio en Internet que reúne la información taxonómica existente sobre el orden Orthoptera (Cigliano & Eades, 2010; Eades et al., 2014) y cuenta con datos completos sobre la taxonomía y las sinonimias de más de 25.370 especies válidas, citas y referencias bibliográficas, fotos de ejemplares de colección (incluyendo tipos) y de caracteres diagnósticos y de ejemplares en su hábitat, grabaciones de sonidos, registros de especímenes y mapas de distribución geográfica, así como claves de identificación para los taxones. También provee LSIDs para los nombres de taxones.
OSF se fundó a partir del catálogo mundial "Orthoptera Species File", cuyos volúmenes fueron publicados entre 1994 y 2000 por Daniel Otte de la "Academy of Natural Sciences", Filadelfia, EEUU. A partir de la colaboración con Piotr Naskrecki del "Museum of Comparative Zoology, Harvard University", EEUU, surgió la primera versión de OSF Online, publicada en Internet en 1997. En 1999, David Eades ("Illinois Natural History Survey" INHS, University of Illinois at Urbana-Champaign", EEUU) realizó un nuevo diseño y programación, se mejoró la interface con el público, se facilitó la edición de la misma permitiendo el ingreso de datos aplicando en forma automatizada las reglas del Código Internacional de Nomenclatura Zoológica, evitando así la incorporación de errores de nomenclatura. A partir de ese momento, el perfeccionamiento del conjunto de programas sobre el que se estructura OSF se realiza en forma permanente. Esto ha llevado a que en la actualidad esta base de datos se asiente sobre un sofisticado sistema de programas: "Species File Software" (http://software.speciesfile.org) utilizado en otros grupos de insectos (Blattodea, Mantodea, Phasmida, Plecoptera, Coreoidea, Aphidomorpha, Lygaeoidea, entre otros). El desarrollo de programas así como el servidor de OSF se encuentran en el INHS. Desde marzo 2010, la actualización, edición de datos y parte de la programación se realiza en la División de Entomología del Museo de La Plata y en el CEPAVE, UNLP.
Un nuevo desarrollo que implicará la migración a una nueva plataforma integrando "Species File Software" con mx (desarrollado por Matthew J. Yoder, INHS, es un programa que permite, entre otras funcionalidades, crear y desarrollar ontologías anatómicas que pueden ser exportadas en formato OBO, ver Cuadro 2) y 3i ("Internet-accesible Interactive Identification", desarrollado por Dmitry Dmitriev, INHS, programa para crear claves interactivas en línea y revisiones taxonómicas virtuales). El objetivo principal de este nuevo desarrollo ("Taxon Works") es la construcción de un nuevo ambiente de trabajo que permitirá replicar las funciones de "Species File Software" expandiendo su alcance y simplificando su uso.
Cuadro 2. Ontologías morfológicas
Las ontologías representan dominios de conocimiento formalizados, donde los conceptos están relacionados entre sí de una manera lógica (Yoder et al., 2010; Deans et al., 2012a). A través de estos modelos se pueden clarificar y definir conceptos y relaciones dentro de un dominio de interés, a fin de lograr un vocabulario unificado (Yoder et al., 2010), (Fig. 3). Numerosas ontologías han sido desarrolladas en el campo de las ciencias biológicas, disponibles desde el repositorio OBO ("Ontological Biomedical Ontologies", http://obo.sourceforge.net/). Dentro de los artrópodos, se han desarrollado ontologías morfológicas para Drosophila [Drosophila Gross Anatomy Ontology (FBbt), Costa et al., 2013)], mosquitos [Mosquito Gross Anatomy Ontology (TGMA) (Topalis et al., 2008)], himenópteros (Hymenoptera Anatomy Ontology, HAO) (Yoder et al., 2010), garrapatas [Tick Gross Anatomy (TADS) (Topalis et al., 2008)] y arañas [Spider Ontology (SPD) (Ramírez et al., 2007)]. Esta última ha sido utilizada como base para vincular imágenes a matrices de datos filogenéticas (Ramírez et al., 2007). La ontología de himenópteros (HAO) fue construida usando las interfaces dentro de mx, software de acceso abierto disponible en la Web, que, entre otras funcionalidades, permite que los especialistas contribuyan simultáneamente al desarrollo de una ontología anatómica (Yoder et al., 2010).
A partir de conceptos incluidos en las ontologías, es posible crear fenotipos semánticos ("semantic phenotypes") que constituyen descripciones estructuradas que representan observaciones del fenoma, en un modelo que facilita el análisis computacional (Mikó & Deans, 2009; Mungall et al., 2010; Dahdul et al., 2010; Deans et al., 2012a; Balhoff et al., 2013). En el campo de la taxonomía, una descripción en lenguaje natural puede ser convertida en una descripción semántica, usando el lenguaje OWL, y basada en el modelo conceptual EQ ("Entity-Quality") (Washington et al., 2009), combinando términos de múltiples ontologías (por ejemplo: HAO, PATO, RO), (Mungall et al., 2010; Mullins et al., 2012; Mungall et al., 2012; Mabee et al., 2012; Balhoff et al., 2013). Por ejemplo, la descripción en lenguaje natural: "Head color" "orange" se convierte en la descripción semántica: "has_part some (head and (is bearer of some orange))" (Mullins et al., 2012).

Fig. 3. Modelo simplificado de los tipos de datos en HAO ("Hymenoptera Anatomy Ontology") modificado de Yoder et al. (2010). Término ("Label"); Concepto ("Class"); Referencia ("Reference"); Comentario ("Tag"); Relación entre conceptos ("Class relation").
Utilización de herramientas cibertaxonómicas
Nuevas funcionalidades permitieron que OSF pueda ser utilizada para el desarrollo de estudios taxonómicos ya que se pueden clonar secciones definidas de la clasificación y hacerlas privadas ("Private Species File") en Internet para usuarios autorizados (Cigliano & Eades, 2010). De esta manera los colaboradores que trabajan en el mismo grupo taxonómico, pueden actualizar la información del taxón a medida que se va desarrollando la investigación. Asimismo, es posible incluir enlaces a OSF en el manuscrito. Una vez que el trabajo es publicado, la "Private Species File" se combina con la base de datos original quedando la información definitivamente incorporada en OSF. De esta manera queda a disponibilidad del público información adicional a la publicada (por ej., imágenes de diferentes morfos o del sexo opuesto al publicado, de distintas estructuras y caracteres), y el trabajo se realza con enlaces a imágenes de ejemplares tipo, a claves pictóricas interactivas, a grabaciones de sonidos, a mapas que se visualizan a través de "Google Maps".
Así como OSF brinda un espacio virtual de colaboración entre taxónomos para trabajar en tiempo real en Internet, a través de las "Private Species Files", también existen otras plataformas para desarrollar estudios taxonómicos en la Web. Algunos proyectos europeos cuyo objetivo es apoyar el desarrollo de las comunidades de investigación virtuales vinculadas a la ciencia de la biodiversidad, proporcionando un marco integrado para usuarios e investigadores que hacen uso y contribuyen con datos de biodiversidad a la Web, han desarrollado la plataforma para la Cibertaxonomía (http://cybertaxonomy.eu/blog/platform/) del programa EDIT ("European Distributed Institute of Taxonomy") y Scratchapds (http://scratchpads.eu/) del proyecto VIBRANT (http://vbrant.eu/). Estos proyectos están incluidos dentro de la asociación europea CETAF ("Consortium of European Taxonomic Facilities" http://www.cetaf.org/). Esta Asociación constituida por 29 museos de historia natural y jardines botánicos europeos tiene como objetivo asegurar la continuidad de los programas antes mencionados y de otros que faciliten el acceso a la información biológica de las colecciones y datos asociados como "Biodiversity Heritage Library-Europe" (http://www.bhl-europe.eu/). En los EEUU la "US National Science Foundation" subvenciona varios programas como Morph-bank (http://www.morphbank.net/) que permite almacenar, debatir y compartir imágenes detalladas de caracteres y ejemplares y Morphobank (http://www.morphobank.org/) que es una aplicación web para realizar estudios filogenéticos basados en morfología. Esta plataforma permite que un equipo de investigadores pueda trabajar en Internet en tiempo real desplegando matrices filogenéticas dinámicas de caracteres morfológicos relacionadas con imágenes que ilustran las homologías.
Otras alternativas van más allá, y proponen establecer una base de datos ("Phenobank") que almacene descripciones semánticas estandarizadas de ejemplares, que puedan ser leídas por las computadoras (Deans et al., 2012). Ya existen ejemplos de aplicación (Balhoff et al., 2013; Mullins et al., 2012) que describen especies aplicando estas nuevas metodologías, donde los conceptos y términos incluidos en las descripciones constituyen fenotipos semánticos que provienen de múltiples ontologías (Mikó & Deans, 2009; Deans et al., 2012a, b; Yoder et al., 2011). En la actualidad se está trabajando en esta temática con la finalidad de aumentar la interoperabilidad entre las distintas ontologías de artrópodos y obtener una correspondencia entre las mismas (Bertone et al., 2013). Aún no es posible saber si este enfoque podrá eventualmente llegar a superar las descripciones clásicas de los taxones, principalmente si se llevan a cabo dentro de la nueva visión de la taxonomía integradora (Padial et al., 2010).
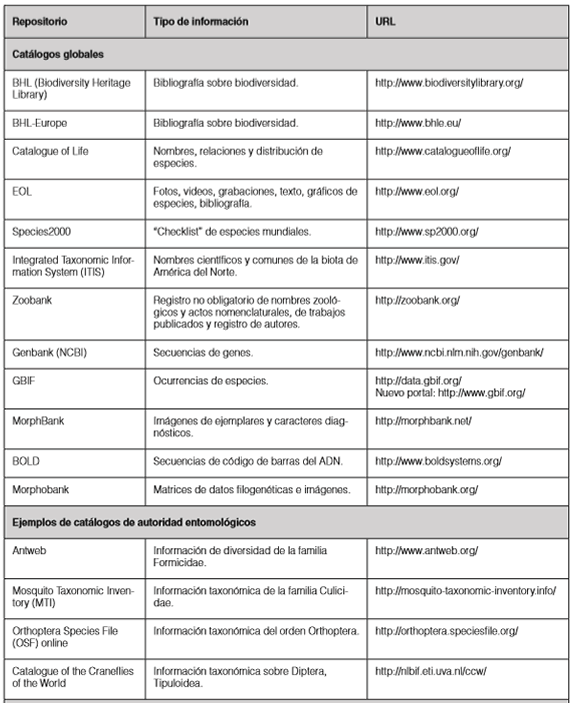
En la Tabla I se brinda una lista de algunos de los principales portales y repositorios de datos de biodiversidad.
Tabla I. Lista de algunos de los principales portales y repositorios de datos de biodiversidad.

La taxonomía es fundamental para comprender y documentar la biodiversidad. Es la ciencia que se ocupa de caracterizar, nominar y clasificar a los taxones. Sin embargo, a pesar de que estas tareas se remontan a más de 250 años, aún quedan por descubrir y describir millones de especies. La sociedad tiene una necesidad creciente de información taxonómica con el fin de entender, conservar, manejar y disfrutar de la naturaleza. El trazado de la distribución de especies y sus atributos es esencial para la comprensión de la historia de la vida. Disponer de un adecuado conocimiento taxonómico de la biodiversidad que nos rodea es necesario para el manejo sustentable de los ecosistemas, el éxito de las políticas de conservación, y la detección de plagas, vectores y especies invasoras. Las herramientas y métodos taxonómicos tradicionales presentan el rigor científico necesario para producir resultados confiables, sin embargo, no responden a los ritmos actuales de la crisis de la biodiversidad. La taxonomía necesita cada vez más de la colaboración internacional entre expertos para poder encarar proyectos de mayor envergadura. Estándares, bases de datos, aplicaciones y herramientas que facilitan la colaboración entre taxónomos ya han sido desarrollados y se encuentran en constante perfeccionamiento. Cada vez más, los avances científicos están potenciados por los nuevos desarrollos en las tecnologías de la informática que permiten manipular y explorar conjuntos de datos masivos. Es muy probable entonces, que la velocidad a la que una determinada disciplina científica avance, también dependa de cuan sólida sea la colaboración entre los investigadores, y con los informáticos, en las áreas de ciberinformática (e-Ciencia) como bases de datos y su visualización. Creemos que a partir de la fusión entre la taxonomía tradicional con la ciber-infraestructura (ciberinformática) es posible que se pueda revitalizar la taxonomía descriptiva para hacer frente a la crisis de la biodiversidad, facilitando el acceso abierto y gratuito a los datos sobre biodiversidad, mundialmente a través de Internet; con el fin de apoyar la investigación científica, la conservación y el desarrollo sustentable del mundo que nos rodea.
Darwin Core: Conjunto de estándares para bases de datos de diversidad biológica. Ha sido desarrollado por la organización internacional TDWG y es el que utiliza el portal biológico GBIF para facilitar el intercambio de información sobre la diversidad biológica entre los distintos portales de biodiversidad.
DOI ("Digital Object Identifier"): identificador único, persistente, que permite la localización de publicaciones electrónicas y otros objetos digitales en Internet.
E-Q ("Entity-Quality"): modelo conceptual donde una entidad (por ejemplo, una estructura anatómica) es descripta por una cualidad (por ejemplo, rugoso, redondo). Los términos de la entidad pueden ser estructuras de ontologías anatómicas u otras ontologías (como por ejemplo, "Gene ontology"); y el término de la cualidad proviene de ontologías de descripciones cualitativas/rasgos fenotípicos (PATO).
GUID ("Globally Unique Identifier"): Es un número único global de referencia usado como identificador en los programas de computación.
LSID ("Life Science Identifier"): Identificador global único y estable usado para rastrear y encontrar datos y metadatos de información de biodiversidad (taxones, datos de ejemplares, etc.) en Internet.
Metadatos: datos que describen otros datos. Proveen información sobre uno o varios aspectos de los datos (autor, fecha de creaci ón, los motivos de registro de los datos, los estándares usados en el registro de los datos, etc.).
OWL (Lenguaje de Ontología Web/ "Web Ontology Language"): es un lenguaje de representación del conocimiento construido sobre semántica lógica formal, y diseñado para ser compatible con la Red Informática Mundial ("World Wide Web").
PATO ("Phenotypic Quality Ontology"): ontología sobre cualidades/atributos fenotípicos (color, tamaño, longitud, etc.), que puede ser usada conjuntamente con otras ontologías para referirse a fenotipos.
RO ("Relation Ontology"): ontología sobre relaciones (es un; parte de; etc.), la cual es empleada por las distintas ontologías del OBO Foundry.
Web semántica: web de datos junto con sus descripciones que permiten vincularlos entre sí y ser interpretados por computadoras. Esa información adicional que describe el contenido, el significado y la relación entre datos presenta una estructura formal (por ejemplo, mediante ontologías) para que las computadoras puedan evaluar y extraer los datos automáticamente. Así se logra una mayor interoperabilidad entre los sistemas informáticos a través de programas de computación que buscan información sin operadores humanos.
AGRADECIMIENTOS
Los autores agradecen a Holger Braun y Klaus Riede por la lectura crítica del manuscrito y a Nélida Caligaris por la ayuda técnica brindada.
BIBLIOGRAFÍA CITADA
1. ACOSTA, L. E. 2007. Nomenclatura Zoológica: oportunidades y desafíos en la era digital. Revista de la Sociedad Entomológica Argentina 66 (3-4): 27-40. [ Links ]
2. AGOSTI, D. 2006. Biodiversity data are out of local taxono-mists' reach. Nature 439: 392. [ Links ]
3. AGOSTI, D. & N. F. JOHNSON. 2002. Taxonomists need better access to published data. Nature 417:222. [ Links ]
4. ANG, Y., J. PUNIAMOORTHY, A. C. PONT, M. BARAK, W.U. BLANCKENHORN,W. G. EBERHARD, N. PUNIAMOORTHY, V .C. SILVA, L. MUNARI & R. MEIER. 2013. A plea for digital reference collections and other science-based digitization initiatives in taxonomy: Sepsidnet as exemplar. Systematic Entomology 38: 637-644. [ Links ]
5. AntWeb. Available from http://www.antweb.org. Accessed 11 April 2014 [ Links ]
6. ARIÑO, A. 2010. Approaches to estimating the universe of natural history collections data. Biodiversity Informatics 7(2): 81-92. [ Links ]
7. BALHOFF, J.P., I. MIKÓ, M. J. YODER, P. L. MULLINS & A. R. DEANS. 2013. A Semantic Model for Species Description Applied to the Ensign Wasps (Hymenoptera: Evaniidae) of New Caledonia. Systematic Biology 62(5): 639-659. [ Links ]
8. BLAGODEROV, V., I. J. KITCHING, L. LIVERMORE, T. J. SIMONSEN & V. S. SMITH. 2012. No specimen left behind: industrial scale digitization of natural history collections. En: Blagoderov, V. & V. S. Smith (eds.), No specimen left behind: mass digitization of natural history collections, ZooKeys 209: 133-146. [ Links ]
9. BERTONE, M. A., R. L. BLINN, T. M. STANFIELD, K. J. DEW, K. C. SELTMANN & A. R. DEANS. 2012. Results and insights from the NCSU Insect Museum GigaPan project. En: Blagoderov, V. & V. S. Smith (eds.), No specimen left behind: mass digitization of natural history collections, ZooKeys 209: 115-132 [ Links ]
10. BERTONE, M.A., I. MIKO, M. J. YODER, K.C. SELTMANN, J. P. BALHOFF & A. R. DEANS. 2013. Matching arthropod anatomy ontologies to the Hymenoptera Anatomy Ontology: results from a manual alignment. Database, Article ID bas057, doi:10.1093/database/bas057. [ Links ]
11. CARBONELL, C.S., M. M. CIGLIANO & C. E. LANGE. 2006. Acridomorph (Orthoptera) species of Argentina and Uruguay/Especies de Acridomorfos (Orthoptera) de Argentina y Uruguay. CD-ROM. Publications on Orthopteran Diversity. The Orthopterists' Society. [ Links ]
12. CIGLIANO, M.M. & D. EADES. 2010. New technologies challenge the future of Taxonomy in Orthoptera. Journal of Orthoptera Research 19: 15-18. [ Links ]
13. COSTA, M., S. REEVE1, G. GRUMBLING & D. OSUMI-SUTHERLAND. 2013.The Drosophila anatomy ontology. Journal of Biomedical Semantics 4:32. [ Links ]
14. DAHDUL, W.M., J.P. BALHOFF, J. ENGEMAN, T. GRANDE, E.J. HILTON, C. KOTHARI, H. LAPP, J.G. LUNDBERG, P.E. MIDFORD, T.J. VISION, M. WESTERFIELD & P.M. MABEE 2010. Evolutionary characters, phenotypes and ontologies: curating data from the systematic biology literature. PLoS One 5: e10708. [ Links ]
15. DEANS, A. R., M. J. YODER, J. P. BALHOFF. 2012a. Time to change how we describe biodiversity. Trends in Ecology and Evolution 27(2): 78-84. [ Links ]
16. DEANS, A.R., I. MIKÓ, B. WIPFLER & F. FRIEDRICH. 2012b. Evolutionary phenomics and the emerging enlightenment of arthropod systematic. Invertebrate Systematics 26: 323-330. [ Links ]
17. DIETRICH, C. H., J. HART, D. RAILA, U. RAVAIOLI, N. SOBH, O. SOBH & C. TAYLOR. 2012. InvertNet: a new paradigm for digital access to invertebrate collections. En: Blagoderov, V. & V. S. Smith (eds.), No specimen left behind: mass digitization of natural history collections, ZooKeys 209: 165-181. doi: 10.3897/zookeys.209.3571 [ Links ]
18. EADES, D. C., D. OTTE, M. M. CIGLIANO & H. BRAUN. 2014. Orthoptera Species File. Version 5.0/5.0. [April 11, 2014]. http://Orthoptera.SpeciesFile.org [ Links ]
19. EOL. Encyclopedia of Life. Available from http://www.eol.org. Accessed May, 14, 2014. [ Links ]
20. FAVRET, C. 2013 Cybertaxonomy to accomplish big things in aphid systematics. Insect Science 21: 1-8. [ Links ]
21. GODFRAY, H. C. J. 2002. Challenges for taxonomy. Nature 417: 17-19. [ Links ]
22. GODFRAY, H. C. J. 2005. Taxonomy as Information Science. Proceedings of the California Academy of Sciences 56: 170-181. [ Links ]
23. GODFRAY, H. C. J., B. R. CLARK, I. J. KITCHING, S. J. MAYO & M. J. SCOBLE. 2007. The Web and the Structure of Taxonomy. Systematic Biology 56 (6): 943-955. [ Links ]
24. HARBACH, R.E. 2013. Mosquito Taxonomic Inventory, http://mosquito-taxonomic-inventory.info/, accessed on April, 2014. [ Links ]
25. HASTON, E., R. CUBEY, M. PULLAN, H. ATKINS & D. J. HARRIS. 2012. Developing integrated workflows for the digitisation of herbarium specimens using a modular and scalable approach. Zookeys 209: 93-102. [ Links ]
26. HAUSER, C. L., A. STEINER, J. HOLSTEIN & M. J. SCOBLE (eds.). 2005. Digital imaging of biological type specimens: A manual of best practice. Staatliches Museum fur Naturkunde, Stuttgart. [ Links ]
27. ICNZ (International Commission on Zoological Nomenclature). 2012. Amendment of Articles 8, 9, 10, 21 and 78 of the International Code of Zoological Nomenclature to expand and refine methods of publication. Zootaxones 3450: 1-7. [ Links ]
28. KELLING, S., W. M. HOCHACHKA , D. FINK , M. RIEDEWALD, R. CARUANA , G. BALLARD & G. HOOKER. 2009. Data-intensive science: a new paradigm for biodiversity studies. BioScience 59: 613-620. [ Links ]
29. Lampe, K. H., Riede, K. & Ingrisch, S. 2005. Repatriation of knowledge about insects and types through the DORSA virtual museum (Digital Orthoptera Specimen Access). Beiträge zur Entomologie 55 (2): 477-484. [ Links ]
30. MABEE P., J.P. BALHOFF, W.M. DAHDUL, H. LAPP, P.E. MID-FORD, T.J. VISION, M. WESTERFIELD. 2012. 500,000 fish phenotypes: the new informatics landscape for evolutionary and developmental biology of the vertebrate skeleton. Journal of Applied Ichthyology 28: 300-305. [ Links ]
31. MANTLE, B. L., J. LA SALLE & N. FISHER. 2012. Whole-drawer imaging for digital management and curation of a large entomological collection. Zookeys 209: 147-163. [ Links ]
32. MIKÓ, I. & A.R. DEANS. 2009. Masner, a new genus of Ceraphronidae (Hymenoptera: Ceraphronoidea) described using controlled vocabularies. ZooKeys 20: 127-153. [ Links ]
33. MULLINS, P., R. KAWADA, J. BALHOFF, A. DEANS. 2012. A revision of Evaniscus (Hymenoptera, Evaniidae) using ontology-based semantic phenotype annotation. ZooKeys 223: 1-38. [ Links ]
34. MUNGALL C.J., G.V. GKOUTOS, C.L. SMITH, M.A. HAENDEL, S.E. LEWIS, M. ASHBURNER. 2010. Integrating phenotype ontologies across multiple species. Genome Biology 11: R2. [ Links ]
35. MUNGALL, C.J., C. TORNIAI, G.V. GKOUTOS, S.E. LEWIS, M.A. HAENDEL. 2012. Uberon, an integrative multi-species anatomy ontology. Genome Biology 13:R5. mx. 2014. http://mx.phenomix.org/index.php/Main_Page [ Links ]
36. NEWMAN, H.B., M. H. ELLISMAN, J. A. ORCUTT. 2003. Data-intensive e-science frontier research. Communications of the ACM 46: 68-77. [ Links ]
37. Nguyen, C.V., Lovell, D.R., Adcock, M. La Salle, J.L. 2014. Capturing Natural-Colour 3D Models of Insects for Species Discovery and Diagnostics. PLOS ONE 9:1-11 [ Links ]
38. NRCNA (National Research Council of the National Academies). 2009. A New Biology for the 21st Century, The National Academies Press. [ Links ]
39. PADIAL, J. M., A. MIRALLES, I. DE LA RIVA & M. VENCES. 2010. The integrative future of taxonomy. Frontiers in Zoology 7: 16. [ Links ]
40. PAGE, L. M. 2008. Planetary biodiversity inventories as models of the new taxonomy. En: Wheeler, Q. D. (ed.), The New Taxonomy. CRC Press, Boca Raton, 55-63 pp. [ Links ]
41. PATTERSON, D. J., J. COOPER, P.M. KIRK, R.L. PYLE & D.P. REMSEN. 2010. Names are key to the big new biology. Trends in Ecology and Evolution 25 (12): 686-691. [ Links ]
42. PATTERSON, D. J., W. EGLOFF, D. AGOSTI, D. EADES, N. FRANZ, G. HAGEDORN, J.A. REES & D. P. REMSEN. 2014. Scientific names of organisms: attribution, rights, and licensing. BMC (BioMed Central) Research Notes 7:79 [ Links ]
43. PENEV, L., D. ROBERTS, V. SMITH, D. AGOSTI & T. ERWIN. 2010. Taxonomy shifts up a gear: New publishing tools to accelerate biodiversity research. ZooKeys 50: 1-4. [ Links ]
44. POLASZEK, A., D. AGOSTI, M. ALONSO-ZARAZAGA, G. BECCALONI, P. de PLACE BJØRN, P. BOUCHET, D.J. BROTHERS, EARL OF CRANBROOK, N. EVENHUIS, H.C.J GOD-FRAY, N.F. JOHNSON, F.-T. KRELL, D. LIPSCOMB, C.H.C. LYAL, G. MACE, S. MAWATARI, S.E. MILLER, A. MINELLI, S. MORRIS, P.K.L. NG, D.J. PATTERSON, R.L. PYLE, N. ROBINSON, L. ROGO, J. TAVERNE, F.C. THOMPSON, J. VAN TOL, Q.D. WHEELER & E.O. WILSON. 2005. A universal register for animal names. Nature 437: 477. [ Links ]
45. PYLE, R. & E. MICHEL. 2008. Zoobank: Developing a nomen-clatural tool for unifying 250 years of biological information. Zootaxones 1950: 39-50. [ Links ]
46. RAMÍREZ, M. J., J. A. CODDINGTON, W. P. MADDISON, P. E. MIDFORD, L. PRENDINI , J. MILLER, C. E. GRISWOLD, G. HORMIGA, P. SIERWALD, N. SCHARFF, S.P. BENJAMIN & W. C. WHEELER. 2007. Linking of digital images to phylogenetic data matrices using a morphological ontology. Systematic Biology 56: 283-294. [ Links ]
47. RAVEN, P. H. (ed.). 1997. Nature and Human Society: The Quest for a Sustainable World. National Academy Press, Washington, DC. [ Links ]
48. SCHMIDT, S., M. BALKE & S. LAFOGLER. 2012. DScan - a high-performance digital scanning system for entomological collections. En: Blagoderov, V., V. S. Smith (eds.), No specimen left behind: mass digitization of natural history collections, ZooKeys 209: 183-191. doi: 10.3897/zookeys.209.3115 [ Links ]
49. SCHRAM, F. 2004. The truly new systematics -- megascience in the information age. Hydrobiologia 519: 1-7. [ Links ]
50. SMITH, V. S., S. D. RYCROFT, K. T. HARMAN, B. SCOTT & D. ROBERTS. 2009. Scratchpads: a data-publishing frame-work to build, share and manage information on the diversity of life. BMC Bioinformatics, 10 (Suppl 14): S6. doi: 10.1186/1471-2105-10-S14-S6 [ Links ]
51. SMITH, V.S. & V. BLAGODEROV. 2012. Bringing collections out of the dark. ZooKeys 209: 1-6. [ Links ]
52. TOPALIS, P., C. TZAVLAKI, K. VESTAKI, E. DIALYNAS, D.E. SONENSHINE, R. BUTLER, R.V. BRUGGNER, E.O. STINSON, F.H. COLLINS, C. LOUIS. 2008. Anatomical ontologies of mosquitoes and ticks, and their web browsers in VectorBase. Insect Molecular Biology 17: 87-89. [ Links ]
53. TULIG, M., N. TARNOWSKY, M. BEVANS, K. ANTHONY & B. M. THIERS. 2012. Increasing the efficiency of digitization work-flows for herbarium specimens. Zookeys 209: 103-113. [ Links ]
54. WASHINGTON N, L., M.A. HAENDEL, C.J. MUNGALL, M. ASHBURNER, M. WESTERFIELD & S.E. LEWIS. 2009. Linking human diseases to animal models using ontology-based phenotype annotation. PLoS Biology 7:e1000247. [ Links ]
55. WHEELER, Q. D. 2007. Invertebrate systematics or spineless taxonomy. Zootaxones 1668: 11-18. [ Links ]
56. WHEELER, Q. D. 2008. Taxonomic shock and awe. En: Wheeler, Q. D. (ed.), The New Taxonomy, Taylor and Francis, Boca Raton, pp. 211-226. [ Links ]
57. WHEELER, Q. D. 2010. What would NASA do? Mission-critical infrastructure for species exploration. Systematics and Biodiversity 8: 11-15. [ Links ]
58. WHEELER, Q. D., S. KNAPP, D. W. STEVENSON, J. STEVENSON, S. D. BLUM, B. M. BOOM, G. G. BORISY, J. L. BUIZER, M. R. DE CARVALHO, A. CIBRIAN, M. J. DONOGHUE, V. DOYLE, E. M. GERSON, C. H. GRAHAM, P. GRAVES, S. J. GRAVES, R. P. GURALNICK, A. L. HAMILTON, J. HANKEN, W. LAW, D. L. LIPSCOMB, T. E. LOVE-JOY, H. MILLER, J. S. MILLER, S. NAEEM, M. J. NOVA-CEK, L. M. PAGE, N. I. PLATNICK, H. PORTER-MORGAN, P. H. RAVEN, M. A. SOLIS, A. G. VALDECASAS, S. VAN DER LEEUW1, A. VASCO, N. VERMEULEN, J. VOGEL, R. L. WALLS, E. O. WILSON1 & J. B. WOOLLEY. 2012a. Mapping the biosphere: exploring species to understand the origin, organization and sustainability of biodiversity. Systematics and Biodiversity 10 (1): 1-20. [ Links ]
59. WHEELER, Q., T. BOURGOIN, J. CODDINGTON, T. GOSTONY, A. HAMILTON, R. LARIMER, A. POLASZEK, M. SCHAUFF, M. A. SOLIS. 2012b. Nomenclatural bench-marking: the roles of digital typification and telemicroscopy. En: Blagoderov, V. & V. S. Smith (ed.), No specimen left behind: mass digitization of natural history collections, ZooKeys 209: 193-202. doi: 10.3897/zookeys.209.3486 [ Links ]
60. WILSON, E. O. 1992. The Diversity of Life. Norton, New York. [ Links ]
61. WILSON, E. O. 2003. The encyclopedia of life. Trends in Ecology and Evolution 18: 77-80. [ Links ] [ Links ]