Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Revista argentina de antropología biológica
versión On-line ISSN 1514-7991
Rev Arg Antrop Biol vol.19 no.1 La Plata jun. 2017
http://dx.doi.org/10.17139/raab.2017.0019.01.03
TRABAJOS DEL DOSSIER
LINAJES MATERNOS EN MUESTRAS ANTIGUAS DE LA PUNA JUJEÑA. COMPARACIÓN CON ESTUDIOS DE LA REGIÓN CENTRO-SUR ANDINA
María B. Postillone*1,2, María L. Fuchs2,3, Cristian M. Crespo1,2, María G. Russo1,2, Héctor H. Varela2,3, Francisco R. Carnese1,4, Sergio A. Avena1,2,4, Cristina B. Dejean1,4
1Equipo de Antropología Biológica. Departamento de Ciencias Naturales y Antropológicas. CEBBAD-Fundación de Historia Natural Félix de Azara. Universidad Maimónides. Argentina
2Consejo Nacional de Investigaciones Científicas y Técnicas (CONICET). Argentina
3Departamento de Ciencias Naturales. Facultad de Exactas, Físico-Química y Naturales. Universidad Nacional de Río Cuarto. Río Cuarto. Argentina
4Sección de Antropología Biológica. Instituto de Ciencias Antropológicas. Facultad de Filosofía y Letras. Universidad de Buenos Aires. Argentina
*Correspondencia a: María Bárbara Postillone. CEBBADFundación de Historia Natural Félix de Azara. Universidad Maimónides. Hidalgo 775. 1405 CABA. E-mail: mbpostillone@gmail.com
Financiamiento: FONCYT (PICT02210/07); CONICET (PIP2511); Fundación Científica Felipe Fiorellino.
Recibido 28 Diciembre 2015; aceptado 13 Julio 2016
doi:10.17139/raab.2017.0019.01.03
RESUMEN Las evidencias arqueológicas proporcionan abundantes pruebas de una activa interacción entre las poblaciones de lo que actualmente es el noroeste de Argentina, el sur de Perú, el oeste de Bolivia y el norte de Chile. Los estudios de ADN en individuos prehispánicos, a su vez, proveen información directa sobre la historia biológica y la dinámica de los grupos humanos, y aportan nuevas perspectivas a la comprensión de la dispersión y variabilidad de los amerindios. El objetivo de este trabajo es analizar los linajes maternos presentes en individuos del período tardío de la Puna jujeña (1000-1450 AD) mediante la comparación de su composición genética con las de otros grupos precolombinos de la región centro-sur andina descriptos por otros autores. A partir de las secuencias de HVR I del ADN mitocondrial se determinó la variabilidad genética inter e intramuestral, además de la distancia genética entre los diversos grupos utilizados. En las muestras analizadas el linaje más frecuente fue el A2, el cual mostró una gran variabilidad haplotípica. Esto lo diferencia de otros estudios de la región andina en los que se ha descripto el predominio de los linajes B2. A una escala local, las distancias genéticas entre la Puna jujeña y otros grupos poblacionales del actual noroeste argentino no fueron significativas, a excepción de Pampa Grande, de la que se encuentra más alejada geográfica y espacialmente. A nivel regional, las diferencias son significativas con respecto a la mayoría de las muestras antiguas de Perú, aunque no evidencian un patrón espacial o temporal determinado.
PALABRAS CLAVE: ADNmt; haplotipos; ADN antiguo; Puna jujeña
ABSTRACT Archaeological records provide abundant evidence of an active interaction between the peoples of what are now northwestern Argentina, southern Peru, western Bolivia, and northern Chile. Likewise, DNA studies in prehispanic individuals provide direct information on the biological history and dynamics of human groups, offering new perspectives to the understanding of dispersion and variability of Native Americans. The aim of this paper is to analyze the maternal lineages present in individuals from the late period of the Puna region (1000-1450 AD) by comparing their genetic composition with other pre-Columbian groups from the south-central Andean region, described by other authors. From mtDNA HVR-I sequences, inter- and intra-sample genetic variability was determined, as well as the genetic distance between the various groups used. In the samples analyzed, A2 was found to be the most common lineage,, showing high haplotype variability, and contrasting with other studies in the Andean region, which described the prevalence of B2 lineages. At a local level, the genetic distances between the Puna population and other groups from presentday northwestern Argentina were not significant, except for Pampa Grande, which is more geographically and spatially remote. At a regional level, the differences are significant in relation to the majority of Peru ancient samples, but do not show a particular spatial or time pattern.
KEY WORDS: mtDNA; haplotypes; ancient DNA; Puna of Jujuy
Los estudios arqueológicos realizados en el Noroeste de Argentina (NOA) señalan que la dispersión humana habría sido un proceso iniciado a finales del Pleistoceno, cerca de los 10800 años AP (Politis et al., 2010). Durante el Holoceno, entre los 10000 y 3000 años AP, se habría incrementado la interacción entre poblaciones de ambos lados de la cordillera. Existe evidencia de que durante este periodo habrían ocurrido diferentes procesos como la ocupación de nuevos ambientes, cambios en la estrategia de formación de asentamientos con diferenciación social y mejoras en el manejo del espacio, intensificación en las redes de intercambio, cría de camélidos como principal actividad ganadera, aumento en las actividades agrícolas y la adopción de nuevas tecnologías. Esto habría dado como resultado un importante crecimiento demográfico y una transición del estilo de vida cazador-recolector al de asentamientos aldeanos (Periodo Formativo) y su posterior complejización en el Periodo de Desarrollos Regionales (1050-500 años AP), (Muscio, 2001). En este último, la actividad agropasto-ril tuvo su apogeo con una economía ganadera basada en la cría de llamas, siendo la zona de Casabindo-Doncellas la de mayor desarrollo (Albeck y Ruiz, 2003), en la Puna de Jujuy. Ésta comprende la porción nororiental de la provincia, siendo una meseta de alta montaña, a más de 3700msm. El tráfico de caravanas asumió un importante rol, permitiendo un mejoramiento en la obtención y regulación de recursos (Tarragó, 1984; Nielsen, 2000; Albeck y Ruiz, 2003; Torres-Rouff et al., 2012). Diversos estudios arqueológicos del NOA señalan que esta región recibió infuencias en diferentes momentos y de magnitud variable desde el sur de Perú, el altiplano boliviano y la foresta tropical (González y Pérez, 1972; Berenguer y Dauels-berg, 1989; Rivera, 1991). Asimismo, las relaciones sociales, el tráfico de materiales, el intercambio y el aprovechamiento de recursos en poblaciones prehispánicas del NOA han sido estudiados en una importante cantidad de trabajos (Albeck, 2007; Escola et al., 2007; Núñez Aten-cio, 2007; entre otros). Sin embargo, la composición genética de las poblaciones y sus afinidades biológicas han sido abordadas en menor medida. Estudios empleando caracteres craneales métricos y no métricos realizados en poblaciones prehistóricas que habitaron el NOA, el norte de Chile y los Valles de Cochabamba (Boli-via) (ca. 3000AC-1500DC) demostraron que la muestra de esta última se ubica en una posición intermedia entre las dos primeras (Varela et al., 2008; Cocilovo et al., 2009). Además, en estos trabajos se plantea un modelo de poblamiento basado en la subdivisión de una población andina ancestral que fue paulatinamente poblando el NOA, los valles de Cochabamba en Bolivia y Norte de Chile. En cada una de estas regiones, el proceso dispersivo originó varias subpoblacio-nes que se diferenciaron gradualmente durante la exploración, conquista y colonización de nuevos ambientes. Por otro lado, a un nivel más regional, otros estudios morfológicos de muestras de 300 a 1480 años DC, proponen que podrían haber existido dos grupos biológicos en el NOA, uno estaría conformado por las poblaciones de Puna y Quebrada de Humahuaca y el otro por pobladores de la Región Valliserrana y Las Pirguas (Varela et al., 2004).
Por su parte, estudios bioantropológicos de diversos marcadores genéticos aplicados a individuos de poblaciones del pasado abrieron las puertas a un nuevo tipo de información. El acceso directo al ADN de restos humanos antiguos (ADNa) provee datos relevantes y específicos acerca de su historia biológica y dinámica pobla-cional (Pääbo et al., 2004; Shapiro y Hofreiter, 2012). Además, permite abordar el estudio de procesos migratorios, patrones de residencia y relaciones genéticas entre poblaciones humanas, teniendo en cuenta así la dimensión temporal de estos procesos (Hofreiter et al., 2001).
Mediante el estudio molecular del ADN mito-condrial se han logrado identificar diversos linajes maternos que conforman los denominados haplogrupos (Hgs), los cuales se definen por compartir un conjunto de mutaciones básicas. Tanto en individuos prehispánicos como en poblaciones autóctonas contemporáneas se han definido como Hgs amerindios mayoritarios en el continente los denominados A2, B2, C1 y D1 y el X2a sólo des-cripto para Norteamérica (Bodner et al., 2012). A su vez, los Hgs se subdividen en haplotipos por la presencia de mutaciones adicionales.
Dentro de la región del NOA, Carnese et al. (2010), analizaron 19 individuos de Pampa Grande (Salta) observando en 9 casos la presencia del Hg B2, 8 del Hg D1 y en sólo 2 del Hg A2. Mendisco et al. (2011) analizaron la secuencia del ADNmt de 23 piezas dentales provenientes de individuos del sitio arqueológico Los Amarillos en la Quebrada de Humahuaca (Jujuy), obteniendo resultados para 18 de las muestras, 13 de las cuales presentaban un linaje particular de A2, proporción muy elevada e inesperada para la región, ya que en la mayoría de las muestras pobla-cionales antiguas y actuales el linaje más representado es el B2. Los individuos restantes fueron asignados a los Hgs D1 (3), C1 (1) y B2 (1). En otro estudio Mendisco et al., (2014) analizaron muestras de sitios arqueológicos de Quebrada de Humahuaca, Valle Calchaquí y Puna correspondientes al período de Desarrollos Regionales. Sus resultados evidenciaron la presencia de los cuatro Hgs amerindios sudamericanos principales, sugiriendo además un origen de la población en los Andes Centrales pero con infuencia genética desde poblaciones de la región de Gran Chaco. La diversidad haplotípica encontrada por estos autores apoya la hipótesis generada a partir de la morfometría craneal en la que el NOA puede dividirse en dos grupos biológicos diferentes, como se mencionó anteriormente.
Dejean et al., (2014) compararon las muestras de Pampa Grande y Los Amarillos estudiadas por Carnese et al. (2010) y Mendisco et al. (2011) y observaron que en ambas están presentes los Hgs A2, B2 y D1, pero a nivel de haplotipos sólo se comparten 2 linajes, un A2 y un D1. Concluyeron que la distancia temporal entre ambos sitios pudo haber sido el principal factor que intervino en la diferenciación mitocondrial encontrada. Motti et al. (2014) compararon datos de poblaciones actuales del NOA y del centro-oeste argentino con los linajes presentes en esas muestras prehispá-nicas y encontraron que sólo comparten cuatro linajes (A216111@, C1b16189-16311, B2b2 y B216142). Además señalan que el linaje de D116129 determinado en Pampa Grande se ha descripto en muestras de Bolivia y Perú, y el linaje B216145-16156-16157 en una muestra de la Amazonia boliviana.
Otro estudio en muestras poblacionales contemporáneas de diferentes regiones de Jujuy, ha demostrado también la presencia de los linajes A2 (13.9%), B4b (56.7%), C1 (17.8%), D1 (8.9%) y D4h3a (1.1%) (Cardoso et al., 2013). La presencia de linajes prehispánicos en muestras actuales daría indicios de continuidad biológica.
En este contexto, el objetivo del presente trabajo es analizar los linajes maternos en individuos de la Puna Jujeña para incorporar nuevos datos al estudio de la diversidad genética de las poblaciones que habitaron el NOA antes de la llegada de los europeos y comparar los resultados con los de otras muestras prehispánicas de la región. De esta manera, se pretende aportar nueva información para la comprensión de la historia evolutiva y del poblamiento local y regional.
MATERIAL Y MÉTODOS
Preparación de la muestra
Se tomaron las precauciones necesarias para el trabajo con ADNa (Gilbert, 2005; Crespo et al., 2010 y Kemp y Smith, 2010). Los cuartos de procesamiento de las muestras pre y post Reacción en Cadena de la Polimerasa (PCR por sus siglas en inglés) se encuentran separados y se utilizó la indumentaria de protección adecuada como cofas, barbijos, guantes descartables y guardapolvos. La indumentaria, los compartimentos y el instrumental a utilizar fueron previamente irradiados con luz UV por al menos 45 minutos (min). Los instrumentos utilizados, antes de ser irradiados fueron lavados con hi-poclorito de sodio al 6%, enjuagado con agua calidad biología molecular y posteriormente lavados con alcohol etílico al 70%.
Muestra
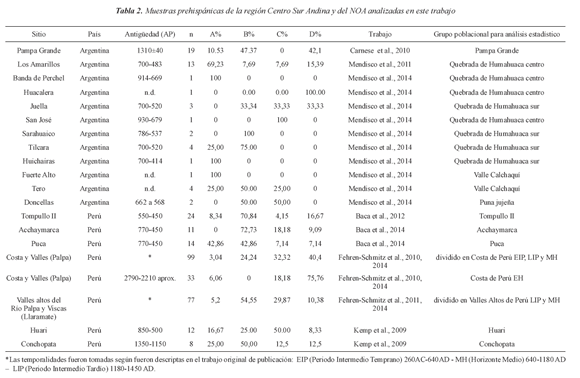
Consistió en 23 piezas dentales provenientes de diferentes individuos de la Puna de Ju-juy, pertenecientes a la colección osteológica depositada en el Museo Etnográfico "Juan Bautista Ambrosetti" (FFyL, UBA). Se incluyeron muestras de los sitios Doncellas, Agua Caliente, Río Negro, Casabindo y Sorcuyo (Fig. 1), excavados por E. Boman a principios de 1900, M.A. Vignati en 1938 y E. Casanova en colaboración con O. Paulotti y P. Haedo durante la década de 1940 hasta 1967 (Alfaro, 1983). Según los fechados radiocarbónicos realizados con anterioridad los sitios presentaron una antigüedad entre 509±42 años y 609± 32 años AP (Fuchs y Va-rela, 2013) correspondiendo al Periodo Tardío (1000-1450 AD). Los estudios craneométricos realizados sobre individuos de esos mismos sitios demostraron una baja diferenciación fenotí-pica entre ellos (Fuchs et al., 2016).
Estos sitios arqueológicos se encuentran ubicados en la cuenca endorreica de Mirafores-Guayatayoc-Salinas Grandes, en el centro de la Puna Jujeña, rodeando a Doncellas, considerado como centro cúltico de la región. La distancia entre los sitios más alejados (Agua Caliente y Sorcuyo) es de aproximadamente 35km (Alfaro, 1983). Estos poblados estaban vinculados con la actividad agropastoril y el contacto con grupos caravaneros, con un patrón de asentamiento característico del tipo semiconglomerado de fácil acceso, con viviendas de planta rectangular, y con relaciones contextuales con la cuenca del Titicaca en Bolivia y San Pedro de Atacama, en el norte de Chile y con una fuerte infuencia de la Quebrada de Humahuaca (Alfaro, 1983; Albeck y Ruiz, 2003). El único sitio que se diferencia en estas características es Sorcuyo por presentar viviendas en forma circular, las cuales pueden ser estructuras realizadas en periodos más tempranos que se mantuvieron en el tiempo, o a un poblado formado a partir de un grupo de individuos provenientes de otras regiones (Albeck y Ruiz, 2003).
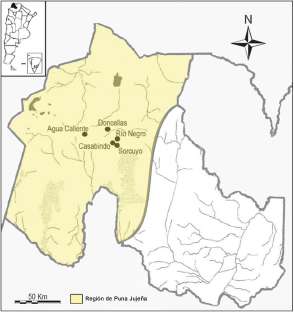
Fig. 1. Ubicación geográfica de los sitios analizados. En color se marca la Puna de Jujuy.
Extracción de ADN
Como primer paso, a cada pieza dental se le realizó un molde con silicona y monómero autocurable, material empleado en odontología convencional. Luego, las piezas dentales fueron descontaminadas superficialmente con hipoclo-rito de sodio al 6%, enjuagadas varias veces con agua calidad biología molecular e irradiadas con luz UV por 45min de cada lado. Se procedió a la obtención de polvo a partir del desbaste de la dentina interior de la pieza con un minitaladro (Dremel). Cuando la pieza conservó su estructura debido a su buen estado de preservación, la misma fue remontada utilizando el monómero antes mencionado para ser restituida a la colección. A cada muestra se le realizaron al menos tres extracciones de ADN utilizando entre 75 y 100 mg de polvo y diferentes métodos. El primero consistió en una extracción mediante columnas de sílica utilizando QIAamp® DNA Investi-gator Kit (QIAGEN). El segundo fue la utilización del equipo Geneclean (MP Biomedics), en ambos casos se llevó a cabo el protocolo especi-ficado por el fabricante. El tercero fue el método de fenol-cloroformo-alcohol isoamílico (25:24:1 v/v) (tomado y modificado de Carnese et al., 2010). Este último culminó con la purificación del ADN mediante columnas de sílica utilizando AccuPrep® PCR Purification Kit (BIONEER), siguiendo el protocolo especificado por el fabricante. En cada extracción se incorporó un "blanco" (tubo sin muestra sometido al mismo procedimiento), que posteriormente se incluyó en los análisis en paralelo al ADN extraído.
Análisis del ADN mitocondrial
En primer lugar se realizó una asignación a los Hgs amerindios A, C y D por Polimorfsmos de Longitud de los Fragmentos de Restricción (RFLP, por su sigla en inglés) y por presencia de la deleción de 9 pares de bases (pb) para el Hg B (Tabla 1). La reacción de amplificación por PCR para cada uno de los cuatro Hgs se realizó en un volumen final de 12,5µl conteniendo: 1x de GoTaq® reaction buffer, 1,5mM de MgCl2 (Promega), 0,2mM de cada dNTP (Thermo Scientific), 0,8µM de cada cebador (Invitrogen), 1,25u de GoTaq® DNA polymerase (Promega) y 5µl de ADNa.
Para los Hgs A, C y D la amplificación fue controlada sometiendo las muestras a electro-foresis en gel de agarosa al 1%. Luego fueron digeridas con la enzima de restricción adecuada (Tabla 1), agregando a cada muestra amplificada 10µl de una solución de digestión que contiene: buffer 1X, 1U de enzima de restricción y agua calidad biología molecular. Esta mezcla se incubó a 37ºC durante 20 horas. Para la detección de la deleción de 9pb característica del Hg B, la amplificación se visualizó directamente en un gel de agarosa al 3%.
Para un estudio más detallado, se procedió con la amplificación por PCR de la Región Hipervariable I (HVR por sus siglas en inglés) en tres fragmentos solapados (1, 2 y 3) que comprenden desde la posición 15984 a la 16410 del genoma mitocondrial utilizando los cebadores: fragmento 1- F15984 (5'-TAGCACC-CAAAGCTAAGATTCTAAT-3', modificado a partir de F15989, Gabriel et al., 2001) y R16167 (Ricaut et al., 2004); segmento 2- F16120 (Ada-chi et al., 2004) y R16239 (Ivanov et al., 1996); fragmento 3- F16204 (5'-GCAAGTACAGCA-ATCAACCCT-3', diseñado por nuestro equipo de trabajo) y R16410 (Gabriel et al., 2001) respectivamente, la temperatura de anidamiento de 55ºC.
Las reacciones de PCR se llevaron a cabo para cada uno de los 3 segmentos y con un volumen de 25µl conteniendo: 1X de Hot Start PCR Buffer (Thermo Scientific), 2mM de MgCl2 (Thermo Scientific), 0,4µM de cada par de cebadores (Invitrogen), 0,2mM de cada dNTP (Thermo Scientific), 1 u de Máxima Hot Start Taq DNA Polymerase (Thermo Scientific) y 5 µl de la muestra de ADNa como templado. En cada amplificación se incluyeron los "blancos" de extracción, un "blanco" de reacción de PCR y controles de ADN moderno.
Preparación de la muestra para secuenciación de HVR I. Los productos de amplificación fueron visualizados en geles de agarosa al 1% ® PCR Purif-cation kit (BIONEER), siguiendo el protocolo descripto por el fabricante. Las reacciones de secuenciación en ambos sentidos fueron llevados a cabo en el servicio de la Unidad Genó-mica del Instituto de Biotecnología del INTA, con los mismos cebadores empleados en la amplificación de cada fragmento y utilizando un secuenciador automático 3130XL Genetic Analyzer con BigDyeT Terminator Sequencing Kit (Applied Biosystems).
Análisis de las secuencias
Se obtuvieron al menos dos por muestra provenientes de diferentes extracciones, las cuales fueron revisadas y corregidas manualmente utilizando el programa Bioedit 2.7.5 (Hall, 2013). En cada muestra el conjunto de mutaciones obtenido en cada secuenciación fue comparado para generar un haplotipo consenso. Estos haplotipos fueron cotejados con la Secuencia de Referencia de Cambridge revisada (rCRS) (Andrews et al., 1999) utilizando el mismo programa. Los linajes obtenidos fueron clasificados utilizando Haplogrep (Kloss-Brandstätter et al., 2011) y datos tomados de la literatura (Tamm et al., 2007; Achilli et al., 2008, 2013; Perego et al., 2009, 2010; Bodner et al., 2012; de Saint Pierre et al., 2012).
Análisis comparativo con muestras de la región. Las secuencias obtenidas fueron comparadas con los datos ya publicados de muestras prehispánicas del NOA y el sur de Perú (Tabla 2; Apendice suplementario 1, Fig. suplementaria 1) con el fin de establecer las posibles relaciones biológicas entre los linajes mitocondriales de las muestras poblacionales en estudio. Además, los linajes de dos individuos analizados por Mendisco et al., (2014) del sitio Doncellas fueron incorporados a la muestra de Puna jujeña analizada en este estudio para todas las pruebas estadísticas empleadas.
Con las secuencias de la HVR I, se llevó a cabo un Análisis de la Varianza Molecular (AMOVA) en el cual algunas de las muestras fueron agrupadas debido a su cercanía geográfica y su bajo número de individuos (Tabla 2) llegando a un total de 16 agrupamientos. También se llevaron a cabo comparaciones entre estos grupos poblacionales a través del índice FST (utilizando 1000 permutaciones) y se calculó la distancia de
Nei con el programa Arlequin 3.11 (Excoffer et al., 2005), utilizando el modelo evolutivo de Ta-mura y Nei (1993) con un valor de Gamma=0,29. Para el caso de la muestra de Puna jujeña, se determinó el número total de sitios nucleotídi-cos variantes (S), el número de haplotipos mito-condriales (h), la diversidad haplotípica (Hd), el número medio de diferencias entre pares de secuencias (k) y la diversidad nucleotídica (π) (Nei, 1987), utilizando el programa DnaSP 5.10.1 (Rozas et al., 2010). Además, se construyó un Neighbor Joining con los FST obtenidos para los grupos muestrales de la Tabla 2 utilizando el programa MEGA 6 (Tamura et al., 2013).
Se utilizaron fuentes bibliográficas de poblaciones actuales del NOA (Cabana et al., 2006; Álvarez-Iglesias et al., 2007; Marrero et al., 2007; Tamm et al., 2007; Sala et al., 2010; Bar-bieri et al., 2011; de Saint Pierre et al., 2012; Cardoso et al., 2013; Taboada-Echalar et al., 2013; Sala y Corach, 2014) para cotejar si los haplotipos encontrados en la muestra antigua de Puna jujeña presentan continuidad en la región.
Análisis del sexo molecular
Se determinó el sexo de los individuos mediante la amplificación del gen de la ameloge-nina, que codifica para una proteína del esmalte dental y se encuentra situado en la región Xp22.3-p22.1 (cromosoma X) y en la región Yp11.2 (cromosoma Y). Durante la amplificación se obtuvo un segmento de 112pb para el cromosoma Y, mientras que el amplicón del cromosoma X presentó 106pb, esa diferencia de 6pb es notoria en la electroforesis en geles de poliacrilamida al 12%. Se utilizaron las mismas condiciones de ciclado que en HVR I pero utilizando los cebadores: F: 5'-CCCTGGGCTCT-GTAAAGAA-3' y R: 5'-ATCAGAGCTTA-AACTGGGAAAGAA-3' creados por el equipo de investigación y una temperatura de anidado de cebadores a 53ºC.

Prueba de inhibición
Para comprobar la presencia de sustancias inhibidoras en cada una de las muestras cuyas amplificaciones resultaban negativas se realizó una PCR de segmento B de HVR I empleando una muestra de ADN moderno para ser amplif-cada. Como control positivo se amplificó solo el ADN moderno. Aquellas reacciones en las que EIP (Periodo Intermedio Temprano) 260AC-640AD - MH (Horizonte Medio) 640-1180 AD tampoco fue posible amplificar la muestra moderna fueron consideradas como inhibidas por la presencia de sustancias en la muestra antigua que interferen en la PCR. En el caso de ampli-ficación positiva se infrió que la cantidad de ADNa era insuficiente o estaba degradado.
RESULTADOS
De las 23 piezas dentales analizadas se obtuvo ADN mitocondrial en 16 (recuperación del 69,6%), lo cual indica un buen estado de preservación. En 13 casos fue posible secuenciar y corroborar los tres segmentos solapantes de HVR I, en 2 se obtuvo secuencia y confrma-ción de los dos primeros segmentos y en uno del segundo y tercer segmento (Tabla 3). De las muestras restantes, 2 presentaron compuestos inhibitorios de la PCR y 5 no poseían ADN de calidad analizable.
Por otro lado se logró establecer el sexo molecular de 4 individuos, determinando que 2 eran masculinos y 2 femeninos (Tabla 3).
En cuanto a los linajes maternos, estuvieron representados los 4 Hgs amerindios. El A2 estuvo presente en un 43,75%, seguido del C1 en un 25%, el D1 en un 18,75% y por último el B2 en un 12,50%. Los resultados obtenidos por RFLP coincidieron con los haplotipos obtenidos por secuenciación de HVR I (Tabla 3).
Los 16 individuos presentaron haplotipos diferentes, dando como resultado una gran diversidad haplotípica en la muestra de Puna Jujeña, en comparación con los resultados de Pampa Grande, Quebrada de Humahuaca centro y sur. Además, el número de sitios polimórficos y la diferencia promedio entre pares de secuencias fueron los más elevados (Tabla 4).
Las comparaciones de los haplotipos obtenidos con las secuencias de otros trabajos dieron como resultado que DC1, DC2, DC12, DC18 y DC19 poseen linajes no descriptos hasta el momento en los estudios de la región. El de DC21 sólo fue hallado en una muestra de la Costa de Perú del Horizonte Temprano (EH) (Fehren-Schmitz et al., 2011). DC4 posee las mutaciones típicas del haplotipo A2 y el individuo DC6 las de C1 nodal (Tabla 3), ambos presentes en varias poblaciones de Sudamérica prehispánicas y actuales (Garcia-Bour et al., 2004; Kemp et al., 2009; Perego et al., 2010; Sala et al., 2010;
Bodner et al., 2012; Baca et al., 2012; 2014; Mendisco et al., 2014; Fehren-Schmitz et al., 2014). El DC14 mostró un linaje típico de la región andina presente en un individuo prehis-pánico de la Quebrada de Humahuaca sur (Men-disco et al., 2014) y en varias poblaciones actuales (Álvarez-Iglesias et al., 2007; Barbieri et al., 2011; de Saint Pierre et al., 2012).
Dentro de los linajes de A2, el individuo DC16 presentó un haplotipo A2+16125A, el cual está presente en una muestra antigua de Los Amarillos junto a la 16111T. DC3 tiene la mutación 16234G descripta en muestras modernas del NOA pero en las cuales está acompañada de otras mutaciones extras a la nodal, como es el caso del DC9 que además posee la 16256T y la 16192T (Tabla 3). El patrón 16234G+16256T está presente en muestras actuales de La Quiaca, una de Tapacari (Taboada-Echalar et al., 2013) y en una muestra de Jujuy (Cardoso et al., 2013). Por su parte el DC7 presentó un haplotipo similar al de individuos guaraníes (Marrero et al., 2007) pero con la mutación extra 16162G.
El resto de los individuos analizados correspondieron al linaje C1. El linaje de DC5 fue descripto en un individuo precolombino de la Costa de Perú del periodo Intermedio Tardío (LIP) (Fehren-Schmitz et al., 2014) y en Ayma-rá (de Saint Pierre et al., 2012) y Mbyá-Guara-níes (Sala et al., 2010) actuales, un individuo de Tucumán (Tamm et al., 2007) y en un Mapuche de Río Negro (Ginther et al., 1993). El de DC10 está presente en una muestra moderna de Brasil (Fridman et al., 2014) y el de DC11 en 3 individuos Tupí de lengua zoro (Ramallo et al., 2013).
Luego de realizar una comparación con las secuencias de 343 individuos prehispánicos divididos en 16 grupos poblacionales indicados en la tabla 2, se obtuvo como resultado que la variación genética promedio dentro de los grupos po-blacionales (84,35%) es mayor a la variación que existe entre ellos (15,65%). Sin embargo, esta última resultó significativa (p<0,00001) indicando que todos estos grupos no pueden considerarse una sola población, sino que la misma se encuentra estructurada y que existe una gran dispersión genética entre grupos, concordando con la amplitud territorial y diferencias temporales de las muestras empleadas en la comparación (Tabla 5).
La comparación de a pares de grupos poblacionales (FST) para determinar cuáles diferían entre sí, mostró que la Puna jujeña se diferencia de Pampa Grande (FST:0,10512; p-valor:0,01953) siendo la única que lo hace del actual territorio argentino. Es de destacar que la primera no presentó una diferenciación significativa con el resto de las muestras cercanas: Quebrada de Hu-mahuaca centro (FST:0,05598; p-valor:0,06934), Quebrada de Humahuaca sur (FST:0,05677; p-valor:0,09961), y Valle Calchaquí (FST:0,04050; p-valor:0,20898), mientras que de las muestras de Perú se diferenció significativamente de Tompullo II (FST:0,20337; p-valor:<0,00001), Acchaymarca (FST:0,11461; p-valor:0,02246), de Costa EIP (FST:0,07155; p-valor:0,00781), LIP (FST:0,07170; p-valor:0,03223), EH (FST:0,14003; p-valor:<0,00001) y de los Valles Altos del MH (FST:0,143109; p-valor:<0,00001) (Apendice suplementario 1; Tabla suplementaria 1). En el análisis de Neighbor Joining pudo observarse un agrupamiento de las muestras de la Tabla 2 según la frecuencia de los cuatro Hgs principales (A2, B2, C1 y D1). La Puna jujeña se encuentra próxima a las muestras de Quebrada de Humahuaca centro y Puca por la elevada proporción de A2 y la presencia de los otros tres linajes, mientras que el Valle Calchaquí quedó ubicado en un clado más alejado probablemente por no presentar D1. El grupo más alejado a Puna quedó compuesto por aquellas muestras con una mayor representación de haplotipos B2 (Fig.2). Este grupo es el más numeroso ya que ese linaje es el más frecuente entre las muestras de los Andes Centro-Sur.
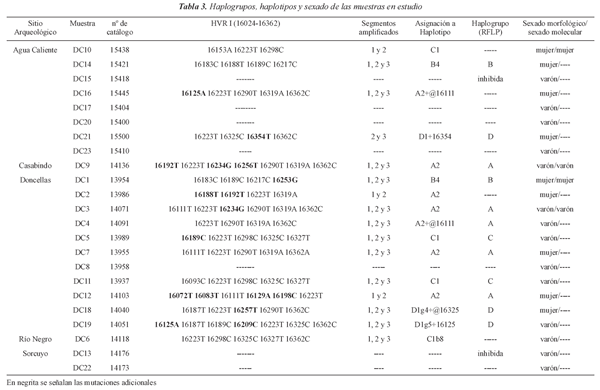

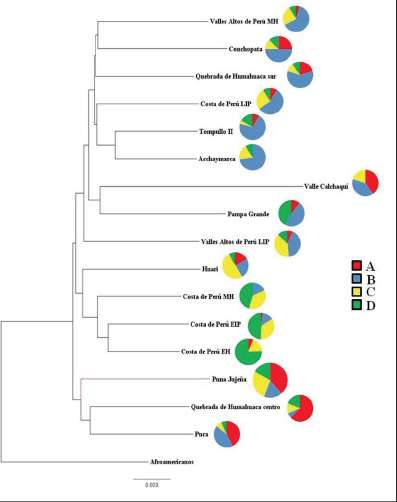
Fig. 2. Neighbor Joining de todas las muestras comparadas con la proporción de linajes que poseen.
DISCUSIÓN
El grado de recuperación de ADN mitocon-drial para la muestra de Puna Jujeña (69,6%) fue satisfactorio, según los cánones de ADNa. El mismo fue similar a los valores de otros estudios en la Quebrada de Humahuaca (66,67%), superior al del Valle Calchaquí (55,56%) (Mendisco et al., 2011; 2014) y menor al del sitio Pampa Grande (90,47%) (Carnese et al., 2010). Esta buena conservación del material genético se debe principalmente a las condiciones de humedad y temperatura de la región y a que se tuvo acceso a piezas dentales, en las cuales el esmalte ayuda a la conservación del ADN y funciona
como barrera física aislante ante el ataque de diversos agentes de degradación (Hofreiter et al., 2001; Gilbert et al., 2005; Crespo et al., 2010; Cardozo et al., 2014). Para el ADN nuclear no se obtuvo la misma eficiencia, lo cual es un problema propio de las investigaciones con muestras arqueológicas, debido a que se presenta un sólo ADN nuclear diploide por célula habiendo menos oportunidades de obtener fragmentos que puedan ser amplificados (Shapiro y Hofreiter, 2012), a diferencia del mitocondrial que está presente entre 2 y 10 copias por mitocondria.
Por otro lado, es importante destacar que los haplotipos de dos muestras del sitio Doncellas descriptos por Mendisco et al. (2014), no fueron encontrados en las muestras analizadas en este trabajo, enfatizando la gran variabilidad genética que podría haber estado presente en esa población. El elevado valor observado de la diversidad haplotípica (Hd=1±0,019), dado que los sitios analizados están cercanos geográfica-mente y fueron enmarcados según los fechados radiocarbónicos en el mismo periodo temporal (1000-1450 AD), podría estar indicando que la Puna de Jujuy habría recibido un importante fujo génico desde otras regiones. A ese respecto, hemos encontrado linajes presentes en varios grupos poblacionales prehispánicos de Perú y la Quebrada de Humahuaca como fue señalado anteriormente. Además, en muchos casos estos linajes se caracterizan por alguna mutación adicional al Hg nodal. Este hallazgo es concordante con la evidencia arqueológica acerca de un activo intercambio de bienes materiales entre distintas regiones del Área Andina Centro Meridional, como ser obsidiana para materia prima, artefactos para inhalación de alucinógenos, objetos de oro, valvas de moluscos marinos y cuentas (Fernández, 1978; Gentile, 1988; Tarragó, 1989; Nielsen, 2011; entre otros).
A una escala local, las distancias genéticas entre la Puna jujeña y otros grupos poblacio-nales del actual noroeste argentino no fueron significativas, a excepción de Pampa Grande, de la que se encuentra más alejada geográfica y espacialmente.
A nivel regional, las diferencias son significativas con 6 de los 11 grupos muestrales antiguos de Perú, a pesar de la existencia de haplotipos compartidos en ciertos casos. No se evidencia una relación directa con la distancia geográfica o temporal.
Es de destacar que este tipo de análisis estadístico no puede escapar a una limitación debida al bajo tamaño muestral frecuente en los estudios de ADNa, lo que afecta a la potencia de la prueba, y también que en algunas muestras no se han podido amplificar los tres segmentos del HVR I. Esta cuestión se intentará solucionar a futuro incorporando nuevas técnicas de amplif-cación. A título de ejemplo, entre la muestra de la Puna jujeña y la del Valle Calchaquí los valores de FST no fueron significativos (FST:0,04050; p-valor:0,20898), sin embargo en el árbol del Neighbor Joining (Fig.2) se posicionaron en grupos distintos.
En cuanto a los linajes hallados en las muestras prehispánicas, la Puna jujeña presenta mayor prevalencia de linajes A2, al igual que Los Amarillos y la muestra peruana de Puca, lo cual no es frecuente en poblaciones surandinas en las cuales suele encontrarse mayor prevalencia de B2 (Rothhammer et al., 2009; Kemp et al., 2009; Fehren-Schmitz et al., 2011; 2014; Baca et al., 2012; Dejean et al., 2014). Un ejemplo claro es Los Amarillos que se diferencia en sus frecuencias haplotípicas del resto de los sitios cercanos geográficamente dentro de la Quebrada y presenta una alta frecuencia de un linaje A2, por lo que Mendisco et al. (2011) consideran que esta población tuvo una historia demo-gráfica particular, diferente a la del resto de la Quebrada. Señalan también que existió intercambio de materiales entre la Quebrada de Hu-mahuaca y la Región de Gran Chaco y sostienen que podría haber estado acompañado de fujo génico (Mendisco et al., 2014). A este respecto, las evidencias fenotípicas mostraron que en la Quebrada de Humahuaca existe una diversidad genética mayor que la esperada para una región geográfica limitada (Cocilovo et al., 1999).
La comparación de los linajes encontrados en Puna antigua con muestras actuales de la región, mostró que ciertos haplotipos descriptos están presentes en éstas últimas, aunque diferan en una mutación adicional. Este es el caso del linaje de DC9, un Hg A2 que presenta las mutaciones 16234G y 16256T encontrado en Ju-juy y Bolivia, y que además posee la mutación 16192T. Otro ejemplo es el haplotipo del individuo DC7 el cual está presente en guaraníes actuales del NEA pero sin la mutación 16162G. Sólo uno de los individuos, DC14, presentó un linaje que ha sido hallado en un individuo precolombino de la Quebrada de Humahuaca (Mendisco et al., 2014), en muestras actuales de la Puna (Álvarez-Iglesias et al., 2007; Cardoso et al., 2013), en Wichis y Qom de la provincia de Formosa (Cabana et al., 2006), en individuos de la cuenca del lago Titicaca en Bolivia (Barbieri et al., 2011) y en Aymará y Ataca-meños de Chile (de Saint Pierre et al., 2012), siendo la única evidencia que hemos encontrado de continuidad genética a lo largo del tiempo. A este respecto, al analizar la continuidad genética deben considerarse que al comparar muestras antiguas y actuales existe la posibilidad de que surja una nueva mutación o una reversión de alguna de las existentes, debido al tiempo transcurrido.
Por otra parte, la posible incidencia de procesos demográficos como la contracción o reemplazo poblacional podría explicar la ausencia de muchos linajes antiguos en la actualidad. Si estimamos en 25 a 30 años el intervalo generacional tendríamos más de veinte generaciones de horizonte temporal para la potencial acción de estos procesos sobre las muestras actuales de la zona. A este respecto señalemos que tampoco puede obviarse el impacto de la conquista y colonización española en la población nativa, lo que incluye, entre sus numerosas consecuencias, una probable pérdida de linajes.
Para investigaciones futuras sería de gran utilidad disponer de mayor número de análisis de ADNa con descripción de linajes maternos en poblaciones prehispánicas de regiones cercanas a la Puna Jujeña como el Altiplano Boliviano, San Pedro de Atacama (Chile), las yungas y diferentes zonas del NEA, y así contribuir a un mejor entendimiento de las relaciones flogenéticas y procesos demográficos que moldearon la diversidad de linajes maternos que ha sido descripta en este estudio.
Por último, la gran ventaja que presenta el ADNa es que tiene el potencial de aportar información en el eje temporal sobre las relaciones biológicas entre las poblaciones del pasado, mientras que los análisis sobre muestras actuales sólo permiten realizar inferencias sobre lo que pudo haber sucedido, pero no es una información directa. Si se toman en cuenta ambos tipos de estudios, la relación entre grupos poblacionales de diferente temporalidad permite un mejor abordaje de los procesos demográficos que acontecieron en cada región.
CONCLUSIONES
Se estudiaron 23 individuos de la Puna de Jujuy (Argentina) obteniéndose resultados positivos para 16 de ellos, lo que representa una elevada proporción de recuperación en estudios de ADNa. Se registraron 16 linajes maternos diferentes, dando como resultado una gran diversidad haplotípica en la muestra de Puna jujeña, en comparación con los resultados de otros estudios, correspondientes a Quebrada de Humahuaca centro y sur y Pampa Grande. El linaje más frecuente fue el A2, a diferencia de otros estudios de la región andina donde se ha descripto el predominio de B2.
A nivel local la Puna Jujeña se diferenció significativamente de Pampa Grande por ser la muestra más alejada geográfica y temporalmente y a nivel regional, presentó también una diferenciación genética significativa con varias muestras de Perú, sin poder determinarse un patrón espacio-temporal específico que dé cuenta de esos resultados. A ese respecto, el bajo número de individuos que presentan la mayoría de los grupos muestrales analizados podría infuir en parte en los casos donde no hubo una significa-tiva diferenciación genética.
Por otro lado, en la comparación de los linajes descriptos con los de las muestras actuales, quedó demostrado que alguno de ellos están presentes en estas últimas, aunque diferan en una mutación adicional, dando evidencia de cierta continuidad biológica en la región.
Las relaciones genéticas mencionadas en este estudio entre la Puna jujeña y el resto de las muestras de los Andes Sur son concordantes, hasta el momento, con la evidencia arqueólogica descripta.
Por último, es importante destacar que el sexado molecular fue coincidente con el morfológico realizado en un estudio anterior, pudiendo ser utilizado como alterativa para tal determinación en individuos subadultos y juveniles.
A futuro se espera poder obtener la secuencia de HVR II del ADNmt o el mitogenoma completo, aumentar el número de marcadores moleculares analizados (STRS autosómicos y del cromosoma Y para aquellas muestras que sean masculinas) y el número de muestras po-blacionales a comparar con el fin de aportar mayor cantidad de datos para el entendimiento de los procesos microevolutivos, los movimientos migratorios y las relaciones biológicas entre las poblaciones que habitaban y habitan el actual NOA.
AGRADECIMIENTOS
Los autores desean agradecer sinceramente la ayuda prestada por el personal del Museo Et-nográfico "Juan Bautista Ambrosetti" y en particular a su Directora Dra. M. Tarragó y a Lic. C. Aranda por su colaboración durante la obtención de las muestras dentales. Asimismo, agradecer especialmente al Dr. José A. Cocilovo por todos sus aportes a esta investigación para el enriquecimiento de la misma. También deseamos agradecer al Dr. J. Valetti y a Diego Salonia por el diseño de las imágenes y a las Instituciones que brindaron su apoyo financiero.
LITERATURA CITADA
Achilli A, Perego UA, Bravi CM, Coble MD, Kong QP, Woodward SR, Salas A, Torroni A, Bandelt HJ. 2008. The phylogeny of the four pan-American mtDNA ha-plogroups: implications for evolutionary and disease studies. PLoS One 3(3):1764. doi:10.1371/journal. pone.0001764
Achilli A, Perego UA, Lancioni H, Olivieri A, Gandini F, Hooshiar Kashani B, Battaglia V, Grugni V, Angerhofer N, Rogers MP, Herrera RJ, Woodward SR, Labuda D, Smith DG, Cybulski JS, Semino O, Malhi RS, Torroni A. 2013. Reconciling migration models to the Americas with the variation of North American native mitogeno-mes. PNAS USA 110(35):14308-14313. doi: 10.1073/ pnas.1306290110
Adachi N, Umetsu K, Takigawa W, Sakaue K. 2004. Phylo-genetic analysis of the human ancient mitochondrial DNA. J Archaeol Sci 31(10):1339-1348. doi: 10.1016/j. jas.2004.02.011
Albeck ME, Ruiz MS. 2003. El tardío en la Puna de Jujuy: poblados, etnias y territorios. Cuadernos FHyCS-UNJu 20:199-221. [ Links ]
Albeck ME. 2007. El intermedio Tardío: interacciones económica y políticas en la Puna de Jujuy. En: Williams V, Ventura B, Callegari A, Yacobaccio H, editores. Sociedades precolombinas surandinas: temporalidad, interacción y dinámica cultural del NOA en el ámbito de los Andes Centro-Sur. Buenos Aires: Artes Gráficas Buschi SA. pp. 125-145.
Alfaro LC. 1983. Investigación arqueológica en la cuenca del río Doncellas (provincia de Jujuy). Integración de la Puna jujeña a los centros cúlticos andinos. Relaciones de la Sociedad Argentina de Antropología XV:25-47.
Álvarez-Iglesias V, Jaime JC, Carracedo Á, Salas A. 2007. Coding region mitochondrial DNA SNPs: targeting east Asian and Native American haplogroups. Forensic Sci Int Genet 1:44-55. doi: 10.1016/j.fsigen.2006.09.001
Andrews RM, Kubacka I, Chinnery PF, Lightowlers RN, Turnbull DM, Howell N. 1999. Reanalysis and re-vision of the Cambridge reference sequence for human mitochondrial DNA. Nat Genet 23(2):147. doi: 10.1038/13779
Baca M, Doan K, Sobczyk M, Stankovic A, Weglenski P. 2012. Ancient DNA reveals kinship burial patterns of a pre-Columbian Andean community. BMC Genet 13:30. doi: 10.1186/1471-2156-13-30
Baca M, Molak M, Sobczyk M, Weglenski P, Stankovic A. 2014. Locals, resettlers, and pilgrims: a genetic portrait of three pre-Columbian Andean populations. Am J Phys Anthropol 154(3):402-412. doi:10.1002/ajpa.22524
Barbieri C, Heggarty P, Castri L, Luiselli D, Pettener D. 2011. Mitochondrial DNA Variability in the Titicaca Basin: Matches and Mismatches with Linguistic and Ethnohistory. Am J Hum Biol 23:89-99. [ Links ] doi:10.1002/ ajhb.21107
Berenguer J y Dauelsberg P. 1989. El Norte Grande en la órbita de Tiwanaku (400 a 1.200 d.C.). En: Hidalgo J, Schiappacasse V, Niemeyer H, Aldunate C, Solimano I. Culturas de Chile. Prehistoria. Desde sus orígenes hasta los albores de la conquista. Santiago de Chile: Editorial Andrés Bello. pp.129-180.
Bodner M, Perego UA, Huber G, Fendt L, Röck AW, Zi-mmermann B, Olivieri A, Gómez-Carballa A, Lancio-ni H, Angerhofer N, Bobillo MC, Corach D, Wood-ward SR, Salas A, Achilli A, Torroni A, Bandelt H-J, Parson W. 2012. Rapid coastal spread of First Americans: Novel insights from South America`s Southern Cone mitochondrial genomes. Genome Res 22:811-820. doi: 10.1101/gr.131722.111
Cabana GS, Merriwether DA, Hunley K, Demarchi DA. 2006. Is the genetic structure of Gran Chaco popula-tions unique? Interregional perspectives on native South American mitochondrial DNA variation. Am J Phys Anthropol 131(1):108-119. doi:10.1002/ajpa.20410
Cardoso S, Palencia-Madrid L, Valverde L, Alfonso-Sánchez MA, Gómez-Pérez L, Alfaro E, Bravi CM, Dipierri JE, Peña JA, de Pancorbo MM. 2013. Mitochondrial DNA control region data reveal high prevalence of Native American lineages in Jujuy province, NW Argentina. Forensic Sci Int Genet 7(3):52-5. doi: 10.1016/j.fsi-gen.2013.01.007
Cardozo DG, Crespo CM, Russo MG, Postillone MB. 2014. Análisis y conservación de ADN antiguo en restos es-queletales humanos de sitios arqueológicos y colecciones de museos de Argentina. En: Esnal AC, Funes ML, Grosso M, Kuperszmit N, Murgo A, Romero G, editores. Entre Pasados y Presentes IV. Estudios Contemporáneos en Ciencias Antropológicas. Ciudad Autónoma de Buenos Aires: Editorial Asociación Amigos del Instituto Nacional de Antropología (AINA): 313-329. ISBN 978-987-25575-3-9
Carnese F, Mendisco F, Keyser C, Dejean CB, Dugoujon JM, Bravi CM, Ludes B, Crubézy E. 2010. Paleogeneti-cal study of pre-Columbian samples from Pampa Grande (Salta, Argentina). Am J Phys Anthropol 141:452-462. doi: 10.1002/ajpa.21165
Cocilovo JA, Varela HH, Baff EI, Valdano SG. 1999. Estructura y composición de la población antigua de la Quebrada de Humahuaca. Análisis multivariado. Rev Arg Antrop Biol 2(1):7-26.
Cocilovo JA, Varela HH, O`Brien TG. 2009. La divergencia genética entre poblaciones del área andina centro meridional evaluada mediante rasgos no métricos del cráneo. Rev Arg Antrop Biol 11 (1):43-59.
Crespo CM, Dejean CB, Postillone MB, Lanata JL, Carnese FR. 2010. Historias en código genético. Los aportes de los estudios de ADN antiguo en antropología y sus implicancias éticas. RUNA XXXI (2):153-174.
Dejean CB, Seldes V, Russo MG, Mendisco F, Keyser C, Ludes B, Carnese FR. 2014. Variabilidad genética mito-condrial: comparación de muestras de dos sitios arqueológicos del noroeste argentino. Rev Arg Antrop Biol 16 (1):5-16. doi: 10.17139/raab.2014.0016.01.02
de Saint Pierre M, Bravi CM, Motti JMB, Fuku N, Tanaka M, Llop E, Bonatto SL, Moraga M. 2012. An alternati-ve model for the early peopling of Southern South America revealed by analyses of three mitochondrial DNA haplogroups. PLoS ONE 7 (9):43486. doi: 10.1371/ journal.pone.0043486
Escola PS. 2007. Obsidianas en contexto: tráfico de bienes, lazos sociales y algo más. En: Williams V, Ventura B, Callegari A, Yacobaccio H, editores. Sociedades precolombinas surandinas: temporalidad, interacción y dinámica cultural del NOA en el ámbito de los Andes Centro-Sur. Buenos Aires: Artes Gráficas Buschi SA. pp. 73-87.
Excoffer L, Laval G, Schneider S. 2005. ARLEQUIN ver. 3.0: an integrated software package for population ge-netics data analysis. Evolutionary Bioinformatics On-line 1:47-50.
Fehren-Schmitz L, Reindel M, Tomasto-Cagigao E, Hummel S, Herrmann B. 2010. Pre-Columbian popu-lation dynamics in coastal southern Peru: a diachronic investigation of mtDNA patterns in the Palpa region by ancient DNA analysis. Am J Phys Anthropol 141:208-221. doi: 10.1002/ajpa.21135
Fehren-Schmitz L, Warnberg O, Reindel M, Seidenberg V, Tomasto-Cagigao E, Isla-Cuadrado J, Hummel S, Herrmann B. 2011. Diachronic investigations of mi-tochondrial and Y-chromosomal genetic markers in pre-Columbian Andean highlanders from south Peru. Ann Hum Genet 75(2):266-283. doi: 10.1111/j.1469-1809.2010.00620.x
Fehren-Schmitz L, Haak W, Mächtle B, Masch F, Llamas B, Tomasto-Cagigao E, Sossna V, Schittek K, Isla-Cuadrado J, Eitel B, Reindel M. 2014. Climate chan-ge underlies global demographic, genetic, and cultural transitions in pre-Columbian southern Peru. Proc Natl Acad Sci USA 111(26): 9443-9448. doi: 10.1073/ pnas.1403466111
Fernandez J. 1978. Los chichas, los lipes y un posible enclave de la cultura de San Pedro de atacama en la Puna limítrofe argentino-boliviana. Estud atacam 6:21-36.
Fridman C, Gonzalez RS, Pereira AC, Cardena MMSG. 2014. Haplotype diversity in mitochondrial DNA hy-pervariable region in a population of southeastern Brazil. Int J Legal Med 128(4): 589-593. doi: 10.1007/ s00414-014-1023-z
Fuchs ML, Varela HH. 2013. Fechados radiocarbónicos de colecciones osteológicas de Puna de Jujuy, Argentina. Relaciones de la Sociedad Argentina de Antropología XXXVIII (2): 553-558.
Fuchs ML, Varela HH, Cocilovo JA. 2016. Kinship and phenotypic divergence in the ancient population of the Puna plateu of northwestern Argentina. Advances in Anthropology 6: 1-10. doi: 10.4236/aa.2016.61001
Gabriel MN, Huffine EF, Ryan JH, Holland MM, Parsons TJ. 2001. Improved mtDNA sequence analysis of fo-rensic remains using a "mini-primer set" amplification strategy. J Forensic Sci 46(2):247-253. doi: 10.1520/ JFS14957J
García-Bour J, Pérez-Pérez A, Álvarez S, Fernández E, López-Parra AM, Arroyo-Pardo E, Turbón D. 2004. Early population differentiation in extinct aborigines from Tierra del Fuego-Patagonia: ancient mtDNA se-quences and Y-chromosome STR characterization. Am J Phys Anthropol 123:361-370. doi: 10.1002/ajpa.10337
Gilbert MT, Bandelt H-J, Hofreiter M, Barnes I. 2005. As-sessing ancient DNA studies. Trends Ecol Evol 20(10): 541-544. doi: 10.1016/j.tree.2005.07.005
Ginther C, Corach D, Penacino GA, Rey JA, Carnese FR, Hutz MH, Anderson A, Just J, Salzano FM, King M-C. 1993. Genetic variation among the Mapuche indians from the Patagonian region of Argentina: mitochondrial DNA sequence variation and allele frequencies of seve-ral nuclear genes. EXS 67:211-219. doi: 10.1007/978-3-0348-8583-6_17
González AR y Pérez JA. 1972. Argentina indígena: vísperas de la conquista. Buenos Aires: Editorial Paidos.
Hall TA. 2013. BioEdit: Biological sequence alignment editor for Win 95/98/NT/2K/XP/7. http://www.mbio.ncsu.edu/bioedit/bioedit.html
Hofreiter M, Davis S, Poinar H, Kuch M, Pääbo S. 2001. Ancient DNA. Nat Rev Genet 2(5):353-359. doi:10.1038/35072071
Ivanov PL, Wadhams MJ, Roby RK, Holland MM, Weedn VW, Parsons TJ. 1996. Mitochondrial DNA sequence heteroplasmy in the Grand Duke of Russia Georgij Romanov establishes the authenticity of the remains of Tsar Nicholas II. Nat Genet 12(4):417-420. doi: 10.1038/ng0496-417
Kemp BM, Tung TA, Summar ML. 2009. Genetic continui-ty after the collapse of the Wari Empire: mitochondrial DNA profles from Wari and post-Wari populations in the ancient Andes. Am J Phys Anthropol 140(1):80-91. doi: 10.1002/ajpa.21037
Kemp BM y Smith DG. 2010. Ancient DNA methodology: thoughts from Brian M. Kemp and David Glenn Smith on "Mitochondrial DNA of protohistoric remains of an Arikara population from South Dakota". Hum Biol 82 (2):227-238. doi:10.3378/027.082.0207
Kloss-Brandstätter A, Pacher D, Schönherr S, Weissens-teiner H, Binna R, Specht G, Kronenberg F. 2011. Ha-ploGrep: a fast and reliable algorithm for automatic classification of mitochondrial DNA haplogroups. Hum Mutat 32(1):25-32. doi:10.1002/humu.21382
Marrero AR, Silva-Junior WA, Bravi CM, Hutz MH, Petzl-Erler ML, Ruiz-Linares A, Salzano FM, Bortolini MC. 2007. Demographic and evolutionary trajectories of the Guarani and Kaingang natives of Brazil. Am J Phys An-thropol 132(2):301-310. doi:10.1002/ajpa.20515
Mendisco F, Keyser C, Hollard C, Seldes V, Nielsen A, Crubézy E, Ludes B. 2011. Application of the iPLEXTM Gold SNP genotyping method to the analy-sis of Amerindian ancient DNA samples: benefts for ancient population-based studies. Electrophoresis 32:386-393. doi: 10.1002/elps.201000483
Mendisco F, Keyser C, Seldes V, Rivolta C, Mercolli P, Cruz P, Nielsen AE, Crubezy E, Ludes B. 2014. Genetic di-versity of a late prehispanic group of the Quebrada de Humahuaca, Northwestern Argentina. Ann Hum Genet 78(5):367-380. doi: 10.1111/ahg.12075
Motti JMB, Ramallo V, Muzzio M, Bailliet G, Bravi CM. 2014. El ADN mitocondrial como indicador de relaciones biológicas entre poblaciones antiguas y actuales del NOA. En: Luna L, Aranda C, Suby J, editores. Avances recientes de Bioarqueología Latinoamericana. Buenos Aires: GIB. pp. 267-283.
Muscio HJ. 2001. Una revisión crítica del Arcaico surandi-no. Ficha de cátedra fundamentos de prehistoria: oficina de publicaciones de la Facultad de Filosofía y Letras (OPFyL). https://ecaths1.s3.amazonaws.com/argenti-na1/Arcaico%20Surandino%20(Muscio).pdf
Nei M. 1987. Molecular evolutionary genetics. Columbia: Columbia University Press.
Nielsen A. 2000. Andean caravans: an ethnoarchaeology. Tesis Doctoral. Universidad de Arizona. http://arizona. openrepository.com/arizona/handle/10150/289098
Nielsen AE. 2011. El tráfico de caravanas entre Lípez y Ata-cama visto desde la Cordillera occidental. En: Núñez Atencio L, Nielsen AE, editores. En ruta: arqueología, historia y etnografía del tráfico sur andino. Córdoba: Encuentro Grupo Editor. Pp.:83-110.
Núñez Atencio L. 2007. Refexiones sobre el tráfico de caravanas y complementariedad circumpuneña. En: Williams V, Ventura B, Callegari A, Yacobaccio H, editores. Sociedades precolombinas surandinas: temporalidad, interacción y dinámica cultural del NOA en el ámbito de los Andes Centro-Sur. Buenos Aires: Artes Gráficas Buschi SA. pp. 33-58.
Pääbo S, Poinar H, Serre D, Jaenicke-Despres V, Hebler J, Rohland N, Kuch M, Krause J, Vigilant L, Hofreiter M. 2004. Genetic analyses from ancient DNA. Annu Rev Genet 38:645-679. doi: 10.1146/annurev.ge-net.37.110801.143214
Perego UA, Achilli A, Angerhofer N, Accetturo M, Pala M, Olivieri A, Kashani BH, Ritchie KH, Scozzari R, Kong QP, Myres NM, Salas A, Semino O, Bandelt HJ, Wood-ward SR, Torroni A. 2009. Distinctive Paleo-Indian mi-gration routes from Beringia marked by two rare mtD-NA haplogroups. Curr Biol 19(1):1-8. doi:10.1016/j. cub.2008.11.058
Perego UA, Angerhofer N, Pala M, Olivieri A, Lancioni H, Ho-oshiar Kashani B, Carossa V, Ekins JE, Gómez-Carballa A, Huber G, Zimmermann B, Corach D, Babudri N, Panara F, Myres NM, Parson W, Semino O, Salas A, Woodward SR, Achilli A, Torroni A. 2010. The initial peopling of the Americas: a growing number of foun-ding mitochondrial genomes from Beringia. Genome Res 20(9):1174-1179. doi:10.1101/gr.109231.110
Politis GG, Prates L, Perez SI. 2010. El poblamiento de América. Arqueología y bio-antropología de los primeros americanos. Buenos Aires: Eudeba. Colección Ciencia Joven 35.
Ramallo V, Bisso-Machado R, Bravi C, Coble MD, Salzano FM, Hünemeier T, Bortolini MC. 2013. Demographic expansions in South America: enlightening a complex scenario with genetic and linguistic data. Am J Phys Anthropol 150(3): 453-463. doi:10.1002/ajpa.22219
Ricaut FX, Keyser-Tracqui C, Bourgeois J, Crubézy E, Ludes B. 2004. Genetic analysis of a Scytho-Siberian skeleton and its implications for ancient central Asian migrations. Hum Biol 76(1):109-125. doi:10.1353/ hub.2004.0025
Rivera M. 1991. The prehistory of northern Chile: a synthe-sis. J World Prehist 5:1-47. doi:10.1007/BF00974731
Rothhammer F, Santoro CM, Poulin E, Arriaza BT, Moraga M, Standen VJ. 2009. Archeological and mtDNA evi-dence for Tropical Lowland migrations during the late Archaic/Formative in northern Chile. Rev Chil Hist Nat 82:543-552. doi: 10.4067/S0716-078X2009000400008
Rozas J, Librado P, Sánchez-DelBArrio JC, Messeguer X, Rozas R. 2010. DnaSP, DNA Sequence polymorphism versión 5.10.1. Universidad de Barcelona. http://www.ub.edu/dnasp/
Sala A, Argüelles CF, Marino ME, Bobillo C, Fenocchio A, Corach D. 2010. Genetic analysis of six com-munities of Mbyá-Guaraní inhabiting Northeastern Argentina by means of nuclear and mitochondrial polymorphic markers. Hum Biol 82(4):433-456. doi:10.3378/027.082.0406
Sala A y Corach D. 2014. Analysis of admixture and genetics structure of two Native American groups of Southern Argentinean Patagonia. Mol Biol Rep 41:1533-1543. doi: 10.1007/s11033-013-2999-z
Shapiro B, Hofreiter M. 2012. Ancient DNA: Methods and Protocols. Methods in molecular biology 840. Nueva York: Huamana Press. Springer Protocols. doi: 10.1007/978-1-61779-516-9
Taboada-Echalar P, Álvarez-Iglesias V, Heinz T, Vidal-Bralo L, Gómez-Carballa A, Catelli L, Pardo-Seco J, Pastoriza A, Carracedo Á, Torres-Balanza A, Rocabado O, Vullo C, Salas A. 2013. The genetic legacy of the pre-Colonial period in contemporary bolivians. PLoS ONE 8(3):58980. doi:10.1371/journal.pone.0058980
Tamm E, Kivisild T, Reidla M, Metspaul M, Glenn Smith D, Mulligan CJ, Bravi C, Rickards O, Martinez-Labarga C, Khusnutdinova EK, Fedorova SA, Golubenko M V, Stepanov VA , Gubina MA, Zhadanova SI, Ossipova LP, Damba L, Voevoda ML, Dipierri JE, Villems R, Malhi RS.2007. Beringian standstill and spread of native American founders. PLoS One 2(9):1-6. doi: 10.1371/journal.pone.0000829
Tamura K y Nei M. 1993. Estimation of the number of nu-cleotide substitutions in the control region of mitochon-drial DNA in humans and chimpanzees. Mol Biol Evol 10(3):512-526.
Tamura K, Stecher G, Peterson D, Filipski A, Kumar S. 2013. MEGA 6: Molecular evolutionary genetics analy-sis version 6.0. Mol Biol Evol 30: 2725-2729. doi: 10.1093/molbev/mst197
Tarragó M.1984. La historia de los pueblos circumpuneños en relación con el altiplano y los Andes Meridionales. Estud Atacam 7:93-104.
Tarragó M. 1989. Contribución al conocimiento arqueológico de las poblaciones de los oasis de San Pedro de Atacama en relación con los otros pueblos puneños, en especial, el sector septentrional del Valle Calchaquí. Tesis Doctoral. Facultad de Humanidades y Artes. Universidad Nacional de Rosario.
Torres-Rouff C, Pimentel G, Ugarte M. 2012. ¿Quienes Viajaban? Investigando la muerte de viajeros prehis-pánicos en el desierto de Atacama (CA. 800 AC- 1536 DC). Estud Atacam 43:167-186. doi:10.4067/S0718-10432012000100009
Varela HH, González MF, Torres MF, Cocilovo JA. 2004. Estructura de las poblaciones prehistóricas del Noroeste Argentino (sector septentrional). Distribución de características epigenéticas. Rev Arg Antrop Biol 6(1):77-102.
Varela HH, O´Brien TG, Cocilovo JA. 2008. The genetic divergence of prehistoric populations of the South-Central Andes as established by means of craniometric traits. Am J Phys Anthropol 137: 274-282. [ Links ] doi:10.1002/ ajpa.20867
Apéndice suplementario 1
















