Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Revista argentina de antropología biológica
versión On-line ISSN 1514-7991
Rev Arg Antrop Biol vol.19 no.1 La Plata jun. 2017
http://dx.doi.org/10.17139/raab.2017.0019.01.04
TRABAJOS DEL DOSSIER
LINAJES MITOCONDRIALES EN MUESTRAS DE ESQUINA DE HUAJRA (JUJUY, ARGENTINA). APORTES AL ESTUDIO DE LA OCUPACIÓN INCAICA EN LA REGIÓN Y LA PROCEDENCIA DE SUS HABITANTES
María G. Russo1,2*, María S. Gheggi2,3, Sergio A. Avena1,2, Cristina B. Dejean1,4, María B. Cremonte2,5
1Equipo de Antropología Biológica. Departamento de Ciencias Naturales y Antropológicas, CEBBAD. Fundación de Historia Natural Félix de Azara. Universidad Maimónides. CABA. Argentina
2CONICET Etnográfico J. B. Ambrosetti. Facultad de Filosofía y Letras. Universidad de Buenos Aires. CABA. Argentina
3Centro Regional de Investigaciones Científicas y Transferencia Tecnológica de La Rioja. Anillaco. Argentina
4Sección de Antropología Biológica. Instituto de Ciencias Antropológicas. Facultad de Filosofía y Letras. Universidad de Buenos Aires. CABA. Argentina 5CONICET CIT-Jujuy. Universidad Nacional de Jujuy. San Salvador de Jujuy. Argentina
*Correspondencia a: M. Gabriela Russo. Hidalgo 775, 6to piso, Laboratorio 602 (C1405BCK) Ciudad Autónoma de Buenos Aires. E-mail: russo.mariagabriela@maimonides.edu
Financiamiento: UBACyT 20020100100072. CONICET PIP2511. ANPCYT PICT 0649. Fundación de Historia Natural Félix de Azara. Fundación Científica Felipe Fiorellino.
Recibido 26 Mayo 2015; aceptado 8 Octubre 2015
doi:10.17139/raab.2017.0019.01.04
RESUMEN El sitio arqueológico Esquina de Huajra (centrosur de la Quebrada de Humahuaca) corresponde a una instalación ubicada cronológicamente en la Fase Inca (ca. 500-420 AP) y la tradicionalmente asignada en el noroeste argentino como Hispano Indígena o de primeros contactos con el español habría sido clave en la explotación y distribución de bienes procedentes de las Yungas Orientales. Además, su cultura material sugiere importantes redes de interacción con las tierras altas, planteando interrogantes sobre la conformación poblacional de sus habitantes.En este trabajo se analizan los linajes maternos de individuos de Esquina de Huajra en comparación con otros sitios del centro-sur andino para evaluar su posible origen foráneo, considerando que el reasentamiento de poblaciones fue una práctica imperial recurrente. El ADN fue extraído de piezas dentales de 6 individuos. Se secuenció la Región Hipervariable I del ADN mitocondrial en cuatro individuos hallándose dos A2 y dos C1. La ausencia de B2 discrepa con lo descripto para gran parte de los sitios andinos, donde este haplogrupo es mayoritario. Dos de los cuatro haplotipos se comparten únicamente con otros sitios de la Quebrada de Humahuaca del Período de Desarrollos Regionales (Los Amarillos y San José); pero una variante nodal C1 fue reportada tanto en Juella como en sitios peruanos preincaicos e incaicos. Estos resultados representan un primer aporte al estudio genético de los habitantes de Esquina de Huajra y una evidencia más de los complejos procesos poblacionales en la región andina.
PALABRAS CLAVE ADN antiguo; haplotipos mitocondriales; Quebrada de Humahuaca; período incaico
ABSTRACT The archaeological site Esquina de Huajra (south-central area of the Quebrada de Humahuaca) is a location chronologically assigned to the Inca phase (ca. 500-420 AP) and to the Hispanic-indigenous phase or initial period of contact with the Spanish (ca. 420-320 AP). In the context of the Incaic economic system, Esquina de Huajra may have been a key settlement in the exploitation and distribution of goods from the eastern forests (Yungas). In addition, its material culture suggests major interaction networks with the highlands, raising questions about its inhabitants' origin. In this work maternal lineages of individuals from Esquina de Huajra were analyzed in comparison to other sites in southcentral Andes to assess possible foreign origins, considering that population resettlement under Inca dominion was a common practice. DNA was extracted from the teeth of six individuals and mitochondrial hypervariable region I (HVRI) was successfully sequenced in four samples, resulting in two of them assigned to lineage A2 and the other two to C1. Absence of B2 disagrees with data from most Andean sites, where this lineage is the most frequent. Two of the four haplotypes are only shared with individuals from other sites located in Quebrada de Humahuaca (Los Amarillos and San Jose), and a nodal C1 variant was reported in samples from Juella and several pre-Inca and Inca Peruvian sites. These results represent the first contribution to genetic studies on the population of Esquina de Huajra and are also further evidence of the complex population processes in the Andean region.
KEY WORDS ancient DNA; mitochondrial haplotypes; Humahuaca Valley; Incaic period
El sitio arqueológico Esquina de Huajra (SjujTum10) está situado aproximadamente a 1900 msnm, en longitud oeste a 65º 27' 20'' y latitud sur a 23º 52' 56,30''. Este asentamiento, localizado en el sector centro-sur de la Quebrada de Humahuaca, posee una ubicación estratégica frente a la entrada de la quebrada subsidiaria de Huajra, uno de los accesos naturales más directos e importantes hacia los valles orientales (Yungas) (Fig. 1). Los resultados de los análisis radiocarbónicos indican que el sitio fue ocupado desde ca. el año 600 AP en adelante, es decir durante el Período Inca, pasando por el Hispano-Indígena y hasta el Colonial temprano (Garay de Fumagalli y Cremonte, 2007; Cremonte y Gheggi, 2012). El análisis estadístico de los nueve fechados de Esquina de Huajra recientemente realizado indica que habría sido ocupado por poco más de dos siglos durante el Período Incaico (ca. 500-420 AP) y el Hispano Indígena (ca. 420-320 AP) (Greco, 2014).
Las investigaciones llevadas a cabo hasta el momento permiten incluir a Esquina de Huajra en un sistema de asentamientos que, junto con el Pukará de Volcán, habrían vinculado el sector meridional de la Quebrada de Humahuaca con enclaves productivos y de frontera localizados en las Yungas Orientales donde se emplazan El Cucho de Ocloyas y sitios de la cuenca del río Ocloyas, cuenca superior del río Corral de Piedras, ríos Tiraxi y Tesorero. Esta vinculación se vería favorecida por la cercanía que existe entre el sector sur de la Quebrada de Humahuaca y los pisos de Yunga y Bosque Montano mediante vías de circulación directas (Cremonte y Garay de Fumagalli, 1997; Cremonte, 2007) (Fig. 1).
Entre la alfarería de Huajra se destacan las formas típicamente incaicas (ollas con pie, arí-balos, platos, etc.) y la vajilla no local (Inca Paya, Inca Pacajes, Chicha, Pucos Bruñidos, Queta Polícromo, etc.) (Cremonte y Scaro, 2011). También se halló un alfler o topu con una rara aleación de cobre y plata sugiriendo un origen lejano, posiblemente de los Andes Centrales (Angiorama, 2013). Adicionalmente, Mengoni Goñalons (2013) ha señalado que posiblemente tanto Esquina de Huajra como el Pucará de Volcán hayan sido lugares de obtención de llamas cargueras, vinculadas con el transporte de mercancías.
Por otra parte, la cerámica del Cucho de Ocloyas presenta escasas vasijas típicas de la Quebrada de Humahuaca en negro sobre rojo, un conjunto importante de recipientes con improntas de cordelería, corrugados complejos, unguiculados, fabricados en el lugar o traídos de zonas más orientales y fragmentos Humahuaca-Inca, Chicha Morado y Chicha Naranja natural, así como fragmentos muy pulidos (rojos, negros, castaños y naranjas) de escudillas o platos incaicos (Garay de Fumagalli y Cremonte, 2007). Debido a las características constructivas y el emplazamiento del sitio, es posible plantear que haya sido construido como una pequeña guarnición de frontera a mediados del S XVI con el objetivo de manejar y ordenar las relaciones con los habitantes de las tierras bajas. Esto permite plantear que en este sector, se habrían establecido fuertes vinculaciones entre las comunidades de la Quebrada de Humahuaca con pobladores de tradición chaqueña bajo el control Inca, que a su vez habrían mediatizado ancestrales interacciones con poblaciones de la llanura oriental (Garay de Fumagalli 2003; Cremonte y Garay de Fumagalli 2013).
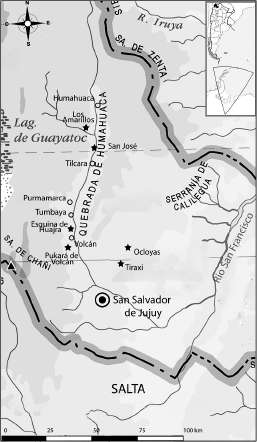
Fig. 1. Localización de los sitios mencionados en el texto. Las estrellas indican los sitios arqueológicos y los círculos los poblados actuales.
Aproximadamente a 20km al oeste de El Cucho de Ocloyas se encuentran los sitios Álvarez Prado 1 y 2 (AP1 y AP2) y Lagunita, que componen el sistema Tiraxi. Estos sitios se localizan a 1900 msmn aproximadamente sobre el piso de las yungas y los fechados obtenidos indican que fueron emplazados durante el momento Inca. Hacia los 1700 msnm, en la base del Bosque Montano, se localizan varios sitios semiconglomerados como Puesto Méndez, La Bolsa, Piedra Parada y Media Loma, ubicados en la cuenca del arroyo Sauzal. El fechado obtenido para La Bolsa, indicaría su contemporaneidad con el sitio AP1. Entre la cerámica hallada en AP1 y en La Bolsa se encuentran atributos incaicos como los diseños en espirales, banderines, bandas reticuladas, superficies pulidas y un fragmento de plato-pato. También se hallaron fragmentos de estilos Yavi o Chicha, Angosto Chico Inciso, corrugados y Humahuaca Negro sobre Rojo. Es posible que estos sitios cumplieran una función extractiva en este sector de Yungas (Cremonte y Garay de Fumagalli 2013).
Esta información permitiría involucrar a Esquina de Huajra dentro de una compleja dinámica poblacional producto de su establecimiento como un sitio que articulaba los objetivos del Imperio en el sector sur de la Quebrada de Hu-mahuaca, los cuales pudieron incluir obtención de mano de obra para la producción de maíz, papa semilla, extracción de recursos de las Yungas como cebil, plumas de colores, plantas medicinales, madera, y el reforzamiento de la frontera oriental. De acuerdo con las dataciones obtenidas hasta el momento, en el sector sur del qollasuyu la estructuración de la frontera oriental parece ser un proceso tardío, que no implicó la conformación de un cordón defensivo, sino más bien un espacio conectivo con una gran dinámica poblacional, cumpliendo objetivos de carácter preventivo y de extracción productiva (Cremonte y Garay de Fumagalli, 2013).
Considerando además que una práctica frecuente del Imperio fue el traslado y reasentamiento de poblaciones, cabe preguntarse acerca de la constitución de la población asentada en Esquina de Huajra. En este sitio pudieron habitar individuos provenientes de la misma quebrada, de las yungas adyacentes o de poblaciones foráneas relocalizadas. Surge entonces el interrogante sobre si esta posible coexistencia de grupos poblacionales de distintas regiones quedó plasmada en el acervo génico de la población de Esquina de Huajra.
Durante la última década se han incrementado los análisis de ADN antiguo (ADNa), es decir aquellos en los que se extrae el material genético de restos arqueológicos, tanto en el actual territorio argentino como en el resto de Sudamérica (e.g. Lalueza-Fox et al., 1997; Rothhammer et al., 2003, 2009; García-Bour et al., 2004; Dejean et al., 2006, 2008; Luciani et al., 2006; Silva et al., 2008; Kemp et al., 2009; Carnese et al., 2010; Fehren-Schmitz et al., 2010, 2011, 2014; Gonçalves et al., 2010, 2013; Casas-Vargas et al., 2011; Figueiro, 2011; Mendisco et al., 2011, 2014; Nores y Demarchi, 2011; Baca et al., 2012, 2014; Sans et al., 2012; Postillone et al., 2014 y referencias allí citadas). Estos trabajos constituyen un aporte fundamental al estudio de la dinámica de las poblaciones en tiempos prehispánicos desde una perspectiva genética, siendo el ADN mitocondrial (ADNmt) la principal herramienta utilizada. Teniendo en cuenta que se ha establecido que solo 8 de sus variantes (denominadas haplogrupos A2, B2, C1b, C1c, C1d, C4c, D1 y D4h3a) han ingresado a Sudamé-rica con los primeros grupos fundadores (Achilli et al., 2013 y referencias allí citadas), los análisis de ADNa se centran en la identificación de estos haplogrupos en poblaciones prehispánicas. Adi-cionalmente, ha aumentado la cantidad de estudios que analizan la variabilidad genética del ADNmt a través de la secuenciación de la Región Hiper-variable I (RHVI), lo que posee un mayor poder resolutivo y comparativo al permitir establecer posibles vínculos entre distintas poblaciones a través de los linajes (haplotipos) mitocondriales de los individuos que las componen.
Con el objetivo de comenzar a responder los interrogantes anteriormente planteados, en este trabajo se estudiaron los linajes maternos mediante el análisis de los haplotipos mitocondria-les de individuos de Esquina de Huajra a fin de compararlos con los encontrados en otras poblaciones con afinidad tanto espacial como temporal, intentando dilucidar los posibles orígenes y movimientos de grupos humanos.
MATERIAL Y MÉTODOS
Muestra
Durante el año 2001 se llevaron a cabo tareas de rescate arqueológico a propósito de las remodelaciones realizadas en la Ruta Nacional Nº 9 que pasa inmediatamente por enfrente del sitio.
Las excavaciones se realizaron en tres niveles (Terraza I, Terraza II y Terraza III, desde la inferior a la superior) sobre la ladera norte del cerro donde se concentran las estructuras. En la Terraza III (la más alta) se ubicaron áreas residenciales y de enterramiento. Los cuatro entierros excavados muestran variaciones en las técnicas constructivas, en las modalidades de inhumación y en sus acompañamientos mortuorios. El número mínimo de individuos hallado fue de 18, entre adultos de ambos sexos y subadultos (Gheggi, 2005-2006, 2012). Se seleccionaron 8 muestras correspondientes a 6 individuos, según lo permitiera la presencia de piezas dentales apropiadas para la potencial extracción y amplificación de ADNmt (Tabla 1).
Previamente al procesamiento de las piezas dentales y a fin de conservar la información morfológica del material, se tomó la impresión de la corona y raíces realizando un molde con silicona (Integra VPS) y a partir del mismo se elaboraron réplicas en acrílico (O'Dent S.R.L y Vaicril S.A.).
Extracción de ADN
Todas las piezas dentales fueron sometidas a un proceso de limpieza de su superficie realizando lavados con una solución de hipoclorito de sodio al 10% y luego enjuagando tres veces con agua de calidad biología molecular. Posteriormente, las muestras fueron irradiadas con luz UV durante un tiempo mínimo de 45 minutos en cada una de sus caras. Todo el procedimiento tiene como fin eliminar fuentes de contaminación superficial con ADN exógeno proveniente principalmente de los investigadores que han tenido contacto con las muestras desde su recolección, así como también de microorganismos que residen en el ambiente de depositación.
Luego del proceso de descontaminación y limpieza, se removió la raíz y se obtuvo la dentina por desbaste mecánico utilizando un taladro (Dremel). Mediante este procedimiento la dentina se obtiene en forma de polvo, aumentando la superficie de contacto con las soluciones de extracción y favoreciendo la recuperación del material genético, al mismo tiempo que permite que la pieza pueda ser remontada para su posterior devolución.
Para cada muestra se realizaron al menos tres extracciones de ADN independientes utilizando dos métodos diferentes. Por un lado, partiendo de 100mg de polvo aproximadamente, se realizó una extracción mediante columnas de sílica utilizando QIAamp® DNA Investigator Kit (QIAGEN) siguiendo el protocolo especif-cado por el fabricante. Por otro lado, se realizó una extracción orgánica con fenolcloroformo-isoamílico (25:24:1 v:v:v). Aproximadamente 100mg de polvo fueron incubados con 1ml de EDTA 0,5M (pH 8) a temperatura ambiente y con agitación durante toda una noche. Posteriormente, se centrifugó a 14000 rpm durante 10 minutos, se descartó el sobrenadante de EDTA y el precipitado fue incubado con 1ml de solución de digestión (Tris-HCl 10 mM, EDTA 5mM, Acetato de Sodio 4% y SDS 2%) y 1mg de Proteinasa K (Promega) toda la noche a 55ºC en agitador. Luego se realizó la extracción orgánica y la fase acuosa se purificó mediante columnas de sílica utilizando AccuPrep® PCR Purification Kit (BIONEER), siguiendo el protocolo especificado por el fabricante.
En cada extracción realizada se incorporó un blanco que consiste en un tubo sin muestra sometido al mismo protocolo, y que posteriormente es analizado en paralelo al ADN extraído, a fin de corroborar que no hubo contaminación durante todo el procedimiento.

Secuenciación de la RHVI del ADN mitocondrial
La amplificación por PCR de la RVHI se realizó en tres fragmentos solapados que comprenden las posiciones 15984 a 16411 del ge-noma mitocondrial, utilizando los cebadores: F15984 (5'-tag cac cca aag cta aga ttc taa t-3', modificado a partir de F15989, Gabriel et al., 2001) y H16167 (Ricaut et al., 2004); L16120 (Adachi et al., 2004) y R16239 (Ivanov et al., 1996); F16204 (5'-gca agt aca gca atc aac cct-3', diseñado por nuestro equipo de trabajo) y R16410 (Gabriel et al., 2001).
Las reacciones de PCR se llevaron a cabo en un volumen total de 25µl conteniendo 1X de Hot Start PCR Buffer (Thermo Scientific), 2mM de MgCl2 (Thermo Scientific), 0,2mM de cada dNTP (Thermo Scientific), 0,4µM de cebadores (Invi-trogen), 1U de Maxima Hot Start Taq DNA Poly-merase (Thermo Scientific) y 5-6µl de la muestra de ADNa. En las reacciones de amplificación se incluyeron controles positivos y blancos.
Las condiciones de ciclado fueron: un ciclo de 5 minutos a 94°C; 45 ciclos de 45 segundos a 94ºC, 45 segundos a 54°C y 45 segundos a 72°C; 1 ciclo de 7 minutos a 72°C; mantenimiento a 15°C.
Los productos de amplificación fueron visualizados en geles de agarosa al 1% y se purif-caron con AccuPrep® PCR Purification kit (BIO-NEER), siguiendo el protocolo descripto por el fabricante. Las reacciones de secuenciación se llevaron a cabo utilizando los mismos cebadores empleados en la amplificación de cada fragmento (tanto sentido como antisentido), en un se-cuenciador automático 3130 XL Genetic Analy-zer con BigDyeT Terminator Sequencing Kit (Applied Biosystems), contratando el servicio prestado por la Unidad de Genómica del Intituto de Biotecnología del INTA.
Para cada muestra se obtuvieron secuencias de al menos dos amplificaciones diferentes por fragmento, correspondientes a extracciones independientes. Las mismas fueron revisadas manualmente utilizando el programa BioEdit 7.2.5 (Hall, 1999) y se corroboraron las mutaciones obtenidas con respecto a la Secuencia de Referencia de Cambridge revisada (rCRS; Andrews et al., 1999). La secuencia consenso de la RHVI obtenida para cada individuo se clasificó como perteneciente a uno de los haplogrupos amerindios mediante Ha-plogrep (Kloss-Brandstätter et al., 2011) y comparando con datos tomados de la literatura (Achilli et al., 2008, 2013; O'Rourke, 2009; Perego et al., 2009, 2010; O'Rourke y Raff, 2010).
Comparación de los haplotipos obtenidos
Los haplotipos determinados a partir de la RHVI se compararon con una base de datos de 333 secuencias reportadas en la bibliografía para sitios prehispánicos de los Andes Centrosur (Tabla 2), a fin de encontrar afinidades que puedan explicar la procedencia de los individuos de Esquina de Huajra. En el caso de no hallar los haplotipos en estas poblaciones prehispánicas, se extendió la búsqueda a poblaciones contemporáneas cuyos individuos se autoadscriben como pertenecientes a poblaciones originarias, habitan en zonas rurales y/o hablan una lengua nativa. En la Tabla 2 se detalla la muestra de poblaciones contemporáneas compuesta en total por 1596 secuencias.
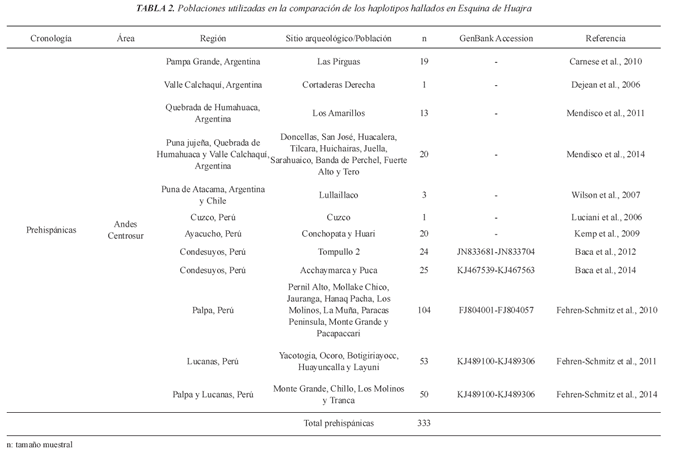

En el caso de que los haplotipos presentes en el sitio bajo estudio tampoco fueran hallados en ninguna de esas poblaciones contemporáneas, se intentó establecer una posible afinidad poblacional a través del estudio de las relaciones flogenéticas con otros linajes mitocondriales. Para ello, se incorporaron todas las poblaciones (Tabla 2) y se realizó una red de haplotipos mediante el método Median Joining con el programa Network 4.6.1.3 (Bandelt et al., 1999; fuxus-engineering.com). Los pesos fueron asignados a cada sitio polimórfico acorde a las tasas mutacionales descriptas en la literatura (Meyer et al., 1999, Soares et al., 2009) y se realizó un postprocesamiento de la red por máxima parsimonia (Polzin y Daneshmand, 2003).
Medidas de autenticidad
Todos los procedimientos se realizaron bajo protocolos estrictos que minimizan la probabilidad de contaminación de las muestras con ADN moderno, tomando como base los criterios de autenticidad existentes en la literatura (e.g. Crespo et al., 2010; Kemp y Smith, 2010). Estos incluyeron la separación física de los cuartos destinados al lavado de las piezas dentales y obtención de la dentina, a la extracción y PCR, y al procesamiento posterior a la PCR; además del uso de indumentaria protectora y del lavado de los materiales utilizados con hipoclorito de sodio al 10% y alcohol etílico al 70%, con posterior irradiación con luz U V.
RESULTADOS
Los métodos de extracción de ADN utilizados no mostraron diferencias en cuanto a su eficiencia. Se obtuvieron al menos dos secuencias de la RHVI a partir de extracciones independientes en cuatro de los 6 individuos analizados, lo cual representa una tasa de recuperación de material genético del 66,7%. La secuencia final de cada individuo se obtuvo a partir del consenso entre todos los fragmentos. La región secuenciada (16024-16365pb) permitió, a partir de la identificación de las mutaciones por comparación con la rCRS, la asignación de dos de ellos como pertenecientes al linaje A2 (50%) y dos a C1 (50%) (Tabla 1).
En dos individuos se contó con dos piezas dentales que fueron analizadas independientemente. En uno de los casos solo se recuperó material genético de una de las muestras, mientras que para el otro individuo ambas secuencias resultaron coincidentes (Tabla 1).
Las secuencias obtenidas indican que los cuatro individuos poseen haplotipos diferentes (Tabla 1). La comparación de los mismos con los publicados para otros sitios prehispánicos del área Centrosur Andina (Tabla 3) revela que la mayoría de los haplotipos hallados se encuentran únicamente en la Quebrada de Humahuaca. En primer lugar, solo uno de los dos haplotipos A2 hallados en Esquina de Huajra fue reportado previamente. Se trata del linaje de la muestra Huaj1 (Tabla 1) que también se encuentra en Los Amarillos (Mendisco et al., 2011), un sitio ubicado al norte de la Quebrada de Humahuaca cuyos individuos analizados corresponden a la ocupación del Período de Desarrollos Regionales (PDR; 700-483 AP). En segundo lugar, en cuanto a los haplotipos C1, una de las variantes fue encontrada solamente en un individuo del sitio San José (Mendisco et al., 2014), también de ese período de la Quebrada de Humahuaca. La otra variante C1, que posee las mutaciones basa-les del linaje, fue igualmente identificada en un individuo de Juella (Mendisco et al., 2014), del PDR de la misma región, pero también se halló en los sitios preincaicos Conchopata y Huari en Ayacucho (Kemp et al., 2009), en la región de Palpa en el valle del Río Grande de Nasca y en los valles altos del Río Viscas (Fehren-Schmitz et al., 2010, 2011, 2014) del actual territorio de Perú. Asimismo, fue detectado en Acchaymarca (Baca et al., 2014), un sitio ubicado en la región de Arequipa (Perú) con evidencias de ocupación durante el período Incaico (Tabla 3).
La red realizada para los haplotipos A2 (Fig. 2) muestra, en primer lugar, que el haplotipo central (que corresponde al basal con las mutaciones 16111T, 16223T, 16290T, 16319A y 16362C con respecto a la rCRS) se encuentra en mayor frecuencia y representado en la mayoría de las poblaciones que se incluyeron en el análisis comparativo. Además, ilustra la ya mencionada exclusividad de las variantes halladas en este trabajo, que se encuentran únicamente en sitios de la Quebrada de Humahuaca. Pero por otro lado se observa que el linaje de Esquina de Huajra (Huaj6/7, Tabla 1), que no se halló en ninguna de las poblaciones compiladas en este trabajo, se ubica como derivado de otro detectado en una población actual de Guaraníes de Brasil y, al mismo tiempo, de él deriva otro linaje presente en dos individuos Kaingang (Marrero et al., 2007; Yang et al., 2010) (Fig. 2).
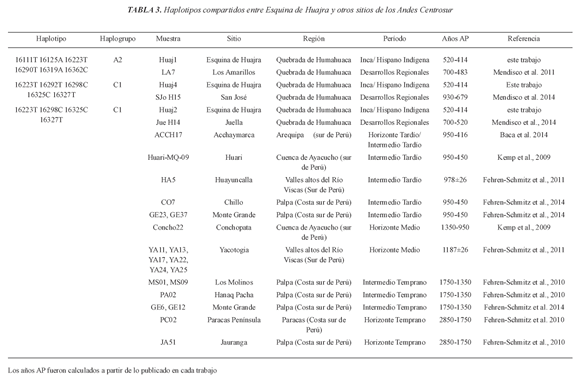

Fig. 2. Red de haplotipos A2 de poblaciones prehispánicas y actuales de los Andes centrosur y de los valles orientales. Los números indican la posición de las mutaciones con respecto a la rCRS. El tamaño de los gráficos de torta es proporcional a la frecuencia de los haplotipos.
DISCUSIÓN
La evidencia arqueológica posiciona a Esquina de Huajra como un sitio clave en relación con los objetivos del imperio Incaico en el sector meridional de la Quebrada de Humahuaca, que habrían incluido la explotación de los recursos de las Yungas y el fortalecimiento de la frontera oriental, entre otros. Considerando que en este si-
tio se encuentran evidencias de cultura material Inca de manufactura local y no local, otras típicas de la Quebrada y alfarería con características de las Yungas Orientales (Cremonte et al., 2005; Cremonte, 2007; Cremonte y Scaro, 2011), es posible hipotetizar una procedencia al menos de tres zonas distintas para los habitantes de Esquina de Huajra. Teniendo en cuenta a la relocalización de poblaciones como una práctica frecuente del Imperio Inca, se presume que los individuos de este sitio podrían provenir de otros sitios de la Quebrada de Humahuaca, de asentamientos bajo el dominio incaico en tierras altas cercanas al centro administrativo, así como de zonas más orientales. Debido a ello, en este sitio podrían encontrarse algunos linajes maternos que son poco frecuentes en el resto del Noroeste Argentino (NOA). La evidencia genética hallada en este trabajo es compatible con estas hipótesis.
Antes de discutir específicamente los resultados obtenidos, es importante destacar que la tasa de recuperación de material genético (66,7%) concuerda con las de otros trabajos de ADNa en muestras del NOA, que se encuentran entre el 65% (Mendisco et al. 2011) y el 90% (Carnese et al, 2010). Este resultado probablemente se deba no sólo a las condiciones de preservación favorables para las muestras en la Quebrada de Huma-huaca (Hofreiter et al., 2001; Fulton, 2012) sino también al hecho de tratarse de piezas dentales (Gilbert et al., 2005; Cardozo et al., 2014).
Acorde a lo hallado hasta el momento, y teniendo en consideración la naturaleza preliminar de los resultados debida al tamaño muestral con el que se trabajó, cabe mencionar que la composición de haplogrupos en Esquina de Huajra, con una predominancia de A2 y C1, se encuentra en discrepancia con lo obtenido para la mayoría de los sitios andinos en los cuales el linaje B2 se presenta como mayoritario (e.g. Kemp et al., 2009; Fehren-Schmitz et al., 2011, 2014; Baca et al., 2012, 2014). Esto ocurre incluso en otros sitios del centro de la Quebrada de Humahuaca (Tilca-ra y Sarahuaico), así como en el Valle Calchaquí (Tero) (Mendisco et al., 2014) y en la serranía Las Pirguas (Carnese et al., 2010). No obstante, en Los Amarillos, ubicado al norte de la Quebrada de Humahuaca, es el haplogrupo A2 el más frecuente (Mendisco et al., 2011).
Realizar un análisis arqueogenético suele implicar frecuentemente una limitación debida al tamaño muestral. En este trabajo, si bien son pocos los individuos analizados, las secuencias de la RHVI obtenidas representan linajes distintos (Tabla 1) y dan cuenta de haplotipos compartidos con sitios (Tabla 3). De particular interés es el caso de aquellos haplotipos A2 y C1 (muestras Huaj1 y Huaj4, Tabla 1) que solamente se encuentran en individuos de otros sitios de la Quebrada de Humahuaca, correspondientes tanto al sector central como norte y con ocupación durante el Período de Desarrollos Regionales, anterior al Incaico. Considerando que los haplotipos hallados en Esquina de Huajra se compararon con 46 secuencias A2 y 85 C1 de más de 35 sitios arqueológicos de los Andes Centrosur, resulta llamativo que estos linajes sólo se encuentren en otros sitios que-bradeños. Es importante destacar que la singularidad del linaje A2 de la muestra Huaj1 (que posee las mutaciones basales adicionalmente a la 16125A, Tabla 1) fue comunicada por Motti et al. (2014) quienes, comparando con más de 6000 secuencias, señalan que no se encuentra en poblaciones actuales del NOA, del centro-oeste argentino ni del resto de Sudamérica, sino únicamente en el sitio Los Amarillos (Mendisco et al., 2011). Esto entraría en concordancia con la hipótesis de un origen local para parte de los habitantes de Esquina de Huajra.
Por otro lado, la otra variante C1 hallada en este estudio también fue detectada en la Quebrada de Humahuaca y en numerosos sitios preincaicos e incaicos del actual territorio de Perú (Tabla 3). Considerando que, a este nivel de resolución, se trata de un linaje nodal ampliamente distribuido en todo el continente (Achilli et al., 2013 y referencias allí citadas), esto no permite distinguir entre un origen en tierras más cercanas a la capital incaica o el hecho de compartir un ancestro poblacional antiguo. Esto remarca la necesidad de, por un lado, incrementar el número de sitios estudiados así como el tamaño muestral en cada uno de ellos con el potencial de encontrar otros linajes compartidos entre ambas regiones, y por otro lado, extender el análisis a otros fragmentos del ADNmt a fin de aumentar el poder resolutivo.
Finalmente, resulta llamativa la singularidad del otro de los haplotipos de A2 hallados en Esquina de Huajra, que no fue encontrado en ninguno de los sitios arqueológicos compilados en el presente trabajo (Huaj6/7, Tabla 1). Es importante tener en cuenta que todas las poblaciones con las que se realizó la comparación corresponden a grupos prehispánicos del área Andina Centrosur puesto que, según nuestro conocimiento, hasta el momento no se cuenta con datos provenientes de sitios arqueológicos de otras regiones más orientales que pudieron haber tenido contacto con la Quebrada de Hu-mahuaca, como por ejemplo las Yungas. Constituyen una excepción los trabajos realizados en sitios del actual territorio uruguayo y del sur de Brasil (Marinho et al., 2006; Figueiro, 2011; Sans et al., 2012), pero los mismos incluyen muestras mayoritariamente de la costa atlántica y para aquellos sitios ubicados al oeste de Uruguay sólo se halló un haplotipo B2 (Figueiro, 2011). Por este motivo, se compilaron adicio-nalmente secuencias de la RHVI existentes en la literatura de poblaciones originarias actuales, habitantes tanto de los Andes Centrosur como de regiones en las tierras bajas del este (Tabla 2). En ninguna de estas poblaciones se halló el haplotipo de A2 encontrado en Esquina de Huajra (Huaj6/7, Tabla 2). Sin embargo, un total de 50 individuos Guaraní tanto Kaiowá como Ñandeva (Brasil) poseen la transición 16209C (Marrero et al., 2007) y, considerando que el sitio 16126 posee una alta tasa mutacional (Meyer et al., 1999; Soares et al., 2009), el linaje hallado en el presente trabajo se presenta como relacionado flogenéticamente al observado en esa población actual (Fig. 2). Cabe destacar que, a pesar de la alta tasa mutacional, la mutación 16126C se encuentra en baja frecuencia en el conjunto de datos analizado, hallándose además sólo en dos individuos Kaingang de Río Grande do Sul (Brasil) que se presentan como variantes haplotípicas derivadas de la presente en Esquina de Huajra. El hecho de haberla hallado en grupos originarios que actualmente habitan regiones del este es importante puesto que dicho polimorfsmo hasta el momento no fue encontrado en otras poblaciones prehispáni-cas del área Centrosur Andina. Debido a esto, resulta evidente la necesidad de incrementar el muestreo en los valles orientales y, en particular, queda de manifesto la gran utilidad que representarían futuros análisis de la RHVI que fueran realizados en poblaciones prehispánicas de las Yungas y del Chaco.
Por último es necesario señalar que siendo el objetivo del presente trabajo intentar responder al interrogante acerca de la procedencia de los individuos en este sitio construido en el período Incaico, no se han incluido comparaciones con poblaciones actuales no nativas de la región, ya que eso implicaría asumir una continuidad geográfica para los habitantes de la zona en los últimos cinco siglos. Por el contrario, durante ese tiempo las poblaciones originarias han sufrido varios eventos que dificultan la asunción de dicha continuidad, como mitas, encomiendas, la "reducción" de distintas comunidades en "Pueblos de Indios" y traslados poblacionales de comunidades enteras, incluso a otras provincias (García Moritán y Cruz, 2012). Teniendo esto en consideración, la comparación con poblaciones contemporáneas se ve limitada a la hora de establecer la posible procedencia de individuos en un asentamiento prehispánico.
CONCLUSIONES
Este trabajo representa el primer análisis genético de individuos provenientes de un con-
texto incaico en la Quebrada de Humahuaca, puesto que hasta el momento solamente dos estudios de ADNa se han enfocado en sitios quebradeños del Período de Desarrollos Regionales (Mendisco et al. 2011, 2014). Asimismo, representa uno de los pocos realizados en el actual Noroeste Argentino, con lo cual adquiere especial relevancia a la hora de interpretar los patrones de poblamiento y movilidad en la región.
Aunque los resultados son preliminares, se encuentran en concordancia con las hipótesis que sostienen una procedencia multirregional para la población de Esquina de Huajra, posiblemente con mayor peso del componente local, y constituyen una evidencia más de la complejidad de los procesos poblacionales ocurridos en toda el área andina en épocas pre-hispánicas.
Se destaca la importancia de continuar con los análisis en los individuos de este sitio incluyendo la secuenciación de la RHVII, lo cual permitirá incrementar el poder resolutivo facilitando la identificación de haplotipos y su comparación, e intentar determinar los linajes paternos que podrían ser comparados con otras muestras prehispánicas de la región (Carnese et al., 2010; Mendisco et al., 2011, 2014). Con el objetivo de incrementar el tamaño muestreal se plantea la posibilidad de realizar análisis de ADNa sobre restos óseos del esqueleto post-craneal, debido a que no todos los individuos hallados poseen representación de piezas dentales.
Para concluir, futuros análisis de ADNa en muestras de las Yungas Orientales podrían aportar, desde la perspectiva genética, mayor información a la discusión acerca de la existencia de fujo génico entre esa región y la Quebrada de Humahuaca.
AGRADECIMIENTOS
Deseamos expresar nuestro agradecimiento al Dr. Stéphane Mazières (Universidad Aix-Marseille, Francia), por facilitarnos las secuencias correspondientes a las comunidades Aché, Guaraní y Kaingang (Yang et al., 2010). Agradecemos a dos revisores anónimos cuyas sugerencias ayudaron a mejorar significativamente este trabajo.
LITERATURA CITADA
Achilli A, Perego UA, Bravi CM, Coble MD, Kong QP, Woodward SR, Salas A, Torroni A, Bandelt HJ. 2008. The phylogeny of the four pan-American MtDNA ha-plogroups: implications for evolutionary and disease studies. PLoS One 3(3): e1764. doi:10.1371/journal. pone.0001764
Achilli A, Perego UA, Lancioni H, Olivieri A, Gandini F, Hooshiar Kashani B, Battaglia V, Grugni V, Angerhofer N, Rogers MP, Herrera RJ, Woodward SR, Labuda D, Smith DG, Cybulski JS, Semino O, Malhi RS, Torroni A. 2013. Reconciling migration models to the Americas with the variation of North American native mitogeno-mes. Proc Natl Acad Sci U S A 110(35):14308-14313. doi:10.1073/pnas.1306290110
Adachi N, Umetsu K, Takigawa W, Sakaue K. 2004. Phylo-genetic analysis of the human ancient mitochondrial DNA. J Archaeol Sci 31(10):1339-1348. doi:10.1016/j. jas.2004.02.011
Andrews RM, Kubacka I, Chinnery PF, Lightowlers RN, Turnbull DM, Howell N. 1999. Reanalysis and revision of the Cambridge reference sequence for human mito-chondrial DNA. Nat Genet 23(2):147.
Angiorama C. 2013. La metalurgia en tiempos del Inca: estudio de objetos metálicos hallados en Esquina de Huajra (Quebrada de Humahuaca, Jujuy). En: Williams V, Cremonte MB, compiladoras. Al borde del imperio: paisajes sociales, materialidad y memoria en áreas periféricas del Noroeste argentino. Buenos Aires: Relaciones de la Sociedad Argentina de Antropología. p 93-108.
Baca M, Doan K, Sobczyk M, Stankovic A, Weglenski P. 2012. Ancient DNA reveals kinship burial patterns of a pre-Columbian Andean community. BMC Genet 13:30.
Baca M, Molak M, Sobczyk M, Weglenski P, Stankovic A. 2014. Locals, resettlers, and pilgrims: a genetic portrait of three pre-Columbian Andean populations. Am J Phys Anthropol 154(3):402-412. doi:10.1186/1471-2156-13-30.
Bandelt H-J, Forster P, Röhl A. 1999. Median-joining networks for inferring intraspecific phylogenies. Mol Biol Evol 16(1):37-48.
Barbieri C, Heggarty P, Castrì L, Luiselli D, Pettener D. 2011. Mitochondrial DNA variability in the Titicaca basin: matches and mismatches with linguistics and ethnohistory. Am J Hum Biol 23(1):89-99. doi:10.1002/ ajhb.21107
Bert F, Corella A, Gené M, Pérez-Pérez A, Turbón D. 2004. Mitochondrial DNA diversity in the Llanos de Moxos: Moxo, Movima and Yuracare Amerindian populations from Bolivia lowlands. Ann Hum Biol 31(1):9-28. doi: 10.1080/03014460310001616464
Cabana GS, Merriwether DA, Hunley K, Demarchi DA. 2006. Is the genetic structure of Gran Chaco popula-tions unique? Interregional perspectives on native South American mitochondrial DNA variation. Am J Phys Anthropol 131(1):108-119. doi:10.1002/ajpa.20410
Cardozo DG, Crespo CM, Russo MG, Postillone MB. 2014. Análisis y conservación de ADN antiguo en restos es-queletales humanos de sitios arqueológicos y colecciones de museos de Argentina. En: Esnal AC, Funes ML, Grosso M, Kuperszmit N, Murgo A, Romero G, editores. Entre pasados y presentes I V. Estudios contemporáneos en Ciencias Antropológicas. Ciudad Autónoma de Buenos Aires: Editorial Asociación Amigos del Instituto Nacional de Antropología (AINA). p 313-329.
Carnese F, Mendisco F, Keyser C, Dejean CB, Dugoujon JM, Bravi CM, Ludes B, Crubézy E. 2010. Paleogeneti-cal study of pre-Columbian samples from Pampa Grande (Salta, Argentina). Am J Phys Anthropol 141:452-462. [ Links ] doi:10.1002/ajpa.21165
Casas-Vargas A, Gómez A, Briceño I, Díaz-Matallana M, Bernal JE, Rodríguez J V. 2011. High genetic diversi-ty on a sample of pre-Columbian bone remains from Guane territories in northwestern Colombia. Am J Phys Anthropol 146:637-649. [ Links ] doi:10.1002/ajpa.21626
Corella A, Bert F, Pérez-Pérez A, Gené M, Turbón D. 2007. Mitochondrial DNA diversity of the Amerindian popu-lations living in the Andean piedmont of Bolivia: Chi-mane, Moseten, Aymara and Quechua. Ann Hum Biol 34(1):34-55. doi:10.1080/03014460601075819
Cremonte MB. 2007. Aspectos económicos, políticos e ideológicos con relación a la ocupación Inka en los valles meridionales de Jujuy, Argentina. En: Williams V, Ventura B, Callegari A, Yacobacio H, editores. Sociedades Precolombinas Surandinas: Temporalidad, interacción y dinámica cultural del NOA en el ámbito de los Andes Centro-Sur. Buenos Aires: Artes Gráficas Buschi. p 109-124.
Cremonte MB, Garay de Fumagalli M. 1997. El Pukara de Volcán en el sur de la Quebrada de Humahuaca ¿un eje articulador entre las yungas y las tierras altas? (Prov. de Jujuy-Argentina). Estudios Atacameños 14:159-174.
Cremonte MB, Garay de Fumagalli M. 2013. Diferentes modalidades de estructuración de la frontera surorien-tal Inka en el actual territorio de Jujuy. En: Williams V, Cremonte MB, compiladoras. Al borde del imperio: paisajes sociales, materialidad y memoria en áreas periféricas del Noroeste Argentino. Buenos Aires: Relaciones de la Sociedad Argentina de Antropología. p 57-74.
Cremonte MB, Garay de Fumagalli M, Sica G. 2005. La frontera oriental al sur de la Quebrada de Humahuaca. Un espacio conectivo. Mundo de Antes 4:51-66. [ Links ]
Cremonte MB, Gheggi MS. 2012. Espacios rituales y cultura material en un sitio arqueológico Humahuaca-Inca (Quebrada de Humahuaca, Jujuy, Argentina). Revista Española de Antropología Americana 42(1):9-27. doi:10.5209/rev_REAA.2012.v42.n1.38633
Cremonte MB, Scaro A. 2011. Consumo de vasijas cerámicas en un contexto público del Pucara de Volcán (Dto. Tumbaya, Jujuy). Rev Mae 20:147-161. [ Links ]
Crespo CM, Dejean CB, Postillone MB, Lanata JL, Carnese FR. 2010. Historias en código genético. Los aportes de los estudios de ADN antiguo en antropología y sus implicancias éticas. RUNA XXXI(2):153-174.
de Saint Pierre M, Bravi CM, Motti JM, Fuku N, Tanaka M, Llop E, Bonatto SL, Moraga M. 2012. An alternative model for the early peopling of southern South Ame-rica revealed by analyses of three mitochondrial DNA haplogroups. PLoS One 7(9):e43486. DOI: 10.1371/ journal.pone.0043486
Dejean CB, Keyser-Tracqui C, Crubézy E, Blandin P, Luna LH, Aranda CM, Acuto FA, Dugoujon JM, Ludes B, Carnese FR. 2006. Análisis paleogenético de tres individuos provenientes del sitio Cortaderas Derecha, Salta (Argentina). Actas del IX Congreso de la Asociación Latinoamericana de Antropología Biológica, Ouro Preto.
Dejean C, Keyser C, Avena S, Dugoujon JM, Ludes B, Crubezy E, Carnese FR. 2008. Análisis genético de muestras esqueletales provenientes del Canal de Bea-gle. Actas del X Congreso Asociación Latinoamericana de Antropología Biológica, La Plata.
Dornelles CL, Battilana J, Fagundes NJ, Freitas LB, Bonatto SL, Salzano FM. 2004. Mitochondrial DNA and Alu insertions in a genetically peculiar population: the Ayo-reo indians of Bolivia and Paraguay. Am J Hum Biol 16(4):479-88. doi:10.1002/ajhb.20038
Fehren-Schmitz L, Reindel M, Tomasto Cagigao E, Hummel S, Herrmann B. 2010. Pre-Columbian popu-lation dynamics in coastal southern Peru: A diachro-nic investigation of mtDNA patterns in the Palpa re-gion by ancient DNA analysis. Am J Phys Anthropol 141(2):208-221. doi:10.1002/ajpa.21135
Fehren-Schmitz L, Warnberg O, Reindel M, Seidenberg V, Tomasto-Cagigao E, Isla-Cuadrado J, Hummel S, Herrmann B. 2011. Diachronic investigations of mitochondrial and Y-chromosomal genetic markers in pre-Columbian Andean highlanders from south Peru. Ann Hum Genet 75(2):266-283. doi:10.1111/j.1469-1809.2010.00620.x
Fehren-Schmitz L, Haak W, Mächtle B, Masch F, Llamas B, Tomasto Cagigao E, Sossna V, Schittek K, Isla Cuadrado J, Eitel B, Reindel M. 2014. Climate chan-ge underlies global demographic, genetic, and cultural transitions in pre-Columbian southern Peru. Proc Natl Acad Sci U S A 111(26):9443-9448. dói:10.1073/ pnas.1403466111
Figueiro G. 2011. Continuidad temporal en la composición genética de las poblaciones indígenas del Uruguay. En: Colección avances de investigación (Docentes). Montevideo: Editorial Facultad de Humanidades y Ciencias de la Educación. p 141-159.
Fuselli S, Tarazona-Santos E, Dupanloup I, Soto A, Luiselli D, Pettener D. 2003. Mitochondrial DNA di-versity in South America and the genetic history of Andean highlanders. Mol Biol Evol 20(10):1682-1691. doi:10.1093/molbev/msg188
Gabriel MN, Huffine EF, Ryan JH, Holland MM, Parsons TJ. 2001. Improved MtDNA sequence analysis of fo-rensic remains using a "mini-primer set" amplification strategy. J Forensic Sci 46(2):247-253.
Garay de Fumagalli, M. 2003. El Cucho de Ocloyas. Control e interacción en los valles orientales de Jujuy. Cuadernos de la Facultad de Humanidades y Ciencias Sociales de la UNJu 133-150.
Garay de Fumagalli M, Cremonte MB. 2002. Ocupaciones agropastoriles tempranas a sur de la Quebrada de Hu-mahuaca (Jujuy, Argentina). Chungara 34(1):35-52.
Garay de Fumagalli, M. Cremonte. MB. 2007. Contextos inkaicos en momentos Hispano-Indígenas. Jujuy, Argentina. Libro de Resúmenes Ampliados del XIII Congreso Nacional de Arqueología Argentina Tomo 1: 507-512.
García-Bour J, Pérez-Pérez A, Álvarez S, Fernández E, López-Parra AM, Arroyo-Pardo E, Turbón D. 2004. Early population differentiation in extinct aborigines from Tierra del Fuego-Patagonia: ancient mtDNA sequences and Y-chromosome STR characterization. Am J Phys Anthropol 123:361-370. doi:10.1002/ajpa.10337
García Moritán M., Cruz M.B. 2012. Comunidades originarias y grupos étnicos de la provincia de Jujuy. Poblac soc 19(2):155-173.
Gayà-Vidal M, Moral P, Saenz-Ruales N, Gerbault P, Tonasso L, Villena M, Vasquez R, Bravi CM, Dugoujon JM. 2011. mtDNA and Y-chromosome diversity in Aymaras and Quechuas from Bolivia: different stories and spe-cial genetic traits of the Andean Altiplano populations. Am J Phys Anthropol 145(2):215-230. doi:10.1002/ ajpa.21487
Gheggi MS. 2005-2006. Más allá de los huesos. El estudio integral de la evidencia de los enterratorios de Esquina de Huajra (Dto. Tumbaya, Quebrada de Humahuaca) en el contexto histórico regional. Arqueología 13:47-78. [ Links ]
Gheggi MS. 2012. Patologías orales, dieta y modo de vida en Esquina de Huajra (Quebrada de Humahuaca, Ju-juy). Rev Arg Antrop Biol 14 (1): 65-78.
Gilbert MTP, Rudbeck L, Willerslev E, Hansen AJ, Smith C, Penkman KEH, Prangenberg K, Nielsen-Marsh CM, Jans ME, Arthur P. 2005. Biochemical and physical co-rrelates of DNA contamination in archaeological human bones and teeth excavated at Matera, Italy. J Archaeol Sci 32:785-793. [ Links ] doi:10.1016/j.jas.2004.12.008
Gonçalves VF, Parra fic, Gonçalves-Dornelas H, Rodrigues-Carvalho C, Silva HP, Pena SDJ. 2010. Re-covering mitochondrial DNA lineages of extinct Ame-rindian nations in extant homopatric Brazilian popula-tions. Investig Genet 1(1):13.
Gonçalves VF, Stenderup J, Rodrigues-Carvalho C, Silva HP, Gonçalves-Dornelas H, Líryo A, Kivisild T, Malaspinas AS, Campos PF, Rasmussen M, Willerslev E, Pena SDJ. 2014. Identification of Polynesian mtDNA haplogroups in remains of Botocudo Amerindians from Brazil. Proc Natl Acad Sci U S A 110(16):6465-6469. doi:10.1073/pnas.1217905110
Greco C. 2014. Análisis estadístico de fechados radiocar-bónicos del sector sur de la Quebrada de Humahuaca. En: Cremonte, MB, Scaro A, editoras. Cerámicas pre-hispánicas de la Quebrada de Humahuaca. Una visión desde las prácticas sociales del pasado y del presente. EdiUnju. Jujuy. En Prensa.
Hall TA. 1999. BioEdit: a user-friendly biological sequen-ce alignment editor and analysis program for Windows 95/98/NT. Nucl Acids Symp Ser 41:95-98.
Hofreiter M, Davis S, Poinar H, Kuch M, Pääbo S. 2001. Ancient DNA. Nat Rev Genet 2(5):353-359.
Ivanov PL, Wadhams MJ, Roby RK, Holland MM, Weedn VW, Parsons TJ. 1996. Mitochondrial DNA sequence heteroplasmy in the Grand Duke of Russia Georgij Romanov establishes the authenticity of the remains of Tsar Nicholas II. Nat Genet 12(4):417-420.
Kemp BM, Tung TA, Summar ML. 2009. Genetic continui-ty after the collapse of the Wari Empire: mitochondrial DNA profles from Wari and post-Wari populations in the ancient Andes. Am J Phys Anthropol 140(1):80-91. doi:10.1002/ajpa.21037
Kemp BM y Smith DG. 2010. Ancient DNA Methodolo-gy: Thoughts from Brian M. Kemp and David Glenn Smith on "Mitochondrial DNA of protohistoric remains of an Arikara population from South Dakota". Hum Biol 82(2):227-238. doi:10.3378/027.082.0207
Kloss-Brandstätter A, Pacher D, Schönherr S, Weissens-teiner H, Binna R, Specht G, Kronenberg F. 2011. Ha-ploGrep: a fast and reliable algorithm for automatic classification of mitochondrial DNA haplogroups. Hum Mutat 32(1):25-32. doi:10.1002/humu.21382
Lalueza-Fox C, Pérez-Pérez A, Prats E, Cornudella L, Turbón D. 1997. Lack of founding amerindian mito-chondrial DNA lineages in extinct aborigines from Tierra del Fuego-Patagonia. Hum Mol Genet 6(1):41-46. doi:10.1093/hmg/6.1.41
Lewis CM Jr, Lizárraga B, Tito RY, López PW, Iannacone GC, Medina A, Martínez R, Polo SI, De La Cruz AF, Cáceres AM, Stone AC. 2007. Mitochondrial DNA and the peopling of South America. Hum Biol 79(2):159-178. doi:10.1353/hub.2007.0031
Luciani S, Fornaciari G, Rickards O, Martínez Labraga C, Rollo F. 2006. Molecular characterization of a pre-co-lumbian mummy and in situ coprolite. Am J Phys An-thropol 129:620-629. doi:10.1002/ajpa.20314
Marinho AN, Miranda NC, Braz V, Ribeiro-Dos-Santos AK, de Souza SM. 2006. Paleogenetic and taphonomic analysis of human bones from Moa, Beirada, and Zé Espinho sambaquis, Rio de Janeiro, Brazil. Mem Inst Oswaldo Cruz 101(2):15-23.
Marrero AR, Silva-Junior WA, Bravi CM, Hutz MH, Petzl-Erler ML, Ruiz-Linares A, Salzano FM, Bortolini MC. 2007. Demographic and evolutionary trajectories of the Guarani and Kaingang natives of Bra-zil. Am J Phys Anthropol 132(2):301-310. doi:10.1002/ ajpa.20515
Mendisco F, Keyser C, Hollard C, Seldes V, Nielsen A, Crubézy E, Ludes B. 2011. Application of the iPLEXTM Gold SNP genotyping method to the analysis of Ame-rindian ancient DNA samples: benefts for ancient population-based studies. Electrophoresis 32:386-393. doi:10.1002/elps.201000483
Mendisco F, Keyser C, Seldes V, Rivolta C, Mercolli P, Cruz P, Nielsen AE, Crubezy E, Ludes B. 2014. Genetic di-versity of a late prehispanic group of the Quebrada de Humahuaca, Northwestern Argentina. Ann Hum Genet 78(5):367-380. doi:10.1111/ahg.12075
Mengoni Goñalons GL. 2013. El aprovechamiento de la fauna en sociedades complejas: aspectos metodológicos y su aplicación en diferentes contextos arqueológicos del NOA. En: Williams V, Cremonte MB, compiladoras. Al borde del imperio: paisajes sociales, materialidad y memoria en áreas periféricas del Noroeste argentino. Buenos Aires: Relaciones de la Sociedad Argentina de Antropología. p 311-343.
Meyer S, Weiss G, von Haeseler A. 1999. Pattern of nucleotide substitution and rate heterogeneity in the hypervariable regions I and II of human mtDNA. Genetics 152(3):1103-1110.
Motti JM, Ramallo V, Muzzio M, Bailliet G, Bravi CM. 2014. El ADN mitocondrial como indicador de relaciones biológicas entre poblaciones antiguas y actuales del NOA. En: Luna L, Aranda C, Suby J, editores. Avances recientes de la Bioarqueología Latinoamericana. Buenos Aires: GIB. p 267-283.
Nores R, Demarchi D. 2011. Análisis de haplogrupos mito-condriales en restos humanos de sitios arqueológicos de la provincia de Córdoba. Rev Arg Antrop Biol 13(1):43-54.
O'Rourke DH. 2009. Human migrations: the two roads taken. Curr Biol 19(5):R203-205. doi:10.1016/j. cub.2009.01.021
O'Rourke DH, Raff JA. 2010. The human genetic history of the Americas: The final frontier. Curr Biol 20(4):R202-207. doi:10.1016/j.cub.2009.11.051
Perego UA, Achilli A, Angerhofer N, Accetturo M, Pala M, Olivieri A, Kashani BH, Ritchie KH, Scozzari R, Kong QP, Myres NM, Salas A, Semino O, Bandelt HJ, Wood-ward SR, Torroni A. 2009. Distinctive Paleo-Indian mi-gration routes from Beringia marked by two rare mtD-NA haplogroups. Curr Biol 19(1):1-8. doi:10.1016/j. cub.2008.11.058
Perego UA, Angerhofer N, Pala M, Olivieri A, Lancioni H, Hooshiar Kashani B, Carossa V, Ekins JE, Gómez-Carballa A, Huber G, Zimmermann B, Corach D, Babudri N, Panara F, Myres NM, Parson W, Semino O, Salas A, Woodward SR, Achilli A, Torroni A. 2010. The initial peopling of the Americas: a growing number of foun-ding mitochondrial genomes from Beringia. Genome Res 20(9):1174-1179. doi:10.1101/gr.109231.110
Polzin T, Daneshmand S V. 2003. On Steiner trees and mini-mum spanning trees in hypergraphs. OPER RES LETT 31(1):12-20. doi:10.1016/S0167-6377(02)00185-2
Postillone MB, Crespo CM, Russo MG, Cardozo DG. 2014. Distribución de haplogrupos mitocondriales amerindios en muestras arqueológicas de Sudamérica. En: Esnal AC, Funes ML, Grosso M, Kuperszmit N, Murgo A, Romero G, editores. Entre pasados y presentes IV. Estudios contemporáneos en Ciencias Antropológicas. Ciudad Autónoma de Buenos Aires: Editorial Asociación Amigos del Instituto Nacional de Antropología (AINA). p 347-362.
Ricaut FX, Keyser-Tracqui C, Bourgeois J, Crubézy E, Ludes B. 2004. Genetic analysis of a Scytho-Siberian skeleton and its implications for ancient Central Asian migrations. Hum Biol 76(1):109-125.
Rothhammer F, Moraga M, Rivera M, Santoro CM, Standen VG, García F, Carvallo P. 2003. Análisis de ADNmt de restos esqueletales del sitio arqueológico de Tiwanaku y su relación con el origen de sus constructores. Chungara 35(2):269-274.
Rothhammer F, Santoro CM, Poulin E, Arriaza BT, Moraga M, Standen VJ. 2009. Archeological and mtDNA evi-dence for Tropical Lowland migrations during the Late Archaic/Formative in northern Chile. Rev Chil Hist Nat 82:543-552.
Sala A, Argüelles CF, Marino ME, Bobillo C, Fenocchio A, Corach D. 2010. Genetic analysis of six com-munities of Mbyá-Guaraní inhabiting Northeastern Argentina by means of nuclear and mitochondrial polymorphic markers. Hum Biol 82(4):433-456. doi:100.3378/027.082.0406
Sandoval JR, Lacerda DR, Jota MS, Salazar-Granara A, Vieira PP, Acosta O, Cuellar C, Revollo S, Fujita R, Santos FR; Genographic Project Consortium. 2013. The genetic history of indigenous populations of the Peruvian and Bolivian Altiplano: the legacy of the Uros. PLoS One 8(9):e73006. doi:10.1371/journal. pone.0073006
Sans M, Figueiro G, Hidalgo P. 2012. A new mitochon-drial C1 lineage from the prehistory of Uruguay: po-pulation genocide, ethnocide, and continuity. Hum Biol 84(3):287-305. doi:10.3378/027.084.0303
Schmitt R, Bonatto SL, Freitas LB, Muschner VC, Hill K, Hurtado AM, Salzano FM. 2004. Extremely limited mi-tochondrial DNA variability among the Aché natives of Paraguay. Ann Hum Biol 31(1):87-94. doi:10.1080/030 14460310001602063
Silva A, Briceño I, Burgos J, Torres D, Villegas V, Gómez A, Bernal JE, Rodríguez JV. 2008. Análisis de ADN mitocondrial en una muestra de restos óseos arcaicos del periodo Herrera en la sabana de Bogotá. Biomedica 28(4):569-577. doi:10.7705/biomedica.v28i4.62
Soares P, Ermini L, Thomson N, Mormina M, Rito T, Röhl A, Salas A, Oppenheimer S, Macaulay V, Richards MB. 2009. Correcting for purifying selection: an improved human mitochondrial molecular clock. Am J Hum Genet 84(6):740-759. doi:10.106/j.ajhg.2009.05.001
Yang NN, Mazières S, Bravi C, Ray N, Wang S, Bur-ley MW, Bedoya G, Rojas W, Parra MV, Molina JA, Gallo C, Poletti G, Hill K, Hurtado AM, Petzl-Erler ML, Tsuneto LT, Klitz W, Barrantes R, Llop E, Ro-thhammer F, Labuda D, Salzano FM, Bortolini MC, Excoffer L, Dugoujon JM, Ruiz-Linares A. 2010. Con-trasting patterns of nuclear and mtDNA diversity in Na-tive American populations. Ann Hum Genet 74(6):525-538. doi:10.1111/j.1469-1809.2010.00608.x
Wilson AS, Taylor T, Ceruti MC, Chavez JA, Reinhard J, Grimes V, Meier-Augenstein W, Cartmell L, Stern B, Richards MP, Worobey M, Barnes I, Gilbert MT. 2007. Stable isotope and DNA evidence for ritual sequen-ces in Inca child sacrifice. Proc Natl Acad Sci U S A 104(42):16456-16461. doi:10.1073/pnas/0704276104














