Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO  uBio
uBio
Share
Revista argentina de antropología biológica
On-line version ISSN 1514-7991
Rev Arg Antrop Biol vol.19 no.1 La Plata June 2017
http://dx.doi.org/10.17139/raab.2017.0019.01.07
TRABAJOS DEL DOSSIER
VARIABILIDAD MITOCONDRIAL EN MUESTRAS PRE-COLOMBINAS DE LA PATAGONIA ARGENTINA. HACIA UNA VISIÓN DE SU POBLAMIENTO DESDE EL ADN ANTIGUO
Cristian M. Crespo1*, María G. Russo1, Adam Hajduk2, José L. Lanata3 y Cristina B. Dejean4
1CONICET-CEBBAD, Fundación de Historia Natural Félix de Azara. Universidad Maimónides. Ciudad Autónoma de Buenos Aires (CABA). Argentina
2CONICET-Administración de Parques Nacionales. Museo de la Patagonia Francisco Pascasio Moreno. San Carlos de Bariloche. Argentina
3CONICET-UNRN, IIDyPCa. San Carlos de Bariloche. Argentina
4Sección Antropología Biológica. Instituto de Ciencias Antropológicas. Facultad de Filosofía y Letras. Universidad de Buenos Aires. CABA. Argentina
*Correspondencia a: Cristian M. Crespo. CONICET-CEBBAD.
Fundación de Historia Natural Félix de Azara. Universidad Maimónides. Hidalgo 775 6°piso. 1405 CABA. Argentina. Email: cristianmcrespo@gmail.com
Financiamiento: UBACyT (F346); CONICET (PIP2850); Universidad de Río Negro (40B064); Fundación de Historia Natural Félix de Azara; Fundación Científica Felipe Fiorellino, Universidad Maimónides.
Recibido 29 Diciembre 2015; aceptado 1 Julio 2016
doi:10.17139/raab.2017.0019.01.07
RESUMEN Distintas disciplinas han intentado explicar el poblamiento y la evolución de las poblaciones cazadorasrecolectoras que habitaron la Patagonia argentina. La arqueología y la morfología craneal propusieron un escenario de poblamiento temprano caracterizado por una baja demografía, eventos de divergencias y efectos fundadores seriales. Estos postulados fueron avalados desde el análisis del ADN mitocondrial procedente de poblaciones originarias actuales, aunque son escasos aún los estudios en poblaciones precolombinas. Con el objeto de aumentar la información disponible, se analizó un total de 50 muestras de individuos de sitios arqueológicos y colecciones de museos de diversas regiones patagónicas con temporalidades desde los ca.5600 14C años AP hasta tiempos históricos. Se logró asignar el linaje materno en 27 de ellas por RFLP y en 22 de las mismas secuencias de la región hipervariable 1. Se detectaron los linajes A2, C1, D1, D1g, D1j y D4h3a. A través de los análisis estadísticos de los resultados obtenidos en este trabajo se observa una tendencia a la disminución de la diversidad mitocondrial hacia el sur. La comparación de las regiones geográficas analizadas sugiere la ausencia de diferencias estadísticamente significativas y de estructuración poblacional, lo cual puede indicar un origen común reciente y/o la existencia de flujo génico entre las regiones durante el Holoceno tardío. Adicionalmente, puede proponerse un cambio en el acervo genético mitocondrial en las poblaciones nativas actuales representado por las altas frecuencias del linaje B2, posiblemente relacionado con el proceso de araucanización a partir del siglo XVI. Rev Arg Antrop Biol 19(1), 2017. doi:10.17139/raab.2017.0019.01.07
PALABRAS CLAVE ADN antiguo; linajes maternos; Patagonia argentina
ABSTRACT Different disciplines have attempted to explain the settlement and evolution of the hunter-gatherer populations who lived in the Argentinean Patagonia. Both archaeology and cranial morphology have suggested an initial settlement stage characterized by low demography, divergence events, and serial founder effects. These assumptions were supported by mitochondrial DNA analysis of extant native populations; studies in pre-Columbian groups being as yet scarce. In order to increase the information available, a total of 50 samples of individuals from archaeological sites and museum collections of various Patagonian regions, dated It was possible to assign maternal lineage in 27 samples by RFLP analysis and in 22 of them hypervariable region 1 was sequenced. Lineages A2, C1, D1, D1g, D1j and D4h3a were detected. Statistical analysis of the results obtained here suggested a tendency towards a lower mitochondrial diversity southward. Comparison of the geographical regions analyzed has shown no statistically significant differences or population structure, which may indicate a recent common origin and the existence of gene flow across regions during the late Holocene. Additionally, a change in the mitochondrial gene pool can be proposed in current native populations due to the high frequency of lineage B2, possibly related to the process of araucanization begun in the sixteenth century. Rev Arg Antrop Biol 19(1), 2017. doi:10.17139/raab.2017.0019.01.0
KEY WORDS ancient DNA; maternal lineages; Argentinean Patagonia
El poblamiento humano de la Patagonia argentina tradicionalmente ha sido estudiado por la arqueología y la importancia de esta región en la discusión de la ocupación del continente se debe principalmente a dos motivos: por un lado, a la existencia de un rico registro arqueológico que posee una gran profundidad temporal en relación a los fechados radiocarbónicos de otras regiones, tanto de Argentina como del continente americano y, por otro lado, porque la misma representa el final del camino de la dispersión continental global del género Homo (Borrero, 2001; Miotti y Salemme, 2004; Prates et al., 2013; entre otros). Los interrogantes sobre la variabilidad biológica presente en los diferentes grupos humanos patagónicos y las posibles relaciones entre los mismos han sido estudiados desde la craneometría y el análisis morfométrico de piezas dentales, así como también a través de marcadores genéticos moleculares uni y biparentales desde hace ya algunas décadas, con el objetivo de identificar posibles procesos microevolutivos y modelar la estructura poblacional pasada.
El ADN mitocondrial (ADNmt) es actualmente el genoma más estudiado a nivel pobla-cional y el que mayores posibilidades de recuperación tiene en restos arqueológicos debido a sus características intrínsecas (Matisoo-Smith y Horsburgh, 2012). El análisis de los linajes mi-tocondriales procedentes de restos humanos precolombinos (ADN antiguo - ADNa) se ha convertido en una herramienta válida para contestar interrogantes vinculados al estudio de procesos de poblamiento, migraciones, relaciones entre poblaciones y cambios y/o continuidad poblacional mediante el cambio o continuidad en las frecuencias de los haplogrupos mitocondriales a través del tiempo y el espacio (Matisoo-Smith y Horsburgh, 2012).
El objetivo de este trabajo es estudiar la distribución y composición de linajes maternos procedentes de restos humanos de diferentes sitios arqueológicos y colecciones de museos de la Patagonia argentina, con el fin de realizar una mejor aproximación a la diversidad genética mi-tocondrial de los grupos cazadores-recolectores que habitaron el área. De esta manera se espera aumentar la información del acervo genético de las poblaciones precolombinas de la región. A su vez, se compararán los resultados obtenidos con las diferentes hipótesis sobre el poblamien-to y variabilidad biológica esbozadas desde la arqueología y bioantropología, contribuyendo de esta manera a ampliar el conocimiento de la evolución de estos grupos humanos.
La arqueología del poblamiento de la Patagonia argentina
La dispersión humana en la región patagónica se produjo hacia los ca. 13000-12000 años
14C AP (Miotti y Salemme, 2004; Prates et al., 2013; entre otros), momento que se encuentra caracterizado por grandes fuctuaciones climáticas producto de la transición Pleistoceno-Holoceno, dando lugar a un paisaje diferente al actual en cuanto a la disponibilidad de territorio habitable debido a la posición de los glaciares, el nivel de las aguas y las condiciones regionales (McCulloch et al., 2000, Rabassa et al., 2009, Ponce et al., 2011).
Borrero (1989-1990, 1996, 2001) fue el primero en proponer un modelo de poblamiento desde una perspectiva biogeográfica y meta-poblacional, fundamentada en la disponibilidad y productividad ambiental (sobre todo por la disponibilidad de agua) y su relación con la distribución espacial y temporal del registro arqueológico. En dicho modelo, la ocupación de la Patagonia se habría dado en tres fases (exploración, colonización y ocupación efectiva del espacio), dependientes de los cambios ambientales y la disponibilidad de sectores para los grupos cazadores-recolectores. En este sentido, los sitios tempranos de la cuenca del río Deseado, la cuenca Magallánica y el norte de Tierra del Fuego serían los primeros núcleos poblaciona-les durante la primer fase (Miotti, 2003, 2006; Miotti y Salemme, 2004; Prates et al., 2013; entre otros).
La colonización comienza aproximadamente a los ca. 8000 años 14C AP, donde se detectan ocupaciones en distintas regiones (Orquera y Piana, 1999; Borrero, 2008; Morello et al., 2012; Cassiodoro et al., 2013; Martínez et al., 2013; García-Guraieb et al., 2015; Zubimendi et al., 2015; entre otros). En este período se registran cambios importantes a nivel geológico, ya que se liberan sectores anteriormente glaciados, se conforma el Estrecho de Magallanes como principal barrera biogeográfica de la región y se detectan eventos transgresivos marinos (Miotti y Salemme, 2004; Miotti, 2006; Rabassa et al., 2009). Se percibe un aumento de sitios arqueológicos y una ocupación más sistémica con mayor variabilidad artefactual posiblemente relacionada con el inicio del desarrollo de adaptaciones regionales, proponiéndose posibles procesos de evolución cultural divergente posiblemente por vicarianza (Borrero 1989-1990, 1996, 2001).
La fase de ocupación efectiva del espacio comienza hacia el Holoceno tardío, aproximadamente a los ca. 3000 14C años AP, caracterizado por un aumento en la cantidad de sitios y la ocupación, por lo menos estacional, de espacios anteriormente vacíos o poco utilizados (Borrero, 2001; Miotti y Salemne, 2004). Se percibe en algunas regiones mayores densidades demográ-ficas, posibles constreñimientos espaciales, la aparición de la territorialidad, la intensificación en ciertos recursos y la reducción de la movilidad con asentamientos más estables y prolongados (Borrero, 2008; Borrero et al., 2009, 2011; Favier Dubois, 2013; Martínez et al., 2013; Bar-berena et al., 2015; García Guraieb et al., 2015; entre otros).
El poblamiento de Patagonia puede ser visto como una progresión no lineal en la ocupación de los ambientes más óptimos (nodos) hacia las regiones menos "deseables", posiblemente visitadas para alguna tarea en particular. Las principales rutas de dispersión habrían sido los cursos de agua permanente y posiblemente, el litoral atlántico (Miotti, 2006).
Antecedentes desde la bioantropología
La morfometría craneal y dental ha aportado mucha información sobre el poblamiento de la región. Incialmente se planteó a la robusticidad como característica de estas poblaciones, producto de la retención de la morfología de los primeros habitantes y una cierta homogeneidad a nivel poblacional cuando se las compara con otros grupos de América (Lahr, 1995). Sin embargo, análisis posteriores han propuesto un origen dual, caracterizado por una morfología pre-mongoloide o paleoamericana y otra de características mongoloides o amerindias (González-José et al., 2001, 2002; Pucciarelli, 2009; Sardi et al., 2005; entre otros), la cual a su vez sería refejo de la diversidad en todo el continente y denotaría posibles eventos de reemplazo (Neves et al., 2003; Pucciarelli 2009; Hubbe et al., 2014; entre otros).
Otros han propuesto a la deriva como el principal proceso microevolutivo y al accionar de factores aleatorios y no aleatorios como principales mecanismos modeladores de la diversidad biológica presente en estas poblaciones (Sardi et al., 2005; Bernal et al., 2006; Pérez et al., 2007). La baja densidad demográfica y el relativo aislamiento biogeográfico podrían haber genera-
do escenarios proclives para la generación de efectos fundadores seriales, lo cual explicaría la correlación entre las distancias biológicas y geográficas observadas en piezas dentales del Holoceno Tardío (Bernal et al., 2010a, b). Este último modelo plantea múltiples expansiones y sucesivas dispersiones de grupos pequeños de cazadores-recolectores, por lo que cada una de ellas estaría refejando sólo parte de la variación genética y morfológica del grupo primigenio.
Los primeros estudios de ADNmt en Patago-nia fueron realizados en poblaciones originarias actuales de Argentina y Chile (Ginther et al., 1993; Baillet et al., 1994; Moraga et al., 2000; García et al., 2004, 2006) encontrando la existencia de un cline norte-sur de los haplogrupos A y B en favor de C y D (estando A y B ausentes en las regiones más meridionales), posiblemente producto del efecto fundador.
Gracias a la secuenciación completa del ADNmt, posteriores investigaciones lograron confeccionar una flogenia de mayor resolución. Bodner et al. (2012), identificaron dos linajes específicos llamados D1g y D1j, que representarían la primera migración por una ruta costera pacífica debido a su ubicación geográfica y tiempos de coalescencia cercanos a los 15.000-18.000 años AP. Por su parte De Saint Pierre et al., (2012a, b) demostraron la existencia de un cline norte-sur de A2, B2, C1 y D1, desapareciendo los dos primeros e incrementándose los dos últimos, con una alta frecuencia de D1g en todos los casos. Adicionalmente define la presencia de dos clados (llamados B2l y C1b13) característicos de estas poblaciones y sólo des-criptos en Sudamérica con tiempos de coales-cencia cercanos a los 15000 años AP. Asimismo, el linaje D4h3a parecería estar confinado al Sur de la Patagonia tanto chilena como argentina. Según estos autores, D1g y B2l aparecieron en cazadores-recolectores del extremo sur, junto a D4h3a, comprendiendo el 87,6% de la variación mitocondrial, sugiriendo extremo aislamiento de las poblaciones más al sur.
En restos humanos antiguos Lalueza et al., (1997) y posteriormente García-Bour et al., (2004) sobre las mismas muestras de grupos Kaweskar, Aonikenk, Yámana y Selk´nam descubrieron la presencia de los linajes C1, D1, D1g y D4h3a, explicando la ausencia de A2 y B2 por efecto fundador. Posteriormente, Moraga et al. (2010), analizaron restos Kaweskar de los archipiélagos chilenos datados entre 1700 y 150 años AP, hallando C1, D1g y D4h3a, avalando las hipótesis de un poblamiento pacífico y el accionar de la deriva como explicación de la reducción de las variedades mitocondriales. Sin embargo, Manríquez et al. (2011), hallaron los linajes C y B en individuos del sitio Baño Nuevo 1 en la Patagonia chilena datados en ca. 9000 años AP, demostrando la pérdida de distintos haplogrupos por efecto de deriva y aislamiento geográfico.
En los últimos años, en el marco de diversos congresos, se han presentado trabajos de ADNa realizados sobre restos esqueletales de diversos sectores y temporalidades de Patagonia argentina (Postillone y Dejean, 2013; Crespo et al., 2014; Postillone et al., 2015, 2016, Crespo et al., 2016), las cuales han avalado las hipótesis planteadas anteriormente sobre la presencia y distribución de alguno de estos linajes maternos.
MATERIAL Y MÉTODOS
Las muestras analizadas
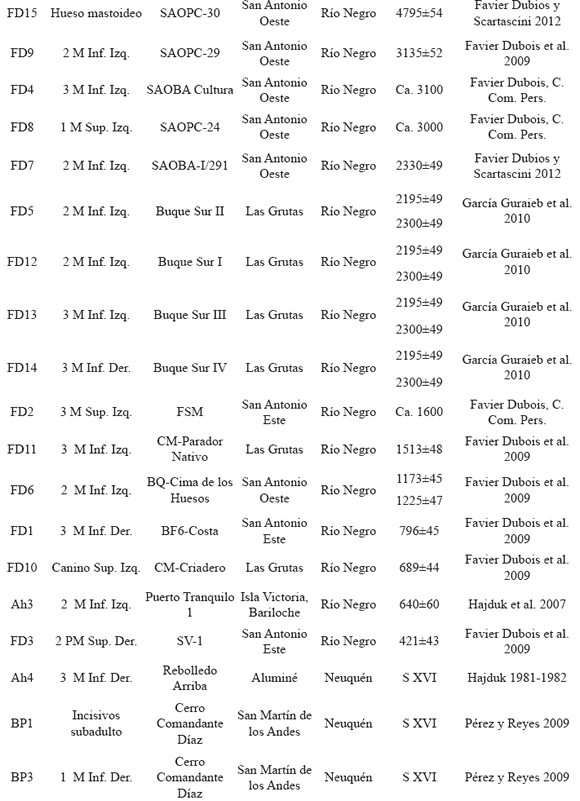
Se analizaron un total de 50 muestras de individuos procedentes de sitios arqueológicos y colecciones de museos de diferentes regiones de la Patagonia argentina, los cuales poseen una cronología desde el Holoceno Medio hasta la época de contacto hispano indígena (Tabla 1 y Fig. 1).
Se seleccionaron piezas dentales de cada individuo para realizar los análisis además de un hueso largo procedente de la colección del museo Rosa Novak, Puerto San Julián, provincia de Santa Cruz. Cada muestra fue elegida siguiendo los lineamientos descriptos en Dejean et al. (2014), excluyendo de los análisis aquellas que mostraran fsuras, quebraduras o excesivo desgaste debido a sus mayores probabilidades de poseer contaminación por exposición de su parte interna y para preservar el material óseo de cada museo y sitio arqueológico.

Fig. 1. Regiones geográficas de donde proceden las muestras analizadas en este trabajo (puntos verdes). NPC: Norpatagonia costera (n: 23). NPA: Norpatago-nia andina (n: 17). CST: Costa de Santa Cruz (n: 6). CdB: Canal de Beagle (n:4).



Todos los análisis fueron realizados en el Centro de Estudios Biológicos, Biotecnológicos, Ambientales y de Diagnóstico (CE-BBAD, Universidad Maimónides, Argentina), el cual cuenta con un laboratorio separado físicamente, y dedicado exclusivamente al análisis de especímenes antiguos. Todos los operadores involucrados utilizaron doble par de guantes, cofias, guardapolvos y batines descartables y su ADNmt fue previamente tipificado con el objeto de identificar posibles contaminaciones con ADN de los investigadores. Se utilizaron materiales plásticos des-cartables, los cuales fueron irradiados con luz UV por 45 minutos antes de ser utilizados. Los reactivos y soluciones fueron alicuotados y de uso exclusivo para ADN antiguo. Todas las superficies y equipos de laboratorio fueron lavados con hipoclorito de sodio 10% y etanol 70% y enjuagados con agua libre de DNAsas y RNAsas, para posteriormente ser irradiados con luz UV por 45 minutos antes y después de cada tarea. Las áreas de trabajo pre y post amplificación se encuentran estrictamente separadas.
Debido a los posibles problemas de contaminación asociados al análisis de muestras humanas antiguas y sobre todo a aquellas que no fueron recuperadas en ambientes controlados y con un registro detallado de su manipulación (Yang y Watt, 2005; Matisoo-Smith y Horsburgh, 2012; entre otros), estrictos protocolos de decontaminación fueron llevados adelante con el fin de remover el ADN exógeno de la superficie de cada muestra. Para las piezas dentales se utilizó hipoclorito de sodio según diversas recomendaciones y posteriormente tandas de 45 minutos de luz UV en cada cara de la pieza (Kemp y Glenn-Smith, 2005; Dissing et al., 2008; Barta et al., 2013; entre otros).
Obtención del material óseo
Se obtuvo la dentina de cada pieza dental por desbaste utilizando la técnica reportada en otros trabajos (Kuch et al., 2007; Dejean et al., 2014). Para el caso del hueso, el acopio del material se realizó siguiendo los protocolos descriptos en Gibbon et al. (2009). Se obtuvo en cada caso aproximadamente 300/400mg de material óseo, realizándose alícuotas de entre 70 y 80mg (Kuch et al., 2007; Dejean et al., 2014) para cada muestra de cada individuo con el fin de poner a prueba diferentes protocolos de extracción y poder autenticar los análisis con diferentes extracciones y tipificaciones de cada individuo. Adicionalmente, para los individuos procedentes del museo de la Patagonia, se realizó una contraprueba mediante el análisis de una segunda pieza dental.
Luego de finalizado el trabajo con cada una de las muestras, las mismas fueron remontadas con un monómero autocurable de uso odontológico preservando de esta manera la morfología externa para posteriores análisis (Dejean et al., 2014).
Extracción y tipificación del ADNmt
El material óseo de cada individuo fue descal-cificado y las proteínas digeridas con Proteinasa K. La solución obtenida fue extraída con solventes orgánicos y el sobrenadante de cada extracto fue purificado y concentrado por medio de columnas de sílica (ver protocolo de extracción en Russo et al., 2016, en este mismo volumen).
Se determinaron los haplogrupos mitocon-driales mediante la técnica de RFLP (restriction fragment length polymorphism) y la secuen-ciación de la Región Hipervariable-1 (RHV-1) del genoma mitocondrial. Para ambas técnicas se utilizaron los cebadores y las condiciones de reacción descriptas por Russo et al., 2016 (en este volumen). Las reacciones de secuenciación se llevaron a cabo utilizando los mismos cebadores empleados en la amplificación de cada fragmento, en un secuenciador automático 3130 XL Genetic Analyzer con BigDyeT Terminator Sequencing Kit (Applied Biosystems), por la Unidad de Genómica del Instituto de Biotecnología del INTA.
Análisis de las secuencias obtenidas
Se consideraron como auténticos sólo aquellos resultados que pudieron reproducirse tanto por RFLP como por la secuenciación de la RHV-1 desde múltiples extracciones y amplif-caciones de cada una de las muestras. Se corroboró que ninguno de los perfles obtenidos coincidiera con el perteneciente a los operadores que procesaron las muestras (CMC y CBD).
Se obtuvo una secuencia consenso de los múltiples fragmentos amplificados de la RHV-1 para cada individuo analizado, las cuales fueron leídos y alineados a la Secuencia de Referencia de Cambridge revisada (Andrews et al., 1999) con el programa MEGA 5.1 (Tamura et al., 2011). Se determinaron los haplotipos de cada individuo mediante el programa Haplogrep (Klöss-Brandstätter et al., 2011) la cual se encuentra basado en Phylotree (Van Oven y Kay-ser, 2008) y mediante búsqueda bibliográfica.
Análisis estadísticos
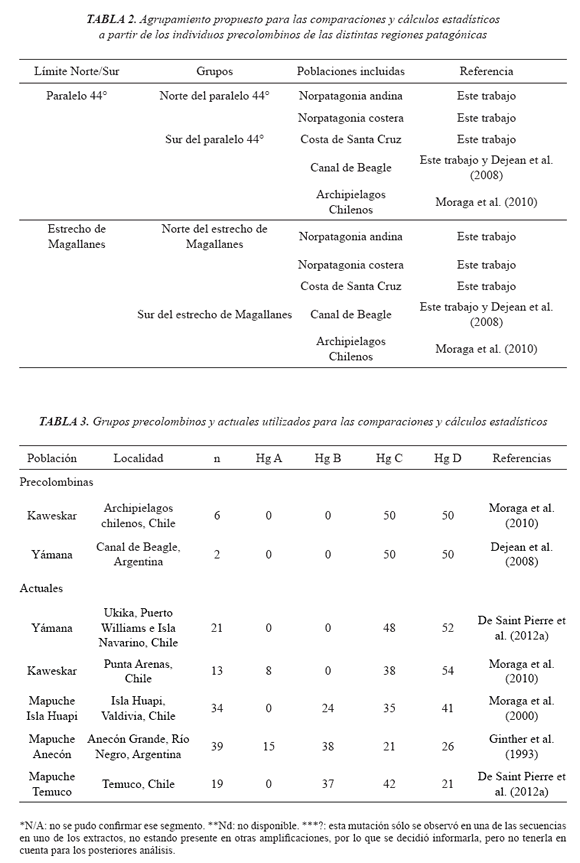
Se realizaron dos agrupaciones Norte-Sur diferentes según las divisiones propuestas por Borrero (2008). La primera estableciendo como límite el paralelo 44° y la segunda por el Estrecho de Magallanes (Tabla 2). En ambos casos, debido a que el tamaño muestral es pequeño en el grupo Sur, se incluyeron secuencias antiguas publicadas por otros investigadores procedentes del Sur de los archipiélagos chilenos y del Canal de Beagle (Tabla 2).
Se investigó si existen diferencias genéticas estadísticamente significativas entre los grupos Norte-Sur propuestos a través del Análisis Molecular de la Varianza (AMOVA) con el programa Arlequin v.3.5.1.3 (Excoffer y Schneider, 2010) y su significancia fue obtenida con 1000 permutaciones. El modelo de evolución molecular utilizado fue TN93+G (Tamura y Nei, 1993) -G=0,36- elegido con el programa MEGA 5.1 (Tamura et al., 2011).
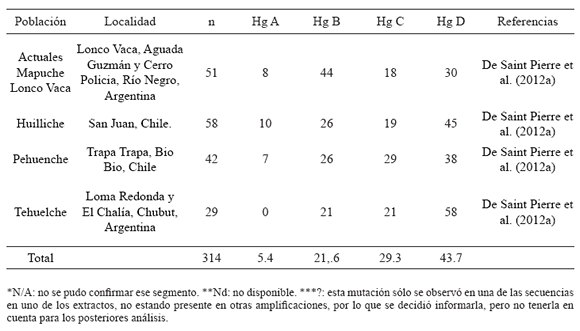
Por otro lado, se estudió la variabilidad genética en la región analizando diversas poblaciones precolombinas y originarias actuales (Tabla 3). Para cada una de las poblaciones y de los grupos Norte-Sur previamente propuestos, se calculó el número de haplotipos, la diversidad haplotípica y la diversidad nucleotídica con el programa DnaSP v.5.10 (Librado y Rozas, 2009). Estos mismos estadísticos se calcularon además para cada haplogrupo (A2, B2, C1 y D1) a fin de comparar la diversidad presente en cada linaje hallado, incluyendo en los mismos a los grupos antiguos y actuales.
Para correlacionar la distancia genética entre pares de individuos con la distancia geográfica de los mismos dentro del conjunto de datos se realizó un análisis de Procrustes. Para ello se utilizaron todas las secuencias precolombinas obtenidas en este trabajo como aquellas disponibles en bibliografía (Tabla 3) y a cada secuencia se le asignó su coordenada geográfica. En caso de poseer puntos exactos por medio de equipos GPS se utilizaron dichas coordenadas. En caso de no poseer este registro detallado, se estimaron sus coordenadas geográficas con la aplicación Google Earth según la información que fgura en el registro museístico de cada individuo analizado. Las distancias genéticas entre pares de secuencias se calcularon con el programa MEGA 5.1 (Tamura et al., 2011), siendo TN93 (Tamura y Nei, 1993) el modelo evolutivo que más se ajustaba al conjunto de datos. A partir de esta matriz de distancias genéticas se realizó un Análisis de Componentes Principales y se extrajeron los dos primeros ejes de variación, utilizando la función pca del paquete vegan (Oksanen et al., 2009) disponible en R 3.0 (R Core Team, 2014). Posteriormente para estos ejes se obtuvo la rotación que mejor se ajusta a las coordenadas geográficas utilizando las funciones procrustes y protest de vegan. Las coordenadas genéticas y geográficas obtenidas fueron mapeadas con la aplicación QGis 2.2.0 (QGis Fundation Project 2011).
Los haplotipos obtenidos de cada individuo fueron comparados con las secuencias de poblaciones precolombinas y originarias actuales de la región (Tabla 2) mediante la construcción de redes medianas para los linajes C1, D1 y D4h3a con el programa NETWORK v.4.6.3.1 (Bandelt et al., 1999; fuxus-engineering.com). Los pesos evolutivos de cada sitio polimórfico fueron aplicados siguiendo los cálculos hechos por Soares et al., (2009). Para los linajes A2 y B2 no se construyeron redes debido a la baja cantidad de secuencias disponibles de los mismos en la región y a la poca representatividad que poseen en el conjunto de las muestras antiguas analizadas.
RESULTADOS
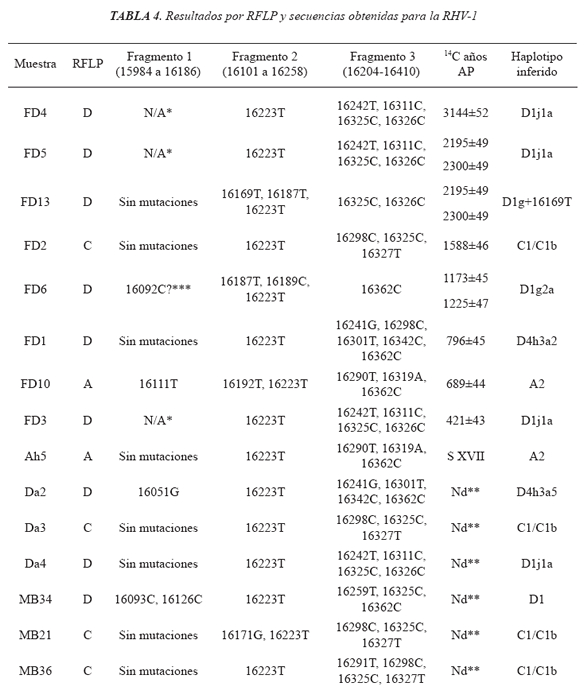
De los 50 individuos analizados en este trabajo pudieron tipificarse por RFLP 27 de ellas (57% de recuperación), mientras que en las 23 restantes (46%) se obtuvieron amplificaciones espurias o fueron negativas. La mayoría de los haplogrupos presentes correspondieron a D (55,6%), seguido de C (37%) y A (7,4%), es- los 1.588 14C años AP también hasta tiempos tando ausente el haplogrupo B. El primero de históricos, mientras que A se encuentra asocia-estos linajes es el que posee mayor antigüedad do a momentos del Holoceno final y de contacto (3.100 14C años AP) y se presenta hasta tiem- hispano-indígena (ver Tabla 4). La distribución pos históricos. El segundo se detecta a partir de espacial demuestra que tanto C como D se encuentran presentes en todas las regiones analizadas, mientras que A sólo se encuentra en Nor-patagonia, tanto en su vertiente atlántica como andina (Fig. 2).
Caracterización de los linajes obtenidos
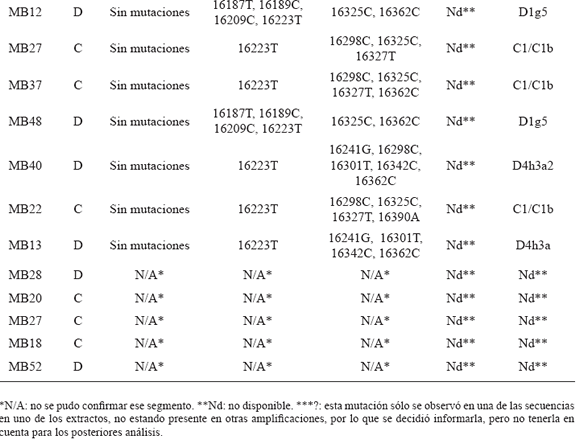
Se identificó el linaje mitocondrial mediante la secuenciación de la RHV-1 en 22 muestras (44% de recuperación), mientras que en las 28 restantes (56%) no se obtuvieron ampificaciones o las mismas fueron espurias y no se pudieron determinar o confrmar el segmento completo de la región analizada. En todos los casos positivos las secuencias fueron concordantes con los resultados obtenidos por RFLP (Tabla 4).
Se observa que dentro del linaje D se encuentran las variedades D1j (18,18%), D1g (18,18%), D4h3a (18,18%) y un D1 (4,54%).
Las muestras correspondientes al linaje C (31,81%) se clasificaron como C1/C1b debido a que su mayoría presentan las mutaciones nodales y la región analizada posee poca resolución para discriminar variedades más específicas de este haplogrupo. El linaje A (9,09%) presentó la variedad nodal en un individuo, mientras que el restante pudo clasificarse como A2. En este caso tampoco pudo discriminarse un clado específico con la secuenciación de la RHV-1.
El linaje D1j presentó la mayor profundidad temporal, seguido por D1g, C1 y A2. A su vez, el mismo fue hallado tanto al norte como al sur de Patagonia, pero en sectores exclusivamente costeros (Fig. 2). Los linajes D4h3a y C1 se detectaron en todas las regiones analizadas. Por su parte, D1g y A2 sólo fueron hallados en Norpatagonia (sólo en el sector costero el primero y en los sectores costero y andino, el segundo).

Fig. 2. Izquierda: distribución geográfica de los haplogrupos obtenidos. 1:Norpatagonia costera (n:23). 2:Norpa-tagonia andina (n:17). 3:Costa de Santa Cruz (n:6). 4: Canal de Beagle (n:4). Derecha: distribución geográfica de los linajes obtenidos por secuenciación de la RHV-1. 1: Norpatagonia costera (n: 11). 2: Norpatagonia andina (n: 4). 3: Costa de Santa Cruz (n: 4). 4: Canal de Beagle (n:3).
Variabilidad y diferenciación genética en Patagonia
Los análisis de variabilidad realizados demuestran que los grupos armados según la región de procedencia de cada muestra poseen niveles similares de diversidad haplotípica y nucleotídi-ca, siendo la de Norpatagonia andina la menor de todas (Tabla 5). De las agrupaciones Norte-Sur propuestas, los niveles de diversidad haplotípica resultaron similares, pero la nucleotídica resultó menor en los sectores meridionales en ambos casos, más allá de la inclusión de los individuos de la cosa de Santa Cruz en cualquiera de los grupos. A su vez, se observan magnitudes similares cuando se comparan con los grupos modernos, pero en este caso la variabilidad menor se encuentra en grupos actuales Kaweskar y Yámana (Tabla 5).

Cuando se comparan los linajes se observa que la menor variación se encuentra en C1, seguida por A2. La diversidad haplotípica de B2 y D1 son las mayores, pero la nucleotídica es superior en D1, lo cual demuestra mayor variabilidad dentro de este linaje (Tabla 5).
El AMOVA realizado no evidencia diferencias genéticas entre los grupos del Norte y Sur de Patagonia, tanto tomando como límite el paralelo 44° como al Estrecho de Magallanes (p=0,15543; Fst=0,03965 y p=0,23851;Fst=0,01924, respectivamente), Esto estaría demostrando la ausencia de estructuración poblacional con los datos de ADNa disponibles hasta el momento. Consistentemente, el análisis de Procrustes demostró una baja correlación entre las distancias genéticas y geográf-cas (r=0,3366) la cual no resultó ser estadísticamente significativa (p=0,061) (Fig. 3).
La red mediana del linaje C1 presenta una topología unimodal en estrella, la cual suele interpretarse como producto de un cuello de botella seguido de expansión poblacional (Fig. 4). La mayor cantidad de los individuos se ubican en la posición nodal de este haplogrupo, estando unos pocos individuos de diversas poblaciones modernas y antiguas diferenciadas por unas pocas mutaciones. De las muestras analizadas en este trabajo sólo dos se diferenciaron por una mutación: una de la costa de Santa Cruz (MB21) con la mutación 16171G y otra procedente del Canal de Beagle (MB36) con la mutación 16291T (Tabla 3).
La red que incluye los linajes D1 y D4h3a presenta una topología multimodal (Fig. 5). El motivo nodal del primero (16223T, 16325C y 16362C) posee una baja representación y sólo se encuentra presente en Yámana. Sólo un individuo antiguo analizado aquí presenta este cla-do (MB34), pero con las mutaciones adicionales 16093C, 16126C y 16259T. Las variedades ma-yoritarias fueron D1j, D1g y D4h3a. Cuatro individuos (FD3, FD4, FD5 del Golfo de San Matías y Da4 de la costa de Santa Cruz) presentaron la variedad D1j (D1+16242T y 16311C) compartida por grupos Mapuche de Anecón y Lonco Vaca. Por su parte, el linaje D1g (D1+16187T) estuvo presente en cuatro individuos del Golfo de San Matías. La muestra FD6 presentó las mutaciones 16189C y @16325C, compartiendo motivos con múltiples grupos actuales. Uno de los extractos de esta muestra presentó la mutación 16092C y compartiría haplotipo con grupos Tehuelche, Mapuche y Pehuenche, pero no pudo confrmarse hasta el momento. Dos individuos (MB12 y MB48) presentaron las mutaciones 16189C y 16209C, la cual es compartida por Mapuche, Tehuelche y Pehuenche. La última muestra perteneciente a este linaje es FD13, la cual posee la mutación 16169T y no es compartida por ninguna población actual o antigua aquí analizada.
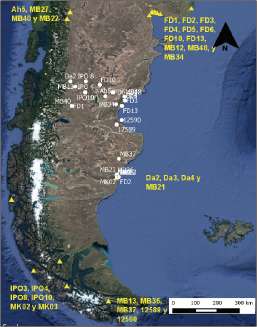
Fig. 3. Mapa de la región patagónica en el cual se marcan los puntos geográficos de las muestras analizadas en este trabajo utilizadas para el análisis de Procrustes (puntos amarillos) y la ordenación de las distancias genéticas en base las distancias geográficas (puntos blancos). La fecha negra indica el norte geográfico.
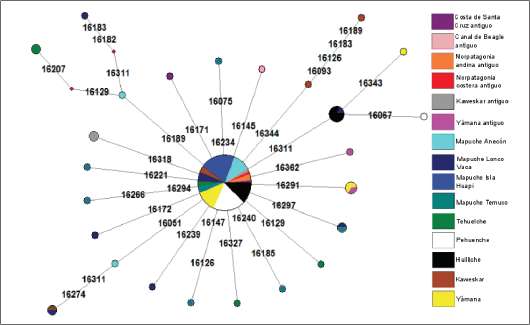
Fig. 4. Red mediana de haplotipos del linaje C1. El tamaño de las tortas es proporcional a la frecuencia de ese haplotipo.
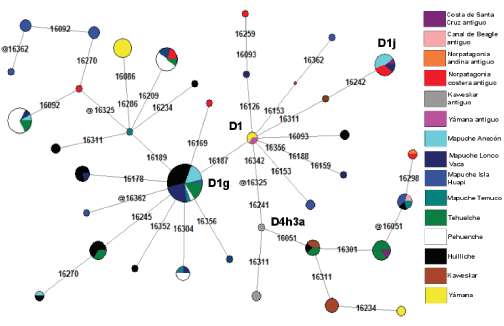
Fig. 5. Red mediana de haplotipos para los linajes D1 (incluyendo las variedades D1g y D1j) y D4h3a. El tamaño de las tortas es proporcional a la frecuencia de ese haplotipo. El @ indica las posiciones revertantes de cada clado
El linaje D4h3a se halló en cuatro individuos. En tanto que FD1 del Golfo de San Matías y MB40 de Norpatagonia Andina presentaron el mismo haplotipo caracterizado por las mutaciones 16298C y 16301T y no compartidas por ninguna población. Por su parte Da2 de la costa de Santa Cruz presentó las mutaciones 16051G y 16301T, compartiendo haplotipo con Tehuel-che. El último individuo perteneciente a este linaje es MB13 procedente del Canal de Beagle y posee la mutación 16301T compartiendo esta variedad con Huilliche y Mapuche.
Sólo las muestras Ah5 (Norpatagonia andina) y FD10 (Norpatagonia costera) presentaron el linaje A2. La primera de ellas presentó su forma nodal, la cual está presente en Mapuche y la segunda de ellas posee las mutaciones 16111T y 16192T y no se encuentra dentro del conjunto de las poblaciones utilizadas para las comparaciones.
DISCUSIÓN
El estudio de los haplotipos mitocondriales a través del espacio patagónico con las muestras antiguas analizadas hasta el momento da cuenta de una ausencia de diferenciación y estructuración poblacional dentro de la Patagonia argentina desde el análisis de este marcador. Tanto los estudios de diversidad genética, como el AMOVA, la correlación entre las distancias biológicas y geográficas y las redes realizadas apuntan hacia una baja diferenciación entre las regiones analizadas y a una escasa cantidad de linajes maternos en todos los grupos antiguos estudiados. Muchos de los haplotipos hallados se encuentran dispersos en distintos lugares de Patagonia y son compartidos por diversos grupos antiguos y actuales, lo cual también apoya la noción de cierta continuidad poblacional. Este hecho también podría estar refejando la baja demografía planteada desde el registro arqueológico (Borrero, 2001; Miotti y Salemme, 2004) y un origen poblacional común con poco fujo génico con otras regiones geográficamente más distantes y posiblemente eventos de efectos fundadores seriales. Los resultados acompañan muchos de los postulados propuestos desde la craneometría en cuanto a la homogeneidad hallada en las poblaciones de diversos sectores de la Patagonia (Lahr, 1995, González-José et al., 2001, 2002). A su vez, también apoya las investigaciones previas en grupos antiguos y actuales en cuanto a la baja diversidad genética mitocon-drial hallada en esta región y la pérdida de diversidad de linajes maternos hacia el Sur refejada en la ausencia del linaje B2 y la muy baja frecuencia de A2 en Norpatagonia y su ausencia en regiones más meridionales (Lalueza et al., 1997, Moraga et al., 2000, García-Bour et al., 2004, Moraga et al., 2010).
Una cuestión llamativa es la distribución encontrada para ciertos linajes. Los modelos de poblamiento de la región han postulado una ruta Pacífica con un ingreso transcordillerano para los clados D1g, D1j y D4h3a (Bodner et al., 2012; De Saint Pierre et al., 2012a). Recientemente se ha postulado para D1j un posible origen en el Centro y Noroeste de Argentina fundamentado en la alta frecuencia hallada de este linaje en la actualidad y el hallazgo del mismo en restos humanos de ca. 4200 14C años AP (García et al., 2012). Sin embargo, los análisis aquí realizados demuestran su presencia en la costa Atlántica desde los 3100 14C años AP, lo cual podría explicarse por un origen poblacional común siguiendo una ruta de poblamiento interior o costera atlántica hasta Patagonia o eventos de fujo génico. Para los linajes D1g y D4h3a se observa una situación similar, los cuales también poseen una locación atlántica. Este hecho puede estar demostrando que la distribución de las variedades mitocondriales y su diversidad en momentos pre-colombinos pudo ser diferente a la que se encuentra en la actualidad, cuestión posiblemente avalada por el hallazgo de dos ha-plotipos (muestras FD13 y MB34, Tabla 3) no descriptos hasta el momento. Desde el estudio de los sitios arqueológicos tempranos también se ha postulado la importancia del litoral atlántico como corredor de dispersión de los primeros grupos humanos (Miotti 2006).
Siguiendo la misma lógica, resulta interesante la frecuencia detectada del haplogrupo B2 en grupos modernos Mapuche, Tehuelche, Huilliche y Pehuenche, ya que en los individuos antiguos de Argentina no se ha encontrado dicho linaje. Este hecho ha sido explicado por De Saint Pierre et al., (2012b) debido a migraciones provenientes del Centro de Chile asociadas con el proceso de Araucanización a partir del Siglo XVI. Adicionalmente, diversos estudios etno-históricos han señalado la existencia de poligi-nia, virilocalidad y posiblemente casamientos mixtos dentro de la estructura social Mapuche (Boccara, 1999). Estos hechos podrían explicar las discrepancias halladas con respecto a la ausencia de dicho linaje en muestras antiguas de Norpatagonia y su presencia en poblaciones actuales o indicar un problema de muestreo en las poblaciones precolombinas y modernas. Seguramente a partir del siglo XVI y como producto del fujo génico desde la Araucania, el acervo genético mitocondrial de Norpatagonia podría haberse modificado, incorporando nuevas variedades (como B2).
Según el modelo arqueológico de pobla-miento, la Patagonia presentaría un escenario proclive para que ocurrieran divergencias po-blacionales, y por ende, sería esperable que se encontraran diferencias genéticas significativas entre las regiones muestreadas debido a procesos de "vicarianza" (Borrero, 1989-1990, 1996), producto de la baja demografía de las poblaciones cazadoras-recolectoras y el establecimiento de barreras biogeográficas. Si aceptamos la propuesta de la fuerte presencia de deriva al momento del ingreso de las primeras poblaciones a la región, la cual habría reducido la variabilidad mitocondrial inicial a sólo unas pocas variantes y consideramos que estos grupos poseían una muy baja demografía hasta el Holoceno tardío, cabría esperar que bajos niveles de variación genética mitocondrial fueran la regla.
Si bien esto puede ser verdad para los primeros momentos del poblamiento, el registro arqueológico del Holoceno tardío muestra grandes radios de movilidad logística y/o redes de interacción amplias, los cuales se encuentran avalados por diferentes evidencias arqueológicas de contacto entre distintas regiones (Cara-cotche et al., 2005; Gómez Otero y Stern, 2005; Tessone et al., 2005; Orquera y Gómez Otero, 2007; Favier Dubois et al., 2009; Martínez et al., 2009; Silveira et al., 2010; Carballo Marina et al., 2011; Stern et al., 2012; entre otros), por lo que es difícil sostener que muchos de estos grupos o metapoblaciones se mantuvieran completamente aislados el tiempo suficiente como para generar una diferenciación biológica de magnitud.
Recientemente, Borrero et al. (2011), ha planteado para el sur de Patagonia a través de indicadores isotópicos y relatos etnográficos la posible existencia de grupos socialmente muy dinámicos y que posiblemente poseyeran sistemas de visitas, intercambio de información y bienes y la búsqueda de pareja o casamientos mixtos, siendo posible la conformación de grupos culturales mezclados y socialmente abiertos a nivel supra-regional a partir de los 2000 años AP. De ser cierta esta posibilidad, podría pensarse si este esquema no es también aplicable a sectores más septentrionales.
Dicho fujo génico entre diferentes regiones de la Patagonia se habría dado a partir de un conjunto muy pequeño de variantes mitocondriales y de origen común relativamente reciente. Con el correr de los milenios habría disminuido la diferenciación inter-poblacional alcanzando una suerte de "homogenización", dando como resultado que la mayoría de los grupos compartieran muchos haplotipos, pero poseyendo alguno de ellos variedades privativas propias de cada región, no siendo suficientes estas últimas para diferenciarlos significativamente.
De todas maneras, el número de individuos analizados aún es pequeño, por lo que podría esperarse que al aumentar el muestreo y ampliar las regiones y temporalidades analizadas puedan observarse patrones diferentes a los aquí hallados.
CONCLUSIONES
Distintas líneas de evidencia han postulado para las poblaciones cazadoras-recolectoras una baja demografía y el accionar de la deriva génica para explicar los patrones observados en cuanto a la distribución del registro arqueológico y la variabilidad biológica encontrada. Las secuencias de la RHV-1 del ADNmt de restos humanos precolombinos analizados en este trabajo parecen avalar estas hipótesis. La mayoría de los haplotipos hallados se encuentran presentes en diferentes sectores de la región y a su vez, compartidos por muchos de los grupos originarios actuales, dificultando la identificación de diferencias genéticas estadísticamente significativas entre los diferentes grupos antiguos analizados, pero siendo siempre mayor la variabilidad de los grupos septentrionales. Por otro lado, la distribución de los linajes D1g, D1j y D4h3a es diferente a la propuesta desde el análisis de grupos actuales, sugiriendo la importancia de los sectores atlánticos en la dispersión humana.
Adicionalmente, se ha detectado en grupos modernos frecuencias considerables del linaje B2, estando el mismo ausente en los individuos precolombinos analizados. Si bien esta ausencia podría vincularse con un problema de muestreo en individuos antiguos, es probable que los procesos de migración trasandina (araucanización) hayan introducido nuevas variedades mitocon-driales. Este hecho podría haber modificado la composición y diversidad del acervo genético de las poblaciones del área.
AGRADECIMIENTOS
Agradecemos el apoyo financiero institucional recibido. También agradecemos al Dr. Favier Dubois, al Lic. Alberto Pérez, al Museo de la Patagonia "Francisco P. Moreno" y al Museo regional "Rosa Novak" por facilitarnos las muestras analizadas en este trabajo y a los dos revisores anónimos por los aportes realizados para mejorar este artículo.
LITERATURA CITADA
Andrews R, Kubacka I, Chinnery P, Lightowlers N, Tur-nbull D, Howell N. 1999. Reanalysis and revision of the Cambridge reference sequence for Human mitochon-drial DNA. Nat Genet 23:147. [ Links ] doi:10.1038/13779
Baillet G, Rothhammer F, Carnese F, Bravi C, Bianchi N. 1994. Founder mitochondrial haplotypes in Amerindian populations. J Hum Genet 54:27-33. [ Links ]
Bandelt H, Forster P, y Röhl A, 1999. Median-joining net-works for inferring intraspecific phylogenies. Mol Biol Evol 16:37-48. doi:10.1093/oxfordjournals.molbev. a026036
Barberena R, Prates L, De Porras M. 2015. The human occupation of North Western Patagonia (Argentina): paleoecological and chronological trends. Quatern Int 356:111-126. doi: 10.1016/j.quaint.2014.09.055
Barta J, Monroe C, Kemp B. 2013. Further evaluation of the efficacy of contamination removal from bone Sur-face. Forensic Sci Int 231:340-348. doi:10.1016/j. forsciint.2013.06.004
Bernal V, Pérez I, González P. 2006. Variation and causal factors of craniofacial robusticity in Patagonian hunter-gatherers from the Late Holocene. Am J Hum Biol 18:748-765. doi: 10.1002/ajhb.20546
Bernal V, Pérez I, González P, Sardi M, Pucciarelli H. 2010a. Spatial patterns and evolutionary processes in southern South America: a study of dental morphometric varia-tion. Am J Phys Anthropol 142:95-104. doi: 10.1002/ ajpa.21206
Bernal V, Pérez S, González P, Diniz-Filho A. 2010b. Eco-logical and evolutionary factors in dental morphologi-cal diversification among modern human populations from southern South America. P Roy Soc Lond B Bio 277:1107-1112. doi: 10.1098/rspb.2009.1823
Boccara G. 1999. Etnogénesis mapuche: resistencia y restructuración entre los indígenas del centro-sur de Chile (siglos XVI-XVIII). Hispanic Am Hist Rev 79:425-461.
Bodner M, Perego U, Huber G, Fendt L, Rock A, Zimmer-mann B, Olivieri A, Gómez-Carballa A, Lancioni H, Angerhofer N, Bobillo M, Corach D, Woodward S, Salas A, Achilli A, Torroni A, Bandelt H, Parson W. 2012. Rapid coastal spread of frst Americans: novel insights from South American´s Southern Cone mitochon-drial genome. Genome Res 22:811-820. doi:10.1101/ gr.131722.111
Borrero LA. 1989-1990. Evolución cultural divergente en la Patagonia austral. Anal Inst Pat 19:133-140.
Borrero LA. 1996. The Pleistocene-Holocene transition in Southern South America. En: Straus L. Eriksen B. Er-landson J Yesner D, editores. Human at the end of the ice age: the archaeology of Pleistocene-Holocene tran-sition. New York: Plenum Press. p 339-354.
Borrero LA. 2001. El poblamiento de la Patagonia. Toldos, milodones y volcanes. Buenos Aires: Emecé editores.
Borrero LA. 2008. Early occupations in the southern cone. En: Silverman H Isbell W editores. Handbook of South American Archaeology. New York: Springer. p 59-77.
Borrero LA, Barberena R, Franco N, Charlin J, Tykot R. 2009. Isotopes and rocks: geographical organization of southern patagonian hunter-gatherers. Int J Osteoar-cheol 19:309-327. doi:10.1002/oa.1036
Borrero LA, Martin F, Barberena R. 2011. Visits, "fue-gians", and information networks. En: Whallon R. Lo-vis W, Hitchcock R, editores. Information and its role in hunter-gatherer bands. Los Angeles: The Cotsen Institu-te of Archaeology Press. UCLA. p 249-265.
Caracotche M, Cruz I, Espinosa S, Carballo Marina F, Be-lardi J. 2005. Rescate arqueológico en el Parque Nacional Monte León (Santa Cruz, Argentina). Magallania 33:143-163. doi:10.4067/S0718-22442005000200010
Carballo Marina F, Belardi J, Saenz J. 2011. Distribución espacial del registro arqueológico en la unidad de paisaje terrazas, cuenca media del Río Coyle (Provincia de Santa Cruz, Argentina). Magallania 39:207-222. doi:10.4067/S0718-22442011000200015
Cassiodoro G, Rindel D, Goñi R, Re A, Tessone A, García-Guraieb S, Belardi J. Espinosa S. Nuevo de la Unay N. Dellepiane J. Flore Coni J. Guichon R. Martínez C, Pasqualini S. 2013. Arqueología del Holoceno medio y tardío en Patagonia meridional: poblamiento humano y fuctuaciones climáticas. Diálogo Andino 41:5-23. doi:10.4067/S0719-26812013000100002
Crespo CM, Postillone MB, Russo MG, Bravi CD, Avena SA, Favier Dubios C, Hajduk A, Arrigoni G, Lanata JL, Dejean CD. 2014. Genetic diversity in maternal ame-rindian lineages in Argentinian Patagonia. Actas XVII Mundial Congress of Prehistoric and Protohistoric Science. Burgos, España.
Crespo CM, Favier Dubois C, Russo MG, Lanata JL, Dejean CB. 2016. First analysis of ancient mtDNA and genetic diversity in northern Patagonia, Río Negro province, Argentina. Manuscrito presentado en la revista Journal of Archaeological Science.
De Saint Pierre M, Bravi C, Motti J, Fuku N, Tanaka M, Llop E, Bonatto S, Moraga M. 2012a. An alternative model for the early peopling of Southern South Ame-rica revealed by analyses of three mitochondrial DNA haplogroups. Plos One 7:e43486. doi:10.1371/journal. pone.0043486
De Saint Pierre M, Gandini F, Perego U, Bodner M, Gómez-Carballa A, Corach D, Angerhofer N, Woodward S, Semino O, Salas A, Parson W, Moraga M, Achilli A, Torroni A, Olivieri A. 2012b. Arrival of Paleo-Indians to the Southern Cone of South America: New Clues from Mitogenomes. Plos One 7:e51311. doi: 10.1371/ journal.pone.0051311
Dejean CB, Keyser C, Avena S, Dugoujon J, Ludes B, Crubezy E, Carnese F. 2008. Análisis genético de muestras esqueletales provenientes del Canal de Beagle. Actas del X Congreso Asociación Latinoamericana de Antropología Biológica, La Plata.
Dejean CB, Crespo CM, Carnese FR, Lanata JL. 2014. An-cient DNA research, scope and limitations. First gene-tic analysis in museum samples from San Julian, Santa Cruz, Argentina. En: Kligmann D, Morales M, editores. Physical, chemical and biological markers in Argentine archaeology: theory, methods and applications. Oxford: British Archaeological Reports. Archaeopress. p:53-62.
Dissing J, Kristinsdottir M, Friis C. 2008. On the elimination of extraneous DNA in fossil human teeth with hypochlorite. Journal Archaeol Sci 35:1445-1452.
Excoffer L, Lischer H. 2010. Arlequin suite ver 3.5: A new series of programs to perform population gene-tics analyses under Linux and Windows. Mol Ecol Res 10:564-567. doi:10.1111/j.1755-0998.2010.02847.x
Favier Dubois C, Stern C, Cardillo M. 2009. Caracterización de la distribución y tipos de obsidiana presentes en la costa rionegrina, Argentina. En: Salemme M, Santiago F, Alvarez M, Piana E, Vázquez M, Mansur ME, editores. Arqueología de Patagonia: una mirada desde el último confín. Ushuaia: Editorial Utopías. p 349-360.
Favier-Dubois C, Scartascini F. 2012. Intensive fshery sce-narios on the north Patagonian coast (Río Negro, Argentina) during the mid-Holocene. Quatern Int 256:62-70. doi:10.1016/j.quaint.2011.07.041
Favier Dubois C. 2013. Hacia una cronología del uso del espacio en la costa norte del Golfo de San Matías (Río Negro), Argentina: sesgos geológicos e indicadores temporales. En: Zangrando A, Barberena R, Gil A, Neme C, Giardina M, Luna L, Otaola C, Paulides S, Salgan C, Tívoli A, editores. Tendencias teórico-metodológicas y casos de estudio en la arqueología de la Patagonia. San Rafael: Museo de Historia Natural de San Rafael y Sociedad Argentina de Antropología. p 87-96.
García F, Moraga M, Vera S, Aspillaga E, Rothhammer F. 2004. Origen y micro-diferenciación de la población humana del archipiélago de Chiloé. Rev Chil Hist Nat 77:539-546. doi: 10.4067/S0716-078X2004000300012
García F, Moraga M, Vera S, Henríquez H, Llop E, Aspi-llaga E, Rothhammer F. 2006. mtDNA microevolution in southern Chile´s archipelagos. Am J Phys Anthropol 129:473-481. doi:10.1002/ajpa.20297
García A, Pauro M, Bravi C, Demarchi D. 2012. Phylogeo-graphy of mitochondrial haplogroup D1: an early spread of subhaplogoup D1j from Central Argentina. Am J Phys Anthropol 149:583-590. doi:10.1002/ajpa.22174
García-Bour J, Pérez-Pérez A, Álvarez S, Fernández E, López-Parra A, Arroyo-Pardo E, Turbón D. 2004. Early population differentiation in extinct aborigines from Tierra del Fuego-Patagonia: ancient mtDNA sequence and Y-chromosome STR characterization. Am J Phys Anthropol 123:361-370. doi:10.1002/ajpa.10337
García Guraieb S, Mariano C, Favier Dubois C. 2010. El buque Sur: un entierro primario múltiple de 2300 años en la costa del golfo San Matías, Río Negro, Argentina. Magallania 38:135-146. doi:10.4067/S0718-22442010000100008
García-Guraieb S, Goñi R, Tessone A. 2015. Paleodemogra-phy of late Holocene hunter-gatherers from Patagonia (Santa Cruz, Argentina): an approach using multiple ar-chaeological and bioarchaeological indicators. Quatern Int 356:147-158. doi:10.1016/j.quaint.2014.09.054
Gibbon V, Penny C, Strkalj G, Ruff P. 2009. Brief commu-nication: minimally invasive bone sampling method for DNA analysis. Am J Phys Anthropol 139:596-599. doi:10.1002/ajpa.21048
Ginther C, Corach D, Penacino GA, Rey JA, Carnese FR, Hutz MH, Anderson A, Just J, Salzano FM, King MC. 1993. Genetic variation among Mapuche indians from the Patagonia region of Argentina: mitochondrial DNA sequence variation and allele frequencies of several nuclear genes. En: Pena SDJ, Chakraborty R, Epplen JT, Jeffreys AJ, editores. DNA fringerprinting: state of the science. Basel: Birkhauser Verlag. p 211-219.
Gómez Otero J, Stern C. 2005. Circulación, intercambio y uso de obsidianas en la costa de la provincia del Chubut (Patagonia argentina) durante el Holoceno tardío. Intersecciones antropol 6:93-108.
González-José R, Dahinten S, Hernández M. 2001. The sett-lement of Patagonia: a matrix correlations study. Hum Biol 2:233-248. doi:10.1353/hub.2001.0019
González-José R, García-Moro C, Dahinten S, Hernández M. 2002. Origin of Fueguian-Patagonians: a using R matrix and matrix permutation methods. Am J Hum Biol 14:308-320. doi:10.1002/ajhb.10033
Hajduk A, 1981-1982. Cementerio "Rebolledo arriba", departamento de Aluminé, provincia del Neuquén. Relaciones 2:125-145.
Hajduk A, Albornoz A. 1999. El sitio Valle Encantado I. Su vinculación con otros sitios: un esbozo de la problemática local diversa del Nahuel Huapi. En: Soplando en el viento.. Actas de las Terceras Jornadas de Arqueología de la Patagonia. Neuquén-Buenos Aires: INAPL-Universidad del Comahue. p 371-391.
Hajduk A, Albornoz A, Lezcano M. 2007. Nuevos pasos en pos de los primeros barilochenses. Arqueología del Parque Nacional Nahuel Huapi. En: Vázquez C, Palacios O, editores. Patrimonio cultural: la gestión, el arte, la arqueología y las ciencias exactas aplicadas. Bariloche: Comisión Nacional de Energía Atómica. p 175-194.
Hubbe M, Okumura M, Bernardo D, Neves W. 2014. Cra-nial morphological diversity of early, middle and late Holocene brazilian groups: implications for human dis-persion un Brazil. Am J Phys Anthropol 155:546-558. doi:10.1002/ajpa.22607
Kemp B, Glenn-Smith D. 2005. Use of bleach to elimi-nate contaminating DNA from the surface of bones and teeth. Forensic Sci Int 154:53-61. doi:10.1016/j. forsciint.2004.11.017
Klöss-Brandstätter A, Sanger T, Lutz-Bonengel S, Parson W, Beraud-Colomb E, Wen B. Kong Q, Bravi C, Ban-delt H. 2005. Phantom mutation hotspots in human mitochondrial DNA. Electrophoresis 26:3414-3429. doi:10.1002/elps.200500307
Kuch M, Grocke D, Knyf M, Gilbert M, Younghusband B, Young T, Marshall I, Willerslev E, Stoneking, M, Poi-nar, H. 2007. A preliminary analysis of the DNA and diet of the extinct beothuk: a systematic approach to an-cient human DNA. Am J Phys Anthropol 132:594-604. doi:10.1002/ajpa.20536
Lahr M. 1995. Patterns of modern human diversification: implications for Amerindian origin. Am J Phys Anthropol 38:163-198. doi: 10.1002/ajpa.1330380609
Lalueza C, Pérez-Pérez A, Prats E, Cornudella L, Turbón D. 1997. Lack of founding amerindian mitochondrial DNA in extinct aborigines from Tierra del Fuego-Patagonia. Hum Mol Genet 6:41-46. doi: 10.1093/hmg/6.1.41
Librado P, Rozas J. 2009. DnasP v5: a software for com-prehensive analysis of DNA polymorphism data. Bio-informatics 25:1451-1452. doi:10.1093/bioinformatics/ btp187
Manríquez G, Moraga M, Santoro C, Aspillaga E, Arriaza B, Rothhammer, F, 2011. Morphometric and ADNmt analyses of archaic skeletal remains from southwestern South America. Chúngara 43:283-292. doi:10.4067/ S0717-73562011000200009
Martínez G, Zangrando F, Prates L. 2009. Isotopic ecology and human palaodiets in the lower basin for the Colorado river basin, Buenos Aires province, Argentina. Int J Osteoarchaeol 19:281-296. doi:10.1002/oa.1057
Martínez G, Flensborg G, Bayala, P. 2013. Chronology and human settlement in Northeastern Patagonia (Argentina): patterns of site destruction, intensity of archaeo-logical signal and population dynamics. Quatern Int 301:123-134. doi: 10.1016/j.quaint.2013.03.027
Matisoo-Smith E, Horsburgh A. 2012. DNA for archaeolo-gist. Walnut Creek: Left Coast Press.
McCulloch R, Bentley M, Purves R, Hulton N, Sugden D, Clapperton C. 2000. Climatic inferences from glacial and palaeoecological evidence at the last glacial termina-tion, Southern South America. J Quaternary Sci 15:409-417. doi:10.1002/1099-1417(200005)15:4<409::AID-JQS539>3.0.CO;2-#
Miotti L. 2003. Patagonia: a paradox for building images of the frst Americans during the Pleistocene/Holocene transition. Quatern Int 109-110:147-173. doi: 10.1016/ S1040-6182(02)00210-0
Miotti L. 2006. La fachada atlántica como puerta de ingreso alternativa de la colonización humana de América del Sur durante la transición Pleistoceno/Holoceno. En: Ji-menez JC editor. 2do Simposio Internacional El Hombre Temprano en América. México: CONACULTA-INAH. p 155-188.
Miotti L, Salemme M. 2004. Poblamiento, movilidad y territorios entre las sociedades cazadoras-recolectoras de Patagonia. Complutum 15:177-206.
Moraga M, Rocco P, Miquel J, Nervi F, Llop E, Chakra-borty R, Rothhammer F, Carvallo P. 2000. Mitochon-drial DNA polymorphisms in Chilean aboriginal popu-lations: implications for the peopling of the Southern Cone of the continent. Am J Phys Anthropol 113:19-29. doi:10.1002/1096-8644(200009)113:1<19::AID-AJPA3>3.0.CO;2-X
Moraga M, de Saint Pierre M, Torres F, Ríos J. 2010. Vínculos de parentesco por vía materna entre los últimos descendientes de la etnia Kawesqar y algunos entierros en los canales patagónicos: evidencia desde el estudio de linajes mitocondriales. Magallania 38:103-114. doi:10.4067/S0718-22442010000200006
Morello F, Borrero LA, Massone M, Stern C, García-Herbst A, McCulloch R, Arroyo-Kalin M, Calás E, Torres J, Prieto A, Martínez I, Bahamonde G, Cárdena, P. 2012. Hunters-gatherers, biogeographic barrier and develop-ment of human settlement in Tierra del Fuego. Antiqui-ty 86:71-87. doi:10.1017/S0003598X00062463
Neves W, Prous A, González-José R, Kipsnir R, Powell J. 2003. Early human skeletal remains from Santa Do Riacho, Brazil: implications for the settlement of the new world. J Hum Evol 45:19-42. doi:10.1016/S0047-2484(03)00081-2
Oksanen J, Kindt R, Legendre P, O'Hara B, Simpson G, So-lymos P, Henry M, Stevens H. Wagner H. 2009. Vegan: community ecology package. R package version 1.15-4. http://CRAN.R-project.org/package=vegan
Orquera L, Piana E. 1999. Arqueología de la región del canal de Beagle. Publicaciones de la Sociedad Argentina de Antropología. Buenos Aires: Sociedad Argentina de Antropología.
Orquera L, Gómez Otero J. 2007. Los cazadores -recolectores de las costas de Pampa, Patagonia y Tierra del Fuego. Relaciones 32:75-99.
Pérez A, Reyes V. 2009. Técnica de impronta de hojas, algunas refexiones acerca de su novedoso registro en la vertiente oriental de la cordillera de los Andes. Magallania 37:113-132. doi: 10.4067/S0718-22442009000100009
Pérez I, Bernal V, González P. 2007. Morphological diffe-rentiation of aboriginal human populations from Tierra del Fuego (Patagonia): implications for South Ame-rican peopling. Am J Phys Anthropol 133: 067-1079. doi:10.1002/ajpa.20633
Ponce J, Rabassa J, Coronato A, Borromei A. 2011. Palaeo-geographical evolution of the Atlantic coast of Pampa and Patagonia from the Last Glacial Maximum to the Middle Holocene. Biol J Linn Soc 103:363-379. doi:10.1111/j.1095-8312.2011.01653.x
Postillone MB, Dejean, CB. 2013. Análisis preliminar de estudios de ADN antiguo e muestras del Museo Regional de Rada Tilly, Chubut. Actas XVIII Congreso Nacional de Arqueología Argentina, La Rioja, Argentina.
Postillone MB, Dejean CB, Serna A, Prates L. 2015. Ances-tría genética de dos individuos inhumados en tiempos de la Campaña del Desierto (Sitio Chimpay, Río Negro). Actas XII Jornadas Nacionales de Antropología Biológica, Corrientes, Argentina.
Postillone MB, Flensborg G, Dejean CB, Martinez G. 2016. Linajes maternos de los individuos del sitio arqueológico Paso Alsina I (Patagones, Buenos Aires).XIX Congreso Nacional de Arqueología Argentina, Tucumán, Argentina.
Prates L, Politis G, Steele J. 2013. Radiocarbon chronology of the early human occupation of Argentina. Quatern Int 301:104-122. doi:10.1016/j.quaint.2013.03.011
Pucciarelli H. 2009. Explicando el Paleoamericano y su probable extinción a través de inferencias comparativas y experimentales. Runa XXX:9-27.
QGis DT. 2011. Quantum Gis geographic information sys-tem. En: Open Source Geospatial Foundation Project.
R Core Team 2014. R: A language and environment for sta-tistical computing. R Foundation for Statistical Compu-ting, Vienna, Austria. URL http://www.R-project.org/.
Rabassa J, Coronato A, Gordillos S, Candel M, Martínez M. 2009. Paleoambientes litorales durante el inicio de la transgresión marina Holocena en Bahía La Pataia, canal de Beagle, Parque Nacional Tierra del Fuego. Rev Asoc Geol Arg 65:648-659.
Russo M, Gheggi M, Avena S, Dejean C, Cremonte M. 2016. Linajes mitocondriales en muestras de esquina de Huajra (Jujuy, Argentina). Aportes al estudio de la ocupación incaica en la región y la procedencia de sus habitantes. Revista Argentina de Antropología Biológica, en este volumen.
Sardi M, Ramirez-Rozzi F, González-José R, Pucciarelli H. 2005. South Amerindian craneofacial morphology: diversity and implications for Amerindian evolution. Am J Phys Anthropol 128:747-756. doi:10.1002/ajpa.20235
Silveira M, López L, Pastorino G. 2010. Movilidad, redes de intercambio y circulación de bienes en el Sudoeste de Neuquén (Norpatagonia, Argentina). Los moluscos marinos del Lago Traful. Intersecciones antropol 11:227-236.
Soares P, Ermini L, Thomson N, Mormina M, Rito T, Röhl A, Salas A, Oppenheimer S, Macaulay V, Richards M. 2009. Correcting for purifying selection: an improved human mitochondrial molecular clock. Am J Hum Ge-net 84:740-759. doi: 10.1016/j.ajhg.2009.05.001
Stern C, Pereda I, Aguerre A. 2012. Multiple primary and secondary source for chamically similar obsidians from the area of Portada Convuco, west-central Neuquen, Argentina. Archaeometry 54:442-453. doi:10.1111/j.1475-4754.2011.00642.x
Tamura K, Nei M. 1993. Estimation of the number of nu-cleotide substitutions in the control region of mitochon-drial DNA in humans and chimpanzees. Mol Biol Evol 10:512-526.
Tamura K. Peterson D. Peterson N. Stecher G. Nei M, Ku-mar S. 2011. MEGA5: Molecular evolutionary genetics analysis using Maximum Likelihood, Evolutionary Distance, and Maximum Parsimony methods. Mol Biol Evol 28: 2731-2739. doi:10.1093/molbev/msr121
Tessone A, Zangrando F, Barrientos G, Valencio S, Panarello H, Goñi R. 2005. Isótopos estables del carbono en Patagonia meridional: datos de la cuenca del lago Salitroso (Provincia de Santa Cruz, República Argentina). Magallania 33:21-28. doi:10.4067/S0718-22442005000200003
Van Oven M, Kayser M. 2008. Updated comprehensive phylogenetic tree of global human mitochondrial DNA. Hum Mutat 30:e386-e394. doi:10.1002/humu.20921
Yang D, Watt, K. 2005. Contamination controls when preparing archaeological remains for ancient DNA analysis. J Archaeol Sci 32:331-336. doi: 10.1016/j. jas.2004.09.008
Zubimendi M. Ambrustolo P. Zilio L, Castro A. 2015. Con-tinuity and discontinuity in the human use of the Nor-th Coast of Santa Cruz (Patagonia Argentina) through its radiocarbon record. Quatern Int 356:127-146. doi:10.1016/j.quaint.2014.09.035














