Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO  uBio
uBio
Share
Revista argentina de antropología biológica
On-line version ISSN 1514-7991
Rev Arg Antrop Biol vol.21 no.1 La Plata Jan. 2019
http://dx.doi.org/10.17139/raab.2019.0021.01.03
doi:10.17139/raab.2019.0021.01.03
TRABAJOS ORIGINALES
Análisis de la estructura genética del Valle de Traslasierra a partir de inserciones Alu
Genetic structure in traslasierra Valley. Analysis of Alu insertions
Gabriela A. Díaz-Rousseau1, Angelina García1 y Darío A. Demarchi1 *
1Instituto de Antropología de Córdoba (IDACOR). CONICET/Universidad Nacional de Córdoba. Córdoba. Argentina
Financiamiento: SECyT Universidad Nacional de Córdoba 2016, FONCyT PICT Nº 711 2012.
* Correspondencia a: Darío Demarchi. Museo de Antropología UNC. Hipólito Yrigoyen 174. 5000 Córdoba. Argentina. E-mail: demarchi@ffyh.unc.edu.ar
Recibido: 3 Diciembre 2017;
Aceptado: 23 Marzo 2018
RESUMEN
La diversidad genética de las poblaciones humanas del centro de Argentina es resultado de un proceso de movimientos migratorios y mestizaje, consecuencia de cinco siglos de contacto entre los nativos americanos y los migrantes principalmente de Europa, Medio Oriente y África. Estos eventos histórico-demográficos no fueron homogéneos, sino que difirieron según condiciones ambientales, económicas y políticas de cada subregión. En este trabajo estudiamos la estructura genética del Valle de Traslasierra a partir del análisis de 8 inserciones Alu (PV92, FXIIIB, APO, TPA25, ACE, A25, B65 y D1). El objetivo es contribuir al conocimiento de su historia evolutiva reciente, principalmente en lo referente a patrones migratorios y a la posible existencia de consanguinidad, empleando metodologías propias de la Antropología Genética. La muestra investigada incluyó a 97 individuos de varias localidades, mayormente rurales, y fue subdividida en tres submuestras, de acuerdo con la orientación norte-sur del valle. Los resultados revelaron la ausencia de diferenciación entre submuestras y un déficit significativo de heterocigotas en 7 de los 8 marcadores utilizados. Los índices de endogamia local (FIS) y total (FIT), que miden el aumento de homocigosis debido a apareamientos consanguíneos, muestran valores positivos y estadísticamente significativos, tanto en la población total como en cada una de las subpoblaciones. Estos resultados confirman la existencia de consanguinidad estimada previamente de forma indirecta a partir de isonimia, distancias maritales y otros estimadores biodemográficos. Finalmente, si bien cinco de las inserciones Alu estudiadas presentaron diferencias altamente significativas entre grupos continentales, la estimación de ancestría genética no refleja la contribución de las poblaciones parentales al acervo genético de la población según lo esperado a partir de evidencias obtenidas de marcadores uniparentales y autosomales o de la historia migratoria reciente. Rev Arg Antrop Biol 21(1), 2019. doi:10.17139/raab.2019.0021.01.03
PALABRAS CLAVE: consanguinidad; homogeneidad genética; estadísticos F
ABSTRACT
The genetic diversity of human populations in central Argentina is the result of a process of migratory movements and admixture, after five centuries of contact between Native Americans and migrants, mainly from Europe, the Middle East, and Africa. These historical and demographic events were not homogeneous; rather, they showed differences related to the environmental, economic, and political conditions of each sub-region. In this work we studied the genetic structure of Traslasierra Valley from the analysis of 8 Alu insertions (PV92, FXIIIB, APO, TPA25, ACE, A25, B65, and D1). Our objective is to contribute to the knowledge of its recent evolutionary history, mainly in relation to migratory patterns and the possible existence of consanguinity, using methodologies of anthropological genetics research. The sample examined included 97 individuals from several localities, mostly rural, and was divided into three subsamples, according to the north-south orientation of the valley. The results revealed the lack of differentiation between subsamples and a significant deficit of heterozygotes in 7 of the 8 markers used Local (FIS) and total (FIT) inbreeding coefficients, which measure the increase of homozygosis due to consanguineous mating, show positive and statistically significant values, both in the total population and in each of the subpopulations. The results confirm the existence of inbreeding, previously estimated indirectly by isonymy, marital distance, and other biodemographic estimators. Although five of the Alu insertions showed highly significant differences between continental groups, the estimation of genetic ancestry does not reflect the contribution of parental populations to the population's gene pool, as expected from evidence obtained from uniparental and autosomal markers, or from recent migration history. Rev Arg Antrop Biol 21(1), 2019. doi:10.17139/raab.2019.0021.01.03
KEYWORDS: inbreeding; genetic homogeneity; F statistics
La diversidad genética de las poblaciones humanas del centro de Argentina es resultado de movimientos migratorios y mestizaje, consecuencia de cinco siglos de contacto entre los nativos americanos y los migrantes principalmente de Europa, Medio Oriente y África, aunque también de los descendientes de países limítrofes (Albarracín, 2005). Estos eventos histórico-demográficos no fueron homogéneos en toda la región, sino que difirieron de acuerdo con las condiciones ambientales, económicas y políticas (García y Demarchi, 2009). De particular interés resulta la región de Traslasierra, con algunas poblaciones geográficamente semiaisladas donde se ha informado la frecuencia inusualmente alta de la enfermedad genética recesiva de Sandhoff (OMIM# #268800) debido a la alta consanguinidad observada (Dodelson de Kremer et al., 1987), condición que también ha sido inferida a través de estudios biodemográficos. Por ejemplo, en el Departamento Pocho (al NO de la región) se observó que a lo largo de los siglos XIX y XX existieron fuertes corrientes emigratorias y escaso aporte de migrantes desde el exterior, lo que condujo a una drástica reducción del tamaño de la población y a contactos reducidos con poblaciones de áreas vecinas (Colantonio, 1996). Estudios de isonimia y de otros indicadores biodemográficos y biológicos, mostraron que en los departamentos Pocho y Minas, al norte, existen poblaciones que pueden ser consideradas como semiaislados poblacionales (Colantonio, López y Demarchi, 1998; Demarchi y Colantonio, 2000; López y Colantonio, 1999). En consecuencia, resulta de suma importancia verificar, a partir del análisis de marcadores genéticos de herencia biparental, la existencia de consanguinidad en estas poblaciones.
La Antropología genética investiga la diversidad genética humana en el presente y la historia evolutiva que la ha generado. Para ello se basa en el estudio de rasgos polimórficos que caracterizan a las poblaciones de acuerdo con su presencia, ausencia o diferencias en la frecuencia (Relethford y Harding, 2001). A través del análisis de marcadores de herencia biparental, ubicados en cromosomas autosómicos, o bien de aquellos transmitidos por vía materna (ADN mitocondrial) o paterna (cromosoma Y), es posible reconstruir la historia migratoria de una región y evaluar la estructura genética de la población (Relethford y Harding, 2001). Las inserciones Alu son elementos genéticos cortos y móviles, típicamente de 300 nucleótidos de longitud, dispersos por todo el genoma, que se replican y se trasladan a diferentes cromosomas y representan más del 10% del genoma humano (Lander et al., 2001). Los polimorfismos se identifican observando la presencia o ausencia de una inserción en loci específicos, siendo la ausencia de la inserción el estado ancestral (Relethford, 2003). La probabilidad de que dos elementos Alu independientes se inserten al azar en la misma posición genómica es prácticamente nula, por lo que las inserciones Alu compartidas por diferentes individuos deberían ser idénticas por descendencia (Batzer et al., 1994). Una de las principales características de las inserciones Alu es su estabilidad evolutiva, debido a la escasa influencia de la mutación y de la selección sobre estos polimorfismos (García-Obregón, Alfonso-Sánchez, Pérez-Miranda, de Pancorbo y Peña, 2007). Todas las propiedades antes mencionadas (estado ancestral conocido, identidad por descendencia y estabilidad evolutiva), hacen que estos marcadores sean particularmente eficientes y confiables para estudios de genética de poblaciones asociados a la reconstrucción de la historia evolutiva humana (Batzer y Deininger, 2002; Batzer et al., 1994; Gómez-Pérez et al., 2010; Herrera, Rojas y Terreros, 2007; Novick et al., 1998). Algunos autores sostienen que aun un número limitado de inserciones Alu puede servir para obtener una buena discriminación entre poblaciones de diferentes continentes por lo que constituyen marcadores eficientes para estudios de mestizaje (Gómez-Pérez et al., 2007).
En este trabajo investigamos la variación genética del Valle de Traslasierra (provincias de Córdoba y San Luis) a partir del análisis de 8 inserciones Alu en una muestra poblacional conformada por diversas localidades de esa región. Los objetivos específicos del presente trabajo fueron: (i) caracterizar genéticamente poblaciones de Traslasierra mediante la tipificación y el análisis de polimorfismos de inserciones Alu; (ii) confirmar la existencia de endogamia y verificar sus posibles efectos en la estructura de la población; (iii) analizar los patrones geográficos de variación genética regional; (iv) investigar las afinidades genéticas entre la población estudiada y otros grupos humanos de América, Europa y África, a partir de datos tomados de la literatura.
MATERIAL Y MÉTODOS
La muestra
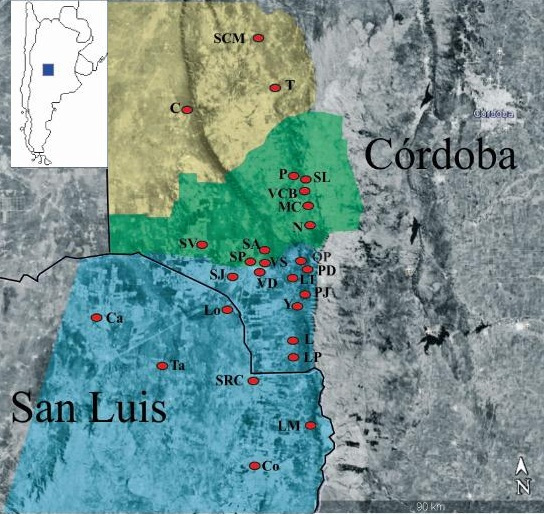
Se trabajó con un total muestral de 97 individuos. De ese total, 51 muestras se obtuvieron durante 2015 en los hospitales regionales de Villa Dolores y Mina Clavero donde concurren pacientes desde diferentes sitios de la región. Las 46 muestras restantes fueron tomadas previamente por nuestro equipo de investigación en diferentes localidades del valle de traslasierra entre los años 2005 y 2009. Previo a la toma de muestras biológicas, consistente en hisopados bucales, se explicó a los participantes los objetivos del proyecto, el significado de los resultados y se les hizo firmar un consentimiento informado. Para cada uno de los donantes se completó además una encuesta que incluyó preguntas relacionadas con la procedencia de sus ancestros (maternos y paternos) hasta tres generaciones atrás. Estas encuestas mostraron que la mayoría de los participantes procedían de parajes rurales distribuidos a lo largo de toda la región de estudio. Por ese motivo, y dado el bajo tamaño muestral de algunas de las localidades incluídas, se reagrupó la muestra total en 3 submuestras. Se siguió un criterio geográfico tomando en cuenta la orientación natural norte-sur del valle de Traslasierra y la división política departamental. La submuestra Norte incluyó individuos nacidos en los departamentos Pocho y Minas (N= 35); la Centro a los individuos procedentes del departamento San Alberto (N=27) y la Sur a aquellos nacidos en el departamento San Javier y en el extremo noreste de San Luis (N= 35). En la Figura 1 se detallan los sitios de muestreo incluidos en cada una de las regiones consideradas.
Fig. 1. Mapa de la región y sitios de procedencia de las muestras. Norte (en amarillo): SCM (San Carlos Minas); C (Chancaní); T (Taninga); Centro (en verde) P (Panaholma), SL (San Lorenzo), VCB (Villa Cura Brochero), MC (Mina Clavero), N (Nono), SV (San Vicente), SA (Sauce Arriba), SP (San Pedro), VS (Villa Sarmiento); Sur (en azul) SRC (Santa Rosa del Conlara), SJ (San José), VD (Villa Dolores), QP (Quebrada de los Pozos), PD (Paraje las Dalias), LT (Las Tapias), PJ (Paraje las Jarillas), Y (Yacanto), L (Luyaba), LP (La Paz), Ca (Candelaria), Lo (Lomitas), Ta (Talita); LM (Las Lomitas), Co (Concarán).
Análisis de laboratorio
Para la extracción de ADN se utilizó el kit comercial Accuprep Genomic DNA Kit (GenBiotech), basado en la afinidad del ADN por las partículas de sílice. Se analizaron 8 inserciones Alu (PV92, FXIIIB, APO, TPA25, ACE, A25, B65 y D1), utilizadas previamente en el análisis de diversas poblaciones de todos los continentes (García-Obregón et al., 2006; Romualdi et al., 2002; Stoneking et al., 1997;) y que se caracterizan por presentar frecuencias marcadamente diferentes entre poblaciones continentales. La tipificación se realizó a partir de la técnica de PCR (Polymerase Chain Reaction) convencional en un volumen final de 10 ul. Las condiciones específicas de amplificación y los cebadores para cada inserción Alu, utilizadas en el presente trabajo fueron las mismas detalladas en los trabajos de García-Obregón et al. (2006, 2007). Todos los productos de PCR fueron analizados por electroforesis en geles de agarosa al 2% y las bandas de ADN se visualizaron por tinción con GelStar (Lonza) bajo luz UV.
Análisis de los datos
Las frecuencias genotípicas y alélicas se calcularon por conteo directo. La existencia de posibles desviaciones del equilibrio Hardy-Weinberg se evaluó mediante el test Exacto según el procedimiento de Guo y Thompson (1992). La estructura genética de la población fue investigada mediante el análisis molecular de la varianza (AMOVA) y de los estadísticos F derivados del mismo, según el procedimiento de Weir y Cockerham (1984). Se empleó, asimismo, el test Exacto de diferenciación muestral (Raymond y Rousset, 1995) basado en las frecuencias genotípicas, para poner a prueba la existencia de diferencias significativas entre submuestras. Estos análisis fueron realizados con el programa Arlequín 3.5 (Excoffier y Lischer, 2010). La similitud relativa entre poblaciones se investigó por medio de las distancias genéticas FST (Reynolds, Weir y Cockerman, 1983), representadas a través de un dendrograma UPGMA, y del Análisis de Componentes Principales, realizado a partir de una matriz de correlación. Para ambos procedimientos se utilizó el programa PAST 3.0 (Hammer, Harper y Ryan, 2001).
La contribución relativa de las poblaciones parentales continentales al acervo genético actual de la población de Traslasierra se estimó a través del método de identidad genética utilizando el programa Admix (Chakraborty, 1985), tomando las frecuencias promedio de las poblaciones de Europa, África y América del Sur listadas en la Tabla 1.
TABLA 1. Distribución de inserciones Alu en 13 muestras poblacionales
RESULTADOS
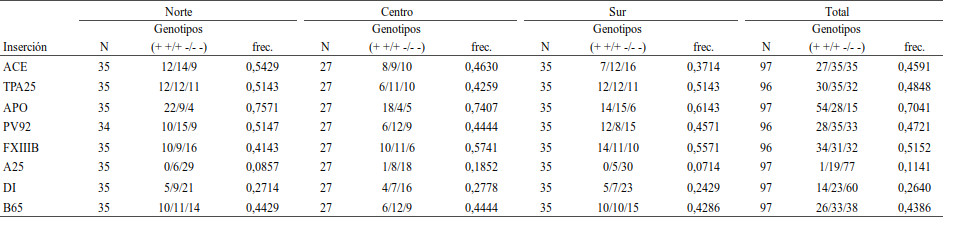
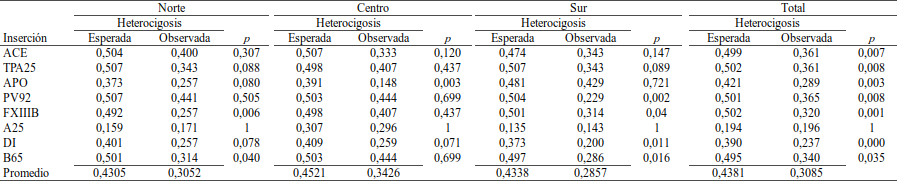
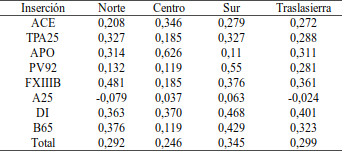
En la Tabla 2 se presentan las frecuencias genotípicas y alélicas de las 8 inserciones Alu incluidas en el análisis para las 3 subregiones y la población total. Todos los marcadores mostraron ser polimórficos en las tres submuestras. Las frecuencias mínimas de inserciones se observaron para A25 (variando desde 0,071 en la submuestra Sur y 0,185 en la Centro) y máximas para APO (desde 0,757 en Norte y 0,614 en Sur). El resto de los marcadores mostraron frecuencias intermedias. En la Tabla 3 se presentan valores de heterocigosis promedio observada y esperada insesgada (Nei, 1978), y valores de significación para el test de Hardy-Weinberg. En todos los sistemas, salvo A25, la frecuencia de heterocigotas observados es sensiblemente inferior a la esperada en una población en equilibrio. En promedio, la heterocigosis observada es sensiblemente menor en la submuestra Sur de Traslasierra, alcanzado los valores más altos en la subregión Centro e intermedios en la Norte. Se encontraron desviaciones significativas en FXIIIB, B65, PV92 y D1en la submuestra Sur. En la submuestra Norte se encontraron desviaciones significativas en FXIIIB y B65 y en la submuestra Centro sólo APO presenta una disminución de heterocigosis que llega a ser estadísticamente significativa. Cuando se analiza la población total, todos los marcadores (excepto A25) muestran desviaciones significativas.
TABLA 2. Frecuencias genotípicas y alélicas de 8 inserciones Alu en la región de Traslasierra
+ corresponde a inserción; - corresponde a no inserción; frec.:frecuencias alélicas.
TABLA 3. Heterocigosis observada y esperada (insesgada, Nei 1978). Valores de significación para el test de Hardy y Weinberg
Significación: p<0,05
El test Exacto revela la ausencia de diferenciación entre las submuestras (p=0,792 ± 0,031). El mismo resultado arrojó el AMOVA (FST=-0,003, p=0,989), confirmando que la distribución de las frecuencias alélicas y genotípicas en la región es homogénea. Dado este resultado, en los análisis interpoblacionales se consideró a Traslasierra como una única población.
De manera complementaria, el AMOVA también proporcionó otros estadísticos F que son de interés para los objetivos de este trabajo. El índice de endogamia local (FIS), que mide el aumento de homocigosis debido a apareamientos consanguíneos en la subpoblación, revela valores positivos y altamente significativos tanto en la población total como en cada una de las subpoblaciones (Tabla 4). Todos son estadísticamente significativos excepto en el caso de A25. El coeficiente de endogamia total de la región (FIT=0,295, p=0,000) también muestra un valor altamente significativo.
TABLA 4. Valores de FIS para cada subregión y total (Traslasierra)
§FSC:variación entre poblaciones dentro de grupos; FCT:entre grupos, FST:interpoblacional total; *p<0.01
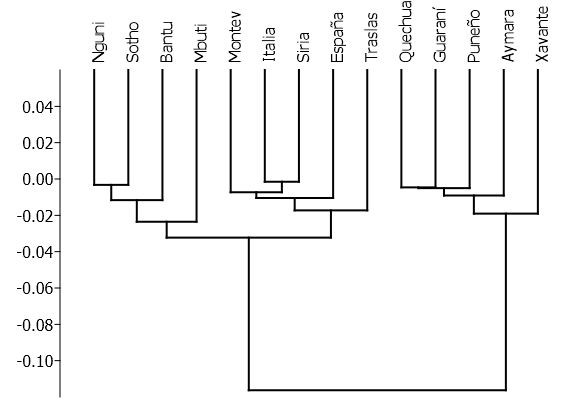
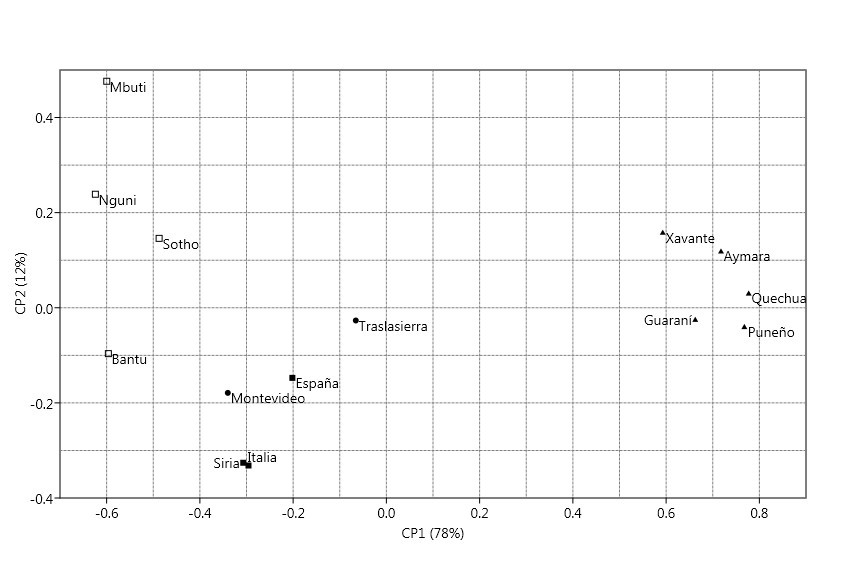
En la Figura 2 se presenta el dendrograma (UPGMA), realizado a partir de las distancias FST, mostrando las similitudes relativas entre las poblaciones estudiadas a partir de las frecuencias de 8 inserciones Alu. El coeficiente de correlación cofenética (r=0,895) indica un buen ajuste entre la matriz de distancias y su representación gráfica. Se observan tres agrupamientos bien diferenciados, el primero, integrado por los pueblos nativos americanos, se separa netamente del resto. Posteriormente se separan las cuatro poblaciones subsaharianas por un lado y por el otro los dos representantes de Europa, Siria (muy cerca de Italia) y las dos muestras de poblaciones neoamericanas, Montevideo y Traslasierra. Al observar la matriz de distancias, se advierte que España y Montevideo son las poblaciones que presentan las menores distancias genéticas con Traslasierra (0,015 en ambos casos). El análisis de componentes principales de la Figura 3 muestra un patrón similar al observado en el dendrograma, con tres grupos bien definidos que se separan principalmente a lo largo del primer componente principal, que acumula el 78% de la variación total. Sin embargo, aquí se advierte que la muestra de Traslasierra ocupa una posición más o menos equidistante de los grupos continentales parentales, cercana al centro del gráfico.
Fig. 2. Árbol UPGMA a partir de distancias FST (Reynolds et al. 1983). Coeficiente cofenético: r =0,895
Fig. 3. Análisis de Componentes Principales
Análisis de ancestría genética
La contribución relativa de las poblaciones parentales continentales (americana, europea y africana) al acervo genético actual de la población de Traslasierra se estimó a través de dos grupos de variables (Tabla 6). En primer lugar, se incluyeron las 8 inserciones estudiadas y, en segundo término, solamente las 5 que presentaron diferencias altamente significativas entre grupos continentales (FCT). Los resultados muestran, en el primer análisis, una contribución casi similar de las tres poblaciones parentales, siendo apenas levemente superior el componente europeo. En el segundo análisis (a partir de 5 inserciones) se observa una contribución algo mayor del componente europeo y americano, en detrimento del componente africano que, de todas maneras, es aún muy superior al que podría inferirse a partir de datos históricos y demográficos y de los resultados previos obtenidos usando marcadores moleculares de herencia uniparental y AIMS autosómicos (García y Demarchi, 2009; García et al., 2015; Pauro, García, Bravi y Demarchi, 2010).
TABLA 6. Análisis de ancestría genética

DISCUSIÁN
En el valle de Traslasierra, particularmente en el norte, correspondiente a los departamentos Pocho y Minas, se ha observado a partir de estudios biodemográficos la existencia de pequeños demes, caracterizados por baja inmigración y altos índices de consanguinidad (Colantonio, 1996; Colantonio et al., 1998; López y Colantonio, 1999; Demarchi y Colantonio, 2000). Estudios epidemiológicos relacionados a la enfermedad de Sandhoff (Dodelson de Kremmer et al., 1987) sugieren que la alta carga genética de esa población, resultante de la consanguinidad sostenida durante varias generaciones, sería responsable de la incidencia, inusualmente alta, de esa enfermedad genética recesiva en toda esa región. En el presente trabajo abordamos el estudio del valle de Traslasierra, a partir del análisis de 8 inserciones Alu autosómicas, evolutivamente neutras, en busca de evidencias genético-poblacionales que nos permitiesen confirmar la existencia de consanguinidad, determinar posible subestructuración poblacional y contribuir al conocimiento acerca de su reciente historia evolutiva.
El primer resultado a destacar de esta investigación es la marcada deficiencia de heterocigotas en todos los marcadores utilizados (con excepción de A25) en las tres submuestras. La razón por la que ésta deficiencia alcanzó significación estadística obedece simplemente al bajo tamaño muestral. Por otra parte, cuando se analizó la población total, todos los marcadores (otra vez con excepción de A25) muestran desviaciones significativas del equilibrio Hardy-Weinberg, en parte debido al mayor tamaño muestral y en parte, probablemente, al efecto Wahlund. A saber, si dos o más subpoblaciones difieren en sus frecuencias alélicas y genotípicas y son analizadas como si fuera una sola, la heterocigosis total puede reducirse significativamente, aunque las subpoblaciones estén en equilibrio.
El coeficiente de endogamia total de una población subdividida (FIT) es resultado de dos componentes, el primero generado por el apareamiento entre individuos consanguíneos dentro de una subpoblación (FIS) y el segundo por el balance entre la deriva génica y el flujo génico entre subpoblaciones (FST) (Wright, 1951). En este estudio se observó que, a pesar de la reducción del tamaño efectivo de la población debido a deficiencia de heterocigotas, no existe estructura geográfica de la variación genética, es decir, no hay subpoblaciones diferenciadas por la deriva, sino que, el flujo génico conduciría a una distribución homogénea de la variación genética dentro de la región, tal como lo muestran los resultados del AMOVA y del Test Exacto. Es decir, el valor de endogamia total FIT, positivo y altamente significativo, se conforma exclusivamente por el componente FIS. En síntesis, los valores observados de FIS y FIT confirman la existencia de consanguinidad en la región, condición que había sido inferida previamente a partir de datos biodemográficos (Colantonio, 1996; Colantonio et al., 1998; Demarchi y Colantonio, 2000). Por otra parte, la región funcionaría como un único deme y no como varias subpoblaciones, al menos para estos marcadores y en base a una muestra poblacional, si se quiere, limitada. Sin embargo, es posible advertir que la subpoblación Centro (voluntarios provenientes principalmente de Mina Clavero y Villa Dolores) muestra valores de consanguinidad algo menores que la del Norte (Chancaní y Minas) y marcadamente inferiores a los del Sur (San Javier y NE de San Luis), seguramente por encontrarse menos aislada geográficamente y provenir de localidades de mayor tamaño censal. Además, la región central de Traslasierra es la que concentra un mayor flujo comercial y, particularmente, turístico, factor que condujo en las últimas décadas a la radicación de numerosos migrantes de otras provincias, según se desprende de las entrevistas realizadas durante el muestreo, donde los participantes manifestaron que llegaron a la región debido a la "belleza y tranquilidad de la zona". Esto explica, en gran medida, la diversidad genética relativamente mayor de esta subpoblación.
El análisis interpoblacional, realizado a partir de distancias genéticas, muestra tres agrupamientos bien diferenciados, que se corresponden con grupos poblacionales continentales. Las poblaciones neoamericanas, Montevideo y Traslasierra, se agrupan en el clúster europeo, de acuerdo con su historia migratoria reciente. Sin embargo, en el Análisis de Componentes Principales se observa que, si bien aparece más cercana a las muestras europeas, Traslasierra tiende a ocupar el centroide de la representación, casi equidistante de los grupos parentales, revelando el origen trihíbrido de su población. La diferenciación extrema de las poblaciones nativas americanas puede ser explicada por efectos fundadores seriales a lo largo de su historia evolutiva (Deshpande, Batzoglou, Feldman y Cavalli-Sforza, 2009). Siendo la deriva génica, consecuencia del pequeño tamaño efectivo de algunas poblaciones nativas, responsable de la rápida diferenciación de las poblaciones (Demarchi, 2009).
Si bien 5 inserciones Alu presentaron diferencias altamente significativas en la distribución de frecuencias a nivel continental, confirmando su utilidad en estudios de ancestría genética, el componente africano está evidentemente sobrerrepresentado, si se considera la información histórica y los estudios genéticos realizados en la región a partir de marcadores de herencia uniparental (García y Demarchi, 2006; 2009; Pauro et al., 2010) y biparental (García et al., 2015). La elección de las poblaciones parentales continentales podría conducir, al menos parcialmente, a un importante error en la estimación de la contribución relativa de las poblaciones ancestrales al acervo genético de la población. Sin embargo, el sesgo observado parece deberse principalmente al bajo número de marcadores utilizados y al error de estimación derivado del mismo. En el trabajo de Russo et al. (2016) se observó una fuerte dependencia de las estimaciones de ancestría respecto al número de marcadores utilizados. Esos autores informan que el incremento en el número de AIMs analizados aumenta la precisión de las estimaciones, puesto que se reduce el desvío estándar. Esto fue particularmente notable en el caso del componente subsahariano, que sólo resultó aceptable cuando la estimación de ancestría se realizó a partir de 30 marcadores. Los autores expresan que la posible explicación a este fenómeno se halla en el método empleado, el cual podría estar estimando el componente minoritario con un mayor error, especialmente cuando se emplea una menor cantidad de marcadores. En su análisis, el componente subsahariano se vio fuertemente sobredimensionado cuando la estimación se hizo con 10 AIMs. De manera contraria, en el trabajo de García et al. (2015) también realizado a partir de 10 AIMS, la estimación del componente subsahariano se ve sensiblemente subrepresentado, probablemente por la misma razón, es decir el mayor error en la estimación debido a la baja frecuencia.
Por otra parte, la intensidad con la que la deriva génica ha actuado sobre la población de la región de Traslasierra, tal como muestran las estimaciones de heterocigosis y los estadísticos F, podría haber modificado aleatoriamente las frecuencias alélicas lo suficiente como para que las contribuciones relativas de las poblaciones parentales no puedan ser estimadas con precisión. Es decir, la deriva podría ser también responsable de los anómalos resultados observados y no sólo el bajo número de marcadores utilizados.
En conclusión: (i) los estimadores de consanguinidad intra e interpoblacional muestran valores altamente significativos que confirman la existencia de consanguinidad, inferida previamente a partir de datos biodemográficos; (ii) a pesar de la reducción del tamaño efectivo de la población debido a deficiencia de heterocigotas, no existe estructura geográfica de la variación genética dentro del área de estudio; (iii) si bien cinco de las inserciones Alu estudiadas presentaron diferencias altamente significativas entre grupos continentales, confirmando su utilidad en estudios de mezcla genética, la estimación de ancestría genética realizada con un número bajo de marcadores no refleja de manera precisa la contribución esperada de las poblaciones parentales al acervo genético de la población.
AGRADECIMIENTOS
Se agradece a todas las personas que hicieron posible esta investigación a través de la donación de muestras biológicas y al Dr. Rodrigo Nores por la lectura crítica del manuscrito.
LITERATURA CITADA
Albarracín, J. (2005). Inmigración en la Argentina moderna: ¿un matrimonio en la salud y en la enfermedad con los europeos? En E. Domenech. (Comp.), Migraciones contemporáneas y diversidad cultural en la Argentina (pp.19-40). Córdoba, Argentina: Centro de Estudios avanzados UNC. [ Links ]
Battilana, J., Fagundes, N. J. R., Heller, A., Goldani, A. y Freitas, L. B., Tarazona-Santos, E., Bonatto, S. L. (2006). Alu insertion polymorphisms in Native Americans and related Asian populations. Annals of Human Biology, 33(2), 142-160. doi:10.1080/03014460500487347 [ Links ]
Batzer, M.A. y Deininger, P.L. (2002). Alu repeats and human genomic diversity. Nature Reviews Genetics 3, 370-379. doi:10.1038/nrg79 [ Links ]
Batzer, M.A., Stoneking, M., Alegria-Hartman. M., Bazan, H., Kass, D., Shaikh, T., Novick, G., Ioannou, P., Scheer, D., Herrera, R. y Deininger, P. (1994). African origin of human-specific polymorphic Alu insertions. Proceedings of the National Academy of Sciences of the United States of America, 91, 12288-12292. doi:10.1073/pnas.91.25.12288 [ Links ]
Chakraborty, R. (1985). Gene identity in racial hybrids and estimation of admixture rates. En Neel, J.V. y Ahuja, Y. (Ed.), Genetic microdifferentiation in man and other animals. (pp. 171-180). Delhi, Indian: Anthropological Association. [ Links ]
Colantonio, S.E. (1996). Análisis histórico-demográfico de un semiaislado en la provincia de Córdoba. Revista de Antropología Biológica, 1(1), 69-85. [ Links ]
Colantonio, S.E., López, M.A. y Demarchi, D.A. (1998). Isonimia y estructura poblacional en un semiaislado humano actual. Mendeliana,13(1), 27-35. [ Links ]
Demarchi, D.A. (2009). Microsatélites, diversidad y distancias génicas en poblaciones nativas sudamericanas. Revista Argentina de Antropología Biológica, 11(1), 73-88. [ Links ]
Demarchi, D.A. y Colantonio, S.E. (2000). Apellidos como alelos de un sistema polimórfico. Ensayo en una población aislada. En Tendencias actuales de Investigación en la Antropología Física Española (265-271). León, España: Universidad de León. [ Links ]
Deshpande, O., Batzoglou, S., Feldman, M. W. y Cavalli-Sforza, L. L. (2009). A serial founder effect model for human settlement out of Africa. Proceedings of the Royal Society Biological Sciences, 276, (1655), 291-300. doi: 10.1098/rspb.2008.0750. [ Links ] 10.1098/rspb.2008.0750
Dodelson de Kremer, R., Depetris de Boldini, C., Paschini de Capra, A., Pons de Veritier, P., Goldenhersch, H., Corbella, L., Sembaj, A., Martín, S., Kremer, I. y Mass, L. (1987). Estimation of heterozygote frequency of Sandhoff disease in a high risk Argentinian population. Predictive assignment of the genotype through statistical analysis. Medicina, 47(5), 455-63. [ Links ]
Excoffier, L. y Lischer, H. E. (2010). Arlequin suite v. 3.5: A new series of programs to perform population genetics analyses under Linux and Windows. Molecular Ecology Resources,10, 564-567. doi:10.1111/j.1755-0998.2010.02847.x [ Links ]
García, A. y Demarchi, D. A. (2006). Linajes parentales amerindios en poblaciones del norte de Córdoba. Revista Argentina de Antropología Biológica, 8(1), 57-72. [ Links ]
García, A. y Demarchi, D. A. (2009). Incidence and distribution of Native American mtDNA haplogroups in central Argentina. Human Biology, 81(1), 59-69. doi:10.3378/027.081.0105 [ Links ]
García, A., Dermarchi, D. A., Tovo-Rodrigues, L., Pauro, M., Callegari-Jacques, S., Salzano, F. M. y Hutz, M. H. (2015). High Interpopulation Homogeneity in Central Argentina as Assessed by Ancestry Informative Markers (AIMs). Genetics and Molecular Biology, 38(3), 324-331. doi:10.1590/S1415-475738320140260 [ Links ]
García-Obregón, S., Alfonso-Sánchez, M. A., Pérez-Miranda, A. M, Vidales, C., Arroyo, D. y Peña, J. A. (2006). Genetic position of Valencia (Spain) in the Mediterranean basin according to Alu insertions. American Journal Human of Biology, 18, 187-195. doi:10.1002/ajhb.20487 [ Links ]
García-Obregón, S., Alfonso-Sánchez, M. A., Pérez-Miranda, A. M., de Pancorbo, M. M. y Peña, J. A. (2007). Polymorphic Alu insertions and the genetic structure of Iberian Basques. Journal of Human Genetics, 52, 317-327. doi:10.1007/s10038-007-0114-9 [ Links ]
GayÁ -vidal, M., Dugoujon, J. M., Esteban, E., Athanasiadis, G., Rodríguez, A., Villena, M. y Moral, P. (2010). Autosomal and X chromosome Alu insertions in Bolivian Aymaras and Quechuas: two languages and one genetic pool. American Journal of Human Biology, 22(2), 154-162. [ Links ]
Gómez-Pérez, L. G., Alfonso-Sánchez, M. A., Pérez-Miranda, A. M., Pancorbo, M. M. y Peña, J. A. (2007). Utilidad de las inserciones Alu en los estudios de mestizaje. Antropo, 14, 28-36. [ Links ]
Gómez-Pérez, L., Alfonso-Sánchez, M. A., Pérez-Miranda, A. M., García-Obregón, S., Builes, J. J., Bravo, M. L., De Pancorbo, M. M. y Peña, J. A. (2010). Genetic admixture estimates by Alu elements in Afro-Colombian and Mestizo populations from Antioquia, Colombia. Annals of Human Biology, 37(4), 488-500. doi:10.3109/03014460903433810 [ Links ]
GómezâPérez, L., AlfonsoâSánchez, M. A., Dipierri, J. E., Alfaro, E., GarcíaâObregón, S., De Pancorbo, M. M. y Peña, J. A. (2011). Microevolutionary processes due to landscape features in the Province of Jujuy (Argentina). American Journal of Human Biology, 23(2), 177-184. doi:10.1002/ajhb.21098 [ Links ]
Guo, S.W. y Thompson, E. A. (1992). Performing the exact test of Hardy-Weinberg proportion for multiple alleles. Biometrics, 48, 361-372. doi:10.2307/253229 6 [ Links ]
Hammer, Á., Harper, D. A. T. y Ryan, P. D. (2001). PAST: Paleontological statistics software package for education and data analysis. Palaeontologia Electronica 4(1), 9. [ Links ]
Herrera, R. J., Rojas, D. P. y Terreros, M. C. (2007). Polymorphic Alu insertions among Mayan. Journal of Human Genetics, 52, 129-142. doi:10.1007/s10038-006-0089-y [ Links ]
Hidalgo, P.C., Mut, P., Ackermann, E., Figueiro, G. y Sans, M. (2014). Questioning the "melting pot": Analysis of Alu inserts in three population samples from Uruguay. Human Biology, 86(2), 83-92. doi:10.3378/027.086.0202 [ Links ]
Lander, E. S., Linton, L. M., Birren, B., Nusbaum, C., Zody, M. C., Baldwin, J....... y Chen, Y. J. (2001). International human genome sequencing consortium Initial sequencing and analysis of the human genome. Nature, 409, 860-921. doi:10.1038/35057062 [ Links ]
López, A.M. y Colantonio, S. E. (1999). Análisis demográfico de un semiaislado en la provincia de Córdoba, Argentina. Revista de Antropología Biológica, 2(1), 67-78. [ Links ]
Nasidze. I., Risch, G.M., Robichaux, M., Sherry, S.T., Batzer, M.A. y Stoneking, M. (2001). Alu insertion polymorphisms and the genetic structure of human populations from the Caucasus. European Journal of Human Genetics, 9(4), 267-272. [ Links ]
Nei, M. (1978). Estimation of average heterozygosity and genetic distance from a small number of individuals. Genetics, 89(3), 583-590. [ Links ]
Novick, G. E., Novick, C. C., Yunis, J., Yunis. E., Antunez de Mayolo, P., Scheer, W. D., Deininger, P. L., Stoneking, M., York, D. S., Batzer, M. A y Herrera, R. J. (1998). Polymorphic Alu insertions and the Asian origin of Native American populations. Human Biology, 70, 23-39. [ Links ]
Pauro, M., García, A., Bravi, C. y Demarchi, D. A. (2010). Distribución de haplogrupos mitocondriales alóctonos en poblaciones rurales de Córdoba y San Luis. Revista Argentina de Antropología Biológica, 12(1), 47-55. [ Links ]
Raymond, M. y Rousset, F. (1995). An exact test for population differentiation. Evolution, 49(6), 1280-1283. doi:10.1111/j.1558-5646.1995.tb04456.x [ Links ]
Relethford, J. H. (2003). The Human Species. An introduction to Biological Anthropology. New York, USA: McGraw & Hill. [ Links ]
Relethford, J. H. y Harding, R. M. (2001). Population genetics of modern human evolution. Encyclopedia of Life Sciences. New York, USA: John Wiley & Sons. doi:10.1038/npg.els.0001470 [ Links ]
Reynolds, J., Weir, B. S. y Cockerman, C. C. (1983). Estimation of the coancestry coefficient: bases for a short term genetic distance. Genetics, 105, 767-779. [ Links ]
Romualdi, C., Balding, D., Nasidze, I. S., Risch, G., Robichaux, M., Sherry, S. T., Stoneking, M., Batzer, M. A. y Barbujani, G. (2002). Patterns of human diversity, within and among continents, inferred from biallelic DNA polymorphisms. Genome Research, 12, 602-612. doi:10.1101/gr.214902 [ Links ]
Russo, M. G., Di Fabio Rocca, F., Doldán, P., Cardozo, D. G., Dejean, C., Seldes, V. y Avena, S. (2016). Evaluación del número mínimo de marcadores para estimar ancestría individual en una muestra de la población argentina. Revista del Museo de Antropología, 9(1), 49-56. doi:10.31048/1852.4826.v9.n1.12579 [ Links ]
Santovito, A., Selvaggi, A., Cervella, P., Castellano, S., Bigatti, M. P., Sella, G. y Delpero, M. (2007). Polymorphic Alu insertions in five NorthâWest Italian populations. American Journal of Human Biology, 19, 589-592. doi:10.1002/ajhb.20605 [ Links ]
Stoneking, M., Fontius, J. J., Clifford, S. L., Soodyall, H., Arcot, S. S., Saha, N., Jenkins, T., Tahir, M. A., Deininger, P. L. y Batzer, M. A. (1997). Alu insertion polymorphisms and human evolution: Evidence for a larger population size in Africa. Genome Research, 7, 1061-1071. doi:10.1101/gr.7.11.1061 [ Links ]
Weir, B. S. y Cockerham, C. C. (1984). Estimating F-Statistics for the Analysis of Population Structure. Evolution 38(6), 1358-1370. doi:10.2307/2408641 [ Links ]
Wright, S. (1951). The genetical structure of populations. Annals Eugenics, 15, 323-354. [ Links ]