Hasta hace pocas décadas se consideraba que la población indígena del Uruguay había sido exterminada sin dejar descendientes, tal como se manifestaba en la visión “oficial” plasmada en el Libro del Centenario de 1925 (López Campaña y Castells Carafi, 1925) y que aún continúa vigente en algunos sectores de nuestra sociedad. La identidad nacional enfatizaba el etnocidio Charrúa en los episodios que culminaron en la matanza de Salsipuedes en 1831, aun existiendo referencias a repartos de indígenas en diversas oportunidades (Cabrera Pérez, 1983; Padrón Favre, 1986), o a caciques que escaparon de la matanza y cuyos descendientes se han identificado en la población actual (Acosta y Lara, 1981). Por otra parte, la identidad nacional no incluía a otros grupos indígenas, si bien es conocido que el General Rivera trajo de las Misiones Jesuíticas, entre 8.000 y15.000 indígenas misioneros (estos indígenas serían principalmente Guaraníes, pero también de otras etnias; Curbelo y Barreto, 2010); Rivera instaló estos indígenas en Santa Rosa del Cuareim (actual Bella Unión). Además, diversas fuentes mencionan Chanás y Guenoas hasta el siglo XVIII (Cabrera Pérez, 1992).
Actualmente, el 4,5% de la población uruguaya reconoce tener ancestros indígenas (INE, 2012), valores que muestran una clara subdeclaración si se considera que aproximadamente el 14% del genofondo de la población es de origen indígena, y entre 34 y 37% si se considera la ancestría materna, con máximos que superan el 60% en el norte del país (Bonilla et al., 2004; 2015; Sans et al., 2015a). Estos estudios han sido posibles, fundamentalmente, por el avance en las técnicas moleculares que permiten estas cuantificaciones de modo confiable.
En relación a la herencia materna, el empleo de genomas mitocondriales completos (mitogenomas) ha contribuido a aumentar la resolución de la información, que se basaba inicialmente en el análisis de las regiones hipervariables (HVR) I y II. A partir de los mitogenomas, hasta el presente se ha definido un número creciente de haplogrupos fundadores para las poblaciones americanas, siendo los más frecuentes A2, B2, C1b, C1c, C1d, D1, D4h3 y C4c que se extienden por toda América, mientras que en América del Norte se agregan otros (Tamm et al., 2007, entre otros).
Los subhaplogrupos regionales o locales permiten una mejor identificación de lugares de origen y rutas migratorias durante y después del poblamiento de América. En Uruguay, Sans y colaboradores (2015a) encontraron algunos subhaplogrupos previamente definidos para la región, como por ejemplo D1j, inicialmente encontrado en Chile y Argentina, pero extendido hasta Brasil y Uruguay (Bodner et al., 2012; García et al., 2012). Por otra parte, se identificó un nuevo subhaplogrupo, C1d3, al que se le estimó una antigüedad de 8.974 años (intervalo de confianza 95% 5.748-12.261) (Figueiro et al., 2011; Sans et al., 2012, 2015b). No se han encontrado casos bien documentados de este subhaplogrupo fuera del país. Sin embargo, hasta la fecha se contaba con menos de una decena de mitogenomas de haplogrupos indígenas de Uruguay, impidiendo un análisis detallado de afinidades interpoblacionales a nivel regional y continental.
Con el objetivo de aportar al conocimiento del poblamiento prehistórico e histórico del territorio uruguayo y sus relaciones con otras poblaciones indígenas o cosmopolitas, se analizan mitogenomas de los cuatro haplogrupos principales de origen indígena de la población uruguaya actual.
MATERIAL Y MÉTODOS
Muestra
Se seleccionaron 32 muestras de ADN de origen indígena a partir de distintos proyectos ya realizados (Sans et al., 2011; Bonilla et al., 2015; Flores Gutiérrez, 2019; Negro, 2019), de investigaciones que se realizan en el Polo de Desarrollo Universitario “Diversidad Genética Humana” (Centro Universitario de Tacuarembó, Universidad de la República) y del Proyecto Urugenomes del Institut Pasteur Montevideo. Las 32 muestras, tomadas de distintos puntos del país (Fig. 1), corresponden a los siguientes haplogrupos: A2: 7 individuos; B2: 8 individuos; C1:11 individuos; D1: 6 individuos (Tablas Suplementarias S01-S05).
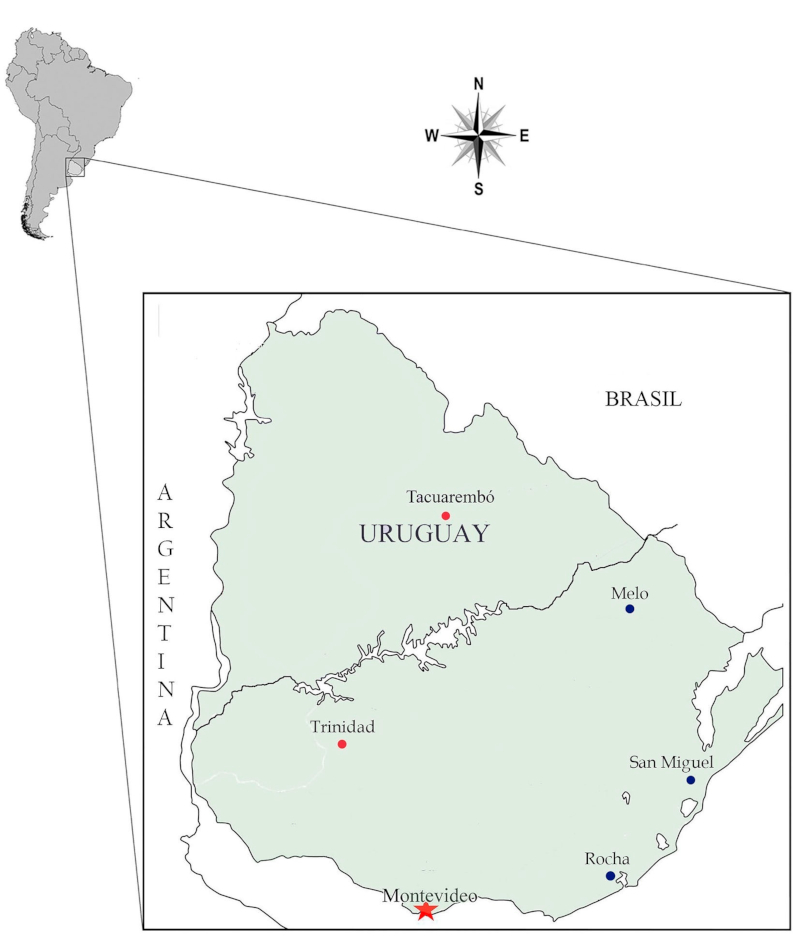
Fig. 1 Localización de los lugares de origen de los individuos cuyas secuencias se mencionan en este trabajo.
Debe señalarse que no se trata de una muestra representativa, y que se siguieron dos criterios de selección a partir de la HVRI a fin de reflejar la variabilidad existente en el país: muestras sin secuencias similares a nivel regional, y muestras con claras afinidades regionales, buscando establecer la profundidad temporal de los eventos microevolutivos.
Obtención de las secuencias
A partir de ADN previamente extraído en diversos proyectos y clasificado en los haplogrupos A2, B2, C1, o D1, se construyeron bibliotecas siguiendo el protocolo de Illumina Inc. (2016) con dos modificaciones: el enriquecimiento fue realizado con cebadores previamente disponibles en el laboratorio (9230F-2007R y 14856F-9397R, Rieder et al., 1998) en vez de los especificados en el protocolo, y se emplearon, en caso de dificultades de amplificación, tres o cuatro fragmentos (9230F-14268R, 13233F-2007R, 14856F-3346R y 3169F-9397R, Rieder et al., 1998; Martínez-Cruzado et al., 2005). La cuantificación de los productos del enriquecimiento y de las bibliotecas terminadas se realizó mediante una combinación de electroforesis en gel de agarosa y cuantificación con fluorómetro Qubit (Thermo Fisher Scientific). Se utilizó la secuenciación de extremos pareados (paired-end) con un kit de 300 ciclos en un equipo Illumina MiSeq en el Institut Pasteur de Montevideo. La profundidad de secuenciación promedio de las muestras fue de entre 200x y 4000x aproximadamente (Tabla Suplementaria S05).
En los distintos proyectos de los cuales se tomaron las muestras se había solicitado consentimiento informado y se siguieron las pautas de las declaraciones formuladas en Helsinky. Cuentan con el aval de la Comisión de Ética de la Facultad de Humanidades y Ciencias, UdelaR, cuyo consentimiento informado incluye la conformidad a participar de estudios de ancestría (121900-000087-14,121900-000140-14 y 121900-000211-16), a excepción de las muestras cuyo código inicia por KC o URU. En el primer caso (KC), son parte de un estudio caso-control sobre cáncer de mama, que fue avalado por la Facultad de Medicina, UdelaR, y cuyo consentimiento informado incluye el estudio de la ancestría (071140-000439-06). En el segundo caso (URU), corresponden al proyecto Urugenomes, del Institut Pasteur Montevideo, y cuentan con aval ético de dicha institución (IP011-17/CEI/LC/MB); el estudio abarcó ancestría en genomas completos.
Análisis de las secuencias
Se realizó el mapeo de las lecturas con relación a la secuencia de referencia de Cambridge corregida (rCRS -Andrews et al., 1999) mediante Bowtie2 (Langmead y Salzberg, 2012), y se procesaron los archivos de alineamiento y mapeo (SAM y BAM) usando Samtools (Li et al., 2009). Se buscaron variantes (polimorfismos de un solo nucleótido, inserciones y deleciones) con respecto a la rCRS mediante SNVer (Wei 2011). Las variantes presentes en cada mitogenoma fueron empleadas para la confirmación de los haplogrupos y la determinación de los subhaplogrupos más probables empleando HaploGrep 2 (Weissensteiner et al., 2016). En todos los casos, las definiciones de haplogrupos y subhaplogrupos fueron tomadas de van Oven y Kayser (2009). Todas las secuencias analizadas fueron incorporadas a GenBank, códigos MW057658-MW057689.
Se utilizan los términos de haplogrupo y subhaplogrupo considerando haplogrupo a los grupos de haplotipos fundadores que ingresaron a América (Tamm et al., 2007, Perego et al., 2010) y subhaplogrupos a los derivados de éstos, como por ejemplo, A2ah o C1d1.
Análisis de afinidades regionales y construcción de filogenias
Se confeccionaron bases de datos de variantes para los haplogrupo A2, B2, C1 y D1, que incluyeron todas las secuencias mitocondriales completas disponibles en el repositorio GenBank de acuerdo a MITOMAP (http://www.mitomap.org- Lott et al., 2013), y se alinearon las secuencias utilizando GeneDoc versión 2.7.000. (Nicholas y Nicholas, 1997). La preparación de las bases de datos a partir de MITOMAP y la posterior preparación de los datos para la elaboración de los networks se realizó mediante R 3.6.1 (R Development Core Team, 2003). Los sitios variables fueron ponderados en base a su tasa de mutación relativa (Soares et al., 2009), excluyéndose del análisis las inserciones y deleciones en los trechos 303-315, 514-523 y 16184-16193 (Bandelt et al., 1999). En virtud del alto número de secuencias y sitios, se elaboraron median-joining networks en dos etapas (Bandelt et al., 1999), una para seleccionar las secuencias de mayor afinidad con las muestras uruguayas, y otra únicamente con las secuencias seleccionadas. Los networks creados en la segunda etapa fueron utilizados para la confección manual de las filogenias, que incluyeron los mitogenomas uruguayos y los sudamericanos que presentaron mayor similitud (Figs. 2, 3, 4 y 5, correspondientes a los haplogrupos A2, B2, C1 y D1).

Fig. 2 Localización geográfica de las secuencias que presentaron similitud con los mitogenomas uruguayos secuenciados, incluyéndose los nuevos subhaplogrupos y D1+15519C+16187A.

Fig. 3 Asignación y relaciones filogenéticas de los mitogenomas uruguayos del haplogrupo A2. Las etiquetas en rojo corresponden a denominaciones nuevas propuestas en este trabajo.

Fig. 4 Asignación y relaciones filogenéticas de los mitogenomas uruguayos del haplogrupo B2. La etiqueta en rojo corresponde a una denominación nueva propuesta en este trabajo. La secuencia 35 (DQ112790 - Kivisild et al., 2006) no dispone de la región control.
Estimación de edad de subhaplogrupos
En el caso de subhaplogrupos de interés que no contaban con estimativos de edad anteriores -o donde la incorporación de nuevas secuencias ameritaba una nueva estimación de edad-, la edad del ancestro común más reciente (TMRCA por su nombre en inglés) se calculó a partir del estadístico ρ (Forster et al., 1996; Morral et al., 1994) de las secuencias divergentes. Se ignoraron inserciones y deleciones, las mutaciones 16182C y 16183C, y las variantes en la posición 16519. El intervalo de confianza del 95% se estableció sumando y restando 1,96 errores estándar de ρ. La estimación de la edad se realizó utilizando la tasa de sustitución de todo el genoma mitocondrial con corrección por profundidad temporal (Soares et al., 2009), resultando en una asimetría positiva en el intervalo.
RESULTADOS
La Figura 6 muestra la localización de las secuencias con las que se halló similitud, separadas por haplogrupo. Se describen las principales características de los motivos de las 32 secuencias (Tablas Suplementarias S01-05), sus panoramas clasificatorios y sus afinidades regionales. Se establecen nuevos clados o subhaplogrupos en los casos de secuencias que comparten al menos una mutación en la región codificante o dos en la región hipervariable, y forman dos o más ramas derivadas. Se discutirán los criterios de las asignaciones en la siguiente sección, que atañe también a algunos subhaplogrupos previamente definidos.

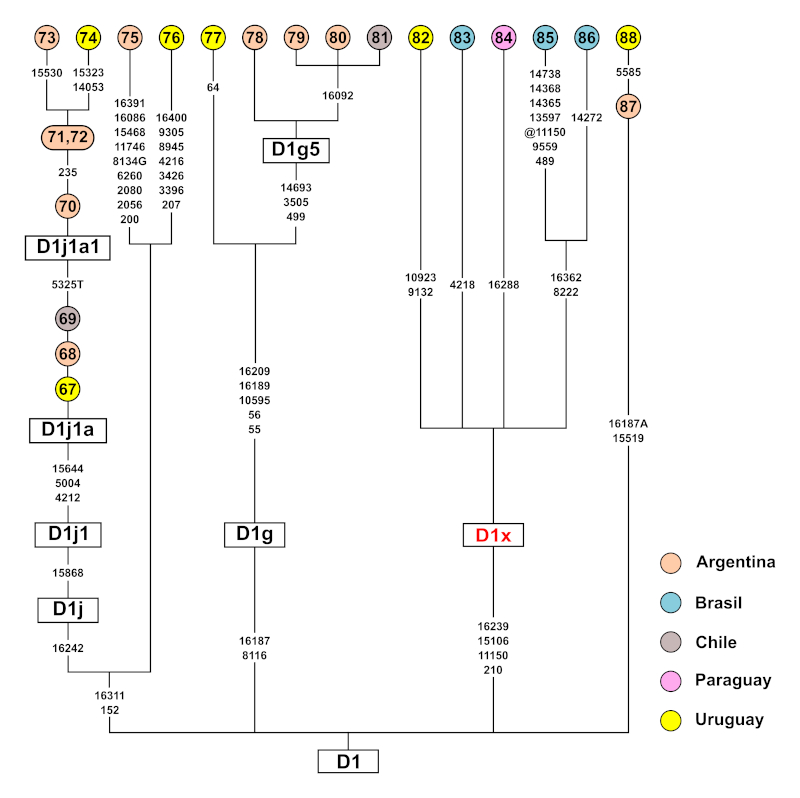
Fig. 6 Asignación y relaciones filogenéticas de los mitogenomas uruguayos del haplogrupo D1. La etiqueta en rojo corresponde a una denominación nueva propuesta en este trabajo.
Haplogrupo A2
De las secuencias del haplogrupo A2 (Fig. 2), el único caso asignable a un subhaplogrupo anteriormente definido fue el de la secuencia (1), de Trinidad (Sans et al., 2011), que pertenece a A2ah, con un tronco común con la secuencia (2), de Perú (Brandini et al., 2018) y con dos secuencias (3, 4) de las provincias bolivianas de Santa Cruz y El Beni respectivamente (Taboada-Echalar et al., 2013).
Dos secuencias de Tacuarembó (6 y 7) y una de Riberão Preto, Brasil (8; de Araujo et al., 2015) comparten con una secuencia de Montevideo (5) de la cual derivan, un motivo caracterizado por once variantes con respecto al motivo ancestral de A2. Se propone, para el subhaplogrupo compartido por estas 4 secuencias y caracterizado por las once mutaciones comunes a éstas, la denominación de A2be (59C, 64T, 152C, 297G, 3535C, 12618A, 15929G, 15930A, 16111C@, 16126C, 16278T - Tabla 1), lo cual será discutido en la siguiente sección.
Tabla 1 Subhaplogrupos propuestos en este trabajo
| Sublaplogrupos | Secuencias* | Mutaciones características |
| A2be | 5 a 8 | 59C, 64T, 152C, 297G, 3535C, 12618A, 15929G, 15930A, 16111C@, 16126C, 16278T |
| A2bf | 9 a 23 | 4616T, 16051G |
| B2an | 36 a 38 | 103A, 152C, 14470C, 16241G |
| C1b30 | 57 a 62 | 2393T, 16126C, 16270T |
| C1b31 | 63 a 65 | 146C, 10993A, 12813T, 14587G, 15728T, 16136C, 16519C |
| C1d1g** | 42 a 46 | 16287T, 16311C |
| C1d1h | 47 a 50 | 7269A |
| D1x | 82 a 86 | 210G, 11150A, 15106A, 16239T |
(*) Los números corresponden a las Figuras 3, 4, 5 y 6.
(**) El haplogrupo C1d1g fue definido por Perego et al. (2010) como C1d1d.
Otras dos secuencias de Tacuarembó (9 y 10), comparten un motivo de secuencia derivado de A2 por seis mutaciones, dos de las cuales se presentan en varias secuencias del Chaco (11-23; Sevini et al. 2014), la mayoría de las cuales comparten otras dos mutaciones. También en este caso, es posible proponer un nuevo subhaplogrupo, A2bf, caracterizado por dos mutaciones compartidas por las secuencias uruguayas y chaqueñas: 4616T, 16051G (Tabla 1).
Por último, una secuencia de Montevideo (24), presenta un motivo de secuencia altamente derivado con respecto a A2 sin que se haya podido establecer relaciones con otras secuencias del haplogrupo.
Haplogrupo B2
Los ocho mitogenomas correspondientes a este haplogrupo se muestran en la Figura 3. Tres secuencias fueron determinadas como pertenecientes a subhaplogrupos ya definidos (dos B2b -uno de ellos B2b3a2-, y uno B2e), mientras que dos casos corresponden a una nueva rama del haplogrupo B2 denominada B2an (Tabla 1) y otros tres son únicos.
La secuencia (25) de Montevideo, clasificada como B2b6, comparte mutaciones con otras dos secuencias, (26), Xavante (Fagundes et al., 2008) y (27) de Loja, Ecuador (Brandini et al., 2018). La pertenencia al subhaplogrupo B2b6 será discutida en la siguiente sección.
La secuencia (28) de Tacuarembó, fue clasificada como B2b3a2, presentando similitud de secuencia con tres individuos de Brasil: uno de Sergipe (29), otro de Ceará (30; Gómez-Carballa et al., 2018), y un indígena Kayapó/Kubenkokre (31; Fagundes et al., 2008).
La secuencia identificada como B2e (32) de Montevideo difiere de las otras tres secuencias encontradas de este subhaplogrupo, que a su vez difieren entre sí: las primeras dos, (33) Waiwai - norte de Brasil (Fagundes et al., 2008) y (34), Colombia (Hartmann et al., 2009) fueron empleadas para definir el subhaplogrupo (van Oven y Kayser, 2009). La tercera secuencia, caracterizada como indígena de Colombia (35; Kivisild et al., 2006), carece de región control, pero comparte las dos variantes que caracterizan el subhaplogrupo en la región codificante.
Dos secuencias, una de Montevideo (37), y otra de Trinidad (36; Sans et al., 2011), ambas del haplogrupo B2, pero sin pertenecer a los subhaplogrupo ya definidos, comparten cuatro mutaciones entre ellas y con una tercera secuencia (38) Xavante (Fagundes et al., 2008). También en este caso se propone un nuevo subhaplogrupo, B2an, caracterizado por las cuatro mutaciones compartidas por las tres secuencias (103A, 152C, 14470C, 16241G - Tabla 1).
Por último, tres secuencias del haplogrupo B2 (39, 40 y 41) no están relacionadas con secuencias conocidas.
Haplogrupo C1
Se analizaron 11 mitogenomas correspondientes al haplogrupo C1, siete de los cuales corresponden al subhaplogrupo C1b y el resto a C1d (Fig. 4).
Las siete secuencias clasificadas como C1b presentan situaciones diferentes. Una de éstas (57) de Montevideo comparte una mutación con una secuencia del nordeste de Brasil (58; Mendes da Silva y Greenspan, 2012). Ambas presentan un tronco común con dos secuencias de Paraguay y Argentina (Simão et al., 2019; Sevini et al., 2014); éstas dos últimas son ancestrales a otra secuencia uruguaya, (61) de Tacuarembó. Otra secuencia de Tacuarembó (62) solo comparte el tronco común. Se propone un nuevo subhaplogrupo, denominado C1b30, caracterizado por las tres mutaciones compartidas por las seis muestras (2393T, 16126C, 16270T - Tabla 1).
Otras tres secuencias identificadas como C1b (63-65), no presentan relación con otras secuencias americanas, aunque comparten entre sí cinco mutaciones; pese a la escasa variación entre estas secuencias, por la cantidad de mutaciones compartidas se designa un nuevo subhaplogrupo, C1b31, restringido a Uruguay.
Por último, una secuencia de Montevideo (66) está poco relacionada con otras secuencias.
Dos individuos C1d, (42) de Montevideo y (46) de Tacuarembó integran un subhaplogrupo denominado como C1d1d, caracterizado por 16311C y 16287T por Perego et al. (2010). Debe notarse que este subhaplogrupo difiere del mencionado por van Oven y Kayser (2009) en la última versión de Phylotree (versión17, febrero de 2016, https://www.phylotree.org/), quienes caracterizan como C1d1d a las secuencias portadoras de 16172C, dejando sin denominación al subhaplogrupo con las mutaciones 16311C y 16287T. En este artículo hemos seguido el criterio de van Oven y Kayser (2009) por lo cual utilizaremos una nueva denominación para este último subhaplogrupo, que pasa a llamarse C1d1g (Tabla 1). La primera secuencia (42) resultó idéntica a la de otro individuo de Uruguay ya analizado (43; Perego et al., 2010), mientras que la otra (46) comparte tronco común con una secuencia de Rio Grande do Sul, Brasil (44) y con una de Buenos Aires, Argentina (45), de la cual deriva, ambas de Perego et al. (2010).
Cuatro individuos: (47) de Tacuarembó, un indígena Kaingang (48; Rieux et al., 2014), y dos individuos de Perú (49 y 50; Perego et al., 2010), caracterizados por 7269A, integran un nuevo subhaplogrupo denominado C1d1h (Tabla 1).
Por último, entre las secuencias del haplogrupo C1d, una (51) de Montevideo corresponde a C1d3 y es idéntica a otra previamente secuenciada (52; Sans et al., 2015b), manteniendo la variabilidad del haplogrupo registrada por Sans et al. (2015b), representada por las secuencias (52 a 56), todas de Uruguay.
Haplogrupo D1
Dos de las seis secuencias del haplogrupo D1 se pueden asignar al subhaplogrupo D1j y una, al D1g, ambos frecuentes en el Cono Sur sudamericano; en los tres casos restantes no es posible asignar las secuencias a subhaplogrupos conocidos, pero tienen similitudes con secuencias de distintas poblaciones de Sudamérica (Fig. 5).
Las secuencias uruguayas correspondientes al subhaplogrupo D1j (67 y 74) se corresponden con el subtipo D1j1a; la primera, de Tacuarembó, tiene solamente las variantes que definen el subhaplogrupo, situación compartida con una secuencia de Córdoba, Argentina, y una de Valparaíso, Chile, mientras que la otra, de Trinidad (Sans et al., 2011), es la secuencia más derivada de D1j1a registrada a la fecha. Una tercera secuencia, (76) también de Trinidad (Sans et al., 2011), es derivada de una forma ancestral de D1j, lo cual apoya lo planteado por García et al. (2012) quienes señalan que16242T sería posterior a 152C y 16311C; si bien hay múltiples secuencias de la región control con estas características (García et al., 2012 y referencias allí citadas), la secuencia mitocondrial completa más similar a la secuencia (76) corresponde a un individuo del Chaco (75).
En cuanto al subhaplogrupo D1g, la secuencia (77), de Trinidad (Sans et al., 2011) evidencia un panorama complejo ya que presenta cinco de las ocho mutaciones que definen al subhaplogrupo D1g5 (Bodner et al., 2012), ancestral a las secuencias D1g5 descritas por Bodner et al. (2012) y García et al. (2012). Asignar un nuevo subhaplogrupo en este caso es complejo, ya que debería ser intermedio a D1g y D1g5, para contemplar las secuencias que comparten esta situación ancestral con respecto a D1g5 como actualmente se define. Este nuevo subhaplogrupo podría denominarse pre-D1g5, pero una solución más adecuada implica redenominar D1g5 al subhaplogrupo que posee las cinco mutaciones compartidas por todos los integrantes del mismo.
Cinco secuencias: una (82), de Tacuarembó, una de un individuo Poturujara del norte de Brasil (83; Fagundes et al., 2008), una del Alto Paraná, Paraguay (84; Simão et al., 2019) y dos de São Paulo, Brasil (85 y 86; de Araujo et al., 2015), comparten cuatro mutaciones: 210G, 11150A, 15106A, 16239T (Tabla 1). Se propone en este caso un nuevo subhaplogrupo de D1, D1x, con amplia distribución que incluye norte y sudeste de Brasil, Paraguay y Uruguay.
Por último, la secuencia (88) de Montevideo, se relaciona estrechamente con una secuencia de Buenos Aires (87; Bodner et al., 2012), de la cual deriva.
Edades de los subhaplogrupos analizados
Las estimaciones de edad (TMRCA) de los subhaplogrupos (Tabla 2) presentan una amplia variabilidad, con edades entre 3.900 y 20.736 años calendario. Las implicancias de estas edades serán discutidas en relación con las afinidades geográficas entre los integrantes de cada subhaplogrupo.
Tabla 2 Edades de origen (TMRCA) de los subhaplogrupos citados o propuestos en este trabajo.
| Subhaplogrupo | Límite inferior IC95% | Estimación puntual | Límite superior IC95% | Referencia |
| A2ah | 7945,6 | 10610,1 | 13314,9 | Este trabajo |
| A2be | 4489 | 6548,6 | 8634,3 | Este trabajo |
| A2bf | 6938,1 | 7532,4 | 8128,9 | Este trabajo |
| B2b6 | 12390 | 14560 | 16742 | Brandini et al., 2018 |
| B2b3a2 | 3300 | 3900 | 4800 | Gómez-Carballa et al., 2018 |
| B2e | 5589,6 | 8793,1 | 12057,3 | Este trabajo |
| B2an | 6323 | 9699,2 | 13141,8 | Este trabajo |
| C1d1g | 17642,1 | 20735,6 | 23873,2 | Este trabajo |
| C1d1h | 9138,4 | 11985,1 | 14876,6 | Este trabajo |
| C1d3 | 5747,9 | 8973,9 | 12261,4 | Sans et al., 2015 |
| C1b30 | 9154,7 | 10610,1 | 12077,5 | Este trabajo |
| D1j1a | 2060 | 5000 | 7940 | Bodner et al., 2012 |
| D1g | 13820 | 19700 | 25580 | Bodner et al., 2012 |
| D1g5* | 6545,3 | 8431,9 | 10339,7 | Este trabajo |
| D1x | 2851,2 | 4554,4 | 6276,5 | Este trabajo |
(*) La edad de D1g5 fue calculada considerando a VA35 (77) como un integrante primitivo del subhaplogrupo.
No se calculó la edad de C1b31 por su falta de variabilidad.
DISCUSIÓN
Un problema general con el cual se ha enfrentado este estudio, y posiblemente muchos de los estudios que analizan mitogenomas, es la nomenclatura. Hasta el presente, haplogrupos y subhaplogrupos han sido definidos ad hoc por un número variable de mutaciones. Esto lleva a que distintos subhaplogrupos derivados, por ejemplo, de C1d, tengan diferentes cantidades de variación acumulada y diferentes antigüedades, como es el caso de C1d1 y C1d3 (Fig. 4). Por otra parte, en algunos casos no resulta posible o al menos coherente, clasificar secuencias que, teniendo parte de las mutaciones que definen un subhaplogrupo ya descrito, serían ancestrales a éste, como es el caso del haplogrupo D1g5, definido por ocho mutaciones (Bodner et al., 2012). En otros casos, los haplogrupos han sido definidos solamente por una o pocas mutaciones de las regiones hipervariables, lo cual incluye “sitios rápidos” bien documentados (Meyer et al., 1999; Soares et al., 2009), como es el caso de C1d1c y C1d1d (sensuPerego et al., 2010), basados respectivamente en 16362C y 16311C. Por último, se debe tener en cuenta que la última actualización de Phylotree -que se emplea como estándar de nomenclatura de haplogrupos y subhaplogrupos mitocondriales- fue hecha en 2016 (van Oven y Kayser, 2009). Es posible que, luego de esa fecha, se hayan propuesto denominaciones para algunos subhaplogrupos que resulten redundantes con las utilizadas en este trabajo. Durante la última revisión de este artículo se publicó un estudio acerca de mitogenomas de la región, de García et al. (2021). Hemos modificado los nombres de los subhaplogrupos de forma acorde a dicho trabajo, pero la revisión de las filogenias incluyendo las secuencias publicadas en ese artículo, en particular del haplogrupo D1, será objeto de un trabajo futuro. Hechas estas salvedades, comenzaremos la discusión por los nuevos subhaplogrupos propuestos en este trabajo (Tabla 1).
Por otra parte, debemos mencionar que los resultados y afinidades presentados conducen a diversas hipótesis filogeográficas, pero sin presentar un análisis filogeográfico en sentido estricto (sensuAvise et al., 1987). Un análisis estricto requiere el registro de las frecuencias de los linajes a nivel geográfico, pero actualmente no existe una densidad de muestreo representativa a nivel de mitogenomas que permita realizarlo.
De los siete subhaplogrupos propuestos, cuatro están caracterizados por dos a cuatro mutaciones e incluyen al menos tres individuos que integran tres o más ramas, por lo cual parecen bien definidos: A2be, B2an, C1b30 y D1x. El primero (A2be - caracterizado por 4616T, 16051G) está integrado fundamentalmente por secuencias del Chaco argentino, a la que se agregan dos secuencias del noreste uruguayo. Si bien las características no permiten determinar el origen geográfico de este subhaplogrupo, su mayor frecuencia y variedad (dos ramas) indicaría un origen chaqueño. Sin embargo, y a pesar de la fuerte afinidad regional, la divergencia de las secuencias uruguayas con respecto al clado chaqueño sugiere una evolución separada relacionada a la antigüedad del origen del subhaplogrupo A2be (estimada en 7.532 años - ver Tabla 2). El segundo subhaplogrupo (B2an - 103A, 152C, 14470C, 16241G) presenta una situación diferente ya que las dos secuencias uruguayas y la Xavante son aproximadamente equidistantes y separadas por un mínimo de tres mutaciones. Se han encontrado en el país varias secuencias con 16241G, la mutación de la HVRI que caracteriza a este nuevo subhaplogrupo: dos de Tacuarembó (sin publicar), una de Montevideo y otra de Mercedes (MN108509 y MN108556; Sans et al., 2020) y una secuencia de Bella Unión (Ar45; Sans et al., 2015b) que comparte también las mutaciones de la HVRII. Otras secuencias de HVRI de habitantes de la región de la Cuenca del Plata también presentan 16241G: en Rio Grande do Sul, Brasil (Marrero et al., 2007a), en el nordeste brasileño (Alves Silva et al., 2000), en Buenos Aires, Argentina (Catelli et al., 2011), y en Mbyá-Guaraní en Misiones, Argentina (Sala et al., 2010). Aunque la variación del subhaplogrupo sugiere una gran antigüedad (estimada en 9.699 años - Tabla 2), su presencia en Uruguay y regiones próximas se podría relacionar a la dispersión Guaraní hacia el sur, de la cual hay registro en el actual territorio de Uruguay en tiempos inmediatamente anteriores a la llegada del europeo (López Mazz y López Cabral, 2020). El subhaplogrupo C1b30 (2393T, 16126C, 16270T) presenta seis secuencias. Todas menos una, de Tacuarembó, comparten una cuarta mutación, 3197C, formando dos ramas principales. La primera incluye secuencias de Paraguay y Argentina (Simão et al., 2019; Sevini et al., 2014), que serían ancestrales a la secuencia uruguaya 61, mientras que otra secuencia uruguaya forma una segunda rama junto a una secuencia del nordeste brasileño (Mendes da Silva y Greenspan, 2012). La tercera rama de la filogenia incluye solamente una secuencia uruguaya. Debe agregarse que al analizar en secuencias uruguayas las dos mutaciones de la HVRI que están en la base de este nuevo subhaplogrupo y 3197C, compartida por una de las ramas, se encontró una secuencia con las tres mutaciones (MN108739), y dos con 16126C y 3197C (MN10635, MN108761; Sans et al., 2020). En Rio Grande do Sul, Brasil, en el análisis de la HVRI también se encontró una secuencia C1 con 16126C y 16270T (Marrero et al., 2007a). El origen geográfico de este haplogrupo es aún incierto y deberá ser analizado con mayor número de mitogenomas, pero la diversidad del mismo apunta a un origen en el Holoceno temprano, con un estimado de edad de 10.610 años (Tabla 2), similar a la del último subhaplogrupo considerado (B2an). Por último, D1x (210G, 11150A, 15106A, 16239T), con una amplia distribución geográfica, presenta las dos secuencias más derivadas en São Paulo, Brasil. En Uruguay se han encontrado varias secuencias D1 con la única mutación de la HVR1 común a este subhaplogrupo: una publicada por Pagano et al. (2005), y MN108648 y MN108657 (Sans et al., 2020), entre otras. Algunas de éstas presentan también la variante 16288C, encontrada en la secuencia de Alto Paraná, Paraguay, incluida en la filogenia, lo cual muestra la vinculación entre las dos regiones, aunque no explica la región de origen. La edad de este nodo, calculada en 4.554 años, es de las más recientes de este trabajo (Tabla 2).
En lo referente al subhaplogrupo A2bf, las once mutaciones del tronco común (59C, 64T, 152C, 297G, 3535C, 12618A, 15929G, 15930A, 16111C@, 16126C, 16278T) indicarían una relación lejana con A2, pero posiblemente se encuentren en el futuro secuencias intermedias que compartan parte de estas mutaciones. Pese a lo derivado de estas secuencias, su relativamente reducida variación hace que su edad estimada sea intermedia entre las calculadas en este trabajo: 6.549 años (Tabla 2). Es interesante notar que hasta el momento, el motivo 16111C@+16126C+16278T ha sido hallado en Córdoba, Argentina (Catelli et al., 2011), diversas regiones de Brasil (Badano et al., 2018; Marrero et al., 2007a; Marrero et al., 2007b; Ramallo et al., 2013; Cárdena et al., 2013) y en varias zonas de Uruguay (Sans et al., 2020). En algunos casos se agrega alguna variante al motivo señalado. Se requeriría un análisis de los mitogenomas de las secuencias que comparten el motivo de la HVRI para inferir el origen de este haplogrupo, ya que si bien la mayor frecuencia se encuentra en Uruguay, podría deberse a un efecto fundador. Un caso similar pero más restringido geográficamente es el subhaplogrupo propuesto C1b31, localizado solo en Uruguay y caracterizado por siete mutaciones compartidas y dos ramas poco diferenciadas.
El último subhaplogrupo nuevo propuesto es C1d1h, caracterizado por la mutación 7249T, figuraba sin denominación en la filogenia confeccionada por Perego et al.. (2010). Se agrega ahora una secuencia uruguaya, por lo que se forman tres ramas; la antigüedad del nodo fue calculada en 11.985 años (Tabla 2), fecha cercana a las estimadas para el inicio del poblamiento del territorio uruguayo hace aproximadamente 12.800 años cal. (Suárez Sainz, 2019).
Otros subhaplogrupos estaban previamente definidos pero se plantea una revisión o cambio de denominación. El primer caso a considerar es el de B2b6, clasificación dada a tres secuencias que comparten 2056A, de Uruguay, Brasil y Ecuador (Fagundes et al., 2008; Brandini et al., 2018). Brandini et al. (2018) han incluido a la última secuencia dentro del subhaplogrupo B2b6b1, junto a otras secuencias de Perú y Ecuador, que incluye también 16266T, de la cual carecen las secuencias 25 y 26. De momento, son escasos los datos que permitan modificar la filogenia planteada por Brandini y colaboradores, o descartar afinidades entre la secuencia uruguaya y la brasileña y las de Perú y Ecuador. Para otro subhaplogrupo, C1d1g, se propone un cambio de denominación, ya que había sido mencionado por Perego et al. (2010) como C1d1d. Con las tres nuevas secuencias uruguayas queda mejor definido, con cinco secuencias que comparten 16287C y 16311T y con dos ramas principales. Las características de este subhaplogrupo llaman la atención por dos motivos: por una parte, la distribución regional (Uruguay, sur de Brasil, y zona aledaña de Argentina), y por la gran profundidad temporal, puesto que su antigüedad fue estimada en este trabajo en 20.736 años (17.642 - 23.873 años) (Tabla 2). Esta fecha es mayor a las estimadas por Perego et al. (2010) de 18.700 ± 1.400 para C1d y 16.200 ± 1.100 para C1d1, aunque queda dentro del rango de la primera. La edad estimada para C1d1g podría relacionarse con una rápida dispersión del haplogrupo fundador C1d1, como ya ha sido señalado para otros haplogrupos como es el caso de D1g y su derivado D1g4 (Bodner et al., 2012), con antigüedades comparables a la calculada en este trabajo.
Se agrega una secuencia al subhaplogrupo C1d3, de carácter más local (Figueiro et al., 2011; Sans et al., 2012, 2015a) y con una antigüedad estimada de 8.974 años, lo cual no se modifica ya que la secuencia descrita en este trabajo es idéntica a otra ya publicada (Sans et al., 2012).
Con relación al subhaplogupo A2ah (64T, 16097C, 16098G), llama la atención la gran divergencia de la secuencia uruguaya respecto al motivo ancestral, que resulta en una antigüedad calculada de 10.610 años (Tabla 2). Se han encontrado varias secuencias de la HVRI que presentan el motivo característico, concretamente, una secuencia prehistórica del noroeste argentino, dos secuencias de Bolivia, una del Chaco, Argentina, y una de Minas Gerais, Brasil (Russo et al., 2018), a las que se agregan dos secuencias de Paraná, Brasil (individuos BRPR002 y BRPR015; Poletto et al., 2019). La secuenciación de más mitogenomas A2ah permitirá arrojar luz sobre la variabilidad del subhaplogrupo.
Por último, debemos referirnos a otros dos subhaplogrupos también identificados previamente: D1j y D1g. Las dos secuencias clasificadas dentro del subhaplogrupo D1j1a presentan situaciones muy diferentes, ya que una (67) forma parte del tronco común a la base de D1j1a1 mientras que la otra (74) es la más derivada de las descritas hasta el momento. García et al. (2012) encuentran D1+152C+16311C en las Sierras Pampeanas en secuencias de la HVR, postulando que 16242T sería posterior y, por lo tanto, ese sería el lugar de origen del subhaplogrupo, con posterior distribución a diversas regiones de Argentina, Chile, Bolivia y Uruguay. El motivo 16311C+152C con ausencia de 16242T también se encuentra en una de las secuencias D1 obtenidas en este trabajo (76). Esta secuencia se agrupa con una del chaco argentino (75, Sevini et al., 2014) y estaría relacionada a una etapa previa a D1j. Por otra parte, el motivo nodal D1+16242T+16311C en la HVRI es frecuente en Uruguay (Sans et al., 2015a, 2020). Si bien el motivo compartido 152C-16311C puede no deberse a una ancestría común por corresponder a sitios con alta tasa de mutación (Soares et al., 2009), se debe mencionar que García et al. (2012) lo señalan como ancestral a D1j por su alta frecuencia en las Sierras Pampeanas del centro de Argentina (García et al., 2012). En cuanto al subhaplogrupo D1g, la secuencia (77) de Trinidad (Sans et al., 2011) presenta cinco de las ocho mutaciones que definen al subtipo D1g5. Es poco plausible que las restantes se deban a reversiones, por lo que debe postularse que sería una forma ancestral a las secuencias D1g5 registradas a la fecha en Chile y Argentina (Bodner et al., 2012; García et al., 2012). En el trabajo recientemente publicado, mencionado al comienzo de esta discusión, se incluye una secuencia idéntica a la uruguaya (77), correspondiente a la región central de Argentina (García et al., 2021). La existencia de una forma intermedia entre D1g y D1g5 estaría avalada por las cronologías estimadas para estos subhaplogrupos (18.300 y 8.432 años respectivamente; Bodner et al., 2012 y Tabla 2). El subhaplogrupo D1g5 es característico de una amplia región que abarca desde el extremo norte hasta el extremo sur de la Argentina así como la región central de Chile (Motti et al., 2019), ante lo cual es posible plantear dos escenarios. El primero, que tenga su origen en la región oriental del Cono Sur, en las cercanías o dentro del actual territorio de Uruguay, y de allí se haya dispersado por Argentina y Chile, lo cual parece poco probable dada la extensión que ocupa. El segundo escenario es que se haya originado en la región central de Argentina, y que sea un hallazgo fortuito en la población uruguaya como producto de migraciones tempranas de representantes de ese subhaplogrupo.
CONCLUSIONES
Este estudio incorpora 32 nuevos mitogenomas al total de secuencias de origen indígena de Sudamérica. Once secuencias pudieron ser incluidas en subhaplogrupos ya definidos, incluyéndose en este grupo dos C1d1g (previamente incluidos en C1d1d sensuPerego et al., 2010) y uno, B2b6 (de adscripción discutida). Seis secuencias fueron adscritas a los haplogrupos fundadores (A2, B2, C1b y D1), sin que haya sido posible para ellos, determinar subhaplogrupos ni definir nuevos. Por último, quince secuencias fueron incluidas en nuevos subhaplogrupos, presentados en este trabajo, y cuyas antigüedades fueron calculadas entre 4.554 y 11.985 años. En virtud del diseño del estudio, que partió de secuencias con motivos de la HVR considerados de especial interés, no deben tomarse estas proporciones como indicativas de la variabilidad mitocondrial existente en Uruguay. Sin embargo, debe enfatizarse que aproximadamente dos tercios de las secuencias no responden a subhaplogrupos determinados con anterioridad.
Al igual que parece ser el caso del subhaplogrupo C1d3 (Sans et al., 2012, 2015b), varias secuencias reflejarían la existencia de poblaciones territorialmente restringidas. Esto se apoya en la antigüedad estimada de los subhaplogrupos, cuatro los cuales superan los 10.000 años, aunque el intervalo de confianza en estos casos es amplio. Sin perjuicio de las migraciones y eventual flujo génico evidenciados en los motivos de secuencia compartidos, aquéllos sin similitud registrada apuntan a escenarios de baja movilidad por parte de las poblaciones portadoras de los mismos.
Nuevas secuencias mitocondriales completas, tanto del Uruguay como de la región, permitirán un análisis más profundo acerca de la antigüedad, lugar de origen, y distribución de los subhaplogrupos nuevos y de los previamente definidos a los cuales refiere este trabajo.












 uBio
uBio