Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Agriscientia
versión On-line ISSN 1668-298X
Agriscientia vol.35 no.2 Córdoba dic. 2018
COMUNICACIóN
Evaluación de la técnica de amplificación por recombinasa y polimerasa (RPA) para la detección de begomovirus presentes en cultivos de soja y poroto en Argentina
Evaluation of the RPA technique for the detection of begomoviruses present in soybean and bean crops in Argentina
Reyna, P.1,2 y RodrÍguez Pardina, P.1
1Instituto de PatologÍa Vegetal (IPAVE, CIAP, INTA), Camino 60 Cuadras, Km 5 1/2, X5020ICA Córdoba, Argentina.
2Consejo Nacional de Investigaciones CientÍficas y Técnicas (CONICET), Godoy Cruz 2290 (1425 FBQ), CABA, Argentina.
Correspondencia a: rodriguez.patricia@inta.gob.ar
Fecha de recepción: 30/01/2018; fecha de aceptación: 05/12/2018
RESUMEN
Los métodos tradicionales de diagnóstico son inespecÍficos para la identificación de begomovirus. Actualmente se usan técnicas moleculares, que requieren equipamientos sofisticados o procedimientos complejos. La técnica de amplificación mediante recombinasa y polimerasa (RPA) es similar a la reacción en cadena de la polimerasa (PCR), sensible, especÍfica, pero opera a temperatura constante. Con el fin de evaluar la utilización de esta técnica para la detección de begomovirus presentes en soja y poroto en Argentina, se diseñaron iniciadores especÍficos. Se probaron inicialmente por PCR, con clones de virus detectados en el paÍs, y se logró amplificar una banda de 371 pb. La RPA se llevó a cabo con el Twist Amp® Basic kit utilizando muestras de soja y poroto, sanas y enfermas y malezas infectadas. Se probaron dos métodos de conservación de muestras: hojas liofilizadas y hojas mantenidas a -70 °C; y dos de obtención de ADN: CTAB y molido de la muestra en 0,5M OHNa. La reacción se incubó a 37 °C durante 30 min, y luego a 65 °C durante10 min. Se visualizaron bandas del tamaño esperado en plantas infectadas y no en testigos sanos. No hubo diferencias según los métodos de conservación de material ni con los de extracción de ADN utilizados. Se logró ajustar la RPA para la detección de begomovirus en cultivos de soja y poroto de Argentina.
Palabras clave: Glycine max; Phaseolus vulgaris; Amplificación mediante polimerasa recombinasa.
SUMMARY
Traditional diagnostic methods are non-specific for begomovirus identification. At present molecular techniques are used, which require sophisticated equipment or complex procedures. The Recombinase polymerase amplification technique (RPA) is similar to the Polymerase chain reaction (PCR), sensitive and specific, but operates at constant temperature. In order to evaluate the use of this technique for the detection of begomovirus present in soybeans and beans in Argentina, specific primers were designed. They were initially tested by PCR, using clones of the different begomoviruses detected in our country and a 371pb band was successfully amplified. RPA was carried out using the Twist AMP ® Basic Kitto test soybean, bean and weed infected samples, as well as healthy soybean and bean controls. Two conservation methods were tested: lyophilized leaves and leaves maintained at-70 °C; and two DNA extraction methods: CTAB and crude extract grounded in OHNa 0.5M. The reaction was incubated at 37 °C/30 min. and at 65 °C/10 min. Bands of the expected size were visualized in infected samples, and not in healthy controls. There were no differences in the results due to either method of conservation or DNA extraction. RPA technique was successfully optimized for the detection of begomoviruses infecting soybean and bean crops of Argentina.
Keywords: Glycine max; Phaseolus vulgaris; Recombinase Polymerase Amplification.
INTRODUCCION
La familia Geminiviridae está constituida por virus vegetales con genoma conformado por uno o dos componentes de ADN de cadena simple (scADN) de aproximadamente 2500 pb (Fauquet et al., 2008). Esta familia se divide en nueve géneros, de acuerdo al rango de hospedantes, vector, composición del genoma viral y nivel de homologÍa de nucleótidos (Zerbini et al., 2017). En el género Begomovirus se incluyen virus que infectan dicotiledóneas y son transmitidos por la mosca blanca Bemisia tabaci (Hemiptera, Aleyrodidae) de manera circulativa. Este género, con más de 300 especies descriptas, es el más importante y reúne a algunos de los virus más devastadores de las regiones tropicales y subtropicales, entre los que podemos mencionar al African cassava mosaic virus (ACMV) en áfrica, Bean golden mosaic virus (BGMV) en América y Tomato yellow leaf curl virus (ToYLCV) en áfrica, América, Asia y Europa (Inoue-Nagata, Lima y Gilbertson, 2016).
En Argentina, especialmente en la región del noroeste (NOA), se han detectado numerosas especies de begomovirus infectando cultivos de soja (Glycine max) y poroto (Phaseolus vulgaris),entre ellas BGMV, Bean dwarf mosaic virus (BDMV), Tomato yellow spotvirus (ToYSV), Soybean blistering mosaic virus (SBlMV), Sida golden mosaic Brazil virus (SiGMBRV), Tomato yelllow vein streak virus (ToYVSV), Euphorbia mosaic virus (EuMV) y Tomato mottle wrinkle virus (ToMoWrV). Varios de estos virus se encontraron, además, en malezas y especies silvestres que actúan como reservorios (Laguna, Fiorona y RodrÍguez Pardina, 2009; RodrÍguez Pardina, Hanada, Laguna, Zerbini y Ducasse, 2011; Avalos, Laguna y RodrÍguez Pardina, 2016).
La detección rápida y especÍfica de los patógenos vegetales es un aspecto importante para el correcto manejo de las enfermedades. Los métodos tradicionales como rango de hospedantes y serologÍa son muy utilizados en la detección de numerosas especies virales. De hecho, la técnica serológica de ELISA, utilizando anticuerpos monoclonales, ha sido empleada para la detección de varios geminivirus como BGMV (Cancino et al., 1995), ACMV (Givord et al., 1994) y Tomato leaf curl virus (ToLCV) (Devaraja et al., 2004). Sin embargo, la detección especÍfica de begomovirus mediante serologÍa tiene ciertas limitaciones; por un lado existe una estrecha relación serológica entre los distintos miembros del grupo, por lo que la producción de antisueros especÍficos para geminivirus, resulta complicada. Además, la concentración de virus en la planta es muy baja, razón por la cual su purificación se ve dificultada. Por otra parte, las partÍculas tienen escasa capacidad inmunogénica y la concentración de anticuerpos especÍficos obtenidos también es baja, por lo que con frecuencia, los sueros resultan poco aptos para su empleo en la técnica de ELISA (Kushwaha, Singh, Chattopadhyay y Chakraborty, 2010). Por todo ello, las pruebas más utilizadas en la detección e identificación de geminivirus son: reacción en cadena de la polimerasa (PCR, por sus siglas en inglés) (Deng, McGrath, Robinson, Harrison, 1994; Potter, Nakha, Mejia y Maxwell, 2003; Rojas, Gilbertson, Russell y Maxwell, 1993) e hibridación de ácidos nucleicos (Hajimorad et al., 1996; Rivas Platero y Lastra, 1993; RodrÍguez Pardina et al., 2011). La técnica de PCR es altamente especÍfica, rápida, sensible, pero posee algunas desventajas para su utilización a gran escala como es la alta frecuencia de aparición de falsos positivos o falsos negativos; lo primero es debido fundamentalmente a contaminaciones en los reactivos e instrumental, fácilmente detectables por la alta sensibilidad del método; mientras que los falsos negativos se originan, principalmente, por la presencia de sustancias inhibidoras en ciertos extractos vegetales. Aparte, tanto para PCR como para las sondas de hibridación molecular, se requieren equipos sofisticados, que no están siempre disponibles en los laboratorios de diagnóstico.
Algunas de las desventajas del PCR fueron subsanadas con el desarrollo del Loop mediated isothermal amplification (LAMP), una técnica que posee una sensibilidad y especificidad similares a las del PCR. Mediante este método se puede amplificar ADN bajo condiciones de temperatura constante (65 °C), lo que permite el uso de equipos de bajo costo. Pero entre sus desventajas, podemos mencionar que requiere de cuatro a seis cebadores, que en general son difÍciles de diseñar. Además, al igual que la PCR, necesita extractos de ADN libres de contaminantes (Fukuta et al., 2003; Kapoor et al., 2017).
Piepenburg, Williams, Stemple y Armes (2006) desarrollaron una nueva tecnologÍa denominada Recombinase polymerase amplification (RPA), que fue introducida por TwistDx Ldt (Reino Unido). En la RPA, la amplificación de fragmentos especÍficos de ADN se logra a través de una combinación de proteÍnas (gp32, usvX y usvY) y la enzima Bacillus subtilis DNA polimerasa I, en presencia de ATP y un agente quelante (generalmente polietilenglicol de alto peso molecular) (Lobato y O'Sullivan, 2018; Valasevich y Schneider, 2017). El producto de RPA puede ser visualizado en geles de agarosa después de la purificación o desnaturalización de las proteÍnas involucradas en el proceso, aunque también existen métodos alternativos como fluorescencia y/o hibridación (Mekuria, Zhang y Eastwell, 2014; Silva, Bomer, Nkere, Lava Kumar y Seal, 2015; Zhang et al., 2014). Este método tiene una serie de ventajas con respecto a los mencionados anteriormente. En primer lugar, todo el proceso de amplificación se completa en 20 minutos, a temperatura constante entre 25 y 42 °C. Además, se ha comprobado que la enzima utilizada no es sensible a la presencia de inhibidores en el extracto de ADN, por lo que se pueden usar métodos más sencillos de extracción de ácidos nucleicos. Se han citado numerosos trabajos de la aplicación de esta técnica para la detección de patógenos vegetales, entre ellos podemos mencionar Little cherry virus 2 (LChC2) (Mekuria et al., 2014), Plumppox virus (PPV) (Zhang et al.,2014), Banana bunchy top virus (BBTV) (Kapoor et al., 2017) y candidatus Phytoplasma mali (Valasevich y Schneider, 2017). Recientemente, se ha publicado un artÍculo en el que se describe la utilización de RPA para la detección de tres begomovirus: Bean golden yellow mosaic virus (BGYMV), Tomato mottle virus (ToMoV) y TYLCV En este, se menciona el diseño de oligonucleótidos especÍficos para cada uno de los virus evaluados (Londoño, Harmon y Polston, 2016). El objetivo del presente trabajo fue evaluar la posibilidad de utilizar la técnica de RPA para la detección inespecÍfica de los begomovirus que afectan a los cultivos de soja y poroto de Argentina.
MATERIALES Y MéTODOS
Se trabajó con plantas de soja (S12) con sÍntomas de clorosis y ampollado generalizado, poroto (P118) con bajo porte, acortamiento de entrenudos, ampollado de hojas y mosaico dorado generalizado. También se incluyó la maleza (Leonurus sibiricus) con clorosis internerval y mosaico cálico (M7). Todas las muestras fueron recolectadas durante la campaña agrÍcola 2016 y mantenidas a -70 °C y liofilizadas. Estas fueron previamente analizadas mediante hibridación molecular con sondas especÍficas para las diferentes especies de begomovirus citadas en el paÍs, de acuerdo al protocolo descripto por RodrÍguez Pardina etal. (2011). Según esta técnica, la muestra S12 estaba infectada con SBIMV, P118 con BGMV, mientras que M7 tenÍa una infección mixta entre cuatro virus ToYSV, ToYVSV, ToMoWrV y SiGMBRV Además, como controles negativos, se emplearon plantas sanas de soja y poroto.
Para la extracción de ADN fueron utilizadas tanto las muestras liofilizadas como las conservadas a -70 °C, evaluando dos métodos de obtención de ADN: extracción con CTAB (Doyle y Doyle, 1987) y homogenización con hidróxido de sodio, de acuerdo al siguiente protocolo: se molió material vegetal en 0,5N OHNa, en relación 1/3 (p/v) para el caso de muestras mantenidas a -70 °C y 1/18 (p/v) para las liofilizadas. Los extractos crudos fueron clarificados por centrifugación a 12.000 rpm por 3 min y se recolectó el sobrenadante para ser empleado en el ensayo (Londoño et al., 2016).
Se diseñaron oligonucleótidos que hibridan en una región conservada de la cápside proteica de los begomovirus (Figura 1). Según lo recomendado para RPA (Piepenburg et al., 2006), los iniciadores eran > 29pb, ricos en GC en el extremo 3', en el 5'contenÍan pirimidinas y amplificaban fragmentos menores a 500 pb (Tabla 1).

Figura 1. Alineamiento de la cápside proteica de diferentes especies de begomovirus detectadas en soja y poroto. Las flechas indican la posición de los iniciadores, en rojo los nucleótidos discrepantes en la secuencia de los cebadores
Tabla 1.Oligonucleótidos utilizados en este estudio

En una primera instancia, los oligonucleótidos fueron evaluados mediante PCR, utilizando como ADN molde, clones de SiGMBRV, BGMV, ToYSV, ToYVSV y ToMoWrV y agua como control negativo. Las reacciones de PCR se llevaron a cabo en un volumen final de 25 pl, conteniendo: 5 pl de tampón de PCR 5X; 1 pl de cloruro de magnesio 2,5 mM; dNTPs mezcla 2,5 mM concentración final; 1 pl de iniciadores (10pM), más una unidad de Taq polimerasa (Promega Corporation Madison, Wisconsin, Estados Unidos) y 1 pl (500 ng a 1 pg) de ADN molde. Las condiciones de PCR fueron las siguientes: una primera etapa de desnaturalización de 95 °C por 2 min; una segunda etapa de 35 ciclos de: desnaturalización a 95 °C por 30 s, hibridación a 60 °C por 30 s; y extensión a 72 °C por 1 min, más una etapa final extensión a 72 °C por 5 min.
Los productos de PCR se analizaron por electroforesis en geles de agarosa (2% p/v), teñidos con Gel Red. BiotiumT (2,5X), a fin de verificar la presencia de banda del tamaño esperado.
El ajuste del RPA se hizo con el Twist Amp® Basic kit, de acuerdo a las especificaciones del fabricante (TwistDx limitated, Cambridge, Reino Unido). En este caso se probaron las muestras S12, P11 y M7, y los controles negativos previamente descritos. Cada reacción se llevó a cabo en un volumen final de 50 pl, que contenÍa 29,5 pl de tampón de rehidratación; 2,4 pl de iniciadores Fw y Rv (10 pM); 2 pl de ADN molde; 11,2 pl de agua y 2,5 pl de acetato de magnesio 280 mM. Se evaluaron dos tiempos de incubación a 37 °C: 40 y 30 min y, en ambos casos, según lo sugerido por Londoño et al. (2016), se realizó una segunda incubación a 65 °C por 10 min para degradar las proteÍnas contenidas en la reacción de amplificación. Los amplicones se visualizaron por electroforesis, de acuerdo a las condiciones especificadas anteriormente.
RESULTADOS Y DISCUSIóN
Con la técnica de PCR se pudo amplificar la banda de tamaño adecuado (371 pb), para todos los clones evaluados (Figura 2), comprobándose que los iniciadores diseñados son capaces de detectar estas cinco especies de begomovirus.
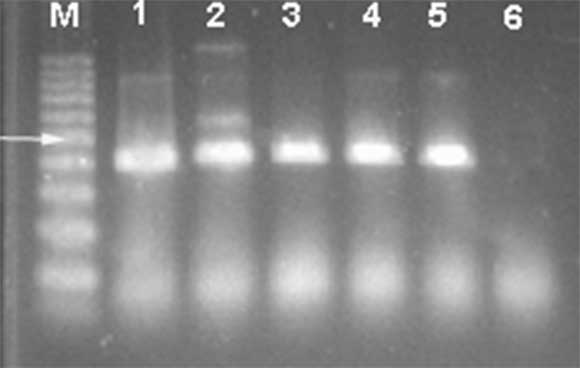
Figura 2.Productos de PCR de clones de diferentes especies de begomovirus con los iniciadores diseñados para RPA: M-Marcador 100 pb Inbio Highway. 1) Sida golden mosaic Brazil virus. 2) Bean golden mosaic virus. 3) Tomato yellow spot virus.4) Tomato yellow vein streak virus. 5) Tomato mottle wrinkle virus.6) Agua. La flecha indica la posición de la banda de 500pb en el marcador de peso molecular.
Cuando se aplicó la técnica de RPA se visualizaron, en todos los casos, bandas del tamaño esperado (371 pb) en las plantas infectadas y no en los testigos sanos. Si consideramos que las muestras evaluadas estaban infectadas, ya sea en forma simple o múltiple, con al menos seis virus distintos, pudimos comprobar que mediante esta técnica es posible detectar diferentes especies, con un solo par de oligonucleótidos, aun considerando que en el diseño de los mismos, especialmente el Rv, se observan varios nucleótidos no coincidentes en las secuencias alineadas (Figura 1). Este hecho es consistente con lo reportado por otros autores, que observaron que la RPA puede tolerar hasta nueve cambios en la secuencia de los iniciadores, permitiendo la detección de diferentes razas de virus (Lobato y O'Sullivan, 2018) o, como en este caso, diferentes especies virales, que compartan una región relativamente conservada.
Por otro lado, se observó que no hubo diferencias según los tratamientos (muestras liofilizadas o mantenidas a -70 °C) ni con los dos métodos de extracción utilizados (Figura 3). Tampoco se encontraron diferencias cuando se probaron los dos tiempos de incubación (resultados no mostrados), por lo que se recomienda, para ahorro de tiempo, incubaciones a 37 °C por 30 min.
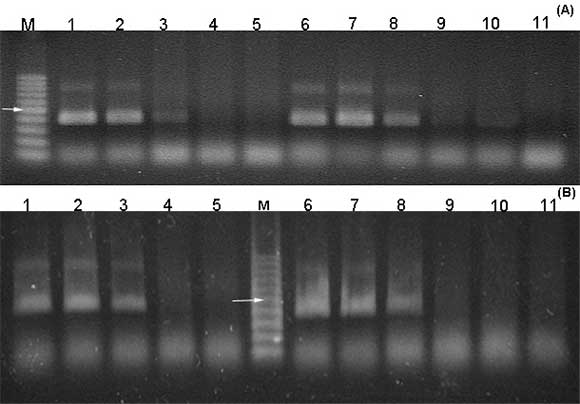
Figura 3.Productos de RPA. (A) Material liofilizado y (B) Material conservado a -70 °C. 1) Maleza (M7). 2) Soja (S12). 3) Poroto (P118). 4) Soja sana. 5) Poroto sano (a partir de extracciones por CTAB). 6) Maleza (M7). 7) Soja (S12). 8) Poroto (P118). 9) Soja sana.10) Poroto sano (a partir de extracciones en OHNa 0,5M). M: Marcador 100pb Inbio Highway. 11) Control negativo (agua). Las flechas indican las posiciones de las bandas de 500pb en los marcadores de peso molecular.
El RPA ha sido ampliamente usado para la detección de numerosos patógenos en humanos y animales (Bonney et al., 2017; Boyle et al., 2013; Gao, Jiang, Wang y Wei, 2018; Kim y Lee, 2017; Law et al., 2018; Wang, Liu, Wang, Pang y Yuan, 2018), y más recientemente para enfermedades en plantas, incluyendo la detección de begomovirus (Londoño et al., 2016) y, como ya se ha mencionado, ha sido sugerido como una buena alternativa de la PCR en laboratorios de baja complejidad, ya que no requiere de equipamientos sofisticados y el desarrollo de la técnica es más rápido y de bajo costo, comparado con la PCR. En este trabajo, se logró ajustar la técnica de RPA para la detección inespecÍfica de begomovirus citados en cultivos de soja y poroto de Argentina. De este modo, se cuenta con una técnica de diagnóstico inicial rápida y confiable para un grupo de virus emergentes en cultivos de gran importancia económica.
AGRADECIMIENTOS
Este trabajo fue financiado por el proyecto Programa Nacional de Protección Vegetal 1135022 del Instituto Nacional de TecnologÍa Agropecuaria. Los autores agradecen a la Dra. Liliana Di Feo, por la revisión crÍtica del manuscrito.
BIBLIOGRAFÍA
1. Bonney, L. C., Watson, R. J., Afrough, B., Hewson, R., Mullojonova, M., Dzhuraeva, V. y Tishkova, F. (2017). A recombinase polymerase amplification assay for rapid detection of Crimean-Congo Haemorrhagic fever Virus infection. PLoS Neglected Tropical Diseases, 11(10). doi:10.1371/journal.pntd.0006013. [ Links ]
2. Boyle, D. S., Lehman, D. A., Lillis, L., Peterson, D., Singhal, M., Armes, N., Parker, M., Piepenburg, O. y Overbaugh, J. (2013). Rapid detection of HIV-1 proviral DNA for early infant diagnosis using recombinase polymerase amplification. mBio, 4(2), e00135-00113. doi:10.1128/mBio.00135-13. [ Links ]
3. Cancino, M., Abouzid, A. M., Morales, F. J., Purcifful, D. E., Polston, J. y Hiebert, E. (1995). Generation and characterization of three monoclonal antibodies useful in detecting and distinguishing bean golden mosaic virus isolates. Phytopathology, 89, 818-828. [ Links ]
4. Deng, D., McGrath, P. F., Robinson, D. y Harrison, B. D. (1994). Detection and differentiation of whitefly-transmitted geminiviruses in plants and vector insects by the polymerase chain reaction with degenerated primers. Annals of Applied Biology, 125, 327-336. [ Links ]
5. Devaraja, B., Gangatirkar, P., Sunitha, S. N., Narayanaswamy, K., Karande, A., Muniyappa, V. y Savithri, H. S. (2004). Production of monoclonal antibodies to Tomato leaf curl Bangalore virus. Annals of Applied Biology, 144(3), 333-338. doi:10.1111/j.1744-7348.2004.tb00348.x. [ Links ]
6. Doyle, J. J. y Doyle, J. L. (1987). A rapid DNA isolation procedure for small amounts of fresh leaf tissue. Phytochemical Bulletin, 19, 11-15. [ Links ]
7. Fauquet, C. M., Briddon, R. W., Brown, J. K., Moriones, E., Stanley, J., Zerbini, M. y Zhou, X. (2008). Geminivirus strain demarcation and nomenclature. |-#91;journal article|-#93;. Archives of Virology, 153(4), 783-821. doi:10.1007/s00705-008-0037-6. [ Links ]
8. Fukuta, S., Kato, S., Yoshida, K., Mizukami, Y., Ishida, A., Ueda, J., Kanbe, M. y Ishimoto, Y. (2003). Detection of tomato yellow leaf curl virus by loop-mediated isothermal amplification reaction. J Virol Methods, 112(1-2), 35-40. [ Links ]
9. Gao, F., Jiang, J. -Z., Wang, J. -Y. y Wei, H. -Y. (2018). Real-time isothermal detection of Abalone herpes-like virus and red-spotted grouper nervous necrosis virus using recombinase polymerase amplification. journal of Virological Methods, 251, 92-98. doi:10.1016/j.jviromet.2017.09.024. [ Links ]
10. Givord, L., Fargette, D., Kounounguissa, B., Thouvenel, J. C., Walter, B. y Van Regenmortel, M. H. V. (1994). Detection of gemihiviruses from tropical countries by a double monoclonal antibody ELISA using antibodies to African cassava mosaic virus. Agronomie, 14, 327-333. [ Links ]
11. Hajimorad, M. R., Kheyr Pour, A., Alavi, V., Ahoonmanesh, A., Bahar, M., Rezaian, M. A. y Gronenborn, B. (1996). Identification of whitefly transmitted tomato yellow leaf curl geminivirus from Iran and survey of its distribution with molecular probes. Plant Pathology, 45, 418-425. [ Links ]
12. Inoue-Nagata, A. K., Lima, M. F. y Gilbertson, R. L. (2016). A review of geminivirus diseases in vegetables and other crops in Brazil: current status and approaches for management. Horticultura Brasileira, 34, 8-18. [ Links ]
13. Kapoor, R., Srivastava, N., Kumar, S., Saritha, R. K., Sharma, S. K., Jain, R. K. y Baranwal, V. K. (2017). Development of a recombinase polymerase amplification assay for the diagnosis of banana bunchy top virus in different banana cultivars. |-#91;journal article|-#93;. Archives of Virology, 162(9), 2791-2796. doi:10.1007/s00705-017-3399-9. [ Links ]
14. Kim, J. Y. y Lee, J. -L. (2017). Development of a multiplex real-time recombinase polymerase amplification (RPA) assay for rapid quantitative detection of Campylobacter coli and jejuni from eggs and chicken products. Food Control, 73(Part B), 1247-1255. doi:10.1016/jrfoodcont.2016.10.041. [ Links ]
15. Kushwaha, N., Singh, A. K., Chattopadhyay, B. y Chakraborty, S. (2010). Recent advances in geminivirus detection and future perspectives. The Journal of Plant Protection Sciences, 2(1), 1-18. [ Links ]
16. Laguna, I. G., Fiorona, M. y RodrÍguez Pardina, P. (2009, 9-15 de agosto). First report of Euphorbia mosaic virus-Peru a Begomovirus, infecting soybean crops in Argentina. Paper presented at the VIII Soybean Research Conference, Beinjing. China. [ Links ]
17. Law, I. L. G., Loo, J. F. C., Kwok, H. C., Yeung, H. Y., Leung, C. C. H., Hui, M., Wu, S. Y., Chan, H. S., Kwan, Y. W., Ho, H. P. y Kong, S. K. (2018). Automated real-time detection of drug-resistant Mycobacterium tuberculosis on a lab-on-a-disc by Recombinase Polymerase Amplification. Analytical Biochemistry, 544, 98-107. doi:10.1016/j.ab.2017.12.031. [ Links ]
18. Lobato, I. M. y O'Sullivan, C. K. (2018). Recombinase polymerase amplification: Basics, applications and recent advances. TrAc Trends in Analytical Chemistry, 98, 19-35. doi:10.1016/j.trac.2017.10.015. [ Links ]
19. Londoño, M. A., Harmon, C. L. y Polston, J. E. (2016). Evaluation of recombinase polymerase amplification for detection of begomoviruses by plant diagnostic clinics. |-#91;Article|-#93;. Virology Journal, 13(1). doi:10.1186/s12985-016-0504-8. [ Links ]
20. Mekuria, T. A., Zhang, S. y Eastwell, K. C. (2014). Rapid and sensitive detection of Little cherry virus 2 using isothermal reverse transcription-recombinase polymerase amplification. journal of Virological Methods, 205, 24-30. doi:10.1016/j.jviromet.2014.04.015. [ Links ]
21. Piepenburg, O., Williams, C. H., Stemple, D. L. y Armes, N. A. (2006). DNA Detection Using Recombination Proteins. PLoS Bio, 4(7), e204. doi:10.1371/journal.pbio.0040204. [ Links ]
22. Potter, J. L., Nakha, M. K., Mejia, L. y Maxwell, D. P. (2003). PCR and DNA hybridization methods for specific detection of bean-infecting begomoviruses in the Americas and Caribbean. Plant Disease, 87(10), 1205-1212. [ Links ]
23. Rivas Platero, G. G. y Lastra, R. (1993). Detección no radiactiva de geminivirus en tomate mediante hibridación de ácidos nucleicos. Manejo Integrado de Plagas, 30, 7-10. [ Links ]
24. RodrÍguez Pardina, P., Hanada, K., Laguna, I. G., Zerbini, F. y Ducasse, D. (2011). Molecular characterization and relative incidence of bean- and soybean-infecting begomoviruses in northwestern Argentina. Annals of Applied Biology, 158(1), 69-78. [ Links ]
25. Rojas, M. R., Gilbertson, R. L., Russell, D. R. y Maxwell, D. P. (1993). Use of degenerate primers in the polymerase chain reaction to detect whitefly-transmitted geminiviruses. Plant Disease, 77, 340-347. [ Links ]
26. Silva, G., Bomer, M., Nkere, C., Lava Kumar, P. y Seal, S. E. (2015). Rapid and specific detection of Yam mosaic virus by reverse-transcription recombinase polymerase amplification. journal of Virological Methods, 222, 138-144. doi:10.1016Zj.jviromet.2015.06.011. [ Links ]
27. Valasevich, N. y Schneider, B. (2017). Rapid detection of "Candidatus Phytoplasma mali" by recombinase polymerase amplification assays. Journal of Phytopathology, 165(11-12), 762-770. doi:10.1111/jph.12616. [ Links ]
28. Varela, G., Avalos, V., Laguna, I. G. y RodrÍguez Pardina, P. (2016, 7-10 de noviembre). Identification and molecular characterization of begomoviruses infecting bean crops in Northwestern Region of Argentina (NOA). Paper presented at the 8th International Geminivirus Symposium & the 6th International ssDNA Comparative Virology Workshop, New Delhi, India. [ Links ]
29. Wang, J., Liu, L., Wang, J., Pang, X. y Yuan, W. (2018). Real-time RPA assay for rapid detection and differentiation of wild-type pseudorabies and gE-deleted vaccine viruses. Analytical Biochemistry, 543, 122-127. doi:10.1016Zj.ab.2017.12.012. [ Links ]
30. Zerbini, F. M., Briddon, R. W., Idris, A., Martin, D. P., Moriones, E., Navas-Castillo, J., Rivera-Bustamante, R., Roumagnac, P., Varsani, A. y Consortium, I. R. (2017). ICTV Virus Taxonomy Profile: Geminiviridae. Journal of General Virology, 98(2), 131-133. doi:10.1099/jgv.0.000738. [ Links ]
31. Zhang, S., Ravelonandro, M., Russell, P., McOwen, N., Briard, P., Bohannon, S. y Vrient, A. (2014). Rapid diagnostic detection of plum pox virus in Prunus plants by isothermal AmplifyRP® using reverse transcription-recombinase polymerase amplification. journal of Virological Methods, 207, 114-120. doi:10.1016/j.jviromet.2014.06.026. [ Links ]














