Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
InVet
versión On-line ISSN 1668-3498
InVet v.11 n.1 Ciudad Autónoma de Buenos Aires ene./jun. 2009
ARTÍCULO DE REVISIÓN
Receptores similares a Toll en peces: el inicio de la divergencia
Rondón-Barragán1, IS, MVZ, CMSC
1Grupo de Inmunología y Fisiopatología Animal, Universidad del Tolima, Departamento de Sanidad Animal, Facultad de Medicina Veterinaria y Zootecnia. Santa Helena, Ibagué, Tolima. Fax: (57)2652506. Email: iangrondon@gmail.com
Recibido: 12/08/09
Aceptado: 16/02/10
Resumen
La inmunidad innata utiliza mecanismos de basados en el reconocimiento no clonal de componentes microbianos, denominados PAMPs, mediante el uso de receptores de reconocimiento de patrón genéticamente codificados (e.g. receptores similares a toll). Los PAMPs son estructuras esenciales para la fisiología y supervivencia de los microbios. Los receptores similares a Toll son una familia de receptores de transmembrana tipo I, con un dominio rico en leucina aminoterminal y una cola intracelular carboxiterminal; los cuales son expresados en diversas células incluyendo neutrófilos, macrófagos, células dendríticas, entre otras. Las vías de activación incluyen reclutamiento de proteínas citoplasmáticas adaptadoras que activan kinasas y factores asociados al factor de necrosis tumoral, conllevando a la activación de factores nucleares y a la producción de citoquinas y expresión de moléculas coestimuladoras de superficie en las células presentadoras de antígenos. El conocimiento acerca de los receptores similares a Toll permite el entendimiento de la dinámica de la respuesta inmune así como los procesos involucrados en el reconocimiento de los agentes microbianos. Esta revisión se enfoca en la identificación de los diversos receptores similares a Toll en peces, sus funciones e interacciones con otros así como la homología e identidad entre animales.
Palabras clave: Inmunidad innata; Peces; Receptores similares a Toll; Reconocimiento molecular.
Toll-like receptors in fish: the beginning divergence
Summary
Innate immunity initiates mechanisms based on the non-clonal recognition of microbial components, so-called PAMPs by using a variety of gene-encoded pattern recognition receptors (e.g. toll-like receptors). PAMPs are essential structures for the physiology and survival of the microbes. Toll-like receptors are transmembrane receptors family type I, with an aminoterminal leucin-rich repeat domain and a carboxiterminal intracellular tail; which are expressed in cells such as neutrophils, macrophages, dendritic cells, as well as others. Activation pathways including the recruitment of cytoplasmic adaptor proteins that activate kinases and tumor necrosis factor- associated factors, lead to the activation of nuclear factors and the cytokine production and surface co-stimulatory molecules expression in antigen-presenting cells. The knowledge about Toll-like receptor allows both the understanding of immune response dynamic and the processes involved in the recognition of microbial agents. This review focuses on the identification of several fish Toll-like receptor, them functions and them interactions with others as well as the homology and identity in animals.
Keywords: Innate immunity; Fish; Toll-like receptors; Molecular recognition.
1. Introducción
El sistema inmune se comporta como un "supersistema", similar al sistema nervioso, y se caracteriza por generar sus propios elementos de un progenitor único, autoregula sus funciones a través de procesos de adaptación (selección), establece un autoreconocimiento molecular y determina su propio comportamiento y posee una relación bidireccional con otros sistemas y tejidos48, 74. La inmunidad innata inicia los mecanismos de defensa inmediatos basados en reconocimiento no clonal de los componentes microbianos (7) usando una variedad de receptores de reconocimiento de patrón codificados genéticamente, los cuales reconocen estructuras microbianas conservadas o patrones moleculares asociados al patógeno (PAMPs) 6. Los PAMPs son productos internos del metabolismo microbiano que son conservados en la evolución, redistribuidos en una amplia variedad de patógenos y no hallados en células del hospedero 23.
Además, los PAMPs son estructuras esenciales para la fisiología, replicación y sobrevivencia de los microorganismos, por lo cual el ataque del sistema inmune es efectivo y poco susceptible de ser inactivado por fenómenos de selección o evolución 48. Los PAMPs, así como los ligandos endógenos (i.e. moléculas de colágeno, fibronectina, entre otras), son reconocidos por una clase de moléculas inmunosensoras llamadas receptores de reconocimiento de patrón (PRR), los cuales son parte de amplios mecanismos homeostáticos que permiten a los organismos multicelulares mantener un ambiente interno constante, en términos del control de los agentes patógenos 17, 62, 76. Los PRR incluyen receptores similares a Toll (TLR), receptor del complemento tipo-3 (CR3), receptores scavengers (SR), Dectina-1 (receptores de â-glucano) 21, 44, lectina de unión a manano (MBL), proteína C-reactiva (CRP), proteína amiolide sérica (SAP) 36, CD14 72 y proteínas de dominio de oligomerización unidas a nucleótidos (Nod) 7.
2. Receptores similares a Toll (TLR)
La proteína Toll está presente en los invertebrados y es conservada en vertebrados superiores (incluyendo los peces) así como en plantas 33. Los receptores relacionados con Toll son caracterizados por un dominio intracitoplasmático de 150 aminoácidos llamado TIR (dominio homólogo Toll/IL-1R), dominios que son compartidos con los miembros de receptores de la familia de la Interleuquina 1 (IL-1) y los genes de enfermedad en plantas 33, 39 y es un módulo conservado involucrado con interacciones bimoleculares que son necesarias para la traducción de las señales intracelulares 65. Sin embargo, la interacción de los ectodominios de los PAMPs han evolucionado más rápidamente que la estructura de los TIR, lo cual podría haber inducido cambios en la afinidad y especificidad del ligando entre especies y presumiblemente refleje la demanda de un ambiente microbiano altamente cambiante 65. Por otro lado, aunque el receptor de la IL-1 (IL-1R) y los TLRs tienen diferentes dominios extracelulares, sus dominios TIR le permiten a ambos receptores activar vías de transducción de señales similares 6, compartiendo de esta manera rutas metabólicas y codificando respuestas conservadas que son efectivas y redundan en una mayor eficiencia de la respuesta.
Los TLRs son una familia de receptores huérfanos (i.e. la porción C-terminal es intracelular y la porción N-terminal es extracelular con un solo dominio de transmembrana), de transmembrana tipo I, caracterizados por pesos moleculares entre 90- 115 kD 12, 53. Estos poseen tanto dominios de repetición ricos en leucina (LRR) amino terminales y una cola intracelular carboxiterminal con una región conservada llamada dominio de homología de receptor Toll/ Interleuquina-1 6. Los dominios LRR tienen una flexibilidad conformacional y se caracterizan por poseer hojas â que pueden participar en interacciones hidrofóbicas fuertes 40. Mientras en mamíferos los receptores TLR contienen un solo grupo de motivos de cisteína C-terminal flanqueando el extremo C-terminal en los LRRs yuxtapuestos de la membrana plasmática, la mayoría de los Tolls de Drosophila contienen dos o más grupos, incluyendo el motivo Nterminal 33. Por otro lado, los factores antimicrobianos codificados genéticamente en insectos muestran motivos flujo arriba de elementos de respuesta de fase aguda conocidos por unirse a factores de transcripción del NF-kB en mamíferos 68. De otra parte, se ha descrito que las interacciones entre los PAMPs y los TLRs, depende principalmente de inserciones aminoacídicas u otras irregularidades en la superficie convexa del LRR, estas modificaciones permiten una mayor diversidad de reconocimiento por parte los TLRs 12.
Los TLRs poseen funciones inmunes de respuesta frente a los PAMPs similar a lo encontrado en invertebrados (33), no obstante, en la Drosophila las proteínas Toll participan además en la formación embrionaria entre otros procesos 14, 45, 54, 77. En invertebrados, la proteína Toll no reconoce directamente los PAMPs sino que función como un receptor de citoquinas y no como un receptor de reconocimiento de patrón 33.
2.1. Sitios de expresión
Los miembros de la familia de los TLR son expresados por células involucradas en la defensa del hospedero, incluyendo neutrófilos, macrófagos, células dendríticas, células epiteliales mucosas, células B, células T 6, células trofoblásticas (expresando TLR2 y TLR4) 1, plaquetas 18; 70 y células megacariocíticas (TLR1 y TLR6) 70.
A nivel celular y tisular, los TLRs poseen diferentes sitios de expresión, y este sitio depende del tipo de PAMP que ellos reconocen (Figura 1). Algunos estudios han clasificado algunos TLRs de acuerdo con su patrón de expresión en: ubicuos (TLR1), restringidos (TLR2, TLR4 y TLR5), y específicos (TLR3) 53. Recientemente, han sido divididos en dos grupos basados en su localización subcelular: TLR1, 2, 4, 5 y 6 los cuales residen en la membrana celular, mientras los TLR3, 7, 8 y 9 son intracelulares y realizan la señalización desde un endosoma/ lisosoma 47.
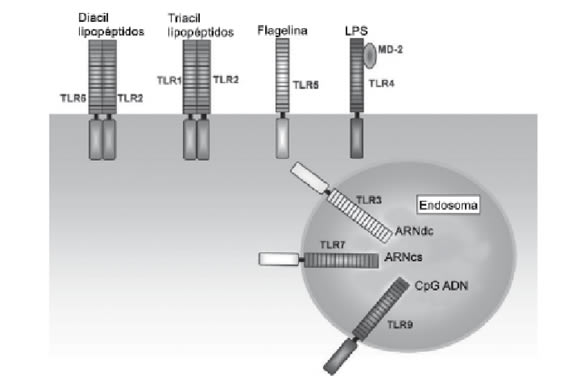
Figura 1. Sitios celulares de expresión de TLRs. El reconocimiento de los diferentes PAMPs requiere mas de un TLR o la presencia de otras moléculas como la proteína MD-2 en el reconocimiento del LPS.
Por ejemplo, los fagosomas que contienen TLR2 son capaces de discriminar entre bacterias gram positivas y gram negativas. Estos procesos son importantes debido a la expresión de TLR en los compartimentos intracelulares que les permite expresarse en sitios donde la bacteria reside y responde a la infección 43. En otros estudios, Shiraki et al. 70 argumentan que la diferenciación de los megacariocitos a plaquetas es modulada por la expresión de TLRs. Además, la expresión de TLR5 ha sido observada en la zona lateral más no en la apical de la superficie de las células epiteliales intestinales. Esta forma de expresión puede constituir una estrategia a través de la cual el hospedero solo responde a microorganismos que lo invaden a través de la barrera superficie mucosa 39 evitando una respuesta exagerada por sobre-estimulación con la flora bacteriana normal.
En goldfish (Carasius auratus), aparentemente sanos, los análisis de RT-PCR mostraron que tejidos como el bazo y el riñón, pero no el corazón o el hígado, expresan niveles detectables de TLR 72. Estas diferencias de niveles están relacionadas con la necesidad de reactividad de ciertos tejidos, la cual debe ser menor en órganos vitales.
La expresión de TLR puede ser también un importante indicador de la aparición temprana de macrófagos u otras células presentadoras de antígeno 37. Jault et al., 37 detectaron TLR en zebrafish (Danio rerio) al estado de 6-8 somitas (12 horas postfertilización), lo cual corresponde al periodo de formación de los progenitores de macrófagos 28, sin embargo, algunas moléculas de la cascada de respuesta (e.g. proteína adaptadora MyD88) no están presentes aun, por lo cual los TLRs deben usar otra vía de respuesta.
Athman & Philpott 7 demostraron que el TLR4 en las células epiteliales de la cripta intestinal es expresado en el aparato de Golgi. En un contexto fisiológico, los PAMPs luminales podrían inducir una redistribución dinámica de los TLRs en los compartimientos intracelulares para mantener un fenotipo hiporreactivo frente a la microflora 7, 60 asegurando una respuesta sesgada contra patógenos (los cuales invaden/atacan células epiteliales). De las misma manera, Muzio et al. 52 sugirieron que la expresión de TLR4 y TLR2 puede ser regulada diferencialmente en los sitios de infección/inflamación, directamente por componentes bacterianos o indirectamente por citoquinas.
Las expresión del TLR5S (TLR soluble)de catfish (bagre de canal, Ictalurus punctatus) fue detectado predominantemente en el riñón craneal, piel, hígado, ovario, cerebro y branquias 10, similar a lo descrito en los tejidos del Takifugu rubripes 55. El TLR5S de la trucha arco iris (Onchorhynchus mykiss) solo fue detectada en el hígado 79.
Los ligandos del TLR7 y TLR9 son incorporados en el endosoma temprano, donde el TLR7 y el TLR9 se mueven desde el retículo endoplásmico - RE 39. En el pez lenguado japonés (Paralichthys olivaceus), el gen del TLR9 fue marcado fuertemente en sangre periférica, branquias, intestino, riñón, bazo y estómago y débilmente expresado en cerebro, corazón, hígado, musculo y piel 75. La expresión de TLR9 en salmón (Salmo salar) fue demostrada en la mayoría de los tejidos, sin embargo, el hígado evidenció los niveles más bajos 71.
El TLR20 y TLR21 del catfish se expresaron en riñón craneal, bazo, intestino, estómago, hígado, riñón caudal, cerebro y branquias. El TLR21 además detectó en músculo, siendo similar al comportamiento registrado en Takifugu 55. De otro lado, en trucha arcoíris, el TLR22 y TLR22L son expresados predominantemente en tejidos inmunes relevantes tales como el bazo, riñón craneal y riñón caudal 64. Sin embargo, Rebl et al. 64 demostraron que la cantidad de mARN declina en casos de estimulación por patógenos.
Aunque la influencia hormonal sobre la expresión de TLR no ha sido descrita en peces; en humanos, Aflatoonian et al., 4 detectaron genes tlr7-10 y proteínas TLR7- 10 a través de inmunotinción, en tejido endometrial humano. Además, ellos demostraron la existencia de al menos una diferencia relativa en la expresión de estos genes de al menos dos veces entre las fases secretoria y proliferativa del ciclo menstrual.
2.2. Homologías e identidades de TLRs. Mamíferos vs. Peces
La familia de los TLR comprende 13 TLR en mamíferos, 10 de los cuales son expresados en humanos 10. Las especificidades de los ligandos de muchos de los TLRs de mamíferos están aun sujetas a investigación. Se han descrito en la literatura homologías (identidades de secuencia) de algunos TLRs en animales domesticos (Tabla 1).
Tabla 1. Porcentajes reportados de homología entre los diferentes animales domésticos
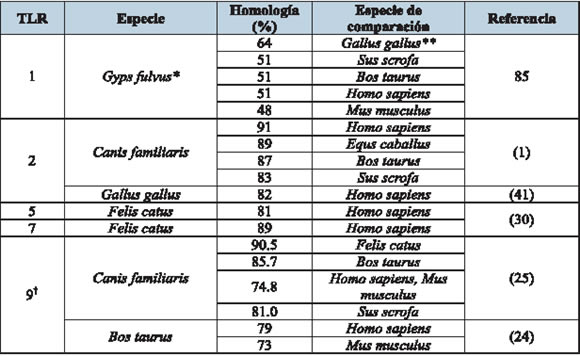
*Buitre leonado, no es una especie doméstica;** En aves a la fecha no existen reportes de ortólogos de TLR9 de mamíferos descritos 41. Sin embargo, ha sido demostrada la activación funcional de los heterófilos por CpG ODN (oligodeoxinucleótidos), el agonista especifico del TLR9 26. †Recientemente, una isoforma del TLR1 de ave (Ch-TLR1 tipo 2) fue identificada 85.
Hasta ahora, existen pocos estudios acerca de homologías entre los vertebrados superiores y los peces. El TLR3 del zebrafish (Danio rerio) muestra una alta similaridad al TLR3 humano con un 63 % de similaridad de aminoácidos en toda la secuencia de aminoácidos 59. De la misma manera, el TLR2 del catfish (Ictalurus punctatus) muestra una identidad de aminoácidos en la secuencia completa y los dominios TIR, de 60,2% y 82.2 % con el Zebrafish, 20,1% y 43,1% con la trucha arco iris, 46,6% y 65,8% con el Takifugu rubripes, 47,7% y 65,1% con el lenguado y de 40-42% y 70-72,4% con los mamíferos, respectivamente 9.
Los análisis filogenéticos de diferentes secuencias de proteínas TLR agruparon el gen del TLR9 del salmón así como el del TLR9 de otros teleósteos 71. Muchos de los sitios funcionales del TLR9 son conservados entre peces y mamíferos principalmente, un motivo de unión a CpG el cual consiste de ácido aspártico (Asp535) y tirosina (Tyr537) en el LRR17 del TLR9 murino reportado por Rutz et al. 69 es conservado en todas las secuencias de TLR9 de los peces 71. Además, fueron demostradas dos inserciones de pares de bases específicas en los LRRs de las secuencias de TLR9 de los peces 71, lo cual puede conferirle una especificidad de reconocimiento diferente.
En un estudio de Rebl et al., 64 los dendogramas filogenéticos muestran una ramificación específica de los peces junto con los TLRs de los mamíferos, en aquellos localizados dentro de la célula (TLR3, 7, 8 y 9), más que en los receptores de transmembrana.
Los estudios filogenéticos han guiado a proponer que los peces podrían ser capaces de responder a los agonistas de los TLR mamíferos y que la respuesta de los peces induciría producción de citoquinas diferentes de las observadas en animales domésticos 62. Así mismo, la radiación de los TLRs toma lugar en un ancestro común a todos los vertebrados, sugiriendo fuertemente que la evolución de las funciones de los diferentes TLRs en la respuesta inmune innata habría ocurrido en paralelo con la evolución de las funciones específicas del sistema inmune adaptativo de los vertebrados 49. Sin embargo, en contraste con los homeotermos, el sistema inmune adaptativo en peces es altamente dependiente de la temperatura del ambiente y no poseen cambios de clase de los anticuerpos más que generación de diferentes alotipos de IgM 83. El sistema de TLR de los peces ha mostrado diferencias comparado con el sistema de TLRs de los mamíferos: i) existe una forma soluble del ortólogo TLR5 en peces pero no en humanos, ii) no existe ortólogo de TLR4 en peces y iii) existen nuevos miembros de los TLRs llamados TLR21, TLR22 y TLR14 en peces 79. Así mismo, Jault et al., 37 demostraron que todos los ortólogos de los genes de TLR de mamíferos están presentes en el zebrafish (Danio rerio) y comparten la mismas características en estructura así como el zebrafish posee 19 variantes putativas de TLR y dos ortólogos de proteínas adaptadoras. Ellos han encontrado 5 ortólogos de TLR21, el cual parece formar una rama especial de la evolución, solo presente en peces pero completamente desaparecida en mamíferos. Además, ellos confirmaron que el TLR1, 6 y 10 tienen un ancestro común entre los mamíferos y los peces. Sumado a esto, los TLR7-9 han evolucionado de un ancestro común, proximal al TLR8. El TLR4, 1 y 7 del zebrafish presumiblemente existen como dos variantes splicing en los dominios TIR, permitiendo que un tipo de receptor pueda tener dos vías de señalización de la traducción 37, limitando la capacidad de inhibición de la respuesta inmune por parte de los agentes potencialmente patógenos así como incrementando las posibilidades de respuesta.
Recientemente, un grupo de TLRs han sido identificados sin ortólogos fácilmente discernible en los mamíferos. Estos incluyen los miembros del TLR21-23 del Tetraodon nigroviridis y Takifugu rubripes 55; 67 y un número de genes TLR 19-22 del Danio rerio 37; 49. Este grupo ha sugerido un posible receptor alternativo para el reconocimiento del LPS (en ausencia de TLR4) y puede estar relacionado con genes divergentes TLR11-13 en ratones 55; 67.
Baoprasertkul et al., 10 han identificado tres miembros de la familia de los TLRs en catfish que están ausentes en mamíferos: TLR5S, TLR20 y TLR21.
El TLR5S contiene 2185 bp (pares de bases) con una región no traducida-50 de 74 bp y una región traducida-30 de 167 bp (GenBank DQ529273). La traducción de la secuencia nucleótida de este TLR genera 647 aminoácidos y su secuencia es similar a la descrita para otros teleósteos. Carece de un dominio TIR y un dominio de transmembrana, como se ha descrito para los TLR5 solubles de otras especies 78; contiene 21 LRRs bien conservados. Sin embargo, el dominio Cterminal del LRR (LRR-CT) en el catfish muestra 4 residuos de cisteína conservados, al igual que en los mamíferos, y difiere de los demás peces en los cuales solo se conservan 2 residuos 10. Bell et al. 12 demostraron que los 4 residuos de cisteína son necesitados en el TLR5 de los mamíferos para mantener el dominio extracelular cerca a la membrana.
El TLR20 consiste de 2185 bp con una región no traducida-50 de 4o bp y una región no traducida-30 de 757 bp (GenBank DQ529275). La traducción de la secuencia nucleótida de este TLR genera 351 aminoácidos y su secuencia es similar a la descrita para el TLR20 de Danio rerio, la única especie para la cual ha sido informada.
El TLR21 contiene 3409 bp con una región no traducida-50 de 225 bp y una región no traducida-30 de 223 bp (GenBank DQ529277). La traducción de la secuencia de nucleótidos de este TLR genera 986 aminoácidos y su secuencia es similar a la descrita para otros teleósteos 10.
2.3. PAMPs y TLRs
Algunos TLRs son activos como complejos homoméricos, aunque ellos han demostrado formar heterodímeros, ampliando así su espectro de reconocimiento 33. Por ejemplo, el TLR2 en heterófilos de gallina requiere la cooperación del TLR6 para inducir la explosión respiratoria por estas células 41. Además, Ozinsky et al. 56 demostraron que los TLRs pueden establecer un repertorio de combinaciones para discriminar entre un gran número de PAMPs, independientemente de su naturaleza. Ellos demostraron que el TLR2 puede formar un par funcional con el TLR6 o el TLR1 y esto conlleva a la inducción de citoquinas; tambien se ha descrito la interacción del TLR2 con el TLR10 11. El CD36 ha demostrado ser requerido para las respuestas ante los lipopéptidos diacilados dependientes de TLR2/TLR6 29. El TLR9 requiere de la catepsina para inducir respuesta 47. Este proceso es denominado "hipótesis de código de barras" 3. Sin embargo, Kogut et al., 41 demostraron que el TLR2 puede responder tanto a lipoproteínas como a LPS (lipopolisacárido) (los cuales son reconocidos por dos TLR diferentes en mamíferos - TLR2 y TLR4); este fenómeno es probablemente una separación evolutiva. De esta manera, la identificación de TLRs y otros receptores de reconocimiento de patrón ha refutado el paradigma que la respuesta inmune innata es "no especifica" 30. Además del proceso de LRR que modifica la afinidad de unión al PAMP, algunos genes TLR están sujetos a splicing alternativo 65, lo cual facilitaría la diversidad molecular. Además, Bell et al., 12 propusieron que para todos los TLRs; las variaciones estructurales consecuencia del cambio de la secuencia de aminoácidos en el TLR proveen la especificidad necesaria para reconocer una lista creciente de PAMPs.
Los TLRs pueden asociarse con moléculas accesorias para mediar un reconocimiento eficiente y responder a los patrones moleculares microbianos. Wallin et al. 82 asociaron la función de las proteínas de choque térmico con las de unión al LPS y las proteínas de incremento en el reconocimiento que liberan LPS al complejo TLR-MD2 y CD14. Además, los TLRs pueden reconocer moléculas generadas por estrés o daño tisular de la matriz extracelular tales como proteoglicanos, acido hialurónico, fibronectina o péptidos antimicrobianos; por tanto, los TLR pueden ser activados sinérgicamente por moléculas derivadas de lo no propio pero también de lo propio dañado. Esto puede facilitar al sistema inmune distinguir entre los comensales inocuos y los microorganismos infecciosos que generan daño tisular 33. De la misma manera, algunos mediadores pueden participar en los diferentes pasos de las respuesta del TLR e.g. el interferón â fue demostrado que era requerido para la expresión de genes selectos que no podrían ser inducidos vía engranaje del TLR2, incluyendo sintasa inducible de óxido nítrico o la proteína inducible de interferón- IP10 6. En goldfish, la exposición al LPS de Aeromonas salmonicida atenuada con calor o Mycobacterium chelonei vivas puede sobre expresar TLRs en macrófagos en comparación con las células no tratadas, pero existe variación en la cinética de activación entre ellas 72. Meijer et al., 49 demostraron que el zebrafish sometido a la infección con micobacterias mostró unos niveles de sobre expresión del gen zTLR2 y los genes zTLR1 y zTLR18 que están estrechamente relacionados con el gen TLR1 humano. De otra parte, las respuestas mediadas por TLR también inducen la producción de anticuerpos tipo IgG1 los cuales son importantes para la opsonización de microbios para facilitar la toma por parte de los fagocitos y también para la activación del complemento 57. Este fenómeno no ha sido reportado en peces, los cuales poseen solo IgM e IgD 83.
El TLR4 puede reconocer el LPS, específicamente la forma hexa-acilada del lípido A el cual es un componente del LPS 61. En mamíferos, la activación del TLR4 genera la respuesta endotoxémica mediada por proteínas de unión al LPS y dispara en el citotrofoblasto del primer trimestre de gestación y en las células vellosas del trofoblasto una respuesta clásica de TLR (Abrahams & Mor, 2005). Los experimentos in vivo han sugerido que los peces son menos sensibles a la toxicidad del LPS que otras especies 13. Es posible que los TLR4 de los peces hayan evolucionado para reconocer estructuras del LPS que son distintas de aquellas de los patógenos de animales terrestres; factores séricos, tales como la proteína de unión al LPS pueden ser requeridas para el reconocimiento del LPS 62. Iliev et al., 31 demostraron que los fagocitos mononucleares de la trucha arco iris (Oncorhynchus mykiss) expuestas a bajas concentraciones de LPS no dependen de la presencia de factores séricos para la activación lo cual sugiere que podrían existir diferencias fundamentales entre el mecanismo de reconocimiento del LPS en peces y mamíferos. Las diferencias en la respuesta biológica al LPS entre los peces y los mamíferos están probablemente determinadas por diferencias en su reconocimiento mediado por receptor del LPS. Es decir, parece que las moléculas asociadas al TLR4 (CD14, LY96 (MD-2) y la TICAM2 (TRAM)) que participan en la respuesta mediada por el TLR4 en el fenómeno endotóxico en mamíferos, pueden estar ausentes o desarrollar diferentes funciones en peces 32. Este análisis sugiere que otro(s) PRR(s) tales como las beta-2 integrinas, un grupo bien conservadas filogenéticamente puede jugar un papel primario en el reconocimiento del LPS en el sistema inmune de los peces.
Por otro lado, un análisis de la familia TLR en el genoma del pufferfish (pez globo), Fugu rubripes, sugiere que el TLR4 está ausente en el linaje de los peces 55 acorde con otras hipótesis de diferentes autores que sugieren que los peces pueden carecer de proteína de unión al LPS funcional 73; 34. Sin embargo algunos autores argumentan que esta ausencia puede ser explicada por una perdida específica de este gen en el linaje del Fugu o por una falta de conocimiento de la secuencia del genoma del Fugu 49. En contraste, Jault et al., (2004) argumentan que los peces poseen dos homólogos de TLR4, probando que el TLR no ha desaparecido en peces, sino por el contrario muestran dos formas de expresión diferentes.
El TLR5 media el reconocimiento de la flagelina en los extremos N- y C-terminal que forman el centro hidrofóbico 36. Estudios in vivo sugieren que la flagelina ejerce propiedades similares a la endotoxina en peces 79 y puede inducir las citoquinas IL-1â2 pero no el IFN tipo 1 62. El TLR5 soluble de los peces genera una curva de retro alimentación positiva en respuesta a la flagelina guiando a una activación fuerte del NF-kB en células TLR5 positivas independiente de los tipos celulares y parece estar distribuida en una variedad de especies de peces 79. Las función del TLR5 soluble puede causar una alta susceptibilidad de los peces a la flagelina; y aunque los mamíferos carecen de TLR5 soluble, el TLR5 aun conserva la capacidad de aumentar la respuesta a la flagelina en concierto con el TLR5 soluble del pez; lo cual evidencia un prototipo de inmunidad humoral que esta conservada en los peces 79; este fenómeno abre nuevas opciones terapéuticas, siendo el TLR5 soluble de los peces un adyuvante para la activación del sistema inmune mediada por flagelina.
El TLR3 y TLR5 han demostrado estar involucrados en la respuesta inmune innata contra la Edwarsiella ictaluri en el bagre de canal 16. Bilodeau et al., 15 demostraron en este bagre inoculado con E. ictaluri, que el TLR5 es expresado en mayor proporción que el TLR3 en hígado y riñón. Así mismo, en el estomago el TLR3 evidenció una mayor expresión que el TLR5, demostrando que la expresión de TLR3 es inducida y de mayor expresión en el estómago que la del TLR5 en presencia de patógenos bacterianos.
Purcell et al., 62 demostraron la presencia de TLR7 y TLR8 en trucha arcoiris, mediante el uso de resiquimod® (R848), un agonista de estos receptores; aunque los genes TLR de la trucha no han sido identificados.
Mientras la respuesta inmune contra proteínas virales ha sido asociada con TLR2 y TLR4, los ácidos nucleicos virales pueden ser reconocidos por otras proteínas TLR 20. Por ejemplo, debido al hecho de que algunos virus ADN poseen motivos CpG demetilados, ellos son capaces de estimular las células a través del TLR9 20. Además, algunos virus, pueden usar la señalización TLR para disminuir la capacidad de la célula hospedera para responder frente a la infección 51, 63, 8.
El TLR9 tiene dos ligandos: los CpG de ADN comúnmente encontrados en las bacterias y los CpG de tipo D de ADN o CpGA, los cuales son menos comunes. Los CpGA tienen una potente habilidad para inducir la producción de IFNã, pero son débiles en activar células B y requieren altas concentraciones para inducir cantidades comparables de IL-12 que los CpG ADN 42, 81; 27, 35. Jorgensen et al., 38 demostraron que los motivos CpG inducen a los leucocitos de salmón y trucha a la producción de IL-1â y respuestas similares a IFN tipo 1. De la misma manera, Purcell et al., 62 mostraron que el TLR9 existe en la trucha arcoiris usando oligonucleótidos de CpG. Takano et al. 75 demostraron que el TLR9 en el lenguado japonés (Paralichthys olivaceus) tiene un dominio de unión al CpG-ADN que es similar a la secuencia en los dominios ricos en leucina (LRD) del TLR9 del ratón y contiene dos residuos de aminoácidos esenciales para la interacción con el CpG-ADN. Estos residuos de aminoácidos también son conservados en los LRD de los TLR9s del lenguado japonés (Asp562 y Tyr564), pez globo (Asp544 y Tyr546) y humano (Asp534 y Tyr536). Los mismos autores encontraron que el TLR9 del lenguado japonés también tiene dos motivos CXXC separados por seis residuos aminoácidos. Esta región se espera este unida directamente a las secuencias de dinucleótidos demetilados de CpG 12. La secuencia de aminoácidos de la proteína TLR9 del lenguado japonés comparte homología de secuencia con el humano (NP 059138), pez globo (AAW69377) y zebrafish (XP 691003) en 35.0%, 60.7% y 49.5%, respectivamente. El numero de las repeticiones ricas en leucina (LRRs) del TLR9 en el lenguado japonés, humano, pez globo y zebrafish son de 12, 18, 14 y 14, respectivamente.
No obstante, a pesar de estos resultados, An et al. 5 demostraron que la respuesta del TLR puede ser inespecífica, pues encontraron que los genes TLR9 sensibles a los CpG fueron inducidos siguiente a la estimulación con LPS de macrófagos murinos, indicado que los productos microbiales pueden también inducir otros TLRs más de los específicos para su propio reconocimiento. También, Takano et al., 75 demostraron un incremento marcado en la expresión de TLR9 en células de lenguado japonés después de una exposición a Edwardsiella tarda, in vivo e in vitro. De la misma manera, Phelan et al., 59 demostraron que el TLR3 del zebrafish, el cual está relacionado con el reconocimiento de patógenos virales en mamíferos, está involucrado en la respuesta inmune ante infecciones con E. tarda. Además, en estudios de Inoue & Aramakii 35 fue demostrado que el TLR9 puede ser expresado después de una injuria en la piel o trauma físico en ratones. Recientemente, trabajos de Skjæveland et al. 71 demostraron al IFN-ã como un inductor mayor de expresión de TLR9 que los CpG (tipo A y B) en salmón del atlantico, lo cual puede ser explicado como parte de un feedback positivo, necesario para respuestas inmunes dependientes de CpG en peces.
Rebl et al. 64 encontraron que el genoma de la trucha arcoíris codifica dos factores TLRs "gemelos" altamente relacionados generando dominios TIR altamente conservados. Allí, solo el 6.3% de los residuos de aminoácidos que establecen estos factores son encontrados intercambiados lo cual sugiere que su origen viene de un evento de duplicación bastante reciente. Estos hallazgos en salmónidos son esperados debido a que son poliploides. Estos autores encontraron un enriquecimiento altamente excéntrico de intercambios en la mitad N-terminal del dominio LRR el cual puede contribuir a las propiedades de unión diferencial al ligando de ambos factores. Además, ejemplo de factores TLR gemelos han sido encontrados en gallinas hasta ahora (TLR2, tipo 1 y 2 [41]), pero no han sido identificados en humanos o ratones, aun cuando sus genomas han sido totalmente secuenciados.
El TLR11 ha sido reconocido en perros, ratones, ratas, gallinas, pez fugu y en ranas 67. Así mismo, Yaronvinsky & Sher, 84 describen que el TLR11 reconoce al Toxoplasma gondii a través de la interacción con la profilina y que moléculas homologas a la profilina existen en un amplio rango de protozoos apicomplexa lo cual genera el interrogante de si los TLRs sirven como receptores de reconocimiento de patrón generales para una clase de organismos; y se ha especulado que la detección de moléculas con la función motil (e.g. polimerización de actina) es una estrategia general para el reconocimiento general de los patógenos móviles.
2.4. Vías de señalización
La señalización de los TLRs guía a la activación de los fagocitos y la eliminación directa de los agentes infecciosos. Además, la señalización de los TLRs en los macrófagos y las células dendríticas (DC) inducen la secreción de IL-12 que sesga la respuesta T resultante a través de un fenotipo Th1 66. Además, los procesos activados por los TLRs en las DC causan una sobrerregulación de las moléculas MHC cargadas con péptidos derivados del patógeno y la expresión de moléculas de superficie CD80 y CD86 57.
Aunque los linfocitos T y B expresan receptores de enorme diversidad, su activación depende de las señales derivadas del sistema inmune innato 57. El comprometimiento in vivo de los TLRs en las DCs es requerido para inducir la estimulación primaria de las células T a través de 2 mecanismos: inducción de moléculas co-estimuladoras en una interacción células T - células dendríticas emparentadas y por la inducción de IL-6 y otras citoquinas que permiten sobreponer los efectos supresivos de las células Tr (reguladoras) 57.
La señalización de los TLR proviene de un domino intracitoplasmático. La activación de la señalización a través de los dominios TIR resulta en el reclutamiento de proteínas adaptadoras citoplasmáticas MyD88 (proteína 88 de respuesta primaria de diferenciación mieloide) y TOLLIP (proteína de interacción Toll) 6. Meijer et al. 49 demostraron que el zebrafish y el Fugu contienen homólogos conservados de 4 proteínas adaptadoras de dominios TIR que han sido identificadas en humanos (MyD88, MAL, TRIF y SARM) sugiriendo que las vías de señalización empleadas por los diferentes TLRs y los receptores de interleuquina (IL-R) de mamíferos y teleósteos son probablemente altamente conservadas. La MyD88 es una proteína adaptadora al TIR que interactúa con los TLRs o IL-1Rs, reclutando las kinasas asociadas al IL-1R (IRAK) y el factor asociado al factor de necrosis tumoral (TRAF6) generando la activación del NFkB, necesario para la síntesis de citoquinas 39; 60. El MyD88 en peces tiene un porcentaje de identidad del 60% comparado con los tetrápodos 62.
Tras la unión del TLR, MyD88 es reclutada al complejo receptor y su dominio TIR Cterminal media su interacción homofílica con el receptor y el dominio de muerte N-terminal que compromete al dominio de la muerte de su blanco corriente abajo IRAK. El IRAK, subsecuentemente se fosforila, además, el RING-dedo contiene funciones adaptadoras TRAF6 como una ligasa de E3 a ubiquinato una hasta ahora no identificada, que es necesaria para la IkB kinasa â mediada por TLR/IL1R (IKK â activación). La IKK â fosforila y es blanco de degradación del inhibidor del NFkB, IkB, liberando de esta manera el NFkB 36 (Figura 2). En el genoma de zebrafish, Meijer et al. 49 identificaron homologías de cinco miembros de la familia de factores de transcripción NF-kB que son blancos centrales de la señalización de TLR en mamíferos, lo cual soporta su alta conservación.
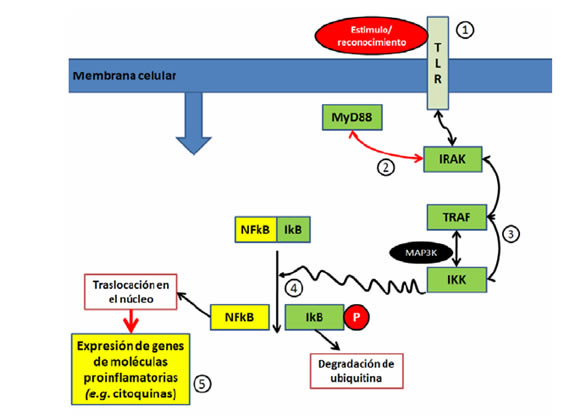
Figura 2. Esquema general de la cascada intracelular de la estimulación de TLRs (dependiente de la proteína de diferenciación mieloide 88 - MyD88).1) Se presenta el reconocimiento de PAMPs o estímulos externos por un TLR o una combinación de TLRs que genera una cascada de señalización intracelular, 2) bajo estimulación, la MyD88 recluta la kinasa asociada al receptor de interleukina 1 (IRAK) a través de la interacción de los dominios de muerte de ambas moléculas. El IRAK es activado por la fosforilación y se asocia con el factor asociado al receptor del factor de necrosis (TRAF), el TRAF6; 3) la oligomerización del TRAF6 activan la proteína kinasa activada por mitógeno (MAP3K), la cual indirectamente genera la activación de la kinasa IkB (IKK) 4) la IKK es capaz de fosforilar los residudos de serina de la IkB para su degradación y liberación del factor nuclear kappa B (NFkB) y 5) la activación del NFkB estimula la expresión de genes de moléculas proinflamatorias (citoquinas).
Además, el TLR activa también al factor de transcripción AP1 guiando a la producción de citoquinas inflamatorias, tales como el factor de necrosis tumoral á (TNF á ) y la interleuquina 6 (IL-6), y a la sobrerregulación de la moléculas co-estimulatorias CD80 y CD86 en las células dendríticas 36.
Adicionalmente, la activación de los TLRs sobre las células presentadoras de antígenos (APC) induce la producción de citoquinas y la expresión de moléculas coestimulatorias de superficie, ayudando en la elaboración de la respuesta inmune adaptativa 19. La señalización por TLR activa principalmente a las células dendríticas a producir citoquinas inductoras de respuestas tipo Th-1 tales como la IL-12 o IL-18, instruyendo a las células T a diferenciarse a células Th1. De la misma manera, los TLRs pueden inducir la liberación de interferón tipo 1 (á/â) 39.
El TLR es capaz de modular la respuesta inmune innata por diferentes vías. Mann et al., 46 hipotetizaron que el TLR4 regula la salida temprana de productos de genes que son vitales para la respuesta inmune innata contra la Bordetella bronchiseptica, e.g. TNFá. Por otra parte, los TLRs pueden inducir la presentación cruzada, involucrando el escape de los antígenos de las vías endosomales al citosol; este escape ocurre antes de la acidificación 19. Los TLR3 y TLR9 son capaces de directamente activar células dendríticas a la presentación cruzada de antígenos soluble a las células T CD8+ 19. La señalización de TLR9 es dependiente de pH y su localización en los endosomas es crítica para su función 71.
La respuesta de los TLRs puede mediar procesos apoptóticos. La apoptosis mediada por el TLR2 ocurre a través de la activación de las caspasas y el inicio de su vía intracelular depende del reclutamiento de dominios de muerte asociados a Fas (FADD) por parte de la MyD88; y la apoptosis mediada por el TLR3 ocurre en una manera dependiente de FADD, pero independiente de la MyD88 1. Goshima et al. 22 demostraron que el LPS disminuye la apoptosis de neutrófilos principalmente a través del TLR2 mas no por el TLR4, los factores asociados a la apoptosis A1, Mcl-1, Bcl-Xl y Bak no parecen jugar un papel importante en este proceso. Por otro lado, los ligandos del TLR2, el ácido lipoteicoico, peptidoglucano y el LPS, pueden inducir apoptosis de células trofoblásticas 1; 2.
Una hipótesis que puede explicar la observación de que los componentes de las vías de señalización de los TLRs estuvieron presentes como simples copias en las especies de vertebrados hasta la fecha, es que puede existir una presión de selección que evita los excesos en los estados inflamatorios que pueden estar asociados con una dosis genética incrementada. Los grandes cambios en la presión de selección en el tiempo y entre los subfilums tienden a invalidar la mayoría de los modelos de la evolución de las proteínas que son usados para computar las distancias moleculares 62.
3. Conclusiones y perspectivas
Los TLRs pueden ser divididos en dos grupos principales: los TLRs 1, 2, 4, 5 y 6 los cuales reconocen componentes microbiales y los TLRs 3, 7, 8 y 9 los cuales reconocen ácidos nucleicos de bacterias o virus. Sin embargo, otros TLRs (e.g. TLR12 y TLR13) han sido identificados solo muy recientemente y su expresión espacial no ha sido caracterizada aun.
La identificación de TLRs mamíferos y de peces tiene un gran impacto en nuestro entendimiento de las patogénesis microbianas y de la regulación del sistema inmune. Además, permite el desarrollo de moléculas (sintéticas o naturales) como adyuvantes o herramientas terapéuticas. El conocimiento de las características de los TLRs, homologías y diferencias, entre los humanos y animales (vías de reconocimiento y activación) permitirán el desarrollo de modelos experimentales en medicina veterinaria y humana.
Por otro lado, la determinación de las interrelaciones entre los TLRs y moléculas/ accesorias puede permitir el entendimiento de la dinámica del algunas respuestas, e.g. enfermedades autoinmunes o enfermedades emergentes.
La investigación de las formas de TLRs en peces puede abrir puertas a estrategias terapéuticas para la profilaxis humana y animal. Adicionalmente, el entendimiento de la función de los TLRs en peces y sus especificidades de patógenos puede guiar al desarrollo de mejores inmunoestimulantes para el uso en acuicultura comercial así como agonistas pertinentes para el control y prevención de enfermedades, lo cual es nulo en especies de cultivo en el país.
Por último, el estudio de los genes relacionados con las formas cortas y largas del TLR20 pueden aclarar los mecanismos de reconocimiento de PAMPs a través de los vertebrados.
1. Abrahams, VM.; Mor, G. Toll-like receptors and their role in the trophoblast. Placenta. 2005; 26:540-547. [ Links ]
2. Abrahams, VM.; Straszewski, SI.; Romero, R.; Mor, G. Toll-like receptor 2 induces apoptosis in trophoblast Cells: a novel mechanism for first-trimester pregnancy Failure. Am J Obstet Gynecol. 2003; 189: S147. [ Links ]
3. Aderem A. Phagocytosis and the inflammatory response. J Infect Dis. 2003; 187:S340-S345. [ Links ]
4. Aflatoonian, R.; Tuckerman, E.; Elliott, SL.; Bruce, C.; Aflatoonian, A.; Li, TC.; Fazeli, A. Expression of toll-like receptors in endometrium during the menstrual cycle. J Reprod Immunol. 2006; 71:152. [ Links ]
5. An, H.; Xu, H.; Yu, Y.; Zhang, M.; Qi, R.; Yao, X.; Liu, S.; Wang, W.; Guo, Z.; Qin, Z.; Cao, X. Up-regulation of TLR9 gene expression by LPS in mouse macrophages via activation of NFkappaB, ERK and p38 MAPK signal pathways. Immunol Lett. 2002; 81:165-169. [ Links ]
6. Armant, MA.; Fenton, MJ. Toll like receptors: a family of pattern recognition receptors in mammals. Genome Biol. 2002; 3:Reviews 301.1-3011.6 [ Links ]
7. Athman, R.; Philpott, D. Innate immunity via Toll-like receptors and Nod proteins. Curr Opin Microbiol. 2004; 7:25-32. [ Links ]
8. Bafica, A.; Scanga, CA.; Schito, M.; Chaussabel, D.; Sher, A. Influence of coinfecting pathogens on HIV expression: evidence for a role of Toll-like receptors. J Immunol. 2004; 172:7229- 7234. [ Links ]
9. Baoprasertkul, P.; Peatman, E.; Abernathy, J.; Liu, Z. Structural characterization and expression analysis of Toll-like receptor 2 gene from catfish. Fish & Shellfish Immunol. 2007; 22:418-426 [ Links ]
10. Baoprasertkul, P.; Xu, P.; Peatman, E.; Kucuktas, H.; Liu, Z. Divergent Toll-like receptors in catfish (Ictalurus punctatus): TLR5S, TLR20, TLR21. Fish & Shellfish Immunol. 2007; 23:1218- 1230. [ Links ]
11. Bazzocci, C.; Montarino, M.; Comazzi, S.; Bandi, C.; Francheschi, A.; Genchi, C. Expression and function of Toll-like receptor 2 in canine blood phagocytes. Vet Immunol Immunopathol. 2005; 104:15-19. [ Links ]
12. Bell, JK.; Mullen, GED.; Leifer, CA.; Mazzoni, A.; Davies, DR.; Segal, DM. Leucine-rich repeats and pathogen recognition in Toll-like receptors. Trends Immunol. 2003; 24:528-533. [ Links ]
13. Berezi, I.; Bertok, T.; Bereznai, T. Comparative studies on the toxicity of Escherichia coli lipopolysaccharide endotoxin in various animal species. Can J Microbiol. 1966; 12:1070- 1071. [ Links ]
14. Bilak, H.; Tauszig-Delamasure, S.; Imler, JL. Toll and Toll-like receptors in Drosophila. Biochem Soc Trans. 2003; 31:648- 651. [ Links ]
15. Bilodeau, AL.; Peterson, BC.; Bosworth, BG. Response of tolllike receptors, lysozyme, and IGF-I in back-cross hybrid (F1 male (blue x channel) x female channel) catfish challenged with virulent Edwarsiella ictaluri. Fish & Shellfish Immunol. 2006; 20:29-39. [ Links ]
16. Bilodeau, AL.; Waldbieser, GC. Activation of TLR3 and TLR5 in channel catfish exposed to virulent Edwarsiella ictaluri. Develop Comp Immunol. 2005; 29:713-721. [ Links ]
17. Boehme, KW.; Compton, T. Innate sensing of viruses by tolllike receptors. J Virol. 2004; 78:7867-7873. [ Links ]
18. Cognasse, F.; Hamzeh, H.; Chavarin, P.; Acquart, S.; Genin, C.; Garraud, O. Evidence of Toll-like receptor molecules on human platelets. Immunol Cell Biol. 2005; 83:196-198. [ Links ]
19. Datta, SK.; Raz, E. Induction of antigen cross-presentation by Toll-like receptors. Springer Semin Immunol. 2005; 26:247-255. [ Links ]
20. Finberg, RW.; Kurt-Jones, EA. Virus and toll-like receptors. Microbes Infect. 2004; 61:1356-1360. [ Links ]
21. Gordon, S. Pattern Recognition receptors: Doubling up for the innate immune response. Cell. 2002; 111:927-930. [ Links ]
22. Goshima, M.; Kotani, J.; Lowry, SF.; Calvano, SE. LPS-induced decrease of neutrophil apoptosis is regulated through TLR2, not through TLR4. J Am Coll Surg. 2004; 199:38. [ Links ]
23. Govind, S.; Nahm, RH. Innate Immunity in fruit flies: a textbook example of genome recycling. PLoS Biol. 2004; 2:e276. [ Links ]
24. Griebel, PJ.; Brownlie, R.; Manuja, A.; Nichani, A.; Mookherjee, N.; Popowych, Y.; Mutwiri, G.; Hecker, R.; Babiuk, LA. Bovine toll-like receptor 9: A comparative analysis of molecular structure, función and expression. Vet Immunol Immunopathol. 2005; 108:11-16. [ Links ]
25. Hashimoto, M.; Asahina, Y.; Sano, J.; Kano, R.; Moritomo, T.; Hasegawa, A. Cloning of canine toll-like receptor 9 and its expression in dog tissues. Vet Immunol Immunopathol. 2005; 106:159-163. [ Links ]
26. He, H.; Crippen, TL.; Farnell, MB.; Kogut, MH. Identification of CpG oligodeoxynucleotide motifs that stimulate nitric oxide synthase and cytokine production in avian macrophage and peripheral blood mononuclear cells. Comp Dev Immunol. 2003; 27:621-627. [ Links ]
27. Hemmi, H.; Takeuchi, O.; Kawai, T.; Kaisho, T.; Sato, S.; Sanjo, H.; Matsumoto, M.; Hoshino, K.; Wagner, H.; Takeda, K.; Akira, S. A Toll-like receptor recognizes bacterial DNA. Nature. 2000; 408:740-745. [ Links ]
28. Herbomel, P.; Thisse, B.; Thisse, C. Ontogeny and behaviour of early macrophages in the zebrafish embryo. Development. 1999; 126:3735-3745 [ Links ]
29. Hoebe, K.; Georgel, P.; Rutschmann, S.; Du, X.; Mudd, S.; Crozat, K.; Sovath, S.; Shamel, L.; Hartung, T.; Zahringer, U.; Beutler, B. CD36 is a sensor of diacylglycerides. Nature. 2005; 433:523-527. [ Links ]
30. Ignacio, G.; Nordone, S.; Howard, KE.; Dean, GA. Toll-like receptor expression in feline lymphoid tissues. Vet Immunol Immunopathol. 2005; 106:229-237 [ Links ]
31. Iliev, DB.; Liarte, CQ.; MacKenzie, S.; Goetz, FW. Activation of rainbow trout (Oncorhynchus mykiss) mononuclear phagocytes by different pathogen associated molecular pattern (PAMP) bearing agents. Mol Immunol. 2005; 42:1215-1223. [ Links ]
32. Iliev, DB.; Roach, JC.; Mackenzie, J.; Planas, JV.; Goetz, FW. Endotoxin recognition: In fish or not in fish?. FEBS Letters. 2005; 579:6519-6528. [ Links ]
33. Imler, JC.; Zheng, L. Biology of Toll receptors: Lessons from insects and mammals. J Leukoc Biol. 2003; 74:000-000. [ Links ]
34. Inagawa, H.; Honda, T.; Kohchi, C.; Nishizawa, T.; Yoshiura, Y.; Nakanishi, T.; Yokomizo, Y.; Soma, G-I. Cloning and characterization of the homolog of mammalian lipopolysaccharide-binding protein and bactericidal permeabilityincreasing protein in rainbow trout Oncorhynchus mykiss. J Immunol. 2002; 168:5638-5644. [ Links ]
35. Inoue, J.; Aramaki, Y. Toll-like receptor-9 expression induced by tape-stripping triggers on effective immune response with CpG-oligodeoxinucleotides. Vaccine. 2007; 25:1007-1013. [ Links ]
36. Janeway, CA.; Medzhitov, R. Innate immune recognition. Ann Rev Immunol. 2002; 20:197-216. [ Links ]
37. Jault, C.; Pichon, L.; Chluba, J. Toll-like receptor gene family and TIR-domain adapters in Danio rerio. Mol Immunol. 2004; 40:759-771. [ Links ]
38. Jørgensen, JB.; Johansen, A.; Stenersen, B.; Sommer, A-I. CpG oligodeoxynucleotides and plasmid DNA stimulate Atlantic salmón (Salmo salar L.) leucocytes to produce supernatants with antiviral activity. Dev Comp Immunol. 2001; 25:313-321. [ Links ]
39. Kaisho, T.; Akira, S. Pleiotropic función of Toll-like receptors. Microbes Infect. 2004; 6:1388-1394. [ Links ]
40. Kobe, B.; Deisenhofer J. A structural basis of the interactions between leucine-rich repeats and protein ligands. Nature. 1995; 374:183-186. [ Links ]
41. Kogut, MH.; Iqbal, M.; He, H.; Philbin, V.; Kaiser, P.; Smith, A. Expression and function of Toll-like receptors in chicken heterophils. Dev Comp Immunol. 2005; 29:791-807. [ Links ]
42. Krug, A.; Rothenfusser, S.; Hornung, V.; Jahrsdorfer, B.; Blackwell, S.; Ballas, ZK.; Endres, S.; Krieg, AM.; Hartmann, G. Identification of CpG oligonucleotide sequences with high induction of IFN alpha/beta in plasmacytoid dendritic cells. Eur J Immunol. 2001; 31:2154-2163. [ Links ]
43. Krutzik, S.; Modlin, R. The toll-like receptors in combating mycobacteria. Semin Immunol. 2004; 16:35-41. [ Links ]
44. Levitz, S. Interactions of Toll-like receptor with fungi. Microbes Infect. 2004; 6:1351-1355 [ Links ]
45. Luo, C.; Shen, B.; Manley, JL.; Zheng, L. Tehao functions in the Toll pathway in Drosophila melanogaster: possible roles in development and innate immunity. Insect Mol Biol. 2001; 10:457-464. [ Links ]
46. Mann, PB.; Elder, K.; Kennet, MJ.; Harvill, ET. Toll-like receptor 4-dependent early elicited tumor necrosis factor alpha expression is critical for innate host defense against Bordetella bronchiseptica. Infect Immun. 2004; 72:6650-6658. [ Links ]
47. Matsumoto, F.; Saitoh, S-I.; Fukui, R.; Kobayashi, T.; Tanimura, N.; Konno, K.; Kusumoto, Y.; Akashi-Takamura, S.; Miyake, K. Cathepsins are required for Toll-like receptor 9 responses. Biochem Biophys Res Comm. 2008; 367:693-697. [ Links ]
48. Medina, ME.; Patiño, PJ. Moléculas, células y acciones del sistema inmune. In: Rugeles, MT.; Patino, PJ. Edits. Inmunología: Una ciencia activa. Tomo I. Fondo Editorial Biogénesis, Colombia. 2004. p. 17-35. [ Links ]
49. Meijer, AH.; Gabby, KS.; Medina, IA.; Ile, S.; Bitter, W.; Snaar- Jagalska, BE.; Spaink, HP. Expression analysis of the Toll-like receptor and TIR domain adaptor families of zebrafish. Mol Immunol. 2004; 40:773-783. [ Links ]
50. Menzies, M.; Ingham, A. Identification and expression of Tolllike receptors 1-10 in selected bovine and ovine tissues. Vet Immunol Immunopathol. 2006; 109:23-30. [ Links ]
51. Monick, MM.; Yarovinsky, TO.; Powers, LS.; Butler, NS.; Carter, AB.; Gudmundsson, G.; Hunninghake, GW. Respiratory syncytial virus up-regulates TLR4 and sensitizes airway epithelial cells to endotoxin. J Biol Chem. 2003; 278:53035-53044. [ Links ]
52. Muzio, M.; Bosisio, D.; Polentarutti, N.; D'amico, G.; Stopacciaro, A.; Mancinelli, R.; van't Veer, C.; Penton-Rol, G.; Ruco, LP.; Allavena, P.; Mantovani, A. Differential expression and regulation of Toll like receptors (TLR) in human leukocytes; selective expression of TLR3 in dendritic cells. J Immunol. 2000; 164:5998- 6904. [ Links ]
53. Muzio, M.; Mantovani, A. The Toll receptor family. Allergy. 2001; 56:103-108. [ Links ]
54. Ooi, JY.; Yagi, Y.; Hu, X.; Ip, YT. The Drosophila Toll-9 activates a constitutive antimicrobial defense. EMBO Rep. 2002; 3:82- 87. [ Links ]
55. Oshiumi, H.; Tsujita, T.; Shida, K.; Matsumoto, M.; Ikeo, K.; Seya, T. Prediction of the prototype of the human Toll-like receptor gene family from the pufferfish, Fugu rubripes, genome. Immunogenetics. 2003; 54:791-800. [ Links ]
56. Ozinsky, A.; Underhill, DM.; Fontenot, JD.; Hajjar, AM.; Smith, KD.; Wilson, CB. The repertoire for pattern recognition of pathogens by innate immune system is defined by cooperation between Toll-like receptors. Proc Natl Acad Sci USA. 2000; 97:13766-13771. [ Links ]
57. Pasare, C.; Medzhitov, R. Toll-like receptors and acquired immunity. Semin Immunol. 2004; 16:23-26. [ Links ]
58. Pérez, JM.; De la Fuente, J. 2007, Molecular cloning and characterization of the griffon vulture (Gyps fulvus) toll-like receptor 1. Dev Comp Immunol. 2007; 31:511-519. [ Links ]
59. Phelan, PE.; Mellon, MT.; Kim, CH. Functional characterization of full-length TLR3, IRAK-4, and TRAF6 in zebrafish (Danio rerio). Mol Immunol. 2005; 42:1057-1071. [ Links ]
60. Philpott, DJ.; Girardin, SE. The role of Toll-like receptors and Nod proteins in bacterial infection. Mol Immunol. 2004; 41:1099- 1108. [ Links ]
61. Portnoy, DA. Manipulation of innate immunity by bacterial pathogens. Curr Opin Immunol. 2005; 17:25-28. [ Links ]
62. Purcell, MK.; Smith, KD.; Aderem, A.; Hood, L.; Winton, JR.; Roach, JC. Conservation of Toll-like receptor signalling pathways in teleost fish. Comp Biochem. Physiol. Part D. 2006; 1:77-88. [ Links ]
63. Rassa, JC.; Meyers, JL.; Zhang, Y.; Kudaravalli, R.; Ross, SR. Murine retroviruses activate B cells via interaction with toll-like receptor 4. Proc Natl Acad Sci USA. 2002; 99:2281-2286. [ Links ]
64. Rebl, A.; Siegl, E.; Köllner, B.; Fischer, W.; Seyfert, H-M. Characterization of twin toll-like receptors from rainbow trout (Oncorhynchus mykiss): Evolutionary relationship and induced expression by Aeromonas salmonicida salmonicida. Dev Comp Immunol. 2007; 31:499-510. [ Links ]
65. Rehli, M. Of mice and men: species variation of toll-like receptor expression. Trends Immunol. 2002; 23:375-378. [ Links ]
66. Reis e Sousa, C. Toll-like receptors and dendritic cells: for whom the bug tolls. Semin Immunol. 2004; 16:27-34. [ Links ]
67. Roach, JC.; Glusman, G.; Rowen, L.; Kaur, A.; Purcell, MK.; Smith, KD.; et al. The evolution of vertebrate Toll-like receptors. Proc Natl Acad Sci USA. 2005; 102:9577-9582. [ Links ]
68. Rock, FL.; Hardiman, G.; Timans, JC.; Kastelein, RA.; Bazan, F. A family of human receptors structurally related to Drosophila Toll. Proc Natl Acad Sci USA. 1998; 95:588-593. [ Links ]
69. Rutz, M.; Metzger, J.; Gellert, T.; Luppa, P.; Lipfford, G.; Wagner, H.; et al. Toll-like receptor 9 binds single-stranded CpG DNA in a sequence- and pH-dependent manner. Eur J Immunol. 2004; 34:2541-50 [ Links ]
70. Shiraki, R.; Inoue, N.; Kawasaki, S.; Takei, A.; Kadotani, M.; Ohnishi, Y.; Ejiri, J.; Kobayashi, S.; Hirata, K-I.; Kawashima, S.; Yokoyama, M. Expression of toll-like receptor on human platelets. Thromb Res. 2004; 113:379-385. [ Links ]
71. Skjæveland, I.; Iliev, D.; Zou, J.; Jørgensen, T.; Jørgensen, J. A TLR9 homolog that is up-regulated by IFN-ã in Atlantic salmon (Salmo salar). Dev Comp Immunol. 2008; 32:603-607. [ Links ]
72. Stafford, JL.; Ellestad, KK.; Magor, KE.; Belosevic, M.; Magor, BG. A toll-like receptor (TLR) gene that is up-regulated in activated goldfish macrophages. Dev Comp Immunol. 2003; 27:685-698. [ Links ]
73. Stenvik, J.; Solstad, T.; Strand, C.; Leiros, I.; Jorgensen, TT. Cloning and analyses of a BPI/LBP cDNA of the Atlantic cod (Gadus morhua L.). Dev Comp Immunol. 2004; 28:307-323. [ Links ]
74. Tada, T. The immune system as a supersystem. Ann Rev Immunol. 1997; 15:1-13 [ Links ]
75. Takano, T.; Kondo, H.; Hirono, I.; Endo, M.; Saito-Taki, T.; Aoki, T. Molecular cloning and characterization of Toll-like receptor 9 in Japanese flounder, Paralichthys olivaceus. Mol Immunol. 2007; 44:1845-1853. [ Links ]
76. Takeuchi, O.; Akira, S. Genetic approaches to the study of Tolllike receptor function. Microbes Infect. 2002; 4:887-895. [ Links ]
77. Tauszig, S.; Jouanguy, E.; Hoffmann, JA.; Imler, JL. Toll related receptors and the control of antimicrobial peptide expression in drosophila. Proc Natl Acad Sci USA. 2002; 97:10520-10525. [ Links ]
78. Tsoi, S.; Park, KC.; Kay, HH.; O'Brien, TJ.; Podor, E.; Sun, G.; et al. Identification of a transcript encoding a soluble form of tolllike receptor 5 (TLR5) in Atlantic salmon during Aeromonas salmonicida infection. Vet Immunol Immunopathol. 2006;109:183-187 [ Links ]
79. Tsujita, T.; Ishii, A.; Tsukada, H.; Matsumoto, M.; Che, F-S.; Seya, T. Fish soluble toll-like receptor (TLR)5 amplifies human TLR5 response via physical binding to flagellin. Vaccine. 2006; 24:2193-2199. [ Links ]
80. Tsujita, T.; Tsukada, H.; Nakao, M.; Oshiumi, H.; Matsumoto, M.; Seya, T. Sensing bacterial flagellin by membrane and soluble orthologs of toll-like receptor 5 in rainbow trout (Oncorhynchus mykiss). J Biol Chem. 2004; 279:48588-45597. [ Links ]
81. Verthelyi, D.; Ishii, KJ.; Gursel, M.; Takeshita, F.; Klinman, DM. Human peripheral blood cells differentially recognize and respond to two distinct CpG motifs. J Immunol. 2001; 166:2372-2377. [ Links ]
82. Wallin, RPA.; Lundqvist, A.; Moré, SH.; von Bonin, A.; Kiessling, R.; Ljunggren, HG. Heat shock proteins as activators of the innate immune system. Trends Immunol. 2002; 23:130-135. [ Links ]
83. Watts, M.; Munday, BL.; Burke, CM. Immune response of teleost fish. Aust Vet J. 2001; 79:570-574. [ Links ]
84. Yarovinsky, F.; Sher, A. Toll-like receptor recognition of Toxoplasma gondii. Int J Parasitol. 2006; 36:255-259. [ Links ]
85. Yilmaz, A.; Shen, S.; Adelson, DL.; Xavier, S.; Zhu, JJ. Identification and sequence analysis of chicken toll-like receptors. Immunogenetics. 2005; 56:743-53. [ Links ]














