Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO  uBio
uBio
Share
InVet
On-line version ISSN 1668-3498
InVet vol.18 no.2 Ciudad Autónoma de Buenos Aires Dec. 2016
ARTÍCULO DE INVESTIGACIÓN
Virus de Distemper Canino: detección molecular de diferentes aislamientos provenientes de perros de la provincia de santa fe, argentina, entre los años 2000 y 2010
Pinotti, M.1; Gollan, A.1; Canavesio, M.1; Passeggi, C.1; Larrateguy, M.V.1; Paz, M.E.1; Formentini, E.2
Universidad Nacional del Litoral, Facultad de Ciencias Veterinarias,
1Cátedra de Microbiología.
2Cátedra de Farmacología.
Correspondencia e-mail: Mario Andrés Pinotti mpinotti@fcv.unl.edu.ar
Recibido: 10/01/2016
Aceptado: 01/09/2017
Resumen
En la ciudad de Santa Fe, Argentina, durante los años 2000 y 2010, se aislaron 13 cepas autóctonas del virus Distemper Canino. Las muestras se tomaron de caninos con signos clínicos de la enfermedad y consistieron en hisopados nasales, oculares, faríngeos (tonsilas) y conjuntivales. Los aislamientos se realizaron en células MDCK, las cuales mostraron efecto citopatogénico característico. Los virus provenientes de la cosecha, se detectaron por RT-PCR de una región que codifica la nucleoproteína NP asociada al RNA (posición 769 a la 1055 del genoma) obteniéndose en todos los casos un producto de 287 pb comparable con el obtenido con cepas vacunales (cepas Ondersterpoort y Lederle), lo que indica presencia viral en todas las muestras procesadas. Las cepas aisladas se sometieron a pruebas de neutralización en células MDCK con el anticuerpo monoclonal frente a la proteína H y sólo 6 de ellas fueron neutralizadas. Este hallazgo sugiere la incapacidad de las cepas vacunales para proteger a los caninos frente a la circulación de algunas cepas virales autóctonas.
Palabras clave: Virus; Distemper canino; Estudio molecula; RT-PCR.
Canine distemper virus; molecular detection of isolates in Santa Fe, Argentina during the years 2000-2010
Summary
In the city of Santa Fe, Argentina, during years 2000 and 2010, 13 wild type strains of canine distemper virus were isolated. All the samples were obtained from dogs with clinical signs of disease. Isolations were performed in MDCK cells which shown characteristic cytopathogenic effect. Identification of the wild type strains was performed by RT-PCR of a region that encodes NP nucleoprotein associated to RNA. The amplified product was a 287-bp corresponding to positions 769-1055 of the Onderstepoort and Lederle vaccine strains genome, which confirmed presence of the virus in all the samples. The isolate strains were tested by neutralization on MDCK cells with the monoclonal antibody against protein H, and only 6 of them were neutralized. This finding suggests the inability of the vaccine strains to protect the canines against the circulation of some wild type strains.
Key words: Virus; Canine distemper; Molecular study; RT-PCR.
Introducción
El Distemper Canino fue descripto por Edward Jenner en 1809 y su etiología fue demostrada por Carré en 1906. Es una enfermedad infecto-contagiosa causada por un agente viral del orden Mononegavirales, familia Paramyxoviridae, género Morbillivirus, que produce una infección sistémica con altos índices de morbilidad y apreciable letalidad, principalmente en animales jóvenes y desprotegidos inmunológicamente1. El período de mayor susceptibilidad se da entre los tres y los seis meses de vida, aunque pueden enfermar individuos de todas las edades6,7.
El virus de Distemper Canino (VDC) posee un ARN monocatenario de polaridad negativa, con nucleocápside helicoidal, rodeada de una envoltura lipoproteica que contiene proteínas como Hemaglutinina (H), de Fusión (F) y de Matriz (M), las que participan en los mecanismos de infección, replicación viral y determinan el tropismo. Mundialmente se conoce un solo serotipo. No obstante, varios estudios realizados en distintas latitudes demuestran la existencia de cepas con diferentes genotipos que a su vez difieren de las cepas vacunales5.
La enfermedad reviste dos presentaciones clínicas: una temprana, con compromiso respiratorio, entérico, cutáneo y nervioso agudo; y una tardía, con desarrollo de signos neurológicos por lesiones inmunomediadas1. El virus tiene la particularidad de ser linfotrópico y altamente inmunosupresor. Se lo indica también como agente causal de la encefalitis de perros viejos, una rara encefalomielitis de los perros maduros9. El diagnóstico de esta patología suele ser clínico, con auxilio de métodos complementarios.
Los métodos específicos incluyen el aislamiento en cultivo de líneas celulares como MDCK (Madin-Darby canine kidney) y VERO (Kidney from African Green monkey) y la Inmunofluorescencia Directa (IFD). Pruebas más sensibles son el Enzimainmunoensayo (ELISA) y la Reacción en Cadena de la Polimerasa con Transcriptasa reversa (RT-PCR), existiendo en la actualidad métodos de RT-PCR que detectan diferencialmente cadenas de cepas salvajes y cepas vacunales de manera efectiva4.
Aunque se trata de una enfermedad controlada con vacunas vivas atenuadas, se reportan brotes de distemper canino en todo el mundo5. Por nuestra parte, hemos reportado datos epidemiológicos regionales7. En la actualidad es motivo de preocupación la descripción de casos en individuos vacunados3, lo que podría indicar diferencias entre las cepas circulantes y las cepas vacunales utilizadas.
El objetivo del presente trabajo fue caracterizar genéticamente cepas de campo regionales de virus de Distemper Canino, aisladas a partir de muestras tomadas de perros con diagnóstico clínico de la enfermedad, en la ciudad Santa Fe y zona de influencia de la Facultad de Ciencias Veterinarias de la Universidad Nacional del Litoral (FCV-UNL), Argentina, entre los años 2000 y 2010 y compararlas con cepas vacunales utilizadas en el país. .
Materiales y Métodos
Muestras clínicas
Las muestras provinieron de 29 caninos con diagnóstico clínico de Distemper Canino procedentes de la ciudad de Santa Fe y de la zona de influencia de la Facultad de Ciencias Veterinarias de la Universidad Nacional del Litoral (Argentina) entre los años 2000 y 2010. De estos animales se obtuvieron hisopados nasales, oculares, faríngeos (tonsilas) y conjuntivales en forma estéril. El material así obtenido se extrajo por presión, acondicionándolo en tubos con 0,5 ml de medio de transporte con el agregado de antibiótico-antimicótico. Las muestras fueron procesadas a 4ºC y se centrifugaron por 15 min a 2000 r.p.m. Los sobrenadantes fueron controlados bacteriológica y micológicamente en agar sangre y en agar ogy en condiciones de aerobiosis y microaerofilia durante cinco días.
Aislamientos virales en líneas celulares
Se utilizaron líneas celulares establecidas MDCK, (pasajes 121 y sucesivos) certificadas y conservadas en nitrógeno líquido. Las mismas se obtuvieron del Banco de Células Argentino (ABAC). Luego de descongelarse se sembraron en botellas T25 para cultivos celulares empleando D-MEM sin aminoácidos esenciales y 10% de suero fetal bovino (SFB), con Hepes comercial GIBCO al 1% y antibiótico - antimicótico comercial GIBCO al 1% (100000 U/ml de penicilina, 10000 ug de estreptomicina y 25 μg de Fungizona), manteniéndolas en estufa a 37ºC hasta obtener monocapas de 100% de confluencia.
Las monocapas celulares se inocularon con 0,2 ml del sobrenadante de las 29 muestras y se dejaron una hora a 37ºC para permitir la adsorción del virus. Previamente se descartó el medio de crecimiento y se realizaron tres lavados con buffer fosfato salino pH7 (PBS). Se completó el volumen del frasco con D-MEM sin SFB. Cada capa fue observada diariamente realizándose la cosecha de los sobrenadantes de cultivos en el momento de la aparición del efecto citopatogénico (ECP) o en su defecto a los 7-10 días de incubación a 37 ºC. El material proveniente de las 21 muestras que presentaron ECP se recolectó en viales individuales, conservándolos a -20°C hasta la realización de las restantes pruebas. En todos los casos se realizaron tres pasajes consecutivos hasta considerar al material como negativo.
RT-PCR
La RT-PCR se realizó sobre 13 muestras de cultivos celulares elegidas al azar entre las 21 que produjeron ECP. Como controles positivos se utilizaron 4 vacunas comerciales a virus atenuados conteniendo las cepas de referencia (Lederle 1-2010, Lederle 2-2011, Onderstepoort 1-2010 y Onderstepoort 2-2011). Estos materiales provenían de lotes comerciales diferentes. Los controles negativos internos se obtuvieron a partir de los líquidos sobrenadantes de los cultivos de células MDCK sin infectar.
La extracción del RNA se realizó a partir de 150 μL de los respectivos sobrenadantes o vacunas utilizando RNAzol® (Genbiotech), siguiendo las instrucciones del fabricante. Los primers utilizados para la amplificación de un sector del genoma que codifica la NP (desde la posición 769 a la 1055) fueron descriptos previamente por otros autores2,3. El producto final de amplificación fue de 287 pb.
Para la RT-PCR se utilizó el kit QIAGEN OneStep RT-PCR y un termociclador TECNE modelo TC3000 partiendo de 5 μL del producto de extracción de RNA con el agregado de 1 μL de RNAsin. El programa utilizado fue: un paso de 50°C durante 30 min seguido por un paso de 95ºC de 15 min. Luego 35 ciclos a 94ºC de 1 min, 60ºC de 1 min, 72ºC de 1 min y un paso de extensión final de 72ºC durante 5 min. La reacción se realizó en un volumen de 50 μL.
Los productos obtenidos de la amplificación de controles positivos (cepas vacunales) y controles negativos (células no infectadas) se revelaron por electroforesis en gel de agarosa al 2%, en buffer TBE (Promega), con el agregado de 1x de SBYR Gold nucleic acid gel stain®, durante 50 min a 75 V y 400 mA. Para la visualización de los productos de amplificación de las cepas se realizó el mismo procedimiento.
Neutralización
Las pruebas de microseroneutralización de la glicoproteína H se realizaron en policubetas de 96 pocillos con monocapas de línea MDCK, en su variante suero constante/virus variable. Se utilizó el reactivo comercial VMRD: MoAb- 1 C 42 H 11-IgG 1 que neutraliza el componente H de envoltura de las cepas Rockborn, Snyder Hill y Onderstepoort. Se descartó el medio de crecimiento y se lavaron las células con solución buffer PBS estéril, y se efectuaron diluciones. Luego se tomaron 100 μl de las diluciones 10-1 a 10-5, tituladas por el método de Reed y Muench, a las que se les adicionó 100 μL del anticuerpo monoclonal en dilución 1:20. De esta mezcla se colocaron 50 μL en pocillos, se incubaron durante 60 min a 37º C y posteriormente se adicionaron 100 μL de D-MEM sin SFB. Este procedimiento se realizó por duplicado y durante siete a diez días se observaron y registraron los resultados. La presencia de virus sin neutralizar fue revelada por ECP.
Resultados
A fin de obtener un orden en la identificación de las cepas aisladas, éstas se denominaron en base a criterios tentativos teniendo en cuenta que no existe una estandarización en la nomenclatura de las cepas conocidas de VDC. En la Figura 1 puede observarse que el fragmento 287 pb fue claramente puesto de manifiesto en las cepas vacunales utilizadas como controles positivos Lederle (1 2010) y (2 2011) y Onderstepoort (1 2010) y (2 2011). El mismo estuvo ausente en los dos controles negativos.

Figura 1. Electroforesis en gel de agarosa al 2% de los productos de RT-PCR obtenidos a partir de cepas vacunales para la obtención de controles positivos y negativos de referencia. La distribución de las muestras en la corrida electroforética es la siguiente: Calle 1: Vacuna Lederle (1-2010); Calle 2: Vacuna Lederle (2-2011); Calle 3: Vacuna Onderstepoort (1-2010); Calle 4: Vacuna Onderstepoort (2-2011); Calle 5: Control (-); Calle 6: Control (-); Calle 7: Vacío; Calle 8: Control de PM (cada banda tiene un valor de 100 pb
En la figura 2, puede observarse que el fragmento 287 pb fue evidenciado en las trece cepas estudiadas y que resultó idéntico en peso molecular al fragmento obtenido. Dicho fragmento estuvo ausente en los controles negativos
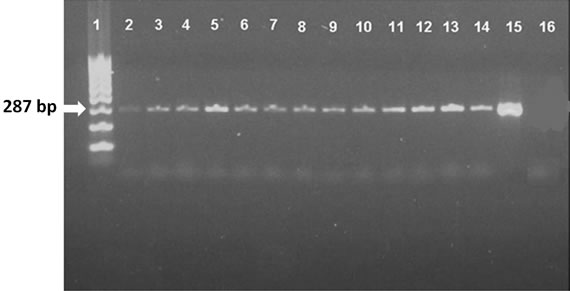
Figura 2. Corrida electroforética de RT-PCR (Región NP- Posición 769-1055) de 13 cepas autóctonas de virus Distemper canino aisladas en la ciudad de Santa Fe, Argentina, durante los años 2000 y 2010. La distribución de las muestras es la siguiente: 1-Marcador de PM (100 pbP); 2-CDV/Esperanza-SFE-ARG/58/2000; 3-CDV/Esperanza-SFEARG/ 104/2003; 4-CDV/Esperanza-SFE-ARG/127/2000; 5-CDV/Esperanza-SFEARG/ 264/2001; 6-CDV/Esperanza-SFE-ARG/268/2001; 7-CDV/Esperanza-SFEARG/ 279/2001; 8-CDV/Esperanza-SFE-ARG/284/2001; 9-CDV/Esperanza-SFEARG/ 289/2001; 10-CDV/Esperanza-SFE-ARG/458/2001; 11-CDV/Esperanza-SFEARG/ 516/2005; 12-CDV/Esperanza-SFE-ARG/579/2006; 13-CDV/Esperanza-SFEARG/ 605/2004; 14-CDV/Esperanza-SFE-ARG/632/2004; 15-Control(+)-Vacuna 1-2011- Cepa Onderstepoort; 16-Control (-).
En las pruebas de Neutralización (NT) en células MDCK de las 13 cepas con el anticuerpo monoclonal frente a la proteína H, se evidenció que solo 6 de ellas pudieron ser neutralizadas (Tabla 1).
Tabla 1. Resultados obtenidos en las pruebas de neutralización de 13 cepas autóctonas de virus de Distemper Canino con anticuerpo monoclonal frente a la proteína H. Las cepas virales fueron aisladas en la ciudad de Santa Fe, Argentina,entre los años 2000 y 2010. Las pruebas se realizaron en células MDCK.

Discusión y conclusiones
En el presente estudio se tomaron 13 cepas del VDC aisladas en células MDCK que mostraron ECP, a las que se les efectuaron pruebas moleculares mediante el método de RT-PCR, confirmándose de este modo los aislamientos. En las mismas se detectó que el fragmento amplificado de 287 pb fue del mismo peso molecular que los controles de las cepas vacunales de referencia8. Si bien no pudo realizarse la secuenciación, el hecho de que algunas cepas no hayan sido neutralizadas sugiere la presencia de variabilidad antigénica como consecuencia de una variabilidad genética de las cepas autóctonas respecto de las cepas vacunales.
El kit QUIAGEN one step RT-PCR y el funcionamiento del termociclador han sido optimizados para su empleo por los fabricantes, por lo que se descarta la presencia de bandas inespecíficas debidas a fallas en la concentración de Cl2Mg y/o temperatura de hibridización. Por otra parte, el kit mencionado utiliza una Hot-Star Taq DNA polimerasa, lo que reduce considerablemente la formación de dímeros de primers. La no aparición de bandas inespecíficas en el control negativo (Fig. 1 calles 5 y 6) indica que no existe contaminación en los reactivos que pudieran dar lugar a la su formación.
Las muestras para la extracción de RNA para los controles positivos (Fig. 1) se tomaron directamente del envase original comercial. Es aquí donde aparecen bandas inespecíficas en las calles 1, 3 y 4. La calle 2, muestra una concentración menor del producto amplificado de 287 pb, no detectándose bandas inespecíficas. Las mismas no aparecen en ninguna de las calles de la Fig. 2 cuyas muestras para la extracción de RNA se tomaron del sobrenadante de cultivos celulares.
La aparición de esas bandas inespecíficas se atribuye a la amplificación de restos de RNA presente en los viales de vacunas comerciales, debido a la conservación de las mismas.
Las modificaciones halladas en las cepas circulantes podrían sugerir la existencia de mecanismos de evasión de la protección inmunitaria otorgada por las vacunas. En diferentes trabajos alrededor del mundo han sido demostradas claras diferencias a nivel antigénico y genético entre las cepas vacunales más utilizadas y las cepas de campo, con aparición de infecciones en animales vacunados3.
Trabajos complementarios, permitirán estudiar la expresión de las proteínas virales tanto in vitro como in vivo. De esta manera se podrá evaluar la capacidad protectora de las vacunas y eventualmente sugerir modificaciones en la composición de las mismas.
1. Appel, M.; Summers, B. Canine Distemper: Current Status. Recent advances in Canine Infectious Diseases.1999; (www.ivis.org) [ Links ]
2. Frisk, A.L.; Konig, M.; Moritz, A., Baumgartner, W. Detection of canine distemper virus nucleoprotein RNA by reverse transcription-PCR using serum, whole blood, and cerebrospinal fluid from dogs with distemper. J. Clin. Microbiol. 1999; 37: 3634-3643. [ Links ]
3. Gallo Calderón, M; Remorini, P.; Periolo, O; Iglesias, M.; Mattion, N.; La Torre, J. Detection by RT-PCR and genetic characterization of canine distemper virus from vaccinated and non-vaccinated dogs in Argentina. Veterinary Microbiology. 2007; 125: 341-349. [ Links ]
4. Li Yi, S.; Hongli, X.; Jianke, W.; Yuening, CH.; Shen, Y.; Bin, L. Development of a combined canine distemper virus specific RT-PCR protocol for the differentiation of infected and vaccinated animals (DIVA) and genetic characterization of the hemagglutinin gene of seven Chinese strains demonstrated in dogs. Journal of Virological Methods. 2012; 179: 281-287. [ Links ]
5. Martella, V.; Martella, G.; Elía, G.; Buonavoglia, A. Canine Distemper virus. Vet .Clin. SINort AmSmall Animal Practice. 2008; 38: 787-797. [ Links ]
6. Morales, M.; Mora, L.; Salazar, J. Distemper canino: sobrevida por edad, sexo raza y estación. Avances en Ciencias Veterinarias. 1997; 12, Nº1. [ Links ]
7. Pinotti, M.; Gollán, A.; Passeggi, C.; Formentini, E.: Aspectos clínicos y epidemiológicos del distemper canino. Estudio de casos diagnosticados en la ciudad de Santa Fe, entre los años 1998 y 2009. Rev. FAVE. Sec. Cs. Vet. ISSN 1666-938X (Esperanza, Arg.). 2012; 11, Nº 1 y 2: 83-94. [ Links ]
8. Scagliarini, A; Battilani, M.; Ciulli, S.; Prósperi ,S..; Morganti, L. Molecular analysis of the NP Gene of Italian CDV Isolates. Veterinary Research Communications. 2000; 27 Suppl. 1: 355-357. [ Links ]
9. Vandevelde, B.; Kristensen, K..; Brand, C.; Greene, L.; Hoerlein, B. Chronic Canine distemper virus encephalitis in mature dogs. Veterinary Pathology. 1980; 17: 17-29. [ Links ]














