Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
RIA. Revista de investigaciones agropecuarias
versión On-line ISSN 1669-2314
RIA. Rev. investig. agropecu. vol.40 no.1 Ciudad Autónoma de Buenos Aires abr. 2014
ARTÍCULOS
Identificación de genes codificantes de enzimas de interés industrial en una cepa de bacteria termofílica aislada de aguas termales de Salta (Argentina)
Navas, L.E.¹; Amadío, A.F.²; Fuxan, I.³; Zandomeni R.O.¹
1CONICET e Instituto de Microbiología y Zoología Agrícola (IMYZA), Instituto Nacional de Tecnología Agropecuaria (INTA), De los Reseros y Las Cabañas s/nro. C.C. 25 (1712), Castelar, Buenos Aires, Argentina. Correo electrónico: lnavas@cnia.inta.gov.ar
2CONICET y EEA Rafaela, Instituto Nacional de Tecnología Agropecuaria (INTA)
3IMYZA, Instituto Nacional de Tecnología Agropecuaria (INTA)
Recibido el 22 de octubre de 2012
Aceptado el 13 de enero de 2014
Publicado online el 12 de febrero de 2014
RESUMEN
Se aislaron dos bacterias termofílicas a partir de aguas termales de la provincia de Salta, Argentina. Estudios filogenéticos permitieron caracterizar los aislamientos como pertenecientes a los géneros Thermus y Geobacillus. Se determinó la secuencia nucleotídica parcial del genoma de Thermus sp. 2.9 con un equipo de secuenciación masiva de ADN de tecnología Roche 454. Se generaron 215.557 lecturas que proveen una cobertura aproximada de 40 veces el tamaño del genoma. Se realizó un análisis preliminar de las secuencias obtenidas para la identificación de regiones codificantes. Mediante el mismo se identificaron y caracterizaron genes que codifican enzimas utilizadas en procesos de transformación de alimentos y relacionadas con la degradación de polímeros, tales como xilanasas, proteasas, esterasas, lipasas, catalasas y galactosidasas. Este primer paso indica que este microorganismo es un potencial productor de enzimas termofílicas que podrían ser aplicadas en la industria alimentaria.
Palabras clave: Bacterias termófilas; Biotecnología; Enzimas termofílicas; Genomas; Microbiología Industrial.
ABSTRACT
Two thermophilic bacteria were isolated from a hot spring in Salta, northwest Argentina. Phylogenic analysis indicates that the isolates belong to the Thermus and Geobacillus genera. We have undertaken the DNA sequencing of the complete genome from the isolate Thermus sp. 2.9 using Roche 454 technology. Two hundred and fifteen thousand readings were obtained providing approximately 40 fold coverage of the genome. A first round of analysis of the contigs was made to identify proteins coded in the genome. We report the identification and characterization of several genes coding for enzymes related to the degradation of polymers such as xylanases, proteases, esterases, lipases, catalase and galactosidases. These enzymes may be useful in processes to transform commodities from agriculture and valuable tools in the food industry.
Keywords: Thermophilic bacteria; Biotechnology; Thermophilic enzymes; Genomes; Industrial microbiology.
INTRODUCCIÓN
Desde un punto de vista antropocéntrico se puede considerar que numerosas especies de microorganismos procariotas sobreviven y proliferan en condiciones fisicoquímicas anormales o extremas, denominándose extremófilos aquellos que pueden crecer en condiciones letales para la mayoría de las especies conocidas (Ferrer et al., 2007). Se han aislado microorganismos en ambientes de: bajas o altas temperaturas (hasta -2º C o 110º C respectivamente); extrema acidez (pH menor a 3) o alcalinidad (pH mayor a 10); elevadas presiones y concentraciones salinas entre 5% a 30% (Niehaus et al., 1999). El estudio de microorganismos extremófilos se ha intensificado en los últimos años, debido al interés por descifrar los mecanismos involucrados con el desarrollo de la vida en estas condiciones y a la posible utilización de estos organismos o sus productos en procesos industriales.
La temperatura de crecimiento de microorganismos es uno de los parámetros físicos mejor estudiados, se han encontrando bacterias que proliferan en ambientes naturales desde -2º C hasta 115º C (Cava et al., 2009). De acuerdo a la temperatura de crecimiento, los microorganismos se clasifican como: psicrófilos, cuando crecen a temperaturas menores a 10º C; mesófilos, si lo hacen entre 10-50º C; termófilos, cuando crecen entre 50-75º C y los hipertermófilos que proliferan a temperaturas superiores a 75º C. Los genomas de microorganismos capaces de proliferar a temperaturas elevadas han recibido gran atención, ya que las enzimas presentes en ellos demuestran generalmente actividad catalizadora a esas altas temperaturas (Herbert, 1992; Madigan et al.,1997). Las enzimas presentan muchas ventajas frente a los catalizadores químicos, entre ellas, la precisión y especificidad por el sustrato, la eficiencia, baja toxicidad y relativo bajo costo (Vieille et al., 2001).
Las aguas termales son la principal fuente de bacterias termofílicas. La primera especie bacteriana en ser aislada, Thermus aquaticus, se obtuvo de fuentes termales del Yellowstone National Park, USA en 1969 (Brock et al., 1969). Las enzimas termofílicas involucradas en la degradación de polímeros son de gran valor por su aplicación industrial, incluyendo a xilanasas, proteasas, esterasas, lipasas, catalasas y galactosidasas que se utilizan en procesos catalíticos de transformación en la industria alimenticia (Bruins et al., 2001).
El objetivo de este trabajo es iniciar la búsqueda de bacterias termófilas en la Argentina, para disponer de aislamientos vernáculos que permitan la identificación y explotación de sus genes con el objetivo final de producir enzimas que habiliten el desarrollo de procesos de transformación industrial de materia prima de origen agropecuario.
MATERIALES Y MÉTODOS
Aislamiento
Se tomaron cinco muestras de 2 litros de aguas termales de Rosario de la Frontera, Salta. Los microorganismos se concentraron filtrando las muestras por membranas de 0,2 μm. Luego se invirtió la membrana y se recuperaron los microorganismos en 5 ml de solución fisiológica. Cien microlitros de esta solución fueron utilizados para inocular por duplicado nueve caldos de cultivo diferentes, los cuales fueron incubados a 60º C y 70º C hasta observar turbidez. Los caldos en los que se observó crecimiento se subcultivaron en el mismo medio solidificado con agar 2,5%. Se realizaron dos rondas de aislamientos de colonias. Los aislamientos fueron conservados a -70º C en medio Luria Bertani con 15% de glicerol. Los aislamientos obtenidos se denominan en adelante Thermus sp. 2.9 y Geobacillus sp. T6.
Medios de cultivo
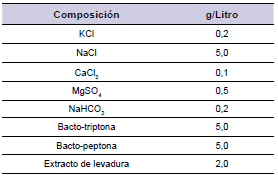
M9: Medio Base para Thermus sp. 2.9
Extracción de ADN genómico Para el aislamiento del ADN genómico se inoculó 500 ml de medio de cultivo "M9" con Thermus sp. 2.9 proveniente de la solución "stock" de glicerol y se incubó a 65º C durante tres días, con agitación constante a 250 rpm. Se centrifugó el cultivo bacteriano a 4.000 xg, durante 15 minutos a 4º C, se resuspendió el "pellet" en 9,5 ml de Buffer A (10 mM Tris, 1 mM EDTA, 1% SDS y 0,040 mg/ml de ribonucleasa "A") y se trató con Proteinasa K (1 mg/ml) por 45 minutos a 37º C. Se agregó 3,5 ml de NaCl 5M, luego 3 ml de Solución B (10% Bromuro de hexadecil-trimetilamonio/0,7 M NaCl) y se incubó durante 20 minutos a 65º C. Se centrifugó a 10.500 xg por 10 minutos, luego de agregar igual volumen de cloroformo/alcohol isoamílico (24:1). La fase acuosa se extrajo dos veces adicionales con fenol/cloroformo/ alcohol isoamílico (25:24:1). Se precipitó el ADN con 0,6 volúmenes de isopropanol y se separó el ovillo de ADN precipitado con un ansa descartable. Se lavó el ADN con etanol 70%, se secó y resuspendió en 200 μl de buffer TE.
PCR y secuenciación de ARNr 16S
El ADN molde se obtuvo por PCR amplificando el gen de la subunidad pequeña ribosomal, 16S, utilizando "primers" consenso para el ADNr bacteriano (Primer F: 5´ CGTCAGATTGAACGCTGGCG 3´ y Primer R: 5´ ACATTTCACA-ACACGAGCTG 3´). La reacción consistió en 35 ciclos (30 seg a 94º C, 30 seg a 55º C, 90 seg a 72º C) seguido por una incubación a 70º C durante 2 min. Los fragmentos amplificados se clonaron en el vector pGem-T Easy (Promega). Los plásmidos resultantes se utilizaron como molde en la reacción de secuenciación de ADN, con la química DYEnamic ET Terminator (Amersham), resolviendo en un secuenciador ABI377 (Applied Biosystems). Como "primers" para la reacción de secuenciación se utilizaron T7, SP6 y adicionales del ADNr 16S (5´ CAGCMGCCGCGGTAA 3´ y 5´ ACGGGCGGTGTGTRC 3´ donde M es A o C; R es G o A). El ensamblado de las secuencias se realizó con el paquete Staden Package (MRC-LMB, Cambridge, UK). Se construyeron los árboles filogenéticos con las secuencias del ADNr 16S utilizando las herramientas disponibles en el sitio web (http://rdp.cme.msu.edu/) del Ribosomal Database Project II (RDP-II) y la interfaz de PHYLIP. El árbol se construyó utilizando el modelo de Jukes-Cantor (Jukes et al., 1969) y el método de Neighbor-Joining (Saitou et al., 1987).
Secuenciación del genoma de Thermus sp. 2.9
Se seleccionó el aislamiento Thermus sp. 2.9. para su secuenciación masiva por tener un tamaño estimado de genoma (2 Mb) menor al de Geobacillus sp. T6, que facilitaría el ensamble de las secuencias e identificación de todos sus genes.
Cinco μg de ADN genómico fueron utilizados conjuntamente con el kit provisto por Roche Applied Science y secuenciado en un equipo GS-FLX 100 (Macrogen). Las lecturas fueron ensambladas de novo con el software Newbler (v2.3) (Miller et al., 2010).
Bioinformática
Para analizar las secuencias obtenidas se utilizaron las herramientas del paquete BLAST (Altschul et al., 1990), Artemis y dos "scripts" creados en el lenguaje Perl. Se utilizó Artemis para identificar los ORFs (Open Reading Frames) de más de cien aminoácidos y se corrió BLAST remoto en el servidor del NCBI (http://www.ncbi.nlm.nih.gov/), utilizando un script de Perl (netblastdir.pl) modificado del original (Rob Edwards, http://salmonella.utmem.edu/cgi-bin/cgi.cgi?submit=script). Se realizaron comparaciones con los programas blastn y blastx, contra las bases de datos no redundantes de ácidos nucleicos y proteínas, respectivamente. Los resultados se procesaron con dos "scripts" de Perl para facilitar su posterior interpretación.
RESULTADOS Y DISCUSIÓN
Aislamiento y caracterización microbiológica de los microorganismos aislados
Se realizó el aislamiento de dos microorganismos con características diferentes, tanto en la temperatura de crecimiento como en sus características morfológicas. Las colonias del aislamiento denominado Geobacillus sp. T6 (figura 1-A) son incoloras o blanquecinas, con aspecto rugoso y mucosas. El tiempo de duplicación, durante la fase logarítmica de crecimiento, es de 30 minutos a la temperatura de crecimiento de 60º C. Las bacterias T6 son Gram positivas y presentan endosporos.

Figura 1. Morfología de las colonias de aislamientos termofílicos. A-T6, colonias de Geobacillusus sp. T6, cultivo 12 horas a 65º C. B-2.9, colonias de Thermus sp. 2.9, cultivo 60 horas a 65º C.
Thermus sp. 2.9 presenta el crecimiento mostrado en la figura 1-B, a los tres días de incubación a 65º C en medio agarizado. Se observan colonias pequeñas, aplanadas, de aspecto rugoso y anaranjadas. Las bacterias de Thermus sp. 2.9 se tiñen como Gram negativas. Las observaciones en microscopio electrónico muestran en promedio bastones de 4 μm de largo por ~0,5-1 μm de ancho, ausencia de flagelos y presencia de fimbrias en toda la superficie de la bacteria (figura 2 A, B y C).

Figura 2. Microfotografía electrónica de Thermus sp. 2.9. Magnitud indicada con barra de referencia.
Clasificación filogenética
La secuenciación del gen ribosomal de los aislamientos Geobacillus sp. T6 y Thermus sp. 2.9 generó fragmentos de 1.391 y 1.370 nucleótidos, respectivamente. El primer aislamiento, denominado T6, se agrupó de acuerdo a la secuencia de su gen ribosomal 16S con especies del género Geobacillus (Nazina et al., 2001). Este género incluye bacilos termofílicos del subgrupo 5, que comprende a bacilos aeróbicos Gram positivos, formadores de endosporos, que crecen a temperaturas mayores a 55º C. De acuerdo a todas estas características compartidas se denominó al aislamiento T6 como Geobacillus sp. T6.
El aislamiento 2.9, se agrupa con especies del género Thermus (figura 3). Su gen ribosomal presenta alta similitud con genes ribosomales de Thermus brockianus y en menor grado con T. aquaticus. Numerosas especies de bacterias clasificadas dentro del género Thermus se han aislado en cada uno de los continentes. El género Thermus agrupa a bacilos aeróbicos, no esporulantes, con temperaturas de crecimiento óptimas entre 70-75º C. Si bien se trata de bacterias Gram negativas, basados en la similitud del gen codificante para la subunidad 16S de ARN ribosómico, tienen una estrecha relación con el género Deinococcus de bacilos Gram positivos (Hensel et al., 1986). Thermus brockianus (Williams et al., 1995), es una bacteria aeróbica, Gram negativa, con forma de bastones cortos. Las colonias presentan pigmentación amarilla más pálida que la de Thermus sp. 2.9 y crece a una temperatura óptima de 70º C en medios con triptona y extracto de levadura.

Figura 3. Árbol filogenético construido a partir de la secuencia del ADNr 16S de Thermus sp. 2.9 y otras especies del género Thermus.
Con respecto a la temperatura óptima de crecimiento y al aspecto y color de las colonias formadas en medio sólido, en las distintas especies se evidencian ciertas diferencias. T. aquaticus presenta un crecimiento óptimo a 70-75º C y, luego de uno o dos días de incubación, forma colonias amarillas compactas (Brock et al., 1969). Thermus thermophilus crece a temperatura óptima ligeramente menor, en el intervalo 65- 72º C y, luego de un día de incubación, pueden observase colonias de 1 mm de diámetro con una superficie lisa, plana y de color amarillo-anaranjado (Oshima et al., 1974).
Thermus sp. 2.9 comparte con especies del género Thermus la formación de colonias de aspecto rugoso y de color anaranjado, su coloración Gram negativa, la forma de bastón y ausencia de flagelos. Para cada una de las especies nombradas los medios de crecimiento requieren peptona y extracto de levadura al igual que para el aislamiento 2.9. Debido a las características microbiológicas y a la clasificación de su gen ribosomal 16S, se denomina Thermus sp. 2.9.
Secuenciación del genoma de Thermus sp. 2.9.
En base a la secuenciación parcial del genoma de Thermus sp. 2.9, utilizando el secuenciador Roche 454, se obtuvieron 215.557 lecturas que corresponden a 81.238.046 pb luego de los cortes por calidad del conjunto de lecturas totales obtenidas. El promedio de nucleótidos por lectura corresponde a 377 nucleótidos que, en total, proveen una cobertura aproximada de 35 a 40 veces el tamaño estimado del genoma de una bacteria del género Thermus. La totalidad de secuencias corregidas por calidad fueron ensambladas de novo por el software Newbler v2.3, utilizado parámetros estándares. Se generaron 137 contigs con más de 500 nucleótidos cada uno y un N50 de 39.906 pb. La cantidad total de nucleótidos ensamblados en los 137 contigs suma 2.460.000 pares de bases totales. La distribución del tamaño de los 62 contigs más grandes obtenidos se representa en la figura 4, el contig más extenso contiene 109.000 pares de bases y el promedio de tamaño de los contigs corresponde a 17.669 nucleótidos.

Figura 4. Distribución de tamaño de los 62 contigs mayores. Tamaño de contigs expresados en pares de bases.
El genoma de Thermus sp. 2.9 de acuerdo a la totalidad de las secuencias ensambladas tiene un contenido GC de 66.7%, similar al determinado para T. aquaticus (68%) y las cepas de T. thermophilus (69%). Actualmente, se dispone de la secuencia genómica completa de cuatro especies de T. thermophilus: HB8 (Brüggemann et al., 2006), HB27 (Henne et al., 2004), JL-18 (Accession: PRJNA162129) y SG0.5JP17-16 (Accession: PRJNA159537) y el centro US DOE Joint Genome Institute (JGI-PGF) avanza en la determinación del genoma completo de T. aquaticus, principalmente, en la unión de secuencias y anotación de sus genes, teniendo ensamblados actualmente 22 contigs.
Se realizó una anotación preliminar de genes del aislamiento Thermus sp. 2.9 sobre los 137 contigs, para identificar las proteínas homólogas en bases de datos y en los genomas de T. aquaticus y de las cepas de T. thermophilus HB8 y HB27. Según la base de datos de NCBI, el genoma de estas tres cepas bacterianas cuenta con 2.593, 2.291 y 2.263 genes, respectivamente. De los genes predichos en Thermus sp. 2.9, 2391 presentaron similitudes con proteínas de la base de datos nr de GenBank y 51 con genes codificantes de ARN. A su vez, 1.986 proteínas presentaron similitudes con proteínas de T. aquaticus y 1.782 y 1.796 con T. thermophilus HB8 y HB27. Un total de 1.755 genes pudieron asociarse a diversos subsistemas presentados en la tabla 1.
Tabla 1. Número de genes asociados a cada subsistema de Thermus sp. 2.9.

Con respecto a la distribución de los genes, se observa una sintenia mayor con el genoma de T. aquaticus, que la observada con HB8 y HB27. Tomando como colineales a un mínimo de 4 genes consecutivos, se observa que 1.687 (71%) de los genes putativos identificados en Thermus sp. 2.9 comparten colinealidad con los genes anotados en los contigs de T. aquaticus. Comparando con los dos genomas completos de T. thermophilus, los genes de Thermus sp. 2.9 comparten colinealidad con 1.496 (63%) y 1.291 (52%) genes de HB8 y HB27, respectivamente. Estudios comparativos indican que Thermus sp. 2.9 pertenece al género Thermus de acuerdo a sus características microbiológicas y moleculares. Thermus sp. 2.9 se encuentra más relacionado a T. brokianus y T. aquaticus por la similitud de la secuencia del gen ribosomal 16S y tiene mayor similitud con T. aquaticus que con los genomas de HB8 y HB27, a nivel del número total de genes homólogos.
Selección de genes relevantes
Analizando la totalidad de los genes de la bacteria, se encontraron diversas utilidades de las enzimas codificadas. En la tabla 3 se mencionan cada una de ellas y el número de genes asociados a dichas aplicaciones.
Tabla 2. Número de genes asociados a diversas utilidades.

Se han preseleccionado 116 genes cuyos homólogos en GenBank y otros genomas de Thermus codifican enzimas relevantes para su utilización en la transformación de alimentos. En una primera revisión de las actividades esperadas para los genes identificados para el aislamiento Thermus sp. 2.9 se encontraron 50 proteasas, 47 esterasas, 10 lipasas, 4 pululanasas, 3 galactosidasas, una catalasa y una xilanasa (figura 5). Es esperable que estas enzimas sean más estables y activas a temperaturas mayores que sus homólogas mesófilas y, de acuerdo a la literatura, pueden ser más resistentes a agentes químicos (Lioliou et al., 2004), propiedad que las haría más atractivas para procesos industriales.

Figura 5. Tipo de actividad en genes seleccionados.
La secuenciación del genoma de Thermus sp. 2.9 ha permitido identificar los genes codificados por la bacteria aislada de las Termas de Rosario de la Frontera en Salta, República Argentina. Más aún, ha demostrado la potencial utilidad de esta bacteria como fuente de numerosas enzimas termoestables para beneficio en investigación básica y aplicada. Futuros estudios de la expresión de estas actividades son necesarios para su explotación y, por tratarse de aislamientos vernáculos, para la protección de la propiedad intelectual sobre el uso de los mismos.
AGRADECIMIENTOS
Agradecemos a Lorena la Fuente por su ayuda en la edición del manuscrito. Este trabajo se encuentra en el marco de los proyectos AETA (Área Estratégica: Tecnología de Alimentos) 1672 y AETA 281721.
BIBLIOGRAFÍA
1. ALTSCHUL, S.F.; GISH, W.; MILLER, W.; MYERS, E.W.; LIPMAN, D.J. 1990. Basic local alignment search tool. J. Mol. Biol. 215:403-410. [ Links ]
2. BROCK, T.D.; FREEZE, H. 1969. Thermus aquaticus gen. n. and sp. n., a nonsporulating extreme thermophile. J Bacteriol; 98(1): 289-97. [ Links ]
3. BRÜGGEMANN, H.; CHEN, C. 2006.Comparative genomics of Thermus thermophilus: Plasticity of the megaplasmid and its contribution to a thermophilic lifestyle. J Biotechnol; 124(4): 654-61. [ Links ]
4. BRUINS, M.E.; JANSSEN, A.E.; BOOM, R.M. 2001. Thermozymes and their applications: a review of recent literature and patents. Appl Biochem Biotechnol; 90(2): 155-86. [ Links ]
5. CAVA, F.; HIDALGO, A.; BERENGUER, J. 2009. Thermus thermophilus as biological model. Extremophiles; 13(2): 213-31. [ Links ]
6. FERRER, M.; GOLYSHINA, O.; BELOQUI, A.; GOLYSHIN, P.N. 2007. Mining enzymes from extreme environments. Curr Opin Biotechnol; 10(3): 207-14. [ Links ]
7. HENNE, A.; BRÜGGEMANN, H.; RAASCH, C.; WIEZER, A.; HARTSCH, T.; LIESEGANG, H.; JOHANN, A.; LIENARD, T.; GOHL, O.; MARTINEZ-ARIAS, R.; JACOBI, C.; STARKUVIENE, V.; SCHLENCZECK, S.; DENCKER, S.; HUBER, R.; KLENK, H.P.; KRAMER, W.; MERKL, R.; GOTTSCHALK, G.; FRITZ, H.J. 2004. The genome sequence of the extreme thermophile Thermus thermophilus. Nat Biotechnol; 22(5): 547-53. [ Links ]
8. HENSEL, R.; DEMHARTER, W.; KANDLER, O.; KROPPENSTEDT, R.M.; STACKEBRANDT, E. 1986. Chemotaxonomic and molecular-genetic studies of the genus Thermus: Evidence for a phylogenetic relationship of T. aquaticus and T. ruber to the genus Deinococcus. Int J Syst Bacteriol; 36: 444-53. [ Links ]
9. HERBERT, R.A. A. 1992. perspective on the biotechnological potential of extremophiles. Trends Biotechnol; 10(11): 395-402. [ Links ]
10. JUKES, T.H.; CANTOR, C.R. 1969. Evolution of protein molecules. En Munro HN, editor, Mammalian Protein Metabolism, pp. 21-132, Academic Press, New York. [ Links ]
11. LIOLIOU, E.E.; PANTAZAKI, A.A.; KYRIAKIDIS, D.A. 2004. Thermus thermophilus genome analysis: benefits and implications. Microb Cell Fact; 3(1): 5. [ Links ]
12. MADIGAN, M.T.; MARRS, B.L. 1997. Extremophiles. Sci Am; 276(4): 82-7. [ Links ]
13. MILLER, J.R.; KOREN, S.; SUTTON, G. 2010. Assembly algorithms for next-generation sequencing data. Genomics; 95(6): 315-27. [ Links ]
14. NAZINA, T.N.; TOUROVA, T.P.; POLTARAUS, A.B.; NOVIKOVA, E.V.; GRIGORYAN, A.A.; IVANOVA, A.E.; LYSENKO, A.M.; PETRUNYAKA, V.V.; OSIPOV, G.A.; BELYAEV, S.S.; IVANOV, M.V. 2001. Taxonomic study of aerobic thermophilic bacilli: descriptions of Geobacillus subterraneus gen. nov., sp. nov. and Geobacillus uzenensis sp. nov. from petroleum reservoirs and transfer of Bacillus stearothermophilus, Bacillus thermocatenulatus, Bacillus thermoleovorans, Bacillus kaustophilus, Bacillus thermodenitrificans to Geobacillus as the new combinations G. stearothermophilus, G. thermocatenulatus, G. thermoleovorans, G. kaustophilus, G. thermoglucosidasius and G. thermodenitrificans. Int J Syst Evol Microbiol; 51(2): 433-46. [ Links ]
15. NIEHAUS, F.; BERTOLDO, C.; KÄHLER, M.; ANTRANIKIAN, G. 1999. Extremophiles as a source of novel enzymes for industrial application. Appl Microbiol Biotechnol; 51(6): 711-729. [ Links ]
16. OSHIMA, T.; IMAHORI, K. 1974. Description of Thermus thermophilus (Yoshida and Oshima) comb. nov., a Nonsporulating Thermophilic Bacterium from a Japanese Thermal Spa. Int J Syst Bacteriol; 24: 102-112. [ Links ]
17. SAITOU, N.; NEI, M. 1987. The neighbor-joining method: a new method for reconstructing phylogenetic trees. Mol Biol Evol.; 4(4): 406-25. [ Links ]
18. VIEILLE, C.; ZEIKUS, G.J. 2001. Hyperthermophilic enzymes: sources, uses and molecular mechanisms for thermostability. Microbiol Mol Biol Rev; 65(1): 1-43. [ Links ]
19. WILLIAMS, R.A.; SMITH, K.E.; WELCH, S.G.; MICALLEF, J.; SHARP, R.J. 1995. DNA relatedness of Themus Strains, description of Thermus brockianus sp. nov., and proposal to reestablish Themus thermophilus (Oshima and Imahori). Int J Syst Bacteriol; 45(3): 495-9. [ Links ]














