Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista veterinaria
versión On-line ISSN 1669-6840
Rev. vet. vol.27 no.2 Corrientes jun. 2016
TRABAJOS DE INVESTIGACIÓN
Espectrofotometría infrarroja transformada de Fourier para identificar bacterias uterinas patógenas en vacas lecheras
Jaureguiberry, M.1,2; Madoz, L.V.1,2; Giuliodori, M.J.3; Drillich, M.4; de la Sota, R.L.1,2
1Cátedra Reprod.Anim., Fac.Cs.Veterinarias, Univ.Nac.La Plata, B1900AVW, La Plata, Argentina, +542214236663, Interno 455. 2CONICET, Buenos Aires, Argentina. 3Cát.Fisiología, FCV-UNLP, La Plata, Argentina. 4Univ.Vet.Med.Vienna, Austria. E-mail: jaureguiberrymaria@gmail.com
Resumen
El objetivo del trabajo fue determinar el grado de acuerdo entre dos métodos de identificación bacteriana, marcha bacteriológica convencional y espectroscopía infrarroja transformada de Fourier (TF-IR), así como determinar si la base de datos construida a partir de bacterias aisladas de úteros de vaca lecheras de Europa podía ser utilizada para identificar bacterias uterinas de vacas lecheras de Argentina. Las muestras bacteriológicas fueron tomadas a vacas post parto (n=64) con distinto grado de descarga vaginal (DV). Inicialmente se efectuó diagnostico bacteriológico por marcha bacteriológica convencional (n=27) y posteriormente por TF-IR. El grado de acuerdo entre ambos métodos de identificación bacteriana fue determinado por el coeficiente kappa y el efecto de la DV sobre el aislamiento de Escherichia coli y Trueperella pyogenes fue analizado por regresión logística. El grado de acuerdo entre ambos métodos tuvo un coeficiente kappa de 0,73 (p<0,001). La probabilidad de aislamiento bacteriano (E. coli y T. pyogenes) tendió a incrementarse junto con el grado de DV (p=0,06), siendo 1,88 veces más alta la probabilidad de encontrar un resultado positivo a E. coli o T. pyogenes en vacas con DV fétida que en vacas con DV normal. En conclusión, TF-IR reveló un buen grado de acuerdo con la marcha bacteriológica convencional. Por otro lado, la base de datos producida a partir de bacterias uterinas de vacas lecheras de Europa pudo ser utilizada para la identificación de bacterias uterinas de vacas lecheras de Argentina.
Palabras clave: vaca lechera, bacterias uterinas, espectrofotometría infrarroja.
Abstract
The objectives of the present study were to test the agreement between routine methods of bacteriological testing and the Fourier transformed infrared spectroscopy (FTIR) method for uterine bacterial identification, and if a database built with uterine bacteria from European dairy cows can be used to identify bacteria from Argentinean dairy cows. Postpartum dairy cows (n=64) with different vaginal discharge (VD) were sampled for bacterial identification. The uterine bacteria were first identified by routine bacteriological testing methods (n=27) and then by FTIR. Statistical analyses were performed by kappas coefficient and by logistic regression analysis. The agreement between the two methods had a kappa`s coefficient of 0.73 (p<0.001). In addition, the likelihood for bacterial growth (E. coli and T. pyogenes) tended to increase with VD score (p=0.06), given that the odds for a positive result to E. coli or T. pyogenes was 1.88 times higher in cows with fetid VD than in herdmates with clear normal VD. In conclution, FTIR spectroscopy has a good agreement with routine bacteriological testing methods and the database built with bacteria from European dairy cows is useful to identify uterine bacteria from Argentinean dairy cows.
Key words: dairy cow, uterine pathogens bacteria, infrared spectroscopy.
Recibido: 10 marzo 2016
Aceptado: 27 mayo 2016
Financiado por MINCyT (Argentina) y BMWF (Austria).
Present.XI Simp.Int.Repr.Anim. IRAC, Córdoba, Argentina (2015)
INTRODUCCIÓN
Muchos trabajos han demostrado que las enfermedades uterinas como metritis (MT), endometritis clínica (EC) y endometritis subclínica (ES), están asociadas a una disminución de la fertilidad y a un incremento de los rechazos por causas reproductivas de las vacas lecheras 10, 17. En la mayoría de los casos la causa es microbiana. Escherichia coli y Trueperella pyogenes son las bacterias patógenas más aisladas de casos de MT, EC y en algunos casos de ES 12 . Otras bacterias importantes en las infecciones uterinas son Prevotella sp, Fusobacterium necrophorum y Fusobacterium nucleatum 17.
Estudios recientes han revelado que las especies bacterianas aisladas del útero de vacas postparto son más de 200 19 . No obstante, todavía no se ha descrito la patogenicidad de las mismas ni la interacción entre ellas. Generalmente, para los casos de MT, EC y ES los veterinarios aconsejan tratamientos con antibióticos de forma empírica. Para los casos de MT se recomienda ceftiofur, penicilina procaínica o tetraciclina (SC o IM). Para los casos de EC y ES se aconseja cefapirina benzatínica (administración intrauterina) 5, 10 . Como inconveniente, se afirma que el uso empírico de antibióticos podría ser un factor importante en la aparición de cepas bacterianas resistentes 14 .
Un estudio reciente reportó que más del 50% de T. pyogenes aisladas eran resistentes a amoxicilina, ampicilina, cloranfenicol, florfenicol, oxitetraciclinas y penicilinas 15 . Asimismo se comunicó que el 35% de E. coli aisladas fueron multidroga resistentes (ampicilinacloranfenicol- florfenicol) 14 . Este problema no solo atañe a la medicina veterinaria, sino también a la medicina humana, ya que muchos de los antibióticos mencionados anteriormente tienen un uso común.
Por ello, el empleo de antibióticos basado en un diagnóstico bacteriológico podría ayudar a reducir la resistencia bacteriana 20 . Por lo tanto, disponer de una
técnica de identificación bacteriana eficaz, relativamente rápida y económica, podría reducir el uso empírico de antibióticos.
La espectroscopia infrarroja transformada de Fourier (TF-IR) es una técnica que permite determinar la composición bioquímica (proteínas, lípidos, ácidos nucleicos, polisacáridos) de cualquier material 11 . De hecho, hace algunos años que se está utilizando como método de identificación bacteriana 7 . Gracias a que los microorganismos son bioquímicamente diferentes unos de otros, el espectro que generan actúa como una huella digital, permitiendo diferenciarlos en género, especie, tipo y serotipo 1. Esta diferenciación se basa en la distancia espectral entre microorganismos, cuanto más distancia haya entre los espectros mayor la diferencia bioquímica entre microorganismos. En tal sentido, es muy importante poseer una base de datos con una gran cantidad de espectros de referencia. Contando con una base de datos completa, TF-IR podría ser un método simple, relativamente rápido y de bajo costo para la identificación de bacterias uterinas patógenas, facilitando el uso racional de antibióticos en lugar del uso empírico. Por lo tanto, la identificación bacteriana por TF-IR podría ser una herramienta muy útil para disminuir la resistencia bacteriana tanto en la práctica veterinaria como en la medicina humana.
Los objetivos del presente trabajo fueron:1) determinar el grado de acuerdo entre marcha bacteriológica convencional y la TF-IR; y 2) determinar si la base de datos generada a partir de bacterias uterinas de vacas lecheras europeas podría ser utilizada para la identificación de bacterias uterinas de vacas lecheras de nuestro país.
MATERIAL Y MÉTODOS
Animales. Para la realización del presente trabajo se utilizaron vacas Holando Argentino (n=64) de tambos comerciales de las localidades de Brandsen (35°17S, 58°23W) y Castelli (36°10S, 57°78W), Provincia de Buenos Aires, Argentina.
Evaluación clínica. La evaluación clínica se realizó en vacas entre 5 y 60 días postparto que no habían sido tratadas con antibióticos durante los 14 días previos al estudio. El examen de las vacas comenzó con la evaluación de la descarga vaginal (DV) a través de la técnica denominada "flujeo" manual. Dicha maniobra consiste en limpiar la zona perineal e introducir suavemente una mano en forma de cuña cubierta por un guante de tacto nuevo y lubricado. El flujo se obtuvo de la porción craneal de la vagina. La DV se clasificó en 4 grados: DV0 correspondió a un flujo normal, DV1 a un flujo con flóculos de pus, DV2 a un flujo purulento sin olor y DV3 a un flujo purulento y con olor fétido. Debido a la amplitud del rango de días post parto de las vacas incluidas en el trabajo, el score de DV fue considerado como una clasificación descriptiva antes que un parámetro de diagnóstico de las enfermedades uterinas.
Muestras bacteriológicas. Las muestras bacteriológicas fueron tomadas a cada una de las vacas incluidas en el experimento, independientemente del grado de DV (0-3). Para la obtención de las muestras se utilizó la técnica cytobrush con el agregado de una "camisa sanitaria" para evitar contaminación proveniente de la vagina. Para realizar la técnica cytobrush se utilizó una pistoleta de acero inoxidable (similar a la que se utiliza para inseminación) que constaba de dos partes, una pieza tubular externa y un vástago interno que en su extremo anterior poseía 1 cm de rosca, al cual se le adosó un cepillito estéril utilizado en ginecología humana (Medibrush XL®, Medical Engineering Co, SA). Posteriormente se colocó una vaina de inseminación (IVM Technologies, Francia) para cubrir el cepillito y la pistoleta. Por encima de ésta se colocó una "camisa sanitaria". Una vez lista la pistoleta se procedió al enhebrado de modo similar a la técnica utilizada en inseminación artificial. En el cérvix, se tiró hacia atrás la "camisa sanitaria" para rasgarla. Pasado el cérvix se expuso el cepillito al endometrio y en contacto con el mismo se lo hizo rotar unas 4 veces. Antes de retirar la pistoleta, el cepillito se retrajo para que quede protegido nuevamente por la vaina plástica de inseminación. Una vez obtenida la muestra, se introdujo el cepillito en un medio de transporte Stuart (Eurotubo, DeltaLab) el cual se colocó en una caja de poliestireno expandido con refrigerantes hasta su llegada al laboratorio. Allí, cada cepillo fue cultivado en un medio agar sangre base, con el agregado de 5% de sangre ovina desfibrinada (Laboratorios Britania, Buenos Aires, Argentina). Las bacterias se identificaron por las siguientes características: morfología de colonia, coloración Gram, tipo de hemólisis y pruebas bioquímicas convencionales 25 . Las colonias bacterianas obtenidas (n=27) fueron congeladas a -80ºC hasta su envío al Instituto de Microbiología de la Universidad de Medicina Veterinaria de Viena, Austria, donde fueron identificadas por TF-IR.
Espectroscopia infrarroja transformada de Fourier (TF-IR). En el Instituto de Microbiología antemencionado, las muestras (n=27) fueron re-cultivadas en placas de agar sangre a 37°C por 48 a 72 h para hacerlas crecer nuevamente. Una submuestra de cada colonia se cultivó a 37°C por 48 h en agar sangre. De las colonias obtenidas se tomó otra submuestra y se cultivó en agar tripticasa soya (Oxoid, TSA, Hampshire, United Kingdom) a 30ºC por 24 h 19 . De las colonias obtenidas en este medio se tomó una "ansada" (1 mm de diámetro) de material celular que fue suspendido en 100 ul de agua estéril desionizada y homogeneizada. Una alícuota de 30 ul de esa suspensión fue colocada en una placa de ZnSe y secada en horno a 40ºC por 40 min. Las mediciones por TF-IR fueron llevadas a cabo cubriendo un rango espectral de 4000 a 500 cm-1 (Bruker Optics). El análisis de los distintos espectros obtenidos fue analizado utilizando OPUS software (versión 5.5; Bruker Optics). La base de datos utilizada para la identificación de bacterias uterinas estuvo formada por más de 700 especies bacterianas, incluyendo más de 200 especies aisladas de vacas lecheras con infección uterina de tambos de Europa12, 18, 19.
Análisis estadístico. El grado de acuerdo entre la marcha bacteriológica convencional y TF-IR fue evaluada mediante coeficiente kappa, y la probabilidad de aislamiento bacteriano según el grado de DV fue evaluado por regresión logística 9 .
RESULTADOS
De las 64 muestras bacteriológicas, 9 (14%, 9/64) fueron excluidas del análisis por contaminación. Por lo tanto, se analizaron los resultados de 55 muestras bacteriológicas correspondientes a 55 vacas. Dieciocho vacas tuvieron DV0 (33%, 18/55), 12 vacas tuvieron DV1 (22%, 12/55), 10 vacas tuvieron DV2 (18%, 10/55), y 15 vacas tuvieron DV3 (27%, 15/55).
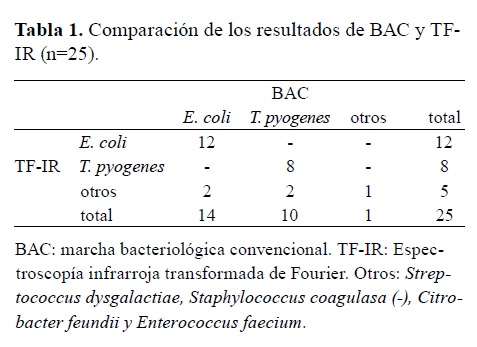
E. coli fue aislada en 14 animales de 55 (25%, 14/55), T. pyogenes en 12 de 55 (22%, 12/55), y coagulase negativo Staphylococci (CNS) en uno de 55 (2%, 1/55). No se observó crecimiento bacteriano en 28 de 55 animales (51%, 28/55). Las muestras positivas a E. coli, T. pyogenes y CNS (n=27) fueron enviadas al Instituto de Microbiología (Viena, Austria), donde fueron nuevamente cultivadas obteniendo un crecimiento bacteriano en 25 de 27 muestras enviadas. Las dos muestras en las que no se obtuvo crecimiento bacteriano correspondían a T. pyogenes. Por lo tanto, 25 muestras fueron evaluadas por TFIR: 14 de E. coli, 10 de T. pyogenes y una muestra de CNS (Tabla 1)
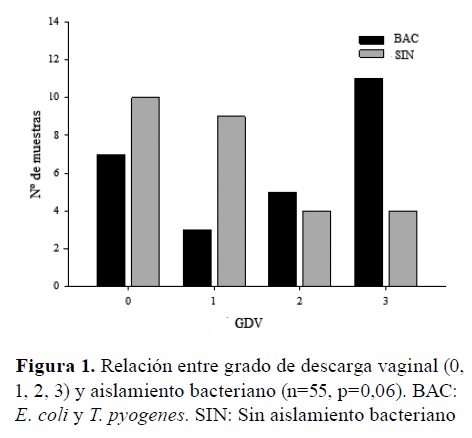
El grado de acuerdo entre la marcha bacteriológica convencional y FT-IR tuvo un coeficiente kappa de 0,73 (95% CI = 0,52-0,95, p<0,001). Si bien no fue significativa, la probabilidad de aislar E. coli y T. pyogenes tendió a incrementarse junto con el grado de DV (p=0,06), siendo el porcentaje de aislamiento de dichas bacterias de 39% (7/18) en vacas con DV0, 25% (3/12) en vacas con DV1, 60% (6/10) en vacas con DV2, y 73% (11/15) en vacas con DV3 (Figura 1). La probabilidad de aislar E. coli o T. pyogenes fue 1,88 veces más alta en vacas con DV3 que en vacas con DV0.
DISCUSIÓN
Si bien la mayoría de los autores concuerda en que TF-IR es una herramienta que combina alta especificidad, bajo costo y fácil procesamiento, sus estudios se han realizado comparando dicho procedimiento con técnicas moleculares complejas y costosas 7, 13 . En cambio, el trabajo aquí realizado comparó TF-IR con la técnica de identificación bacteriana convencional, ampliamente utilizada por los laboratorios, proponiendo una alternativa más eficiente, que brinde resultados en menor tiempo y a bajo costo. Recientemente se ha reportado que FT-IR facilita y mejora la identificación de bacterias causantes de mastitis en vacas lecheras, al ser comparada con la identificación bacteriana por técnicas de rutina 16.
En el presente estudio se determinó que TF-IR tiene un buen grado de acuerdo con la marcha bacteriológica convencional. No obstante, hubo 4 casos en los que el resultado de la marcha bacteriológica convencional no coincidió con los resultados de TF-IR. La razón de dichas diferencias podría ser que las especies bacterianas con características similares se confundan en el procesamiento de la marcha bacteriológica convencional.
Además, el tiempo que lleva obtener el resultado de TF-IR es algo mayor a 3,5 días, mientras que con la marcha bacteriológica convencional se tarda más y requiere más trabajo 2 . Si bien se ha utilizado la TF-IR para la identificación de bacterias uterinas, a nuestro conocimiento, éste es el primer estudio que compara TF-IR con la marcha bacteriológica convencional utilizando muestras uterinas de vacas con distinto tipo de DV 12, 19. Se requieren más estudios con mayor número de muestras y con la identificación de mayor cantidad de especies bacterianas para confirmar los resultados aquí obtenidos.
Varios estudios han demostrado que TF-IR tiene un gran poder de resolución, lo cual le permite diferenciar microorganismos a distintos niveles taxonómicos 6, 22 . De hecho, TF-IR es capaz de identificar cambios en la composición bioquímica de bacterias cultivadas en distintos medios o de bacterias que hayan mutado o hayan variado su composición bioquímica para adaptarse a un nuevo hospedador 1, 3, 4, 8, 21.
El punto clave para que esta técnica sea exitosa es la cantidad de espectros de referencia que contenga la base de datos 8, 22 . Cuantos más espectros de referencia tenga la base de datos, mayor es el poder de resolución de la técnica. La base de datos utilizada en el presente trabajo fue construida con espectros de bacterias uterinas de vacas lecheras europeas. A pesar de que las bacterias a identificar provenían de un origen y sistema de producción distinto, pudieron identificarse las bacterias uterinas de vacas de Argentina.
Por lo tanto, podría postularse que los distintos sistemas de producción varían la prevalencia de infecciones uterinas pero no el tipo de bacterias que se aísla de cada caso. Habría sido importante comparar bacterias uterinas anaerobias provenientes de vacas lecheras europeas con bacterias uterinas anaerobias provenientes de vacas lecheras argentinas, pero las dificultades de la obtención de muestras para la identificación de bacterias anaerobias a campo y la distancia entre los laboratorios impusieron que solo pudieran estudiarse bacterias aerobias.
En futuros trabajos, sería interesante estudiar los cambios en el espectro de las bacterias cuando éstas se vuelven resistentes a un antibiótico. Ello permitiría no solo identificar bacterias de un modo más eficiente sino también identificar cepas resistentes.
En el presente estudio, la probabilidad de aislamiento bacteriano de una muestra (E. coli y T. pyogenes) tendió a incrementarse junto con el grado de DV. Este hecho concuerda con el reportado en otro estudio donde se encontró una correlación entre el aislamiento de bacterias reconocidas como patógenas (E. coli, A. pyogenes, F. necrophorum y Prevotella melaninogenicus) y el grado de DV 24 .
Por otro lado, se halló una correlación significativa entre T. pyogenes y grado de DV (p<0,001), pero no entre E. coli y el grado de DV (p=0,21) 23, 24 . Sorpresivamente, se registró una alta tasa de aislamiento bacteriano en vacas con DV0. No obstante, la tasa de asilamiento bacteriano en vacas con DV0 y DV1 fue menor que en vacas con DV2 y DV3.
Este resultado concuerda con otro estudio donde se aisló E. coli y T. pyogenes en vacas con todos los tipos de DV (0-3), siendo menor en vacas con DV0 y DV1 que en vacas con DV2 y DV3 19 . Si bien la evaluación de la DV podría ayudar a determinar protocolos de tratamiento para reducir el uso empírico de antibióticos (por ejemplo, tratar con antibióticos solo vacas con DV2 y DV3), en muchas vacas con DV2 y DV3 no se asilan bacterias e incluso está demostrado que en muchas vacas se desarrolla un proceso de autocuración sin necesidad de tratamiento.
En conclusión, el estudio demuestra que FT-IR tiene un buen grado de acuerdo con la marcha bacteriológica convencional. La base de datos construida con espectros de bacterias uterinas de vacas lecheras europeas puede ser utilizada para identificar bacterias uterinas de vacas lecheras de argentina.
Agradecimientos. A los productores y veterinarios que ayudaron en los muestreos. A las Doctoras Karen Wagener e Isabella Prunner, de la Universidad de Medicina Veterinaria de Viena, que colaboraron con el procesamiento de las muestras por TF-IR. A los Doctores Tom Gruner y Monica Ehling-Schulz, de la Universidad de Medicina Veterinaria de Viena, que participaron en la coordinación del transporte de las muestras y brindaron el equipamiento necesario para el TF-IR.
1. Beekes M, Lasch P, Naumann D. 2007. A nalytical a pplications of Fourier transform-infrared (FT-IR) spectroscopy in microbiology and prion research. Vet Microbiol 123: 305-319. [ Links ]
2. Bou G, Fernández Olmos A, García C, Sáez JA, Valdezate S. 2011. Métodos de identificación bacteriana en el laboratorio de microbiología. Rev Enferm Infecc Microbiol Clin 29: 601-608. [ Links ]
3. Grunert T, Wenning M, Barbagelata MS, Fricker M, Sordelli DO, Buzzola FR, Ehling M. 2013. Rapid and reliable identification of Staphylococcus aureus capsular serotypes by means of artificial neural network-assisted Fourier transform infrared spectroscopy. J Clin Microbiol 51: 2261-2266. [ Links ]
4. Grunert T, Monahan A, Lassnig C, Vogl C, Muller M, Ehling M. 2014. Deciphering host genotype-specific impacts on the metabolic fingerprint of Listeria monocytogenes by FT-IR spectroscopy. PLoS One 9:12 e115959. [ Links ]
5. Kasimanickam R, Duffield TF, Foster RA, Gartley CJ, Leslie KE, Walton JS, Johnson WH. 2005. The effect of a single administration of cephapirin or cloprostenol on the reproductive performance of dairy cows with subclinical endometritis. Theriogenology 63: 818-830. [ Links ]
6. Kirschner C. et al. 2001. Classification and identification of enterococci: a comparative phenotypic, genotypic, and vibrational spectroscopic study. J Clin Microbiol 39: 1763- 1770. [ Links ]
7. Kuhm AE, Suter D, Felleisen R, Rau J. 2009. Identification of Yersinia enterocolitica at the species and subspecies levels by Fourier transform infrared spectroscopy. Appl Environ Microbiol 75: 5809-5813. [ Links ]
8. Kummerle M, Scherer S, Seiler H. 1998. Rapid and reliable identification of food-borne yeasts by Fourier-transform infrared spectroscopy. Appl Environ Microbiol 64: 2207-2214. [ Links ]
9. Lauritsen J. 2000-2008. EpiData Data Entry, Data Management and basic Statistical Analysis System, EpiData Association, Odense (Denmark), Available from: http:// www.epidata.dk [ Links ]
10. LeBlanc SJ. 2008. Postpartum uterine disease and dairy herd reproductive performance: a review. Vet J 176: 102- 114. [ Links ]
11. Naumann D, Helm D, Labischinski H. 1991. Microbiological characterizations by FT-IR spectroscopy. Nature 351: 81-82. [ Links ]
12. Prunner I, Wagener K, Pothmann H, Ehling M, Drillich M. 2014. Risk factors for uterine diseases on small- and medium-sized dairy farms determined by clinical, bacteriological, and cytological examinations. Theriogenology 82: 857-865. [ Links ]
13. Samelis J, Bleicher A, Delbes C, Kakouri A, Neuhaus K, Montel MC. 2011. FT-IR-based polyphasic identification of lactic acid bacteria isolated from traditional Greek Graviera cheese. Food Microbiol 28: 76-83. [ Links ]
14. Santos TM, Caixeta LS, Machado VS, Rauf AK, Gilbert RO, Bicalho RC. 2010. Antimicrobial resistance and presence of virulence factor genes in Arcanobacterium pyogenes isolated from the uterus of postpartum dairy cows. Vet Microbiol 145: 84-89. [ Links ]
15. Santos TM, Gilbert RO, Caixeta LS, Machado VS, Teixeira LM, Bicalho RC. 2010. Susceptibility of Escherichia coli isolated from uteri of postpartum dairy cows to antibiotic and environmental bacteriophages. Part II: In vitro antimicrobial activity evaluation of a bacteriophage cocktail and several antibiotics. J Dairy Sci 93: 105-114. [ Links ]
16. Schabauer L, Wenning M, Huber H, Ehling M. 2014. Novel physico-chemical diagnostic tools for high throughput identification of bovine mastitis associated gram-positive, catalase-negative cocci. BMC Vet Res 10: 156. [ Links ]
17. Sheldon IM, Cronin J, Goetze L, Donofrio G, Schuberth HJ. 2009. Defining postpartum uterine disease and the mechanisms of infection and immunity in the female reproductive tract in cattle. Biol Reprod 81: 1025-1032.
18. Wagener K, Grunert T, Prunner I, Ehling M, Drillich M. 2014. Dynamics of uterine infections with Escherichia coli, Streptococcus uberis and Trueperella pyogenes in post-partum dairy cows and their association with clinical endometritis. Vet J 202: 527-532. [ Links ]
19. Wagener K, Prunner I, Pothmann H, Drillich M, Ehling M. 2015. Diversity and health status specific fluctuations of intrauterine microbial communities in postpartum dairy cows. Vet Microbiol 175: 286-293. [ Links ]
20. Walsh C, Williams EJ, Evans AC. 2003. Where will new antibiotics come from? Nat Rev Microbiol 1: 65-70. [ Links ]
21. Wenning M, Scherer S, Naumann D. 2008. Infrared spectroscopy in the identification of microorganisms. In: Vibrational spectroscopy for medical diagnosis, Diem MG, Chalmers JM, ed., John Wiley Sons, Chichester, 71-96. [ Links ]
22. Wenning M, Scherer S. 2013. Identification of microorganisms by FT-IR spectroscopy: perspectives and limitations of the method. Appl Microbiol Biotechnol 97: 7111- 7120. [ Links ]
23. Westermann S, Drillich M, Kaufmann TB, Madoz LV, Heuwieser W. 2010. A clinical approach to determine false positive findings of clinical endometritis by vaginoscopy by the use of uterine bacteriology and cytology in dairy cows. Theriogenology 74: 1248-1255. [ Links ]
24. Williams EJ, Fischer DP, Pfeiffer DU, England GC, Noakes DE, Dobson H, Sheldon MI. 2005. Clinical evaluation of postpartum vaginal mucus reflects uterine bacterial infection and the immune response in cattle. Theriogenology 63: 102-117.
25. Winn WC, Koneman EW. 2006. Konemans Color Atlas and Textbook of Diagnostic Microbiology, 6th. ed., Lippincott Williams Wilkins, Philadelphia, 1736 p. [ Links ]