I. INTRODUCCIÓN
Los procedimientos de medicina nuclear tanto de diagnóstico como los utilizados para el tratamiento de diversas enfermedades oncológicas ya se encuentran implementados para el uso diario en clínicas y centros médicos. En particular, aquellos procedimientos teranósticos, capaces de combinar el diagnóstico con el tratamiento, posibilita la opción de tratamiento guiado por imágenes, mejorando así tanto la precisión como la evaluación dosimétrica de los procedimientos.1,2
Actualmente, cuantificar la eficacia de un tratamiento demanda un gran esfuerzo, y aún no se dispone de mecanismos estandarizados de planificación para los procedimientos terapéuticos en medicina nuclear, debido a la variabilidad que existe en la absorción y distribución de los radiofármacos dependiendo del paciente. Para ajustar estos tratamientos a la situación particular de cada paciente se requiere de un seguimiento desde el comienzo de la terapia. Se requiere la estimación de la dosis absorbida, no sólo en el tejido a tratar sino también en los órganos considerados radio-sensibles, como es el caso de riñones o médula ósea.3
El cálculo de la dosis absorbida por el paciente puede abordarse por diversos métodos: i) cálculo Monte Carlo de transporte de radiación,4,5 ii) convolución de núcleos puntuales de dosis (DPK),6 o iii) por el método de valores  propuesto por el MIRD.7 En todos estos casos es necesaria la evaluación precisa de la actividad presente en cada órgano de interés y su evolución temporal en el paciente. El cálculo de la actividad en cuerpo completo a partir de la diferencia entre actividad administrada y la actividad eliminada por orina o sudor resulta entonces insuficiente para estimar la actividad acumulada en cada uno de los órganos de interés, tanto sanos como en riesgo. La utilización de procedimientos teranósticos permite entonces la cuantificación localizada a partir de imágenes metabólicas de Cámara Gamma, PET o SPECT.8
propuesto por el MIRD.7 En todos estos casos es necesaria la evaluación precisa de la actividad presente en cada órgano de interés y su evolución temporal en el paciente. El cálculo de la actividad en cuerpo completo a partir de la diferencia entre actividad administrada y la actividad eliminada por orina o sudor resulta entonces insuficiente para estimar la actividad acumulada en cada uno de los órganos de interés, tanto sanos como en riesgo. La utilización de procedimientos teranósticos permite entonces la cuantificación localizada a partir de imágenes metabólicas de Cámara Gamma, PET o SPECT.8
La segmentación precisa de estas imágenes sirve de guía para delimitar las regiones pertenecientes a cada órgano y tejido de interés, pero para realizarse requiere de personal altamente capacitado, y experimentado, y conlleva tiempos no siempre aceptables en la práctica clínica. Existen métodos semi-automáticos basados en técnicas propias de la ciencia de datos y la inteligencia artificial, como la utilización del clustering K-Means9 o Fuzzy C-Means.10 Estas técnicas no supervisadas son capaces de automatizar gran parte del trabajo, pero en ningún caso permiten prescindir del ojo entrenado del experto a los fines de lograr una segmentación óptima, con los tiempos que esto implica.
Asimismo, estas técnicas de aprendizaje automático se encuentran ampliamente difundidas en el campo del procesamiento digital de imágenes. El reconocimiento de objetos y regiones a partir de modelos previamente entrenados es de uso diario en la mayoría de las ramas de la industria, el comercio y la seguridad.
En este trabajo se estudia la posibilidad de generar un método de segmentación automático a los fines de complementar el trabajo del experto y acortar los tiempos necesarios en la actualidad. Se presenta un algoritmo heurístico automático de segmentación por medio de clustering utilizando dos métodos: K-Means y HDBSCAN.11
Los resultados obtenidos muestran la factibilidad de realizar el segmentado de las imágenes PET de forma automática y en tiempos aceptables para las prácticas clínicas. Éstos resultan promisorios para trabajos futuros en el campo de la segmentación automática de imágenes de medicina nuclear.
II. MÉTODOS Y MATERIALES
Imágenes PET/CT
Para el presente trabajo se utilizó un conjunto de imágenes PET/CT anonimizadas, que corresponden a estudios realizados en pacientes del centro médico INTECNUS en la ciudad de San Carlos de Bariloche. En todos los casos se trata de prácticas que han administrado al paciente radiotrazador basado en  con actividad por unidad de masa de 0.075 mCi/Kg. La adquisición de las imágenes se realizó utilizando un equipo marca General Electric modelo Discovery 710.
con actividad por unidad de masa de 0.075 mCi/Kg. La adquisición de las imágenes se realizó utilizando un equipo marca General Electric modelo Discovery 710.
Se concentró la atención en aplicar la metodología propuesta en imágenes de 4 pacientes, denominados 1, 2, 3 y 4 (ver Fig. 1).
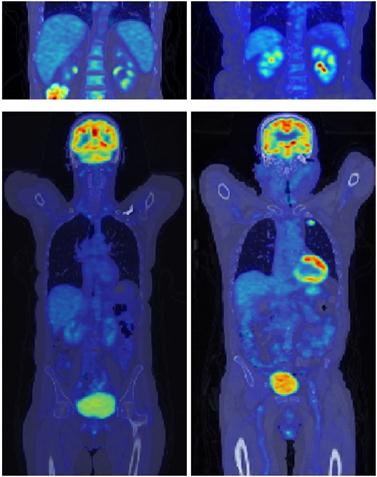
FIG. 1: Imágenes paciente-específico utilizadas. Paciente 1 (arriba-izquierda), paciente 2 (arriba-derecha), paciente 3 (abajo-izquierda) y paciente 4 (abajo-derecha).
La Tabla 1 resume las principales características de las imágenes empleadas. El tamaño de voxel para las imágenes PET es de  m m 3 y para las CT es de
m m 3 y para las CT es de  . A modo de ejemplo, la Fig. 1 reporta algunos cortes típicos.
. A modo de ejemplo, la Fig. 1 reporta algunos cortes típicos.
Algoritmos de Clustering
K-Means
El algoritmo de clustering K-Means consiste en encontrar  particiones de un conjunto de datos
particiones de un conjunto de datos  . En el presente caso, el conjunto de datos a particionar está compuesto por el valor de intensidad
. En el presente caso, el conjunto de datos a particionar está compuesto por el valor de intensidad  de cada voxel de la imagen junto a su posición, es decir que cada elemento del conjunto
de cada voxel de la imagen junto a su posición, es decir que cada elemento del conjunto  es un vector de 4 dimensiones
es un vector de 4 dimensiones  . Un cluster se define como un subconjunto de vóxeles tales que la distancia entre ellos es menor comparada con la distancia a puntos fuera del cluster. Por tanto, el problema de encontrar los
. Un cluster se define como un subconjunto de vóxeles tales que la distancia entre ellos es menor comparada con la distancia a puntos fuera del cluster. Por tanto, el problema de encontrar los  cluster se resume en resolver:
cluster se resume en resolver:
donde  se refiere a la “distancia intercluster".12
se refiere a la “distancia intercluster".12
HDBSCAN
El algoritmo de HDBSCAN11 (agrupación espacial basada en densidad jerárquica de aplicaciones con ruido) es una generalización de DBSCAN (agrupación espacial basada en densidad de aplicaciones con ruido). Ambos algoritmos comienzan buscando la distancia núcleo para cada punto, esta es la distancia entre el punto y un número de vecinos cercanos definidos por el hiperparámetro denominado número mínimo de muestras.
Luego, se forma un dendograma con los posibles clusters. HDBSCAN intenta mantener a los clusters tan grandes como sea posible, por medio del control del hiperparámetro “tamaño mínimo” de cluster.
La búsqueda del valor óptimo para los hiperparámetros que definen el desempeño del algoritmo se realiza maximizando el valor de la validez relativa para una dada configuración de tamaño mínimo de cluster y un número mínimo de muestras.13
Flujo de trabajo
En una primera instancia, al contar con la imagen original (cruda) de PET, es necesario descartar aquellos vóxeles que no pertenezcan a zonas de interés (tejidos), para lo cual se propone la implementación del método de clustering K-Means a los fines de segmentar la imagen en dos grandes regiones, pudiendo determinar aquellos vóxeles que pertenezcan a la zona a evaluar.
Una vez obtenida la primera segmentación, logrando separar los vóxeles entre aquellos que pertenecen a zonas con tejido de aquellos que no pertenecen a tejidos, resulta necesario implementar un nuevo criterio de segmentación para separar los vóxeles pertenecientes a zonas del cuerpo del paciente en dos grupos: a) aquellos que más probablemente pertenezcan a zonas activas de interés, y b) aquellos que puedan ser considerados ruido o fondo. A los fines del presente trabajo, y como resultado de pruebas preliminares, se propone utilizar segmentación por umbralamiento a nivel del tercer cuartil sobre la imagen de tejidos.
Una vez delimitados los tejidos de interés sobre los que se desea calcular la actividad acumulada, la imagen se suaviza utilizando un filtro de mediana que permite eliminar ruido. Asimismo, este filtro permite el relleno de posibles vóxeles “faltantes” en algunas zonas debido a incertezas intrínsecas de la técnica de adquisición de la imagen o la inhomogeneidad de la actividad, entre otras causas. Una vez suavizada, la imagen es re-dimensionada y normalizada a valores entre 0 y 1 para su posterior procesamiento y análisis.
La resolución espacial de las imágenes PET y CT suele ser diferente, incluso en equipamiento de imaging dual. Para esto, se recurre al método de resampling y se crean dos imágenes (anatómica y metabólica) de idéntica resolución espacial. A partir de la imagen anatómica, la que identifica aceptablemente las regiones y tejidos, y la metabólica, la que brinda información de la actividad en cada punto, y ambas imágenes en idéntica resolución espacial, es posible entonces incorporarlas como features en el método HDBSCAN. La matriz de features de entrada del algoritmo se construye a partir de las posiciones espaciales x, y y z, la intensidad del voxel de esa posición en la imagen PET y el correspondiente valor de intensidad (índice de Hounsfield) de la imagen anatómica (CT, en este estudio). Finalmente, se aplican diversas operaciones morfológicas a las etiquetas obtenidas de la aplicación del método HDBSCAN.
En la Fig. 2 se muestra el flujo de trabajo del modelo propuesto e implementado en el presente trabajo enfocado en imágenes PET. Como puede apreciarse, se trata de un método de segmentación automatizado, heurístico, que utiliza principalmente el algoritmo HDBSCAN sobre imágenes previamente procesadas por métodos clásicos (umbralamiento y filtro de mediana) y de primeros vecinos (K-Means).
III. RESULTADOS
Segmentación por K-Means
Se encuentra que, al realizar el primer paso donde se aplica K-Means sobre las imágenes originales, en todos los casos los resultados arrojan regiones donde se conservan las zonas más activas compuestas mayormente de tejidos activos de interés, descartando aquellas zonas que pueden ser consideradas fondo o ruido.
En la Fig. 3 se muestra el resultado de aplicar el segmentado por K-Means a las imágenes PET originales de los cuatros pacientes. Notar, que para una mejor visualización de los efectos de aplicar este método, en la Fig. 3 se muestran imágenes de los mismos cortes que en las imágenes originales de la Fig. 1.
Segmentación por umbralamiento y filtros
A los fines de realizar la siguiente etapa de segmentación por umbralamiento, resulta crítica la selección del valor de umbral. Con el propósito de proveer un abordaje objetivo, se analizaron los histogramas de las imágenes PET, tal como se muestra en la Fig. 4. Los histogramas de cada paciente resultan comparables entre sí para las imágenes localizadas, por un lado, y las de cuerpo completo, por otro.
En la Fig. 5 se muestran resultados de aplicar distintos valores de umbral para el paciente 1, aplicando 4 valores de percentiles distintos de umbral (25%, 50%, 75% y 85%). Como puede observarse, para el primer y segundo cuartil (imagen superior izquierda y derecha en la Fig. 5; respectivamente) el umbral resulta insuficiente, evidenciándose “rastros” de vóxeles activos que no resultan de interés para el estudio o que representan vóxeles de fondo. En cambio, para un valor de umbral muy alto como el del percentil 85 (imagen inferior derecha) se “pierden” vóxeles pertenecientes a tejidos de interés. El tercer cuartil (imagen inferior izquierda) muestra claramente que los vóxeles de fondo se encuetran eliminados y sólo se conservan aquellos pertenecientes a los tejidos de interés. Por ello, se definió para todos los estudios un valor de umbral igual al tercer cuartil. El valor de umbral utilizado para cada paciente se detalla en la Tabla 2.

FIG. 5: Distintos valores de umbral para la imagen PET del paciente 1 para el mismo corte que se ve en laFig. 3
Finalmente, antes de procesar la imagen con el algoritmo de HDBSCAN, es necesario lograr imágenes más realistas, por lo que se procede a un suavizado y un reescalado. En la Fig. 6 se muestran imágenes resultantes de este procedimiento.
Entrenamiento y segmentación definitiva
Una vez obtenidas las imágenes segmentadas por K-Means, descartados los vóxeles caracterizados como ruido y/o fondo a través del umbralamiento y suavizado de las imágenes, se construye la matriz de features que se utiliza como entrada en el entrenamiento del modelo de clustering HDBSCAN.
La matriz de entrada consiste de 5 características (features) ubicadas en columnas: 3 espaciales (x, y, z), intensidad del voxel de la imagen PET e intensidad de voxel de la imagen CT para cada valor (x, y, z), como se muestra en la Fig. 7. En el caso de estudio, debido a la cantidad de vóxeles de cada imagen, estas matrices tienen entre 60000 y 200000 entradas.
La búsqueda de los valores óptimos para los hiperparámetros del algoritmo HDBSCAN se hizo primeramente sobre el tamaño mínimo de cluster y luego, una vez definido este valor, se busca el valor óptimo para el número mínimo de muestras. En la Tabla 2 se muestran los hiperparámetros óptimos utilizados para el entrenamiento.
La Fig. 8 muestra los resultados obtenidos a lo largo de todo el procesamiento de las imágenes, en particular para el paciente 1. En la imagen superior izquierda se ve la imagen resultante de aplicar el segmentado por K-Means. Donde son fácilmente reconocibles los siguientes órganos: hígado, bazo, ambos riñones, médula ósea junto al bulk de actividad. La imagen superior derecha es la resultante de aplicar el umbral igual al tercer cuartil y posteriormente re-escalado y suavizado de la imagen. Finalmente, la imagen inferior muestra la segmentación obtenida de aplicar el algoritmo HDBSCAN, en ésta se puede apreciar cómo el método propuesto es capaz de segmentar e instanciar los distintos tejidos de interés en la imagen.
La Fig. 9 muestra el resultado para los mismos tres cortes para los cuatros pacientes que se presentan en la Fig. 6.
Como puede observarse en los resultados reportados en las Figs. 8 y 9, el modelo propuesto es capaz de segmentar e instanciar los distintos órganos. En el paciente 1 se observa el hígado, el bazo, la médula ósea, los riñones, vejiga; junto al bulk de actividad. En el caso del paciente 2, puede apreciarse que el corazón, el hígado y el riñón izquierdo, los que se segmentan como un sólo órgano, y el riñón derecho. En la figura del paciente 3, se observa el cerebro, el hígado, y la vejiga, entre otros. Para el paciente 4, también se observa el cerebro, el corazón, el hígado, la vejiga, parte de la médula y los riñones. Puede verse que en las imágenes de cuerpo completo la segmentación resulta más compleja, lo que puede asociarse a, al menos, los siguientes motivos: 1) hay un mayor número de tejidos a segmentar y 2) el tamaño de los tejidos a segmentar es muy distinto uno de otro, por ejemplo el cerebro es un tejido que se segmenta mucho más fácil que los riñones o la médula. Esta diferencia en tamaño repercute en la elección del hiperparámetro de tamaño mínimo de cluster. Un valor pequeño de éste ayudará a segmentar mejor la médula o los riñones pero dificultará el segmentado del cerebro o el hígado, caso contrario si se elige un valor muy grande. También puede verse que en la segmentación del hígado y del riñón derecho o del bazo y del riñón izquierdo no siempre se logra una performance óptima.
IV. CONCLUSIONES
El presente trabajo propone, desarrolla e implementa un procedimiento original, y automatizado, para la identificación de tejidos y órganos en imágenes PET-CT. La implementación de la metodología propuesta se realizó por medio de un flujo de trabajo especialmente adaptado. Una vez aplicado a un conjunto de imágenes PET-CT, el flujo de trabajo propuesto mostró ser una herramienta útil para el segmentado automatizado no supervisado de imágenes PET. Así mismo, se verificó la capacidad de HDBSCAN como método de segmentación de imágenes médicas. Dada la capacidad de este algoritmo para procesar grandes cantidades de datos de entrada, se consigue realizar la segmentación en tiempos cortos, esto es un requerimiento importante a la hora de realizar cálculos dosimétricos en ambiente clínico. Sin embargo, el mayor desafío es encontrar los valores de hiperparámetros óptimos que devuelven una segmentación de calidad. ésta es una arista a mejorar en trabajos futuros junto a la optimización del proceso de segmentación en imágenes de cuerpo completo.
























