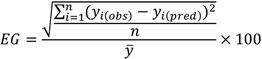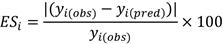INTRODUCCIÓN
Diversos métodos de muestreo pueden ser usados para identificar sitios a muestrear en estudios regionales. En este trabajo se evalúan métodos de muestreo en su capacidad para extraer muestras a partir de las cuales se ajustarán modelos de regresión para explicar la variabilidad de una característica de interés en función de propiedades edáficas y climáticas de los sitios muestreados. En términos generales, el muestreo de sitios en la ciencia de suelos puede orientarse hacia la recolección de datos con dos fines; el de estimar valores promedios poblacionales de una propiedad de interés, o de utilizar las muestras en la construcción de modelos para la predicción de valores en sitios no muestreados (modelos espacialmente explícitos). La metodología de muestreo adquiere especial relevancia para ambos fines, ya que la calidad de los resultados obtenidos dependerá de la selección de sitios muestreados (Carter & Gregorich, 2008). En el contexto del mapeo digital de suelo, donde la predicción de las propiedades de interés se basan en modelos esta- dísticos del tipo de regresión, el uso de estrategias de muestreo robustas se considera fundamental para obtener predicciones con mínima incertidumbre (McBratney et al., 2003).
A través del sensoramiento remoto, las imágenes satelitales y los modelos digitales de elevación, entre otras tecnologías, es posible contar con vasta cantidad de datos o variables auxiliares para mejorar estudios de variabilidad espacial de la característica de interés e incluso realizar predicciones de sus valores en sitios donde ésta no ha sido muestreada (predicción espacial). En ese contexto, es necesario que las estrategias de muestreo se adapten no solo a capturar la variabilidad de la variable de interés sino también, la variabilidad de las propiedades de sitio que será usada en la predicción espacial (Brus, 2019). Los modelos de regresión construidos a partir de estas muestras están siendo usados, por ejemplo, para predicciones de carbono orgánico del suelo (COS) a escalas regionales en todo el mundo (Yigini et al., 2018). Estas predicciones permiten obtener mapas digitales de la variabilidad del COS.
Desde un punto de vista estadístico, los muestreos de suelos pueden ser probabilísticos o dirigidos. En el muestreo probabilístico todas las unidades de la población a muestrear tienen una probabilidad positiva de ser seleccionada y se conoce a priori la probabilidad de selección de cada muestra. El muestreo dirigido, en cambio, se realiza con un diseño condicionado por la conveniencia, acceso a la muestra o incluso por el criterio experto y no se conoce a priori la probabilidad de selección de la muestra. Así, la selección de uno u otro tipo de muestreo es dependiente del fin o uso que tendrá la muestra. La estimación de una media poblacional se considera como un fin distinto a la construcción de un modelo de predicción de la variabilidad espacial de una propiedad edáfica.
En el mapeo digital de suelos (McBratney et al., 2003; Minasny & McBratney, 2016; Zhang et al., 2017) los muestreos para predicción espacial son dirigidos. Existen métodos de muestreos dirigidos que tienen como objetivo capturar la distribución multidimensional del conjunto de variables y así, representar mejor la variabilidad marginal en cada variable (Yigini et al., 2018). La técnica de muestreo denominada Hipercubo Latino condicionado (cLHS), desarrollado por Minasny & McBratney, (2006), es usada para determinar las muestras de sitios a considerar desde una población con abundante información auxiliar sobre cada sitio. Dados N sitios con información multidimensional, cLHS selecciona una muestra tal que existan representantes de todos los intervalos de clase de las distribuciones marginales que componen la distribución multivariada (Minasny & McBratney, 2006). A partir de la distribución multivariada inicial se genera una muestra “ideal” respecto a los rangos de variación de cada variable y, a través de métodos iterativos-heurísticos, se elige la combinación de sitios que mejor reproduce o representa esta muestra ideal. Existen estudios que exploran las bondades de cLHS a escalas regionales (Roudier, 2011; Mulder et al., 2013) en el contexto de estimación de parámetros. Carvalho Júnior et al. (2014) comparó el método con un muestreo aleatorio simple (MAS) logrando no solo capturar la variabilidad de las covariables sino también una mejor distribución geográfica de los sitios muestreados a escala regional. Sin embargo, el desempeño de cLHS en un contexto donde el objetivo es construir un modelo de predicción sitio-especifico ha sido menos explorado.
En la aproximación basada en modelos, a partir de la muestra de sitios, se determinará un modelo estadístico que relaciona las variables explicativas para la predicción espacial de una variable respuesta. Por ejemplo, un modelo estadístico que permita predecir valores de COS, en sitios donde no hay determinaciones de esta variable, en función de variables edáficas y climáticas del lugar y de su posición en el territorio, relativa a otros sitios donde existen determinaciones de COS. Además de existir distintos métodos de muestreos para identificar los sitios a muestrear, existen distintos métodos de regresión que pueden ser usadas para construir este modelo, uno usual es la que deviene de la geoestadística y es basado en un modelo de regresión kriging (Hengl et al., 2007). Otras posibilidades son ajustar un modelo de regresión lineal con errores correlacionados espacialmente (Cressie & Wikle, 2015) o un modelo de regresión de aprendizaje automático, como un árbol de regresión o un algoritmo de bosque aleatorio (random forest), incorporando dependencia espacial en el término residual (Li et al., 2011). Los árboles de regresión (Breiman, 2001) que son particularmente útiles para interpretar relaciones (no necesariamente lineales) en contextos de regresión múltiple con variables explicativas correlacionadas, pueden ser empoderados por la técnica de remuestreo para que los resultados no sean liderados por algunos datos influyentes de la muestra y entonces ampliar la generalización de la inferencia. El algoritmo de aprendizaje automático, conocido como Random Forest (RF) (Breiman, 2001) es un tipo de modelo de regresión que usa la técnica de remuestreo para construir modelos de árboles de regresión y proceder al ensamblaje de éstos para obtener finalmente un modelo de mayor capacidad predictiva que el modelo de árbol que deriva de la muestra original. RF está siendo usado con éxito en el mapeo digital de suelo (Wiesmeier et al., 2011; Guo et al., 2015; Vaysse & Lagacherie, 2015; Hengl et al., 2018). Para la aplicación de este método de regresión con datos espacialmente distribuidos, se combinan las predicciones RF con una interpolación geoestadística de los residuos del modelo realizada con un kriging ordinario (Li et al., 2011). En la mayoría de las implementaciones de RF para mapeo digital, se usan muestras provenientes de un MAS, pero sin explorar las distribuciones de las variables potencialmente usadas como predictoras. Las predicciones obtenidas por RF son luego usadas para construir mapas de variabilidad, continua en el espacio. El objetivo de este trabajo es evaluar el desempeño de la técnica de muestreo por cLHS para determinar las muestras de sitios a seleccionar en una región para construir modelos de regresión que permitan la predicción espacial y el mapeo digital de un atributo de interés, como el COS. Los modelos de regresión son construidos con los métodos de regresión lineal y RF para datos georreferenciados.
MATERIALES Y MÉTODOS
Población
La población que se muestreará se circunscribe en la provincia de Córdoba, Argentina (Figura 1) limitada por los paralelos de 29° y 35° de latitud sur y los meridianos de 61° y 65° de longitud oeste. El paisaje, está constituido predominantemente por planicies (aprox. 60%) y en menor proporción y hacia el oeste del territorio por cordones montañosos con dirección norte sur. La elevación varía entre 79 a 2884 msnm. El territorio es atravesado por las isohietas de 700 mm y de 500 mm determinando un gradiente de humedad en dirección Este-Oeste pasando por: húmedo, subhúmedo, semiárido y árido. La precipitación media anual varía entre 900 y 400 mm y la temperatura media anual oscila entre 10ºC y 24ºC. El balance hidrológico arroja valores de deficiencia hídrica anual que oscilan entre -80 mm y -480 mm. Los suelos son de origen loésico y en base al Soil Taxonomy se los agrupa en Molisoles (61%), Entisoles (13%), Alfisoles (7%) y Aridisoles (5%) (Jarsún et al., 2006). Sobre esta región se realizó una caracterización del horizonte superficial del suelo (0-15 cm) utilizando una grilla de 20 × 20 km2 que comprende un total de 352 sitios que reproducen las proporciones de los distintos órdenes de suelo en la Provincia (Hang et al., 2015). Para cada sitio se usaron datos de 12 variables edafoclimáticas georreferenciadas con coordenadas bidimensionales (Tabla 1).
Muestreos
Se muestrearon sitios de la población de referencia (Figura 1) utilizando dos métodos de muestreo, cLHS y MAS. Se usaron alternativamente 10 tamaños muestrales n=30, 60, 90, 120, 150, 180, 210, 240, 270 y 300 sitios. Para cada una de las 20 combinaciones de método de muestreo y tamaño muestral, se repitió el proceso de muestreo 30 veces, obteniendo 600 muestras en total (dos métodos de muestreo × 10 tamaños muestrales × 30 repeticiones del proceso). Para el muestreo por cLHS, se usaron las variables de la Tabla 1, excepto COS, para decidir los sitios a seleccionar y el paquete cLHS (Roudier, 2011) del software R (R Core Team, 2019). Para el MAS se usó la función sample de R para seleccionar una muestra aleatoria de sitios sin condicionar sobre el valor de las variables auxiliares.
Ajuste de modelos predictivos
Utilizando cada una de las 600 muestras obtenidas se ajustaron modelos de regresión para predecir COS en función de las restantes variables. Los modelos predictivos para COS se obtuvieron usando regresión lineal múltiple suponiendo errores independientes (RL), regresión lineal múltiple suponiendo errores correlacionados espacialmente según un modelo exponencial estimado por máxima verosimilitud restringida (RL-sp), random forest sin considerar correlación espacial (RF) y random forest con kriging ordinario sobre los residuos (RF-sp). Se utilizó el paquete nmle (Pinheiro et al., 2017) del software R para ajustar las regresiones lineales múltiples y los paquetes caret (Kuhn, 2012) y gstat (Pebesma, 2004) para ajustar RF y RF-sp. Para la implementación del algoritmo RF se optimizó, por validación cruzada 10-fold, el parámetro (cantidad de variables predictoras a evaluar en cada nodo del árbol). Para el algoritmo RF-sp además de optimizar el árbol de regresión, se ajustó un modelo espacial exponencial sobre los residuos y la predicción por kriging ordinario del residuo se sumó a la predicción RF según la propuesta de Li et al. (2011).
Criterios de comparación
El desempeño de los muestreos por cLHS y MAS se comparó a través de las predicciones de COS obtenidas para 30 sitios de la población diferentes a los muestreados y por tanto no usados para el ajuste del modelo predictivo. Se calculó, para cada método de muestreo y tamaño muestral, una medida de error de predicción global (promedio de los 30 sitios de validación) y la distribución de los errores sitio específicos.
La raíz cuadrada del error cuadrático medio de predicción fue expresada como porcentaje de la media de COS para obtener una medida de error de predicción global relativo a la media de la variable a predecir,
donde yi(obs) es el valor observado de COS en el sitio i, yi(pred) es el valor predicho de COS para el sitio i, n es el número de sitios de validación (n=30) e la media muestral de COS en los sitios de validación.
El error de predicción sitio-específico (ES) para el i-ésimo sitio, es la distancia entre el valor observado y el valor predicho de COS para ese sitio, que en términos relativos se expresó como,
RESULTADOS Y DISCUSIÓN
En la Figura 2 se muestra la tendencia descendente de los errores de predicción global observados a medida que se incrementa el tamaño muestral. Este decrecimiento del error de predicción con el tamaño de muestra es esperable y se produce con ambos métodos de muestreo. Sin embargo, la disminución del error en función del tamaño muestral no fue la misma en los distintos tipos de modelos de regresión. Mientras que en los modelos basados en árboles (RF y RF-sp) el valor esperado del error de predicción global muestra una proporcional al incremento del tamaño muestral, en los modelos de regresión lineal, las predicciones mejoraron hasta determinados tamaños muestrales (n=180) a partir de los cuales las predicciones se mantienen sin mejoras.
La regresión lineal requiere una muestra que haga evidente las relaciones lineales entre las predictoras y la variable respuesta, mientras que los algoritmos de aprendizaje automático no demandan relaciones lineales y mejoran la tasa de aprendizaje de las relaciones subyacentes a medida que se incrementa la cantidad de datos. Es importante resaltar que, en el ajuste de un modelo para predecir COS a partir de los datos de este trabajo, el desempeño de los métodos que contemplan variabilidad espacial en la estructura residual no fue sustancialmente mejor que el obtenido con modelos sin corrección por espacialidad (comparar RF-sp vs RF y RL vs RL-sp) (Tabla 2). Probablemente, la estructura espacial subyacente que genera variabilidad de COS se explicó directamente a partir de la estructura de variación de las variables explicativas, ya que las propiedades edáficas usualmente presentan altos coeficientes de autocorrelación espacial positiva (Gili, 2013; Zhang et al., 2017). En la predicción de COS realizada en este trabajo, la autocorrelación espacial fue significativa en todas las propiedades edáficas usadas como variables explicativas (índices de Moran entre 0,12 y 0,91, p>0,0001). Comparando los errores de predicción obtenidos con RF respecto a RF-sp (Tabla 2) se observó que la capacidad del algoritmo RF para contemplar distintos tipos de relaciones hizo innecesario la predicción espacial en los residuos del modelo. Luego, cuando el destino de la muestra, es crear un modelo para realizar predicciones espacialmente explícitas, a partir de variables predictoras estructuradas espacialmente, es posible ajustar un modelo adecuado para la predicción como RF, sin necesidad de usar recursos computacionales de modelado de correlaciones espaciales.
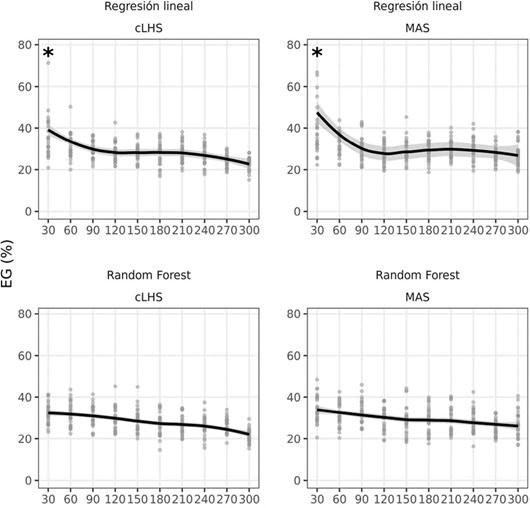
Figura 2 Error de predicción global expresado como porcentaje de la media de la variable que se predice, EG (%), en función del tamaño muestral para dos métodos de muestreo: cLHS (izquierda) y MAS (derecha) y para dos tipos de modelos de predicción sin corrección por espacialidad: Regresión lineal (arriba) y random forest (abajo). El asterisco, para el tamaño muestral n=30, indica que existen observaciones por encima de los intervalos graficados. Figure 2. Global prediction error expressed as a percentage of the predicted mean variable, EG (%), as a function of sample size for two sampling methods: cLHS (left) and MAS (right) and for two prediction types without spatial correction: linear regression (above) and random forest (below). The asterisk, for sample size n = 30, indicates that there are observations above the graphed intervals.
Los resultados observados en el ajuste de modelos de predicción de COS realizados en este trabajo son congruentes con las recomendaciones del método RF para construir predicciones a partir de múltiples capas de información de sitios (Breiman et al., 2017; Hengl et al., 2018)(Breiman et al., 2017; Hengl et al., 2018. Sin embargo, el método RF puede ser más afectado que la RL frente a la presencia de datos raros o en situaciones con poca variabilidad en las variables explicativas (Hengl et al., 2018). Por lo tanto, si bien el método RF puede ser usado para explicar la variabilidad de COS en función de características edafo-climáticas del sitio, también es importante complementar el procedimiento con un método de muestreo optimizado para capturar la variabilidad ambiental entre los sitios que participarán en la calibración del modelo RF.
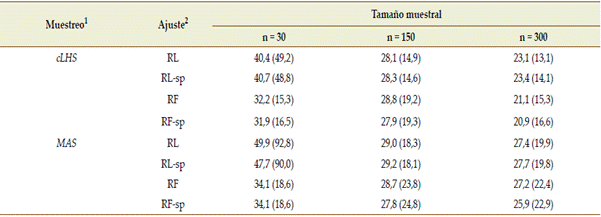
Tabla 2 Promedio de errores de predicción global (en porcentaje de la media a predecir) y coeficiente de variación de los errores de predicción (en paréntesis) para predicciones derivadas de dos métodos de muestreo: cLHS y MAS. Table 2. Global prediction errors average (as a percentage of the mean to predict) and prediction errors coefficient of variation (in parentheses) for predictions derived from two sampling methods: cLHS and MAS.
Comparando los métodos de muestreo cLHS y MAS, se observa que los errores de predicción mostraron mayor variabilidad cuando se usó el método MAS (Figuras 2 y 3). Esta es una característica no deseable ya que en la práctica sólo se obtiene una muestra y se espera que en esa muestra se registre un valor para la variable de interés similar al que podría haberse obtenido de cualquier otra muestra del mismo tamaño y de la misma población. Para tamaños muestrales pequeños, el método cLHS fue más eficiente que MAS. Cuando el modelo de predicción fue ajustado mediante regresión lineal; el error de predicción global (EG) estuvo entre 20,9% y 104% para n=30 con cLHS, mientras que, con muestras obtenidas por MAS del mismo tamaño, el EG varió entre 22,3% y 255%. La mayor amplitud en los rangos de variación del error de predicción global se observó para las muestras de menor tamaño, n=30 y n=60 (Figuras 2). En el ajuste de los modelos RF con muestras pequeñas, cLHS produjo, como es deseable, menos variación entre las predicciones de muestra a muestra que MAS (Figuras 2 y 3).
El método de muestreo cLHS tiende a seleccionar aquellas observaciones que mejor representen las distribuciones de todas las características (variables) del sitio medidas previamente (Mulder et al., 2013). En el contexto predictivo estas variables serán usadas como predictoras. Para el ajuste de modelos de regresión las muestras óptimas son aquellos que tienen mayor variación en las variables explicativas (Drapper & Smith, 1998). Los resultados de nuestro trabajo sugieren que es más probable que esta muestra óptima se produzca con cLHS que con MAS. La determinación de los sitios a muestrear realizada con cLHS produjo modelos con menores errores de predicción y menor variabilidad de muestra a muestra que los obtenidos de ajustes derivados de las muestras de sitios seleccionada por MAS (Figura 3).
Algunas recomendaciones derivadas del uso de modelos de regresión establecen que el tamaño muestral debiera ser mayor a 10×p, siendo p la cantidad de variables explicativas en el modelo de regresión a ajustar. Para el caso de estudio donde COS se predice a partir de 12 variables explicativas, se necesitaría un tamaño mínimo de 120 sitios, por eso a partir del tamaño muestral de 150 sitios los errores de predicción global no cambiaron sustancialmente entre métodos de muestreo. Para el tamaño muestral de 150 sitios, los errores de predicción global promediaron un 28,5% independientemente del método de muestreo usado (Tabla 2). En escenarios de variables respuestas con distribuciones más asimétricas que COS, la diferencia en EG entre cLHS respecto a MAS, ha sido mayor (Mulder et al., 2013). Mapeos digitales publicados para COS a escala regional han reportado EG por encima del 25% (Chai et al., 2008; Gomez et al., 2008; Meersmans et al., 2008; Stevens et al., 2010; Yigini et al., 2018). Los métodos de muestreo y tamaños muestra- les en esos mapeos digitales variaron en función de la escala usada, desde muestreos dirigidos con un sitio por unidad fisiográfica en mapeos a gran escala hasta muestreos sistemáticos. El método de muestreo cLHS garantiza mayor rango de variación en las predictoras producirá mayor estabilidad en los resultados, sobre todo cuando las muestras son pequeñas. Para una caída sensible del EG es importante analizar una a una las predictoras incorporadas. Por ejemplo, para COS menores errores de predicción podrían obtenerse con la incorporación de covariables de sitio con mayor potencialidad para explicar su variabilidad como son aquellas derivadas de sensores espectroscópicos del infrarrojo cercano (Zimmermann et al., 2007; Nocita et al., 2013).
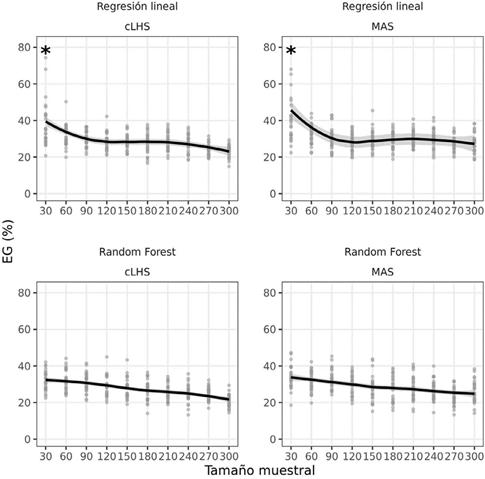
Figura 3 Error de predicción global expresado como porcentaje de la media de la variable que se predice, EG (%), en función del tamaño muestral para dos métodos de muestreo: cLHS (izquierda) y MAS (derecha) y para dos tipos de modelos de predicción con correlación espacial: Regresión lineal con errores correlacionados espacialmente (arriba) y random forest con kriging ordinario sobre los residuos (abajo). El asterisco, para el tamaño muestral n=30, indica que existen observaciones por encima de los intervalos graficados. Figure 3. Global prediction error expressed as a percentage of the predicted mean variable, EG (%), depending on the sample size for two sampling methods: cLHS (left) and MAS (right) and for two types of prediction models with spatial correlation: Linear regression with spatially correlated errors (above) and random forest with ordinary kriging on the residuals (below). The asterisk, for the sample size n = 30, indicates that there are observations above the graphed intervals
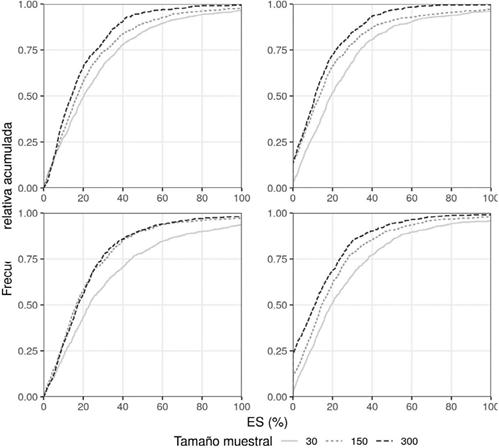
Figura 4 Distribución empírica de errores sitio-específico (ES), expresados como porcentaje de la media de la variable a predecir, para modelos de regresión (con correlación espacial) ajustados con datos obtenidos a partir de dos métodos de muestreo: cLHS (arriba) y MAS (abajo). Regresión lineal múltiple con errores correlacionados espacialmente (izquierda) y random forest con kriging ordinario sobre los residuos (derecha). Figure 4. Site-specific errors empirical distribution (ES), expressed as a percentage of the variable mean to be predicted, for regression models (with spatial correlation) adjusted with data obtained from two sampling methods: cLHS (above) and MAS (below). Multiple linear regression with spatially correlated errors (left) and random forest with ordinary kriging on the residuals (right).

Tabla 3 Indicador de la distribución de errores de predicción sitio específicos (área debajo de la curva de distribución empírica en el intervalo 0-100) para modelos ajustados con muestras de sitios seleccionados por dos técnicas de muestreo: muestreo por hipercubo latino condicionado (cHLS) y muestreo aleatorio simple (MAS) Table 3. Distribution of site-specific prediction errors indicator (area under the empirical distribution curve in the interval 0-100) for fitted models with sampled sites selected by two sampling techniques: conditional latin hypercube sampling (cHLS) and simple random sampling (MAS)
Respecto a la distribución de errores de predicción sitio-específicos (ES), también expresados como porcentaje de la media de COS, se observaron mejoras con el método de muestreo cLHS respecto al MAS (Figura 4). Comparado con el patrón del error de predicción global (Figura 2), se observó que la frecuencia de errores de menor valor fue mayor en los modelos derivados de muestreo por cLHS. En la Figura 4 se presentan las funciones de distribución empírica de los ES para cada método de muestreo, pero para escenarios donde el modelo de predicción se ajustó contemplando correlación espacial. Se observa que, para cLHS el 50% de los casos tuvo un error de predicción sitio-específico menor al 20% de la media de COS. Las formas de las curvas muestran que con cLHS la distribución de los errores mejora más con el incremento del tamaño de muestra que con MAS. En la Tabla 3 se muestran las áreas debajo de cada una de las curvas representadas en la Figura 4 como indicador del desempeño en términos de ES; un mayor valor de este indicador implica una mejor distribución de errores sitio-específicos. Independientemente, del tamaño muestral, el método de muestreo cLHS presenta una mejor distribución de errores de predicción que MAS.
Los resultados de este trabajo sugieren que el método de muestreo cLHS fue mejor que el MAS para predecir valores de COS en sitios sin determinaciones de la variable. El resultado es consistente con la presentación del método cLHS como una forma efectiva de obtener una muestra que representa bien no sólo la distribución de las variables originales sino también la relación entre ellas (Minasny & McBratney, 2006). Para tamaños muestrales pequeños, las muestras obtenidas por cLHS permitieron predecir la COS con menor variabilidad en el muestreo que las muestras obtenidas por MAS.
CONCLUSIONES
El método de muestreo cLHS es recomendable para determinar sitios a muestrear previo al ajuste de modelos de predicción para variables espacialmente distribuidas. El muestreo por cLHS selecciona sitios con mayor variabilidad en las variables explicativas produciendo menor incertidumbre en las predicciones de los valores de interés en lugares donde no fueron medidos. A partir de una muestra de 300 sitios identificados por cLHS, se obtuvieron predicciones adecuadas de COS desde variables edafoclimáticas. El modelo ajustado por el algoritmo Random Forest, a partir de los sitios determinados por cLHS, puede ser usado para el mapeo digital de COS a escala regional.