Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Boletín de la Sociedad Argentina de Botánica
versión On-line ISSN 1851-2372
Bol. Soc. Argent. Bot. vol.48 no.1 Córdoba ene./mayo 2013
GENÉTICA Y EVOLUCIÓN
Estudios citogenéticos en especies de Passiflora subgénero Passiflora (Passifloraceae)
Ana Laura Chiapero1, María Laura Las Peñas1*, María Teresa Amela García2 y Gabriel Bernardello1
1 Instituto Multidisciplinario de Biología Vegetal (UNC-CONICET).
2 DBBE, FCEyN (UBA), PROPLAME-PRHIDEB (CONICET).
* laulaspenas@yahoo.com.ar
Resumen: Passiflora comprende alrededor de 525 especies con x = 9 ó 6. Se realizó un estudio citogenético de cuatro especies del subgénero Passiflora que crecen en la Argentina. Se utilizaron las técnicas de Giemsa, bandeo cromosómico fluorescente CMA/DAPI y FISH (sonda 18-5,8-26S y 5S) en cromosomas mitóticos. P. caerulea, P. tucumanensis y P. cincinnata resultaron diploides (2n = 18) mientras que P. mooreana, tetraploide (2n=36). Los cariotipos fueron altamente simétricos con la fórmula 8 m+ 1 sm, duplicada en P. mooreana. Con CMA/DAPI, todos los diploides presentaron dos o tres pares de bandas terminales CMA + /DAPI - , mientras que la especie tetraploide presentó cinco pares, asociadas a NOR. Con FISH, los loci 18-5,8-26S coincidieron en número y posición con las bandas CMA + /DAPI - . En cada especie, el número de sitios para los genes de 5S ADNr concordaron con el nivel de ploidía. Los datos de P. tucumanensis y P. mooreana se reportan por primera vez, siendo esta última el primer tetraploide conocido para el subgénero Passiflora. Los patrones de FISH y las bandas heterocrómaticas variaron entre las especies de Passiflora estudiadas, principalmente en el número y distribución de las señales.
Palabras clave: Passiflora; Cariotipos; Heterocromatina; ADNr 5S and 18-5,8-26S.
Summary: Cytogenetic studies in species of Passiflora subgenus Passiflora (Passifloraceae). Passiflora has nearly 525 spp. with x = 9 or 6. A cytogenetic study of four species from subgenus Passiflora that grow in Argentina was performed. Giemsa technique, fluorescent chromosome banding CMA/DAPI and FISH (probe 18-5,8-26S and 5S) were used in mitotic chromosomes. P. caerulea, P. tucumanensis and P. cincinnata resulted diploid (2n = 18) while P. mooreana tetraploid (2n = 36). The karyotypes were highly symmetrical, with the formula 8 m+ 1 sm, duplicated in P. mooreana. With CMA/DAPI, diploids showed 2-3 pairs of terminal bands CMA + /DAPI - , whereas the tetraploid five bands, associated to NOR in all cases. With FISH, the loci 18-5.8-26S coincided in number and position with the CMA + /DAPI - bands. In each species, the number of sites for 5S rDNA genes agreed with the ploidy level. Data of P. tucumanensis and P. mooreana are here reported for the first time, being this last species the first known tetraploid for subgenus Passiflora. FISH patterns and heterochromatic bands varied among the Passiflora species studied, mainly with respect to the number and distribution of the signals.
Key words: Passiflora; Karyotypes; Heterochromatin; DNAr 18-5.8-26S and 5S.
Introducción
Passifloraceae Juss. ex Roussell cuenta con 18 géneros y 630 spp. distribuidas en regiones tropicales y subtropicales (Deginani, 2001). Se encuentra dividida en dos tribus: Pariopsae, africana, y Passiflorae, que crece en América, Asia y Oceanía. En ésta se ubica Passiflora, el género más representativo de la familia con aproximadamente 525 spp. americanas y 240 especies nativas del sudeste asiático y Nueva Zelandia (Killip, 1938; Feuillet & MacDougal, 2003, 2007). Son enredaderas herbáceas o leñosas, usualmente trepadoras con zarcillos axilares. El género es considerado monofilético, dividiéndose en 4 subgéneros (Deidamioides, Decaloba, Passiflora y Astrophea), 15 supersecciones, 31 secciones y 13 series, sistema que se adopta en este trabajo.
Las especies de Passiflora pueden ser repartidas en cuatro grupos cariológicos representados por x = 6 (número básico ancestral para el género), 9, 10 y 12 (Melo & Guerra, 2003; Hansen et al., 2006).
Sus dos linajes más importantes son los subgéneros Decaloba y Passiflora. Las especies de Decaloba son 214, en su mayoría enredaderas herbáceas con flores y frutos pequeños; son llamadas el grupo n = 6 (Snow & MacDougal, 1993), por ser este su número básico (Melo et al., 2001; Hansen et al., 2006). Dentro del subgénero Passiflora hay 236 spp., que se caracterizan por ser enredaderas leñosas con flores vistosas y frutos comestibles, siendo n = 9 su número cromosómico haploide más representativo (Hansen et al., 2006). Hasta el momento, se conocen los números cromosómicos de 23% de las especies de Passiflora (Souza et al., 2008) y el 58 % de las 19 citadas para Argentina (Deginani & Cervi, 2008; Bugallo, 2010), siendo diploides en su mayoría. Por otra parte, pocas especies han sido estudiadas con técnicas citogenéticas modernas. Con la técnica de bandeo fueron estudiadas apenas 8 spp. con n = 6 y 9 (Melo et al., 2001; Cuco et al., 2005), mientras que con la técnica de hibridación in situ fluorescente (FISH), analizaron la variación en los sitios ADNr 18-5,8-26S y 5S en 20 spp. de Brasil (Melo & Guerra, 2003).
Con estos antecedentes, el objetivo general de este trabajo es estudiar los cromosomas mitóticos de cuatro especies de Passiflora subgénero Passiflora que habitan en Argentina, con tinción clásica, bandeo fluorescente CMA/DAPI y FISH, atendiendo a sus características cualitativas y cuantitativas a fin de establecer sus cariotipos y las relaciones existentes entre ellas.
Materiales y Métodos
El material estudiado provino de localidades de Argentina: P. caerulea L., Buenos Aires- Cap. Fed., Amela García 517; La Rioja-Chilecito, Las Peñas 515; P. cincinnata Mast., Córdoba- Capital, Las Peñas 516; P. mooreana Hook. f., Córdoba- R. García, Las Peñas 517; P. tucumanensis Hook., Catamarca-Londres Cocucci 4538. Los ejemplares de herbario fueron depositados en el Museo Botánico de Córdoba (CORD) y determinados por Amela García, M. T.
Para la obtención de preparados cromosómicos se utilizaron raíces pretratadas con 8-Hidroxiquinoleína 2 mM durante 24 horas en heladera y fijadas posteriormente en solución Farmer (alcohol etílico:ácido acético glacial, 3:1). Las mismas fueron conservadas en freezer hasta su uso.
Coloración convencional: Los preparados se realizaron utilizando la técnica de HCl/ Giemsa (Guerra, 1983). Las raíces fijadas fueron aplastadas en ácido acético 45%. Posteriormente, los cubreobjetos se retiraron por congelamiento con N2, y finalmente, los preparados fueron coloreados con una solución de Giemsa al 2% preparada en el momento. Los cariogramas de cada célula se realizaron organizando los cromosomas en grupos de acuerdo a su índice braquial, siguiendo la terminología de Levan et al. (1964). La asimetría de los cariotipos fue estimada usando los índices de asimetría intracromosómica (A1) e intercromosómica (A2) propuestos por Romero Zarco (1986) y el índice de asimetría TF% propuesto por Huziwara (1962). Los satélites fueron clasificados según Battaglia (1955).
Bandeo cromosómico fluorescente CMA/DAPI: Las preparaciones se llevaron a cabo mediante digestión enzimática con celulasa 2% y pectinasa 20% a 37ºC durante 90 minutos, y fueron aplastadas con ácido acético 45% y se retiró el cubreobjetos por congelamiento con N2. El bandeo se realizó utilizando los fluorocromos cromomicina A3 (CMA) y 4'-6-diamidino-2-fenilindol (DAPI), siguiendo el protocolo descripto por Schwarzacher et al. (1980) con pequeñas modificaciones: los preparados fueron teñidos con CMA por 90 minutos, posteriormente con DAPI por 30 minutos, y finalmente montados con buffer MacIIvaine: glicerol (1:1).
FISH: Los preparados se llevaron a cabo de la misma manera que para el bandeo. Se siguió el protocolo de Schwarzacher & Heslop-Harrison (2000), utilizando la sonda pTa71 (Gerlach & Bedbrook, 1979) para la localización de los genes ribosómicos 18-5,8-26S y la sonda de ADNr 5S fue obtenida por PCR a partir de ADN genómico de Pereskia aculeata (Cactaceae, Las Peñas et al., 2010). El marcado de las sondas se realizó con Biotina (Bionick, Invitrogen) y Digoxigenina (DIG Nick translation mix, Roche) mediante la técnica de nick translation. Las señales de hibridación fueron detectadas utilizando Avidina-FITC (Sigma) y anti-DIG-Rodamina (Roche). Por último, los cromosomas se colorearon con DAPI.
Las fotomicrografías fueron tomadas en microscopio Zeiss Axiophot y cámara digital Leica DFC300FX. Para la adquisición de fotos de coloración convencional se utilizó contraste de fases, en tanto que para las de bandeo cromosómico y FISH se utilizó epifluorescencia con los filtros correspondientes para cada fluorocromo. Para el análisis morfométrico del cariotipo luego del bandeo y FISH, se efectuaron mediciones de los marcadores (bandas heterocromáticas) en 2-5 placas metafásicas correspondientes a 2-5 individuos de cada taxón, respectivamente. Estos datos se adicionaron a los idiogramas construidos en base a las mediciones cromosómicas de metafases teñidas convencionalmente, donde la medida de cada marcador está representada como porcentaje de la longitud del brazo cromosómico. Se calculó el porcentaje de heterocromatina asociada a regiones organizadoras nucleolares (% HC-NORs) en relación al largo total del genoma.
Resultados
Se encontró que ambas poblaciones de P. caerulea contaron con 2n =18, al igual que P. cincinnata y P. tucumanensis (Fig. 1 A, B); por otro lado, P. mooreana resultó tetraploide, con 2n = 36 (Fig. 1 C). La longitud promedio cromosómica fue de 1,73 μm, la longitud de mayor valor (2,43 μm) fue encontrada en el par 1 de P. caerulea y la de menor valor (1,13 μm) en el par 8 de P. tucumanensis. La longitud del genoma haploide varió entre 17,92 μm en P. caerulea y 13,00 μm en P. tucumanensis, siendo 15,46 μm su promedio. La relación entre el par cromosómico de mayor y de menor tamaño de las especies de Passiflora analizadas varió en un rango de 0,20-0,27 (Tabla 1).
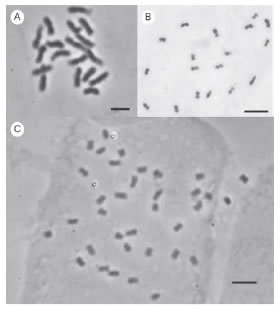
Fig. 1. Fotomicrografías de cromosomas mitóticos de Passiflora teñidos con Giemsa: A: P. caerulea, B: P. tucumanensis, C: P. mooreana. La escala representa 5 μm.
Tabla 1. Características cariotípicas de las especies de Passiflora analizadas. Números cromosómicos (2n), nivel de ploidía (X), fórmula cariotípica (FC), presencia de pares cromosómicos con satélites (Sat), largo total del genoma haploide (Lt), largo cromosómico promedio (C), la relación entre el par cromosómico de mayor y menor tamaño (R), Índice de asimetría intracromosómico (A1) e intercromosómico (A2) (Romero Zarco 1986), índice TF% (Huziwara 1962) y porcentaje de heterocromatina asociada a NORs (%HC NOR). Lt y C están expresados en μm.

P. caerulea y P. tucumanensis mostraron la misma fórmula cariotípica haploide (8 m+ 1 sm), en tanto que la especie tetraploide P. mooreana, presentó una fórmula cariotípica duplicada (16 m + 2 sm) (Fig. 3). En la Tabla 1, se observa que las especies estudiadas cuentan con cariotipos simétricos, lo cual se ve reflejado en los índices de asimetría calculados, con un rango de A1+A2 = 0,29-0,34 y TF % entre 43,16% y 44,73% para P. caerulea y P. tucumanensis, respectivamente. En todas las especies analizadas se encontraron microsatélites, siendo su visualización dificultosa con la técnica clásica, probablemente debido a su tamaño reducido (Figs. 1 y 2).

Fig. 2. Cromosomas metafásicos de Passiflora teñidos con doble tinción fluorescente CMA/DAPI (A-D) y FISH (E-H). A y E: P. caerulea, B y F: P cincinnata, C y G: P. mooreana y D y H: P. tucumanensis . Las flechas indican las bandas CMA+/DAPI-y las escalas representan 5 μm.

Fig. 3. Idiogramas de Passiflora. Los bloques negros indican las regiones de ADNr 18-5,8-26S (CMA+/ DAPI -asociadas a NORs) y los bloques grises representan los sitios 5S. La escala representa 3μm.
Con la técnica de bandeo fluorescente CMA/ DAPI, en todas las especies se detectaron bandas CMA+/DAPI- asociadas a regiones organizadoras nucleolares (NORs). En las dos poblaciones de P. caerulea estudiadas y en P. tucumanensis fueron observados tres pares cromosómicos con bandas CMA+/DAPI- (Fig. 2 A, C), en tanto que P. cincinnata presentó sólo 2 pares (Fig. 2 B). Finalmente, en P. mooreana se identificaron 5 pares cromosómicos con bandas CMA+/DAPI- (Fig. 2 D). Cabe destacar que en todos los casos pudieron observarse al menos un par de bandas de mayor tamaño, mientras que las restantes siempre fueron pequeñas. A partir de las fotografías obtenidas fue calculado el porcentaje de heterocromatina asociado a NORs, en relación al largo total del genoma. El valor más elevado fue 5,18% en P. caerulea y el menor fue 3,69% en P. mooreana (Tabla 1).
Por último, con la técnica de FISH se observó la localización de los genes ribosómicos. El ADNr 18-5,8-26S se colocalizó con el patrón de bandas CMA+/DAPI- asociadas a NORs (Figs. 2 E-H, 3). Los genes 5S se encontraron en número variable y posición paracentromérica en las distintas especies: P. caerulea, P. tucumanensis y P. cincinnata presentaron señales en un par cromosómico, mientras que en P. mooreana pudieron observarse cuatro sitios 5S. Dichas señales fueron siempre de tamaño muy reducido (Figs. 2 y 3) y estuvieron siempre localizados en pares cromosómicos distintos a los que contenían señales 18-5,8-26S (Fig. 3).
Discusión
Passiflora caerulea y P. cincinnata (2n = 18) fueron examinadas previamente, coincidiendo los números cromosómicos aquí hallados con recuentos previos en otras poblaciones (Janaki Ammal, 1945; Guerra, 1986; Melo et al., 2001; Bugallo, 2010). A la vez, se dan a conocer por primera vez los números cromosómicos de P. tucumanensis (2n = 18) y P. mooreana (2n = 36). Esta última resulta una novedad dentro del subgénero Passiflora ya que es la segunda especie poliploide conocida luego de Passiflora sp. con 2n = 72 proveniente del Estado de Pernambuco en Brasil (Melo et al., 2001). Los resultados obtenidos en este trabajo confirman x = 9 como el número básico característico del subgénero (Melo et al., 2001; Melo & Guerra, 2003; Souza et al., 2008).
Las especies con x = 9 (subgénero Passiflora) presentan cariotipos simétricos con 7 m+ 2 sm ó 8 m + 1 sm (Melo & Guerra, 2003; Hansen et al., 2006). Melo et al. (2001) reportan grandes diferencias entre los cariotipos; éstos difieren tanto en posición, como en número de constricciones secundarias, presentándose principalmente en posición terminal o sub-terminal en el brazo largo de dos o tres pares cromosómicos. Nuestros resultados indican que los caracteres cariológicos se ajustan a los descriptos para dicho subgénero (excepto P. mooreana, tetraploide).
La asimetría de los cariotipos fue calculada a través del índice TF% (Huziwara, 1962) en pocas especies de Passiflora. Del subgénero Passiflora previamente fueron analizadas sólo trece especies, con valores entre 46,78% para P. edmundoi y 38,68% para P. quadrangularis (Souza et al., 2003), rango dentro del cual se encontraron las especies de este estudio. En los estudios previos de bandeo en Passiflora, se hallaron bloques de HC CMA+/DAPI- en las regiones terminales de uno a tres pares cromosómicos, coincidiendo generalmente con la posición y número de satélites (Guerra, 2000; Cuco et al., 2005; Melo et al., 2001; Souza et al., 2008). En nuestros resultados, se observa que las especies diploides, efectivamente presentaron 2-3 pares cromosómicos con bloques de HC CMA+/DAPI- ubicados en las constricciones secundarias. Asimismo, no se encontraron diferencias entre las poblaciones de P. caerulea y P. cincinnata de Argentina y Brasil. Por otro lado, P. mooreana (poliploide) presentó cinco bloques coincidentes con el número de satélites. La única especie poliploide (2n = 8x = 72) estudiada hasta el momento, cuya identidad específica se desconoce, mostró quince bandas CMA+/DAPI-, observándose que el número de satélites aumenta junto al nivel de ploidía (Melo et al., 2001). Además, en este trabajo por primera vez en Passiflora se calculó el porcentaje de HC, observándose que dentro de las especies diploides no se observan grandes diferencias, mientras que en la especie poliploide, el porcentaje de HC fue menor.
Con la técnica de FISH, Melo & Guerra (2003) analizaron la variación en los sitios ADNr 5S y 18- 5,8-26S en 20 especies de Passiflora, observando que las diploides con x = 6 presentaron generalmente dos sitios ADNr 5S y dos o cuatro ADNr 18-5,8-26S, en relación con su grado de ploidía. Las especies con x = 9 frecuentemente mostraron más de dos locus 5S y 18-5,8-26S. De acuerdo con estos autores, en general, el número y localización de sitios 5S y 18-5,8-26S es consistente con la hipótesis de x = 6, como el grupo ancestral del género, mientras que los grupos con x = 9, 10 y 12 serían de origen tetraploide con disploidía descendiente y silenciamiento de genes de sitios redundantes, principalmente de ADNr 5S.
Las 11 spp. pertenecientes al subgénero Passiflora estudiadas por Melo & Guerra (2003) fueron diploides y mostraron 2-3 sitios 18-5,8-26S. A la vez, en todas las especies que estudiaron, tanto el número como posición de sitios 18-5,8-26S detectados por FISH, coincidió con las bandas CMA+/DAPI- (Melo et al. 2001, Melo & Guerra, 2003) y constricciones secundarias observadas en los cariotipos. Estos antecedentes concuerdan con los resultados obtenidos aquí para las 3 spp. diploides. Por otro lado, P. mooreana presentó diez loci 18-5,8-26S, también coincidentes con el número de satélites.
En varios géneros de angiospermas se hallaron correspondencias entre el número de locus ADNr 5S y el nivel de ploidía (Moscone et al., 1999; Adams et al., 2000; Taketa et al., 2005). En Passiflora, las especies diploides estudiadas por Melo & Guerra (2003) mostraron un par de loci 5S, que coincide con lo encontrado en P. caerulea, P. tucumanensis y P. cincinnata. Por otro lado, P. mooreana (tetraploide) presentó cuatro sitios 5S, número que se corresponde con su nivel de ploidía. A la vez, los poliploides tienen una tendencia a reducir los sitios duplicados debido a mecanismos de diploidización y silenciamiento génico (Leitch & Bennet, 1997; Levin, 2002). La presión de selección por reducir el número de sitios ADNr parece ser menor en los loci 18-5,8-26S, dada la existencia de mecanismos de homogeneización inter-loci más eficientes para esta secuencia que para la ADNr 5S (Cronn et al., 1996). Por este motivo, la probabilidad de hallar sitios 5S duplicados es mayor en poliploides infraespecíficos o en neopoliploides que en paleopoliploides. Estos antecedentes sugieren que la ploidización de P. mooreana sería de origen reciente.
Las especies del subgénero Passiflora analizadas hasta la fecha, incluyendo las de este estudio, mostraron que los cromosomas portadores de señales ADNr 5S fueron distintos de aquellos con señales ADNr 18-5,8-26S. Los patrones de FISH de genes ribosomales varían en Passiflora principalmente en referencia al número y distribución de las señales (Melo et al., 2001; Melo & Guerra, 2003; Souza et al., 2010). A la vez, la variación en el número de loci ADNr entre angiospermas es atribuida a rearreglos cromosómicos, eventos de transposición y silenciamiento génico (Moscone et al., 1999). De esta manera, mecanismos similares podrían estar actuando dentro del subgénero.
Passiflora tucumanensis fue estudiada por primera vez mostrando características comunes con P. caerulea, sin embargo se diferencian principalmente en el largo total del genoma haploide (Lt) y la posición de bandas de heterocromatina y genes ADNr 18-5,8-26S y 5S. Además, en este trabajo se encontró una especie tetraploide (P. mooreana) siendo este hallazgo raro para el género.
Todas las especies aquí analizadas pudieron ser distinguidas por sus características cariotípicas, distribución y porcentaje de heterocromatina y posición y número de loci ADNr. De esta manera, los hallazgos de este trabajo ponen en evidencia la importancia de la citogenética, tanto clásica como molecular, en la tarea de dilucidar las afinidades sistemáticas de las especies estudiadas, así como también para establecer tendencias evolutivas dentro de Passiflora en general.
Agradecimientos
Los autores desean expresar su agradecimiento a Andrea Cocucci por el material otorgado. Este trabajo ha sido financiado con subsidios otorgados por CONICET (PIP- 185) y SECYT- UNC.
Bibliografía
1. ADAMS, S. P., I. J. LEITCH, M. D. BENNETT, M. W. CHASE & A. R. LEITCH. 2000. Ribosomal DNA evolution and phylogeny in Aloe (Asphodelaceae). Amer. J. Bot. 87: 1578-1583. [ Links ]
2. BATTAGLIA, E. 1955. Chromosome, morphology and terminology. Caryologia 8: 179-187. [ Links ]
3. BERNARDELLO, G., L. STIEFKENS & M. L. LAS PEÑAS. 2008. Karyotype studies in Grabowskia and Phrodus (Solanaceae). Plant Syst. Evol. 275:265- 269. [ Links ]
4. BOWDEN, W. M. 1945. A list of chromosome numbers in higher plants. II. Menispermaceae to Verbenaceae. Amer. J. Bot. 32: 191-201. [ Links ]
5. BUGALLO, V. L. 2010. Hibridación interespecífica en el género Passiflora. Tesis de grado. FAUBA. Univ. Bs. As. [ Links ]
6. CRONN, R. C., S. ZHAO, A. H., PATERSON & J. F.WENDEL. 1996. Polymorphism and concerted evolution in a tandemly repeated gene family: 5S ribosomal DNA in diploid and allopolyploid cottons. J. molec. evol. 42: 685-705. [ Links ]
7. CUCO, S. M., M. L. C.VIEIRA, M. MONDIN, & M. L. R. AGUIAR- PERECIN. 2005. Comparative karyotype analysis of three Passiflora L. species and cytogenetic characterization of somatic hybrids. Caryologia. 58: 220-228. [ Links ]
8. DEGINANI, N. B. 2001. Las especies argentinas del genero Passiflora. Darwiniana 39: 43-129. [ Links ]
9. DEGINANI, N. B. & A. CERVI. 2008. Passifloraceae. In: ZULOAGA, F. O., O., MORRONE & M. J. BELGRANO (Eds.). Catálogo de las plantas Vasculares del Cono Sur. Volumen 2. Syst. Bot. Missouri Bot. Gard. 107. [ Links ]
10. DEGINANI, N. & A. ESCOBAR. 2002. Números cromosómicos de especies de Passiflora (Passifloraceae). Hickenia 3: 143-144. [ Links ]
11. FEUILLET, C & J. M. MACDOUGAL. 2007. In: Passifloraceae. Kubitzki, K. (Ed.). The families & genera of vascular plants. Flowering plants. Eudicots. Vol. 9. Springer. Berlin. [ Links ]
12. FEUILLET, C. & J. C. MACDOUGAL. 2003. A new infrageneric classification of Passiflora L. (Passifloraceae). Passiflora 13: 34-38. [ Links ]
13. GERLACH, W. L. & J. L. BEDBROOK. 1979. Cloning and characterization of ribosomal RNA genes from wheat and barley. Nucleic Acids Res. 7: 1869-1885. [ Links ]
14. GUERRA, M. 1983. O uso da Giemsa na citogenética vegetal. Cienc. Cult. 35: 190-193. [ Links ]
15. GUERRA, M. 1986. Citogenética de angiospermas coletadas em Pernambuco, I. Rev. Bras. Gen. 9, 21-40. [ Links ]
16. GUERRA, M. 2000. Patterns of heterochromatin distribution in plant chromosomes. Genet. Molec. Biol. 23: 1029-1041. [ Links ]
17. HANSEN, A. K., G. LAWRENCE, B. B. SIMPSON, S. R. DOWNIE, R. STEPHEN, A. C. CERVI & R. K. JANSEN. 2006. Phylogenetic relationships and chromosome number evolution in Passiflora. Syst. Bot. 31: 138-150. [ Links ]
18. HARVEY, M. 1966. IOPB chromosome number reports VII. Taxon 15: 155-163. [ Links ]
19. HEITZ, E. 1926. Der Nachweis der Chromosomen. Vergleichende Studien über ihre Zahl, Grösse und Form. Pflanzenreich. I. Z. Bot. 18: 625-681. [ Links ]
20. HUZIWARA, Y. 1962. Karyotype analysis in some genera of Compositae. VIII. Further studies on the chromosome of Aster. Amer. J. Bot. 49: 116-119. [ Links ]
21. JANAKI AMMAL, E. (eds.).1945. Chromosome atlas of cultivated plants. George Allen and Unwin Ltd. London. U.K. [ Links ]
22. KILLIP, E. P. 1938. The American species of Passifloraceae. Field Mus. Nat. Hist. Bot. Ser. 19: 1-613. [ Links ]
23. LAS PEÑAS, M. L., G. BERNARDELLO & R. KIESLING. 2010. Karyotype, heterochromatin, and physical mapping of 5S and 18-26S rDNA genes in Setiechinopsis mirabilis (Cactaceae). Haseltonia 16: 83-90. [ Links ]
24. LEITCH I. J. & M. D. BENNETT. 1997. Polyploidy in angiosperms. Trends Plant Sci. 2: 470-476. [ Links ]
25. LEVAN A., A. SANDBERG, & K. FREDGA. 1964. Nomenclature for centromeric position on chromosomes. Hereditas 52: 201-220. [ Links ]
26. LEVIN, D. A. (ed.). 2002. The role of chromosome change in plant evolution. Oxford University Press, New York. [ Links ]
27. MELO, N. F., A. C. CERVI & M. GUERRA. 2001. Karyology and cytotaxonomy of the genus Passiflora L. (Passifloraceae). Plant Syst. Evol. 226: 69-84. [ Links ]
28. MELO, N. F. & GUERRA, M. 2003. Variability of the 5S and 45S rDNA sites in Passiflora L. Species with distinct base chromosome numbers. Ann. Bot. 92: 309-316. [ Links ]
29. MORETTI, A. 1990. Karyotypic data on North and Central American Zamiaceae (Cycadales) and their phylogenetic implications. Amer. J. Bot. 77: 1016- 1029. [ Links ]
30. MOSCONE, E. A., F. KLEIN, M. LAMBROU, J. FUCHS, & D. SCHWEIZER. 1999. Quantitative karyotyping and dual-color FISH mapping of 5S and 18S-25S. rDNAprobes in the cultivated Phaseolus species (Leguminosae). Genome 42: 1224- 1233. [ Links ]
31. MUSCHNER, V. C., A. P. LORENZ; A.C. CERVI, S. L. BONATTO, T. T. SOUZA-CHIES, F. M. SALZANO & L. B. FREITAS. 2003. A first molecular phylogenetic analysis of Passiflora (Passifloraceae). Amer. J. Bot. 90:1229-1238. [ Links ]
32. ROMERO ZARCO, C. 1986. A new method for estimating karyotype asymmetry. Taxon 35: 556- 530. [ Links ]
33. SCHWARZACHER, T., P. AMBROS & D. SCHWEIZER. 1980. Application of Giemsa banding to orchid karyotype analysis. Plant Syst. Evol. 134: 293-297. [ Links ]
34. SCHWARZACHER, T. & P. HESLOP-HARRISON. 2000. Practical in situ hybridization. Bios Scientific Publishers Limited, Oxford. [ Links ]
35. SNOW, N. & MACDOUGAL, J. M. 1993. New chromosome reports in Passiflora (Passifloraceae). Syst. Bot. 18: 261-273. [ Links ]
36. SOUZA, M. M., T. N. SANTANA PEREIRA, L. DA CRUZ SILVA, D. SOARES DA SILVA REIS, & C. P. SUDRE. 2003. Karyoptype of six Passiflora species collected in the State of Rio de Janeiro. Cytologia (Tokyo) 68: 165-171. [ Links ]
37. SOUZA, M., G. PALOMINO,T. N. S.PEREIRA, M.PEREIRA & A. P. VIANA. 2004. Flow cytometric analysis of genome size variation in some Passiflora species. Hereditas 141: 31-38. [ Links ]
38. SOUZA, M. M., T. N. SANTANA PEREIRA & M. L. CARNEIRO VIEIRA. 2008. Cytogenetic studies in some species of Passiflora L. (Passifloraceae): A Review Emphasizing Brazilian Species. Braz. Arch. Biol. Technol. 51: 247-258. [ Links ]
39. SOUZA, M.M., J.D. URDAMPILLETA & E.R. FORNIMARTIN. 2010. Improvements in cytological preparations for fluorescent in situ hybridization in Passiflora. Gent. Molec. Res. 9: 2148-2155. [ Links ]
40. STEBBINS, G. L. (ed.). 1971. Chromosomal evolution in higher plants. Edward Arnold, London. [ Links ]
41. STOREY, W.B. 1950. Chromosome numbers of some species of Passiflora occurring in Hawaii. Pacific Sci. 4: 37-42. [ Links ]
42. SOLTIS, D., P. SOLTIS, P. ENDRESS & M.CHASE. (eds.). 2005. Phylogeny and Evolution of Angiosperms. Sinauer Associates, Massachusetts, USA. [ Links ]
43. TAKETA, S., H. ANDO, K. TAKEDA, M. ICHII & R.VON-BOTHMER. 2005. Ancestry of American polyploid Hordeum species with I genome inferred from 5S and 18S-25S rDNA. Ann. Bot. 96: 23-33. [ Links ]
44. YOCKTENG, R. & S. NADOT. 2004. Phylogenetic relationships among Passiflora species based on the glutamine synthetase nuclear gene expressed in chloroplast (ncpGS). Molec. Phylogenet. Evol. 31: 379-396. [ Links ]
Recibido el 3 de febrero de 2012,
aceptado el 20 de septiembre de 2012.













