Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Boletín de la Sociedad Argentina de Botánica
versión On-line ISSN 1851-2372
Bol. Soc. Argent. Bot. vol.49 no.4 Córdoba dic. 2014
GENÉTICA Y EVOLUCIÓN
Divergencia histórica en Anadenanthera colubrina var. cebil (Leguminosae) analizando una región intrónica del ADN cloroplástico
Verónica Calonga Solís1,2,3, María Eugenia Barrandeguy1,2,4 y María Victoria García1,2,4*
1 Departamento de Genética, Facultad de Ciencias Exactas, Químicas y Naturales, Universidad Nacional de Misiones. 3300 Posadas, Misiones.
2 Instituto de Biología Subtropical, UNaM - CONICET.
3 Comité Ejecutivo de Desarrollo e Innovación Tecnológica de Misiones (CEDIT).
4 Consejo Nacional de Investigaciones Científicas y Técnicas (CONICET).
* Autor para correspondencia. Genética de Poblaciones y Cuantitativa - Depto. Genética - Facultad de Ciencias Exactas, Químicas y Naturales, Universidad Nacional de Misiones, Félix de Azara 1552, 3300 Posadas, Misiones. E-mail: vgarcia@fceqyn.unam.edu.ar +54-376-4437023 int. 167.
Resumen
Anadenanthera colubrina var. cebil es una especie forestal nativa de América del Sur. Presenta una distribución amplia y disyunta que se corresponde con la distribución de los Bosques Tropicales Estacionalmente Secos. Para estudiar el posible efecto de los cambios climáticos del Pleistoceno sobre la distribución de A. colubrina var cebil se analizó la secuencia del intrón trnL del ADN cloroplástico en 48 individuos provenientes de las Provincias Fitogeográficas Paranaense y de las Yungas. Se identificó un SNP en la posición 501 que permitió definir dos haplotipos fijados cada uno de ellos en la región fitogeográfica de origen de los individuos. Las secuencias del intrón trnL para estos haplotipos fueron contrastadas con secuencias de esta región disponible en la base pública de datos GenBank. A partir de este conjunto de secuencias se definieron otros tres haplotipos. Se construyó una red para determinar las relaciones filogenéticas. Las estimaciones de distancia genética entre los haplotipos tomados de a pares presentaron valores entre 0 y 0,017 en tanto que el tiempo de divergencia entre los haplotipos presentó valores comprendidos entre 0 y 5.600.000 años, aproximadamente.
Palabras clave: Anadenanthera colubrina var. cebil; Cambios climáticos históricos; ADN cloroplástico.
Summary
Historical divergence in Anadenanthera colubrina var. cebil (Leguminosae) analyzing an intronic region of chloroplast DNA. Anadenanthera colubrina var. cebil is a native woody tree from South America which presents a discontinuous distribution which is similar to that observed for Seasonally Dry Tropical Forests (SDTFs). To evaluate the probable effects of climate changes during Pleistocene in A. colubrina var cebil distribution, the chloroplast trnL intron sequence of 48 individuals from Paranaense and Yungas phytogeographic provinces were analyzed. A SNP in the position 501 defined two haplotypes. These haplotypes were fixed according to phytogeographic provinces of individual's origin. The sequences from the trnL intron of these haplotypes were compared with sequences from the same region available in GenBank public database. Three more haplotypes were defined based on the alignment analyses of these sequences. A network was constructed to determine the phylogenetic relationships between haplotypes. The estimation of the genetic distance between haplotypes reached values among 0 and 0.017, while the divergence time between haplotypes was estimated among 0 and 5,600,000 years.
Key words: Anadenanthera colubrina var. cebil; Historical climate changes; Chloroplast DNA.
Introducción
Los eventos históricos pueden tener efectos profundos sobre la distribución de la variación genética ya que la estructuración genética entre las poblaciones se encuentra influenciada tanto por el intercambio genético contemporáneo como por las relaciones ancestrales entre las mismas (Schaal, 1998; Hewitt, 2000). Los componentes espaciales y temporales de la estructuración genética pueden ser descifrados a través de estudios filogeográficos (Beheregaray, 2008) debido a que ellos analizan la estructura genética y demográfica de las poblaciones de una especie en un contexto geográfico. Dichos estudios tienen como objetivo principal el entendimiento de los factores históricos que contribuyeron a la divergencia entre las poblaciones (Eckert, 2011) permitiendo explicar la evolución de las especies dentro de los biomas (Ramos et al., 2007). De este modo, es posible distinguir entre escenarios históricos alternativos para explicar la distribución geográfica contemporánea de la variación genética (Avise, 2009).
Anadenanthera colubrina (Vell.) Brenan var. cebil (Griseb) Altschul es una especie forestal nativa de América del Sur, localmente conocida como curupay, cebil colorado o angico, que pertenece al orden Fabales, familia Leguminosae, subfamilia Mimosoideae, tribu Mimoseae (Cialdella, 2000). Es una especie exclusivamente heliófita, que generalmente forma parte del dosel arbóreo, llegando a alcanzar entre 15 y 30 m de altura (Justiniano& Fredericksen, 1998).
A. colubrina var. cebil presenta una distribución amplia y disyunta, comprendiendo las provincias fitogeográficas Paranaense y de las Yungas (Cialdella, 2000). Su distribución se corresponde con los Bosques Tropicales Estacionalmente Secos (Seasoally Dry Tropical Forests-SDTFs) (Prado& Gibbs, 1993). Se definen tres núcleos en la distribución de A. colubrina var. cebil en estos bosques: El núcleo Caatinga, ubicado en el NE de Brasil, donde la especie es abundante. El núcleo Misiones, el cual se encuentra en el sistema de los Ríos Paraná y Paraguay, donde la especie es común, sin embargo no es dominante. En tanto que el núcleo Pedemonte Subandino se localiza al SO de Bolivia y al NO de Argentina, donde A. colubrina var. cebil se encuentra entre las cinco especies más comunes (Prado& Gibbs, 1993; Prado, 2000) (Fig. 1).

Fig. 1: Distribución geográfica contemporánea de Anadenanthera colubrina var. cebil: 1) Núcleo Caatinga, 2) Núcleo Misiones y 3) Núcleo Pedemonte Subandino. Imagen adaptada desde Prado& Gibbs (1993).
En estudios previos realizados en poblaciones naturales de A. colubrina var. cebil del norte argentino, empleando marcadores de tipo microsatélite, se identificó estructuración genética tanto en el genoma cloroplástico como en el genoma nuclear. Estos resultados reflejarían los efectos de la fragmentación histórica (Barrandeguy et al., 2014).
Prado& Gibbs (1993) sobre la base de un registro de más de 80 especies distribuidas en los SDTFs y antecedentes climatológicos, que señalan un clima seco y frío en América del Sur durante el Pleistoceno, postularon la hipótesis del Arco Pleistocénico. Esta hipótesis plantea que la distribución contemporánea disyunta de estos bosques (Fig. 1) representa remanentes fragmentarios de un bosque cuya distribución era extensa y continua durante el Pleistoceno. De este modo, en el Pleistoceno tardío podría haber ocurrido una sustitución del tipo de vegetación como consecuencia de la contracción y fragmentación de los bosques húmedos y de la expansión de los bosques secos.
Pennington et al. (2000) apoyan y contribuyen a la hipótesis del Arco Pleistocénico señalando la presencia en la Selva Amazónica de especies propias de los SDTFs, con lo cual sugieren que estas especies podrían haber colonizado la cuenca Amazónica durante el período Pleistocénico. Recientemente, Werneck et al. (2011) plantean que los efectos de las fluctuaciones climáticas del Cuaternario han sido sobreestimados. Estos autores elaboraron mapas de estabilidad de los SDTF a partir de modelos de paleodistribución utilizando variables climáticas correspondientes al último máximo glacial (Last Glacial Maximum-LGM), al Holoceno y al presente, los cuales fueron validados mediante registros paleopalinológicos de especies características de estos bosques. De este modo, estos autores sugieren que los SDTFs sufrieron una expansión en el Pleistoceno Temprano o en el Terciario, seguida de una fragmentación hace 21.000 años en el LGM, pasando luego por una expansión secundaria en el Holoceno (6.000 años atrás). Sobre la base de este modelo, estos autores proponen cuatro refugios potenciales de estabilidad actual y a largo plazo: el núcleo Misiones, la región del Chiquitano ubicada al E de Bolivia, un refugio al SE del estado de Mato Grosso en Brasil y el núcleo Caatinga. A. colubrina var. cebil es considerada la especie más paradigmática en los SDTFs por presentar una distribución amplia en los mismos. La superposición de su distribución con la de otras especies forestales ha llevado a la propuesta de una nueva región fitogeográfica llamada Dominio de los Bosques Secos Estacionales Neotropicales (Prado, 2000).
La reconstrucción histórica de los eventos que han operado sobre la distribución geográfica contemporánea de la variación genética requiere de la identificación de variabilidad producida por mutaciones en regiones de ADN que no presenten recombinación meiótica, es decir, identificar variabilidad en aquellas regiones donde la mutación sea su única fuente de variación (Avise, 2009). Bajo este marco teórico, la mayoría de estos estudios en las angiospermas se basan en el análisis de la variabilidad presente en el genoma cloroplástico (ADNcp) debido a que este genoma no sufre recombinación reteniendo, por esto, las huellas de su historia evolutiva en mayor proporción que el genoma nuclear (Ravi et al., 2008). El ADNcp es heredado por vía materna en la mayoría de las angiospermas (Corriveau& Coleman, 1988) determinando, por lo tanto, que el flujo de los genes transmitidos por esta vía ocurra sólo a través de la dispersión de las semillas (Ennos, 1994). De esta manera, los marcadores heredados por vía materna y dispersados únicamente por semilla revelan grupos genéticos distintos cuyo origen puede asociarse a procesos históricos (Petit& Vendramin, 2007; Tollefsrud et al., 2009) siendo el análisis de la variación genética del ADNcp una herramienta adecuada para la reconstrucción de eventos demográficos, tales como expansión y contracción poblacional, migración y colonización (Ramos et al., 2009). Así, los eventos demográficos podrían explicar la distribución geográfica contemporánea de la distribución de la variación genética. Sin embargo, los estudios filogeográficos en plantas son difíciles de desarrollar, debido a que la evolución del genoma cloroplástico es lenta (Petit& Vendramin, 2007).
En este trabajo se estudió el posible efecto de los cambios climáticos del Pleistoceno sobre la distribución de A. colubrina var cebil caracterizando la variabilidad genética cloroplástica y definiendo haplotipos a partir de los cuales se determinaron las relaciones filogenéticas y se estimó el tiempo aproximado de divergencia.
Materiales y Métodos
Area de estudio y metodología de muestreo
Se colectaron hojas jóvenes de 48 individuos localizados en las provincias fitogeográficas Paranaense y de las Yungas. En la provincia fitogeográfica Paranaense se consideraron las poblaciones Candelaria (27° 26' 58,200" S, 55° 44' 20,220" O) y Santa Ana (27° 25' 55,920" S, 55° 34' 16,680" O). Por su parte, en la provincia fitogeográfica de las Yungas se consideraron las poblaciones: Horco Molle (26° 47' 26,100" S, 65° 18' 58,140" O), Calilegua (23° 45' 15,012" S, 64° 51' 12,996" O), Guasayán (28° 08' 13,880'' S, 64° 49' 27,580'' O) y Catamarca (28° 1' 1,595'' S, 65° 41' 54,888'' O). El muestreo se realizó considerando 8 individuos adultos en cada población, ubicados a una distancia de separación mínima de 20 metros. Los individuos fueron geo-referenciados mediante el sistema de posicionamiento global (GPS). Muestras de estos individuos fueron depositadas en el herbario del Museo de Ciencias Naturales de la Universidad Nacional de Salta.
Obtención del intrón trnL en A. colubrina var. cebil
Se analizó la secuencia del intrón trnL del ADNcp dado que fue utilizado en estudios previos realizados en otras leguminosas (Ribeiro et al., 2011). Para su amplificación se emplearon los cebadores universales descriptos por Taberlet et al. (1991), autores que describen este intrón en el genoma cloroplástico de Nicotiana tabacum.
El ADN genómico total fue extraído utilizando el protocolo descripto por Steiner et al. (1995) y modificado por García et al. (2007). La calidad y cantidad del ADN fue verificada en geles de agarosa al 1%. El intrón trnL se amplificó mediante la reacción en cadena de la polimerasa (PCR) en un volumen final de 30 µl empleando 1X de Buffer de ClK, 2 mM de Cl2Mg, 0,2 mM de cada dNTP, 0,6 µM de cada cebador (sentido y antisentido), 1U de Taq Polimerasa y 0,6 ng de ADN. Las condiciones de ciclado fueron: desnaturalización inicial a 94°C por 4 min, seguida de 35 ciclos de: desnaturalización a 94°C por 1 min, hibridación a 58°C por 1 min, elongación a 72°C por 1 min, y una elongación final a 72°C por 10 min utilizando un termociclador marca Techne. Los productos de amplificación fueron purificados aplicando un protocolo de precipitación con polietilenglicol (PEG).
Las secuencias sentido y antisentido de cada individuo fueron controladas y editadas a mano para minimizar errores utilizando el programa Chromas Lite 2.01. Los alineamientos de múltiples secuencias se realizaron empleando el programa Clustal X (Thompson et al., 1997). Las secuencias obtenidas fueron comparadas con secuencias existentes en la base pública de datos GenBank utilizando el algoritmo BLAST para corroborar que los amplicones obtenidos corresponden al intrón trnL.
Búsqueda de secuencias del intrón trnL de A. colubrina en la base pública de datos GenBank
Se seleccionaron secuencias del intrón trnL con un 99% de identidad pertenecientes a Anadenanthera colubrina con el objeto de incrementar los niveles de variación genética a ser analizados. La información disponible en la base pública para cada una de las secuencias incorporadas fue enriquecida mediante consultas a los diferentes herbarios donde se encuentran alojados los ejemplares.
Análisis estadístico de los datos
Se identificaron polimorfismos de nucleótido simple (SNPs) y de tipo indels y mediante la combinación de las variantes polimórficas se definieron los haplotipos correspondientes.
Para cuantificar la variabilidad genética se estimó el número medio de diferencias nucleotídicas entre secuencias mediante la ecuación: k = Σmi=1hi donde, m es el número total de posiciones nucleotídicas (incluyendo las monomórficas, pero excluyendo los sitios con indels), h es la heterocigosis en el sitio i, la cual toma valores entre 0 - 0,5 y se calcula como: hi = [n/(n-1)][1-Σ4j=1 x2ij] donde, n es el número de secuencias analizadas y xij es la frecuencia relativa de la variante nucleotídica j (j = A, C, G o T) en el sitio i (Rozas, 2009).
Para determinar las relaciones filogenéticas entre los haplotipos se construyó una red empleando el algoritmo Median-Joining (MJ) (Bandelt et al., 1999). Este análisis fue realizado empleando el programa Network versión 4.611 (Forster et al., 2001).
Se estimó el tiempo de divergencia entre los haplotipos mediante la ecuación: T = dA/2µ donde, dA es la distancia genética entre secuencias y µ es la tasa de substitución por sitio (Ribeiro et al, 2011). Dado que se estima que la tasa de mutación nucleotídica por año en angiospermas fluctúa entre 1 x 10-9 y 3 x 10-9 (Provan et al., 1999) para este trabajo se consideró un valor de 2 x 10-9. Para estimar la distancia genética (dA) entre los haplotipos se aplicó el modelo Kimura 2-parámetros (Nei& Kumar, 2000) empleando el programa Mega versión 5 (Tamura et al., 2011). Luego mediante cálculo manual se obtuvo el valor del tiempo de divergencia T.
Resultados
Las secuencias obtenidas tuvieron una longitud promedio de 600 pb y presentaron un alto contenido de AT (63,7%), tal lo esperado en los genomas cloroplásticos (Ravi et al., 2008). Mediante la comparación con las secuencias de la base pública de datos GenBank se estableció que dichas secuencias corresponden al intrón trnL, lo cual permitió su inclusión en dicha base de datos (KF787090).
Se obtuvo una secuencia consenso para cada individuo resultante del alineamiento de las secuencias sentido y antisentido. El alineamiento de la totalidad de las secuencias obtenidas permitió identificar un SNP en la posición 501, originado por una transición G - A. Este SNP definió dos haplotipos: Haplotipo Parananense (HP), portado por los individuos provenientes de la provincia fitogeográfica Paranaense, el cual presenta el nucleótido A en la mencionada posición, y el haplotipo Yungas (HY), portado por los individuos provenientes de la provincia fitogeográfica de las Yungas, el cual porta el nucleótido G en dicha posición.
Se hallaron tres secuencias del intrón trnL pertenecientes a A. colubrina a partir de la búsqueda en la base pública de datos GenBank (Tabla 1).
Tabla 1: Secuencias del intrón trnL del ADNcp de A. colubrina disponibles en la base pública de datos GenBank.

1Identidad: Porcentaje de nucleótidos iguales entre dos secuencias en una misma posición del alineamiento.
El alineamiento de las secuencias para los haplotipos HP y HY con las tres secuencias halladas en la base pública de datos definió un fragmento de 537 pb. A lo largo de dichas secuencias alineadas se identificaron 14 sitios polimórficos: tres transiciones, siete transversiones y cuatro indels (Tabla 2). Siendo mayor la ocurrencia de transversiones (50%) que de transiciones (21%). El número medio de diferencias nucleotídicas entre las secuencias fue de 4 y la heterocigosis por sitio fue de 0,4. A partir de este conjunto de secuencias alineadas se definieron otros tres haplotipos: Bolivia (HBo), Brasil (HBr) y Salta (HS) (Tabla 2).
Tabla 2: Sitios polimórficos en la totalidad de las secuencias analizadas.

(.) bases idénticas; (-) deleciones.
En la red construida para determinar las relaciones filogenéticas entre los haplotipos la posición central fue ocupada por HY mientras que, los haplotipos HP, HBo y HBr, se diferenciaron de HY por un único evento mutacional, consistiendo estas diferencias en una transición para el primer caso, un indel para el segundo y una transversión para el tercero. Mientras que el haplotipo HS se diferenció de HY por 11 eventos mutacionales (3 indels, 6 transversiones y 2 transiciones) localizándose en una posición distal al resto de los haplotipos (Fig. 2).
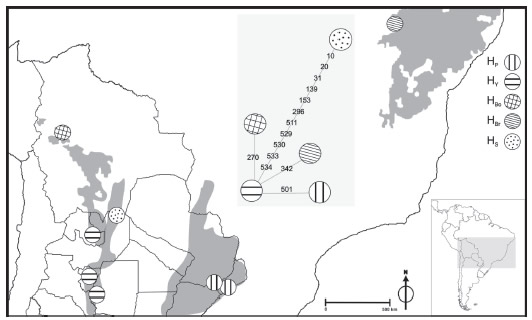
Fig. 2: Red de haplotipos y distribución geográfica de los mismos. Las áreas sombreadas corresponden a los núcleos SDTFs a los que pertenecen las poblaciones de los individuos analizados.
Las estimaciones de distancia genética entre los haplotipos tomados de a pares presentaron valores entre 0,000 y 0,017 en tanto que el tiempo de divergencia entre los haplotipos presentó valores comprendidos entre 0,000 y 5.600.000 años, aproximadamente (Tabla 3).
Tabla 3: Comparaciones de a pares entre los haplotipos.

Por encima de la diagonal se indican las distancias genéticas y por debajo se indican los tiempos estimados de divergencia.
Discusión
El intrón trnL presenta universalidad entre las especies vegetales, desde algas hasta gimnospermas y angiospermas, por lo cual ha sido extensamente utilizado en estudios filogeográficos y filogenéticos.
Esta universalidad puede ser explicada debido a que las regiones intrónicas se encuentran flanqueadas por regiones codificantes altamente conservadas (Haider, 2011).
La variabilidad genética del intrón trnL en las secuencias analizadas fue moderada considerando la baja tasa mutacional del genoma cloroplástico, la cual ha sido estimada como 20 veces menor que la del genoma mitocondrial animal (Palmer et al., 1988). La elevada frecuencia de transversiones (50%), a pesar de que las mismas presentan una menor probabilidad de ocurrencia desde el punto de vista estereoquímico, podría explicarse debido a la existencia de una correlación entre la proporción transición/transversión y la composición de los nucleótidos adyacentes en las regiones no codificantes. Se demostró que los nucleótidos flanqueados por A o por T presentan una tendencia significativa hacia las transversiones (Kelchner, 2000), por lo tanto, dado que el genoma cloroplástico es rico en nucleótidos A y T pueden resultar frecuentes las transversiones en este genoma.
El nivel de polimorfismo se reflejó en la red de haplotipos, la misma representa gráficamente las relaciones entre ellos basándose en los eventos mutacionales ocurridos. El haplotipo HY, portado por los individuos pertenecientes a la provincia fitogeográfica de las Yungas, se encuentra en el centro de la red y comparte la mayor cantidad de conexiones con los haplotipos restantes. Sin embargo, debido a que la realización de esta red no incluyó frecuencias haplotípicas, no es posible confirmar las relaciones de ancestralidad
Esta universalidad puede ser explicada debido a que las regiones intrónicas se encuentran flanqueadas por regiones codificantes altamente conservadas (Haider, 2011).
La variabilidad genética del intrón trnL en las secuencias analizadas fue moderada considerando la baja tasa mutacional del genoma cloroplástico, la cual ha sido estimada como 20 veces menor que la del genoma mitocondrial animal (Palmer et al., 1988). La elevada frecuencia de transversiones (50%), a pesar de que las mismas presentan una menor probabilidad de ocurrencia desde el punto de vista estereoquímico, podría explicarse debido a la existencia de una correlación entre la proporción transición/transversión y la composición de los nucleótidos adyacentes en las regiones no codificantes. Se demostró que los nucleótidos flanqueados por A o por T presentan una tendencia significativa hacia las transversiones (Kelchner, 2000), por lo tanto, dado que el genoma cloroplástico es rico en nucleótidos A y T pueden resultar frecuentes las transversiones en este genoma.
El nivel de polimorfismo se reflejó en la red de haplotipos, la misma representa gráficamente las relaciones entre ellos basándose en los eventos mutacionales ocurridos. El haplotipo HY, portado por los individuos pertenecientes a la provincia fitogeográfica de las Yungas, se encuentra en el centro de la red y comparte la mayor cantidad de conexiones con los haplotipos restantes. Sin embargo, debido a que la realización de esta red no incluyó frecuencias haplotípicas, no es posible confirmar las relaciones de ancestralidad existentes entre ellos. La posición distal de HS, como consecuencia del elevado número de pasos mutacionales, no presenta relación con la distancia geográfica entre los individuos que portan los haplotipos de referencia. Esta observación no puede ser explicada desde los resultados de este trabajo, sin embargo deja planteado un interrogante a resolver en futuros estudios los cuales deberían incluir mayor número de individuos provenientes de este sitio.
Los cambios climáticos pueden alterar el equilibrio demográfico (Hewitt, 2000) siendo una de sus posibles consecuencias la expansión del área de distribución de una especie (Excoffier& Ray, 2008), conllevando cambios en el acervo génico de sus poblaciones (Lehe et al., 2012). Estudios teóricos muestran que la diversidad genética en las poblaciones en expansión disminuye a medida que aumenta el área de distribución, como resultado de efectos fundadores sucesivos (Excoffier& Ray, 2008), debido a que el acervo génico disponible para la nueva población es proporcionado sólo por unos pocos individuos (Hallatschek& Nelson, 2008).
Al ubicar la distribución de los haplotipos HY y HP sobre el mapa de paleodistribución propuesto por Werneck et al. (2011), se observa que éstos quedan ubicados en áreas inestables de baja probabilidad de ocurrencia de los SDTFs durante el Pleistoceno (Fig. 3). Así, debido a esta disposición de los haplotipos, podría inferirse que estas áreas fueron colonizadas con posterioridad al LGM. Cabe recordar que, según Werneck et al. (2011), las áreas inestables habrían sido colonizadas recientemente, y por lo tanto deberían retener las huellas genéticas de las expansiones poblacionales pasadas lo cual explicaría la baja variabilidad genética presente entre ambos haplotipos.

Fig. 3: Distribución de los haplotipos HY (▲) y HP (●) en el mapa de paleodistribución propuesto por Werneck et al. (2011) para el Pleistoceno. En colores claros se representan las áreas de baja probabilidad de ocurrencia de los SDTFs mientras que los colores oscuros representan áreas estables de alta probabilidad de ocurrencia de los SDTFs.
En relación a los tiempos de divergencia estimados y considerando que el Pleistoceno tuvo su inicio 2,6 millones de años atrás, la separación entre los núcleos Misiones y Caatinga podría haber ocurrido durante ese tiempo al igual que la separación entre los núcleos Misiones y Pedemonte Subandino. Estas observaciones concuerdan con los resultados empíricos obtenidos desde diferentes enfoques metodológicos (Caetano et al., 2008; Werneck et al., 2011).
A pesar de los elevados niveles de biodiversidad, el conocimiento de los patrones filogeográficos y genéticos en el Neotrópico es deficiente comparado con el conocimiento que se tiene de las regiones templadas. Además, dentro del Neotrópico, la mayoría de los estudios filogeográficos se concentran en las regiones neotropicales húmedas de estos bosques, mostrando una variedad de patrones históricos complejos en respuesta a las fluctuaciones climáticas (Cavers et al., 2003; Ramos et al., 2007). Caetano et al. (2008) realizaron un estudio filogeográfico en Astronium urundeuva, una especie de los SDTFs, analizando dos regiones no codificantes del ADNcp en individuos provenientes de los núcleos Caatinga, Misiones y Pedemonte Subandino. En dicho estudio se identificaron cinco haplotipos que no presentaron una distribución geográfica aleatoria. Los núcleos Caatinga y Pedemonte Subandino presentaron cada uno de ellos un haplotipo característico fijado mientras que el núcleo Misiones presentó tres haplotipos.
El tiempo de divergencia estimado entre los haplotipos pertenecientes a individuos de A. colubrina var. cebil provenientes de los núcleos Caatinga y Misiones fue de 1.300.000 años, aproximadamente. En tanto que el tiempo de divergencia entre los haplotipos de individuos provenientes de los núcleos Misiones y Pedemonte Subandino fue de 600.000 años, aproximadamente. Estos resultados concuerdan con la propuesta realizada por Naciri et al. (2006) quienes mediante estudios filogeográficos clásicos en A. urundeuva, basándose en la topología de la red y en los niveles de polimorfismo, propusieron que las poblaciones del núcleo Pedemonte Subandino localizadas en el norte argentino son resultado de la expansión de las poblaciones del núcleo Misiones localizadas en Paraguay. De la misma manera, el mayor tiempo de divergencia entre los haplotipos de los individuos provenientes de los núcleos Misiones y Caatinga concuerdan con la propuesta de Caetano et al. (2008), quienes proponen un aislamiento antiguo entre las poblaciones de los núcleos Misiones y Pedemonte Subandino en relación a las del núcleo Caatinga.
Por su parte, dentro del núcleo Pedemonte Subandino, el tiempo de divergencia entre HY y HBo tomó un valor de cero, correspondiéndose con una distancia genética nula. El modelo Kimura 2-parámetros, empleado para el cálculo de la distancia genética, no considera las mutaciones de tipo indels subestimando, de esta manera, la distancia genética entre ellos y como consecuencia el tiempo de divergencia no puede ser establecido.
La distribución contemporánea de la variación genética del intrón trnL en A. colubrina podría asociarse a la distribución discontinua de la especie. La separación histórica conduce a la acumulación de mutaciones, aunque se requiere de un largo periodo de tiempo desde la coalescencia para que dos regiones de ADN no recombinantes muestren grandes diferencias en sus secuencias nucleotídicas (Avise, 2009). De esta manera, la distribución contemporánea de la variación genética de este intrón podría estar reflejando signos de divergencia histórica entre los núcleos de distribución de A. colubrina.
Conclusiones
El análisis de la región trnL en A. colubrina permitió detectar diferencias genéticas entre los núcleos de distribución de esta especie y contextualizarlas en un marco temporal. Las diferencias genéticas observadas resultarían de divergencia histórica asociada a diferentes eventos de expansión - fragmentación ocurridos durante la evolución de los SDTFs.
Este trabajo constituye una aproximación previa a un estudio filogeográfico el cual contribuiría al entendimiento del efecto de los cambios climáticos históricos sobre la distribución geográfica de una especie de los SDTFs.
Agradecimientos
Este trabajo fue parcialmente financiado mediante el subsidio FONCyT-PICTO UNaM 2011 N° 0133 del cual es Investigadora Responsable M. V. García. Parte de los resultados informados aquí corresponden al trabajo final de graduación de V. Calonga Solís para optar el grado académico de Licenciada en Genética.
Bibliografía
1. AVISE, J. C. 2009. Phylogeography: retrospect and prospect. J. Biogeogr. 36: 3-15. [ Links ]
2. BANDELT, H. J., P. FORSTER& A. RÖHL. 1999. Median-joining networks for inferring intraspecific phylogenies. Molec. Biol. Evol. 16: 37-48. [ Links ]
3. BARRANDEGUY, M. E., M. V. GARCIA, K. PRINZ, R. RIVERA POMAR& R. FINKELDEY. 2014. Genetic structure of disjunct Argentinean populations of the subtropical tree Anadenanthera colubrina var. cebil (Fabaceae). Plant. Syst. Evol. 300: 1693-1705 [ Links ]
4. BESSEGA, C., H. E. HOPP& R. H. FORTUNATO. 2008. Toward a Phylogeny of Mimosa (Leguminosae: Mimosoidae): A Preliminary Analysis of Southern South American Species Based on Chloroplast DNA Sequence. Ann. Missouri Bot. Gard. 95: 567-579. [ Links ]
5. BEHEREGARAY, L. B. 2008. Twenty years of phylogeography: the state of the field and the challenges for the Southern Hemisphere. Molec. Ecol. 17: 3754-74. [ Links ]
6. CAETANO S., D. PRADO, R. T. PENNINGTON, S. BECK, A. OLIVEIRA-FILHO, R. SPICHIGER& Y. NACIRI. 2008. The history of Seasonally Dry Tropical Forests in eastern South America: inferences from the genetic structure of the tree Astronium urundeuva (Anacardiaceae). Molec. Ecol. 17: 3147- 59. [ Links ]
7. CAVERS, S., C. NAVARRO& A. J. LOWE. 2003. Chloroplast DNA phylogeography reveals colonization history of a Neotropical tree, Cedrela odorata L., in Mesoamerica. Molec. Ecol. 12: 1451-1460. [ Links ]
8. CIALDELLA, A. M. 2000. Fabaceae. Subfamilia Mimosoideae, Flora Fanerogámica Argentina, Fascículo 67: 1-10. Programa Pro Flora-CONICET, Córdoba. [ Links ]
9. CORRIVEAU J. L.& A. W. COLEMAN. 1988. Rapid screening method to detect potential biparental inheritance of plastid DNA and results for over 200 angiosperm species. Amer. J. Bot. 75: 1443-1458. [ Links ]
10. ECKERT, A. 2011. Seeing the forest for the trees: statistical phylogeography in a changing world. New Phytologist. 189: 894-897 [ Links ]
11. ENNOS, R. A. 1994. Estimating the relative rates of pollen and seed migration among plant populations. Heredity 72: 250-259. [ Links ]
12. EXCOFFIER, L.& N. RAY. 2008. Surfing during population expansions promotes genetic revolutions and structuration. Trends Ecol. Evol. 23: 347-351. [ Links ]
13. FORSTER, J. W., E. S. JONES, R. KOLLIKER, M. C. DRAYTON, J. L. DUMSDAY, M. P. DUPAL, K. M. GUTHRIDGE, N. L. MAHONEY, E. VAN ZIJLL DE JONG& K. F. SMITH. 2001. Development and implementation of molecular markers for forage crop improvement. In: G. Spangenberg (ed.), Molecular breeding of forage crops, pp. 101-133. Springer Netherlands, Dordrecht. [ Links ]
14. GARCÍA, M. V., P. A. BALATTI& M. J. ARTURI. 2007. Genetic variability in natural populations of Paspalum dilatatum Poir. Analyzed by means of morphological traits and molecular markers. Genet. Resources Crop Evol. 54: 935-946. [ Links ]
15. HALLATSCHEK, O.& D. R. NELSON. 2008. Gene surfing in expanding populations. Theor. Populat. Biol. 73:158-170. [ Links ]
16. HAIDER, N. 2011. Chloroplast-specific universal primers and their uses in plant studies. Biol. Pl. 55:225-236. [ Links ]
17. HEWITT G. 2000. The genetic legacy of the Quaternary ice ages. Nature. 405:907-913 [ Links ]
18. JUSTINIANO M. J.& T. S. FREDERICKSEN. 1998. Ecología y Silvicultura de especies menos conocidas: Curupaú. Anadenanthera colubrina (Vell.) Benth. Mimosoideae. Bolfor Proyecto de Manejo Forestal Sostenible 1-30. [ Links ]
19. KELCHNER, S. A. 2000. The evolution of non-coding chloroplast DNA and its application in plant systematics. Ann. Missouri Bot. Gard. 2000: 482- 498. [ Links ]
20. LEHE, R., O. HALLATSCHEK& L. PELITI. 2012. The rate of beneficial mutations surfing on the wave of a range expansion. PLoS comput. biol. 8: 1-13. [ Links ]
21. LUCKOW M., J. T. MILLER, D.J. MURPHY& T. LIVSHULTZ. 2003. A phylogenetic analysis of the Mimosoideae (Leguminosae) based on chloroplast DNA sequence data. Adv. Legum. Syst. PT 10. 197- 220, Royal Botanic Gardens, Kew. [ Links ]
22. NACIRI Y., S. CAETANO, D. PRADO, R. SPICHIGER& R. T. PENNINGTON. 2006. Population Genetics and Inference of Ecosystem History: An Example Using Two Neotropical Seasonally Dry Forest Species. In: PENNINGTON R. T., G. P. LEWIS& J. A. RATER (eds.), Neotropical Savannas and Seasonally Dry Forests: Plant Diversity, Biogeography, and Conservation, Systematics Association Special Vol. 69: 417-432. CRC Press, Boca Raton. [ Links ]
23. NEI, M.& S. KUMAR. 2000. Molecular Evolution and Phylogenetics. Oxford University Press, Oxford. [ Links ]
24. PALMER, J. D., R. K. JANSEN, H. J. MICHAELS, M. W. CHASE& J. R. MANHART. 1988. Chloroplast DNA variation and plant phylogeny. Ann. Missouri Bot. Gard. 75: 1180-1206. [ Links ]
25. PENNINGTON, R. T., D. E. PRADO& C. A. PENDRY. 2000. Neotropical seasonally dry forests and Quaternary vegetation changes. J. Biogeogr. 27: 261-273. [ Links ]
26. PETIT R. J.& G. G. VENDRAMIN. 2007. Plant phylogeography based on organelle genes: an introduction. In: WEISS S. & N. FERRAND (eds.), Phylogeography of southern European refugia 23-97. Springer Netherlands, Dordrecht. [ Links ]
27. PRADO, D. E. & P. E. GIBBS. 1993. Patterns of species distributions in the dry seasonal forests of South America. Ann. Missouri Bot. Gard. 80: 902-927. [ Links ]
28. PRADO, D. 2000. Seasonally dry forest of tropical South America: from forgotten ecosystems to a new phytogeography unit. Edinburgh J. Bot. 57: 437-461. [ Links ]
29. PROVAN, J., N. SORANZO, N. J. WILSON, D. GOLSTEIN& W. POWEL. 1999. A Low Rate For Chloroplast Microsatellites. Genetics 153: 943-947. [ Links ]
30. RAMOS, A. C. S., J. P. LEMOS-FILHO, R. ACÁCIO RIBEIRO, F. RODRIGUES SANTOS & M. B. LOVATO. 2007. Phylogeography of the tree Hymenaea stigonocarpa (Fabaceae: Caesalpinioideae) and the influence of quaternary climate changes in the Brazilian cerrado. Ann. Bot. 100: 1219-28. [ Links ]
31. RAMOS, A. C. S., J. P. LEMOS-FILHO& M. B. LOVATO. 2009. Phylogeographical structure of the neotropical forest tree Hymenaea courbaril (Leguminosae: Caesalpinioideae) and its relationship with the Vicariant Hymenaea stigonocarpa from Cerrado. J. Heredity 100: 206- 16. [ Links ]
32. RAVI, V., J. P. KHURANA, A. K. TYAGI& P. KHURANA. 2008. An update on chloroplast genomes. Plant Syst. Evol. 271: 101-122. [ Links ]
33. RIBEIRO, R. A., J. P. LEMOS-FILHO, A. C. S. RAMOS& M. B. LOVATO. 2011. Phylogeography of the endangered rosewood Dalbergia nigra (Fabaceae): insights into the evolutionary history and conservation of the Brazilian Atlantic Forest. Heredity 106: 46-57. [ Links ]
34. ROZAS J. 2009. DNA Sequence Polymorphism Analysis Using DnaSP, en David Posadas (ed.), Bioinformatics for DNA Sequence Analysis. Methods Mol. Biol. 537: 337-350. [ Links ]
35. SCHAAL, B. A., D. A. HAYWORTH, K. M. OLSEN, J. T. RAUSCHER& W. A. SMITH. 1998. Phylogeographic studies in plants: problems and prospects. Molec. Ecol. 7: 465-474. [ Links ]
36. STEINER, J. J., C. J. POCKLEMBA, R. G. FJELLSTROM& L. F. ELLIOT. 1995. A rapid one tube genomic DNA extraction process for PCR and RAPD analyses. Nucl. Acids Res. 23: 2569-2570. [ Links ]
37. TABERLET, P., L. GIELLY, G. PAUTOU,& J. BOUVET. 1991. Universal primers for amplification of three non-coding regions of chloroplast DNA. Plant Mol. Biol. 17:1105-1109. [ Links ]
38. TAMURA K., D. PETERSON, N. PETERSON, G. STECHER, M. NEI& S. KUMAR. 2011. MEGA5: Molecular Evolutionary Genetics Analysis Using Maximum Likelihood, Evolutionary Distance, and Maximum Parsimony Methods. Molec. Biol. Evol. 28: 2731-2739. [ Links ]
39. THOMPSON, J., T. GIBSON, F. PLEWNIAK, F. JEANMOUGIN& D. HIGGINS. 1997. The ClustalX windows interface: Flexible strategies for multiple sequence alignment aided by quality analysis tools. Nucl. Acids Res. 25: 4876-4882. [ Links ]
40. TOLLEFSRUD, M. M., J. H. SØNSTEBØ, C. BROCHMANN, Ø. JOHNSEN, T. SKRØPPA& G. G. VENDRAMIN. 2009. Combined analysis of nuclear and mitochondrial markers provide new insight into the genetic structure of North European Picea abies. Heredity 102: 549-562. [ Links ]
41. WERNECK, F. P., G. C. COSTA, G. R. COLLI, D. E. PRADO& J. W. SITES. 2011. Revisiting the historical distribution of Seasonally Dry Tropical Forests: new insights based on palaeodistribution modelling and palynological evidence. Global Ecol. Biogeogr. 20: 272-288. [ Links ]
Recibido el 14 de junio de 2014,
aceptado el 14 de octubre de 2014.














