Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Boletín de la Sociedad Argentina de Botánica
versión On-line ISSN 1851-2372
Bol. Soc. Argent. Bot. vol.50 no.3 Córdoba set. 2015
GENÉTICA Y EVOLUCIÓN
Estimación del tamaño del genoma en especies de la tribu Cestreae (Solanaceae) mediante citometría de imagen
Aquiles Frossasco1, Alejandra Trenchi1 y Juan D. Urdampilleta1*
1 Instituto Multidisciplinario de Biología Vegetal (IMBIV), CONICET and Universidad Nacional de Córdoba. Córdoba, Argentina.
* Corresponding Author. E-mail: juanurdampilleta@hotmail.com
Resumen
Los géneros Cestrum, Sessea y Vestia pertenecen a la tribu Cestreae y presentan varias características cromosómicas que los diferencian de otras especies de Solanaceae, como el número y tamaño cromosómico. El tamaño del genoma contribuiría a generar nuevas hipótesis sobre su evolución en los clados basales de Solanaceae. Para estimar el tamaño del genoma en especies de Cestreae fue utilizada la citometría de imagen de núcleos en interfase y telofase teñidos con la técnica de Feulgen. Los valores del tamaño del genoma en las especies analizadas de Cestreae varían entre 17,22 a 24,40 Gpb (2C), siendo los mayores tamaños de genoma de la familia. Además los resultados demuestran que la citometría de imagen es un método alternativo a la citometría de flujo que permite la determinación del tamaño del genoma en forma práctica y económica.
Palabras clave: Cestrum; Sessea; Vestia; Tamaño del genoma; Citometría de imagen.
Summary
Estimation of genome size in species of the tribe Cestreae (Solanaceae) by image cytometry.
Cestrum, Sessea and Vestia genera belong to Cestreae tribe and have several chromosome characteristics that differentiate it from other species of Solanaceae, such as number and size. Genome size would contribute to new hypotheses about evolution in basal clades of Solanaceae. To estimate the genome size Cestreae species was used image cytometry of telophase and interphase nuclei stained with Feulgen technique. The values of genome size in species analyzed ranging from 17.22 to 24.40 Gpb (2C), being the largest genome sizes of the family. Furthermore, the results show that the image cytometry is an alternative method to flow cytometry for the determination of the genome size in practical and economical way.
Key words: Cestrum; Sessea; Vestia; Genome size; Image cytometry.
Introducción
Los géneros Cestrum L., Sessea Ruiz & Pav. y Vestia Willd. pertenecen a la tribu Cestreae (G. Don). Esta tribu es un clado monofilético de la subfamilia Cestroideae (Olmstead et al., 2008), considerada un grupo basal dentro de la familia Solanaceae (Benítez de Rojas & D'Arcy, 1998). Sus representantes son árboles o arbustos que se distribuyen en América tropical y subtropical (Hunziker, 1979).
Cestrum es el género más numeroso de la tribu (Judd et al., 1999), con alrededor de 250 especies distribuidas en las regiones tropicales y subtropicales de América (D'Arcy, 1986), desde el norte de México y el sur de Florida hasta el sur de Chile. Incluye hierbas, arbustos y árboles de hasta 12 m de altura, con flores actinomorfas, corola tubular-infundibuliforme y fruto tipo baya (Cronquist, 1988). Algunas especies de Cestrum presentan importancia económica ya que proporcionan la base para la extracción de compuestos activos como alcaloides, esteroides, saponinas y taninos (Schultes & Raffauf, 1991). Una especie en particular, Cestrum parqui, es temida por su toxicidad para el ganado (McLennan & Kelly, 1984). Sessea está restringida a zonas tropicales de América del Sur, especialmente en la región andina, e incluye unas 15 especies. Vestia es un género monotípico y endémico de la región centro - sur de Chile.
Caracteres florales, tipo de fruto y forma de semillas fueron utilizados para distinguir géneros y especies (Francey, 1934; Benítez de Rojas & Nee, 2001). A menudo las especies están separadas por diferencias morfológicas sutiles, lo que dificulta la delimitación específica y el desarrollo de una clasificación natural. Actualmente no existe un tratamiento taxonómico integral que incluya las especies de la tribu. Los escasos estudios moleculares resultan en ciertos aspectos contradictorios con estudios morfológicos (Montero-Castro et al., 2006). Por ello, resulta necesaria la búsqueda de nuevas fuentes de variabilidad que permitan una mejor comprensión de las relaciones filogenéticas de este grupo.
Desde el punto de vista de los cariotipos, la tribu Cestreae presenta varias particularidades que la diferencian de las otras especies de Solanaceae, lo cual confirma su monofilia: 1) Todas las especies estudiadas de Cestrum, Sessea y Vestia poseen 2n = 16 (Tschischow, 1956; Fedorov, 1974; Goldblatt & Johnson, 1979; Goldblatt, 1984) y los mayores tamaños cromosómicos de la familia (Fregonezi et al., 2006; Las Peñas et al., 2006; Fernandes et al., 2009); 2) Complejo patrón de heterocromatina en Cestreae (Berg & Greilhuber, 1993; Fregonezi et al., 2006), que permite diferenciar especies y grupos de especies; 3) Cromosomas B con diferencias en el patrón de bandeo (Fregonezi et al., 2004) y distribución de ADNr 45S y 5S (Sykorova et al., 2003a; Urdampilleta et al., 2014); y 4) Ausencia de telómeros Arabidopsis-type (TTTAGGG)n, reemplazados por regiones de ADN repetitivo ricos en A/T (Sykorova et al., 2003b). Estos hechos hacen de la tribu Cestreae un excelente modelo para estudios de evolución del genoma.
El tamaño del genoma (valor C) en Angiospermae es variable, aproximadamente 2400 veces su magnitud (Pellicer et al., 2010), lo cual ha estimulado especulaciones con respecto a la evolución del genoma (Soltis et al., 2003). La citometría de imagen combinada con la tinción de Feulgen (Vilhar et al., 2001; Hardie et al., 2002) es uno de los métodos utilizados para estimar la cantidad de ADN por núcleo (Bacic et al., 2007; Acosta et al., 2012). Las reconstrucciones de tamaño del genoma indican que el genoma ancestral de angiospermas y gimnospermas seria pequeño (1C< = 1,4 pg) (Leitch et al., 1998). Estudios realizados en especies pertenecientes a la familia Solanaceae indican que su genoma varía desde 0,63 a 24,80 pg (1C), sugiriendo que la amplia diversidad taxonómica de la familia es acompañada por importantes cambios en la estructura del genoma (Bennett & Leitch, 2012). Sin embargo, el valor C en especies de la tribu Cestreae no fue reportado hasta el momento. Si bien no existe una estricta relación directa, la presencia de cromosomas grandes sugiere que el tamaño del genoma en Cestreae podría ser uno de los mayores de la familia (Fregonezi et al., 2006; Las Peñas et al., 2006; Fernandes et al., 2009; Urdampilleta et al., 2014). La caracterización del tamaño del genoma en Cestreae podría contribuir a generar nuevas hipótesis sobre la evolución del genoma en los clados basales de Solanaceae. Por esta razón el objetivo de este estudio es caracterizar el tamaño del genoma en ocho especies de los tres géneros de la tribu.
Materiales y métodos
Las especies estudiadas de Cestrum, Sessea y Vestia y sus datos de colección son detallados en la Tabla 1. Para la estimación del tamaño fueron utilizados los métodos basados en citometría de imagen (Hardie et al., 2002), utilizando como estándar de referencia a Secale cereale "Dankovske" (2C = 16,19 pg) (Dolezel et al., 1998), cedidos por Jaroslav Dolezel del Institute of Experimental Botany de Czech Republic.
Para corroborar los números cromosómicos y la estructura general del cariotipo de las especies estudiadas fueron utilizados meristemas radiculares pretratados con 8-hidroxiquinolina 2 mM a 14ºC por 4-5 h, fijados en 3:1 (v/v) de etanol: ácido acético durante 24 horas y conservadas a -20ºC. Los preparados cromosómicos fueron realizados mediante aplastado en ácido acético 45%, posteriormente fueron congelados en nitrógeno líquido para eliminación de cubreobjetos y secados al aire. La coloración fue realizada con DAPI (4-6-diamidino-2-fenilindol) 1,5 µg/ml, y montado con Vectashield® (Vector Lab.). Para la obtención de imágenes de cromosomas fue utilizado el software Cytovision® (Leica Biosystems) y cámara JAI® modelo CV-M4+ CL monocromática acoplada a microscopio Olympus BX61.
La estimación del tamaño de genoma por citometría de imagen se realizó mediante el análisis de núcleos en interfases tardías/profases tempranas y telofase coloreados con la técnica de Feulgen (Greilhuber & Temsch, 2001; PraÇa-Fontes et al., 2011). Dichos núcleos fueron obtenidos de meristemas radiculares en activo crecimiento, fijados en 3:1 (v/v) de etanol: ácido acético durante 24 horas y conservadas a -20ºC. Las raíces fueron lavadas en dH2O, hidrolizadas en solución de HCl 1M durante 10 min a 60ºC y nuevamente lavadas en dH2O. Los meristemas fueron sumergidos en reactivo de Schiff durante 24 hs a 4ºC y se lavaron tres veces durante 10 min en agua sulfurosa. La maceración y aplastado del tejido se llevó a cabo en solución de ácido acético glacial al 45%. El cubreobjetos fue retirado mediante congelamiento en nitrógeno líquido, y previo secado al aire, el preparado fue montado con Entellan (Merck®). Las preparaciones citológicas de las muestras fueron realizadas en simultáneo con raíces del estándar de referencia, las cuales fueron fijadas y coloreadas bajo las mismas condiciones. Los núcleos en diferentes fases de la mitosis, en especial telofases e interfases tardías/profases tempranas de muestras y estándar, fueron fotografiados con microscopio Axiophot Zeiss acoplado con cámara digital Leica DFC300FX y sistema de captura de imagen Leica LAS EZ Versión 3.0.
La densidad óptica integrada (IOD) de cada núcleo fue determinada analizando los valores de grises en el canal verde de cada imagen RGB obtenida mediante la utilización del software ImageJ 1.46 (http://rsb.info.nih.gov/ij/index.html) siguiendo el método descripto por Hardie et al. (2002). A partir de estos valores de grises obtenidos se calculan las IOD, mediante el siguiente algoritmo:
![]()
Donde IOD = valor de IOD nuclear; IFi = valor de intensidad del pixel i dentro del núcleo; IBi = valor de intensidad del pixel i de la región libre de núcleos (background). El análisis de las imágenes para las determinaciones de los IODs fue realizado mediante la generación de un macro utilizando parámetros fijos de determinación de umbrales ("Triangle dark") dentro del software Image J.
El tamaño del genoma nuclear de la muestra se midió, en picogramos (pg), utilizando los valores de IOD de la muestra y del estándar primario. Para este propósito, se utilizó la siguiente fórmula:
![]()
En la que el 2Cs = C contenido de ADN nuclear de la muestra; 2Cp = C contenido de ADN nuclear del estándar primario (2C), IODs = valor IOD nuclear de la muestra, e IODp = valor IOD nuclear del estándar primario. Para estimar el tamaño del genoma en nucleótidos fue utilizada la relación 1 pg DNA = 978 Mbp (Dolezel et al., 2003). Los resultados obtenidos de valores de tamaño de genoma (2C) fueron comparados mediante ANOVA y test de Tukey con InfoStat (Di Rienzo et al., 2014).
Resultados y discusión
Todas las especies analizadas son diploides y presentan 2n = 16 (Fig. 1), confirmando los resultados previamente citados (Tschischow, 1956; Sharma & Sharma, 1957; Fedorov, 1974; Moscone, 1992; Fregonezi et al., 2006; Fernandes et al., 2009; Urdampilleta et al., 2014). Todas las especies poseen cromosomas metacéntricos y submetacéntricos grandes, que varían entre 7 y 13,5 mm, uno de los mayores tamaños cromosómicos de la familia Solanaceae. La presencia de cromosomas accesorios es frecuente en algunas especies (Sykorova et al., 2003a; Fregonezi et al., 2004), siendo detectado en C. nocturnum una cantidad variable entre 10 y 14 cromosomas B (Fig. 1D).

Fig. 1. Metafases coloreadas con DAPI de las distintas especies, C. elegans (A), C. fasciculatum (B), C. kunthii (C), C. nocturnum (D) (las flechas indican cromosomas B), C. parqui (E), C. strigillatum (F), S. regnellii (G), V. foetida (H). Escala = 10 mm.
En la Tabla 1 se describen por primera vez los resultados obtenidos en relación al tamaño del genoma de seis especies de Cestrum, una de Sessea y una de Vestia, mediante citometría de imagen. Se analizaron entre 44 y 89 núcleos en telofase tardía (2C) y profase temprana (4C) por especie. Como es de esperar, los valores de IODs en profase temprana fueron aproximadamente el doble de los respectivos valores en telofase. Los valores del tamaño del genoma (2C) en las especies analizadas de Ceastreae calculados por este método varían entre 17,61 pg en S. regnelli, lo que representaría un genoma de 17,22 Gpb, hasta 24,95 pg en C. parqui, un genoma de 24,40 Gpb (Tabla 1).
Tabla 1. Lista de especies estudiadas de Cestrum, Sessea y Vestia, detallando tamaño del genoma y datos de colección. σ: desvío estándar, n: número de núcleos analizados. NLD020: Experimental Garden of the Radboud University of Nijmegen; CORD: Herbario del Museo Botánico de Córdoba.

a Clasificación en grupos con base en el test de Tukey (a = 0,05, DMS = 1,10918).
* Tamaño del genoma en nucleótidos calculado según relación 1 pg DNA = 0,978 Gbp (Dolezel et al., 2003).
El ANOVA de las mediciones permitió detectar diferencias significativas entre las especies (F = 148,28, p < 0,0001), y el test de Tukey permite distinguir 4 categorías (a = 0,05, DMS = 1,10918) con base en la diferenciación de medias (Tabla 1). Desde una apreciación general, los valores del tamaño del genoma permiten observar dos grupos de especies (Tabla 1). Un grupo que presenta los tamaños mayores de genoma, que varía de 24,06 a 24,95 pg y que incluye cuatro especies: C. elegans (Figs. 1A, 2A y 2I), C. fasciculatum (Figs. 1B, 2B y 2J), C. nocturnum (Figs. 1D, 2D y 2L) y C. parqui (Figs. 1E, 2E y 2M). El segundo grupo se caracteriza por tamaños menores de genoma, que varían desde 17,61 a 21,11 pg, incluyendo las especies C. kunthii (Figs. 1C, 2C y 2K), C. strigilatum (Figs. 1F, 2F y 2N), S. regnelli (Figs. 1G, 2G y 2O) y V. foetida (Figs. 1H, 2H y 2P).
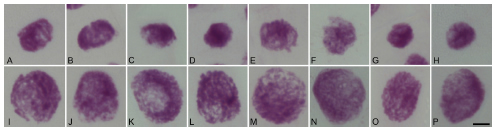
Fig. 2. Aspecto de los núcleos analizados en las distintas especies, C. elegans (A,I), C. fasciculatum (B,J), C. kunthii (C,K), C. nocturnum (D,L), C. parqui (E,M), C. strigillatum (F,N), S. regnellii (G,O), V. foetida (H,P). A-H, núcleos en telofase tardía (2C), I-P, núcleos en profase temprana (4C). Escala = 5 mm.
Dada la constancia en el número de cromosomas podemos considerar que las variaciones en el contenido de ADN serían resultado de diferencias en las cantidades de cromatina intracromosómica y/o por presencia de cromosomas accesorios (cromosomas B). Las especies analizadas demostraron poseer diferencias en cuanto a la cantidad y tipo de heterocromatina (Fregonezi et al., 2006; Fernandes et al., 2009) como así también por la presencia de cromosomas B (Urdampilleta et al., 2014). Sin embargo, esta variación no parece ser la causa de las variaciones del tamaño del genoma observadas ya que, por ejemplo en C. parqui posee el genoma mayor y la menor cantidad de heterocromatina y C. nocturnum posee una gran cantidad de cromosomas B (Urdampilleta et al., 2014) pero no difiere significativamente de otras especies. Por lo tanto, esta variación del tamaño del genoma podría estar relacionada con la variación de secuencias repetitivas dispersas en el genoma.
El tamaño del genoma (1C) en Solanaceae varía de 0,63 pg en S. chacoense a 24,80 en Cyphomandra hartwegii (Bennett & Smith, 1976; Pringle & Murray, 1991) y esta variación es característica de algunos clados. La tribu Cestreae es un grupo monofilético que posee varias características propias, como el número y tamaño de cromosomas, además nuestros resultados sostienen que estas especies, junto con el clado Cyphomandra del género Solanum (Bennett & Leitch, 2012), poseen los mayores tamaños de genoma de la familia.
Los estudios filogenéticos incluyen a Vestia como un clado que ocupa una posición basal en el árbol (Olmstead et al., 1999; Montero-Castro et al., 2006). Comparada con otras especies de Cestreae, V. foetida tiene el cariotipo más simétrico y con menor longitud de complemento cromosómico (Las Peñas et al., 2006). Esto es congruente con las observaciones sobre el tamaño del genoma ya que pertenece al grupo de genomas menores. Estas observaciones permiten suponer que el aumento del tamaño del genoma en Cestreae, es un carácter derivado dentro del grupo.
La variación del valor C es considerada una de las principales causas de divergencia específica (Castro-Jimenez et al., 1989; Slovák et al., 2009) y podría estar relacionada con variaciones en caracteres morfológicos (Poggio & Hunziker, 1986; Knight & Beaulieu, 2008; Slovák et al., 2009), hábitat (Leitch et al., 2009) y distribución (Bennett et al., 1998). Sin embargo, el significado funcional de esta variación y los mecanismos responsables de estos cambios son una incógnita. Modificaciones en el patrón de heterocromatina pueden llevar a variaciones en el tamaño de genoma (Pringle & Murray, 1993; Kao et al., 2001) y las regiones de ADN repetitivo son frecuentemente indicadas como responsables de este cambio (Uozu et al., 1997; She et al., 2012). Secuencias repetitivas dispersas, como elementos transponibles, pueden tornarse más activos en condiciones de stress, llevando a un incremento en la cantidad de DNA y cambios en la estructura del genoma (Kalendar et al., 2000; Raskina et al., 2008; Agren & Wright, 2011).
La reacción de Feulgen, en su formulación inicial, se utilizó para la detección de ADN (Feulgen & Rossenbeck, 1924). La demostración de su especificidad y relación estequiométrica con la cantidad de ADN, permitió utilizar esta tinción para la estimación del tamaño del genoma por microdensitometría. La citometría de imagen es un método que combina las ventajas de la reacción de Feulgen con el análisis de imágenes obtenidas desde un microscopio óptico, permitiendo la estimación del tamaño del genoma en forma confiable y de bajo costo. El análisis de imágenes Feulgen fue utilizado en estudios cuantitativos de ADN y permitió determinar el tamaño del genoma en varias especies de plantas (Venora et al., 1995; Cremonini et al., 1998; Voglmayr & Greilhuber, 1998; Vilhar et al., 2001) incluyendo algunas especies de Solanaceae (Acosta et al., 2012), demostrando ser un método aceptado para la determinación del tamaño del genoma de la plantas (PraÇa-Fontes et al., 2011).
Agradecimientos
Los autores agradecen a las agencias Argentinas CONICET, ANPCyT - FONCyT, MINCyT - Córdoba, and SECyT - UNC por su apoyo financiero.
Bibliografía
1. ACOSTA, M. C., M. GUERRA & E. A. MOSCONE. 2012. Karyological relationships among some South American species of Solanum (Solanaceae) based on fluorochrome banding and nuclear DNA amount. Pl. Syst. Evol. 298: 1547-1556. [ Links ]
2. AGREN, J. A. & S. I. WRIGHT. 2011. Co-evolution between transposable elements and their hosts: a major factor in genome size evolution? Chromosome Res. 19: 786. [ Links ]
3. BACIC, T., N. JOGAN & J. D. KOCE. 2007. Luzula sect. Luzula in the south-eastern Alps-karyology and genome size. Taxon 56: 129-136. [ Links ]
4. BENÍTEZ DE ROJAS, C. & W. G. D'ARCY. 1998. The genera Cestrum and Sessea (Solanaceae: Cestreae) in Venezuela. Ann. Mo. Bot. Gard. 85: 273-351. [ Links ]
5. BENÍTEZ DE ROJAS, C. & M. NEE. 2001. The neotropical genus Sessea (Solanaceae): a preliminary survey. In: VAN DE BERR, R. G., G. W. M. BARENDSE, G. M. VAN DER WERDEN & C. MARIANI (eds.), Solanaceae V: Advances in Taxonomy and Utilization, pp. 153-159. Nijmegen University Press. [ Links ]
6. BENNETT, M. D. & I. J. LEITCH . 2012. Plant DNA C-values Database (Release 6.0, Dec. 2012). http://data.kew.org/cvalues/. [ Links ]
7. BENNETT, M. D., I. J. LEITCH & L. HANSON. 1998. DNA amounts in two samples of angiosperm weeds. Ann. Bot. 82: 121-134. [ Links ]
8. BENNETT, M. D. & J. B. SMITH. 1976. Nuclear DNA amounts in angiosperms. Phil. Trans. R. Soc. B 274: 227-274. [ Links ]
9. BERG, C. & J. GREILHUBER. 1993. Cold-sensitive chromosome regions and heterochromatin in Cestrum (Solanaceae): C. strigillatum, C. fasciculatum, and C. elegans. Pl. Syst. Evol. 185: 133-151. [ Links ]
10. CASTRO-JIMENEZ, Y., R. J. NEWTON, H. J. PRICE & R. S. HALLIWELL. 1989. Drought stress responses of Microseris species differing in nuclear DNA content. Am. J. Bot. 76: 789-795. [ Links ]
11. CREMONINI, R., D. MIOTTO, M. A. NGU, D. TOTA, D. PIGNONE, S. BLANGIFORTI & G. VENORA. 1998. Cytology of Vicia Species. 5. Nuclear Chromatin Structure, Karyomorphological Analysis and DNA Content in Newly Discovered Relatives of Vicia faba L.: Vicia kalakhensis Khattab, Maxted et Bisby and Vicia eristalioides Maxted. Cytologia 63: 371-380. [ Links ]
12. CRONQUIST, A. 1988. The Evolution and Classification of Flowering Plants. New York: New York Botanic Gardens. [ Links ]
13. D'ARCY, W. G. 1986. Solanaceae, Biology and Systematics. Columbia University Press. [ Links ]
14. DI RIENZO J.A., CASANOVES F., BALZARINI M.G., GONZALEZ L., TABLADA M., ROBLEDO C.W. 2014. InfoStat versión 2014. Grupo InfoStat, FCA, Universidad Nacional de Córdoba, Argentina. http://www.infostat.com.ar. [ Links ]
15. DOLEZEL, J., J. BARTOS, H. VOGLMAYR & J. GREILHUBER. 2003. Nuclear DNA content and genome size of trout and human. Cytom. Part A 51: 127. [ Links ]
16. DOLEZEL, J., J. GREILHUBER, S. LUCRETTI, A. MEISTER, M. A. LYSAK, L. NARDI & R. OBERMAYER. 1998. Plant genome size estimation by flow cytometry: inter-laboratory comparison. Ann. Bot. 82: 17-26. [ Links ]
17. FEDOROV, A A. 1974. Chromosome numbers of flowering plants. Reprint by Otto Koeltz, Science Publishers, Königstein, West Germany. [ Links ]
18. FERNANDES, T., L. D. DE ALMEIDA REGO, M. NARDY, P. M. YUYAMA & A. L. L. VANZELA. 2009. Karyotype differentiation of four Cestrum species (Solanaceae) revealed by fluorescent chromosome banding and FISH. Genet. Mol. Biol. 32: 320-327. [ Links ]
19. FEULGEN, R. & H. ROSSENBECK. 1924. Mikroskopisch-chemischer Nachweis einer Nucleisäure vom Typus der Thymonucleinsäure und die darauf beruhende elektive Färbung von Zellkernen in mikroskopischen Präparaten. Hoppe-Seyler's Z. Physiol. Chem. 135: 203-248. [ Links ]
20. FRANCEY, P. 1934. Monographie du genre Cestrum L. Candollea 6: 46-398. [ Links ]
21. FREGONEZI, J. N., T. FERNANDES, J. M. D. TOREZAN, A. O. S. VIEIRA & A. L. L. VANZELA. 2006. Karyotype differentiation of four Cestrum species (Solanaceae) based on the physical mapping of repetitive DNA. Genet. Mol. Biol. 29: 97-104. [ Links ]
22. FREGONEZI, J. N., C. ROCHA, J. M. D. TOREZAN & A. L. L. VANZELA. 2004. The occurrence of different Bs in Cestrum intermedium and C. strigilatum (Solanaceae) evidenced by chromosome banding. Cytogenet. Genome Res. 106: 184-188. [ Links ]
23. GOLDBLATT, P. 1984. Index to Plant Chromosome Numbers 1979-1981. Monographs in Systematic Botany from Missouri Botanical Garden 8, St. Louis. [ Links ]
24. GOLDBLATT, P. & JOHNSON, D. E. 1979. Index to Plant Chromosome Numbers. Missouri Botanical Garden, St. Louis, http://www.tropicos.org/Project/IPCN. [ Links ]
25. GREILHUBER, J. & E. M. TEMSCH. 2001. Feulgen densitometry: some observations relevant to best practice in quantitative nuclear DNA content determination. Acta Bot. Croat. 60: 285-298. [ Links ]
26. HARDIE, D. C., T. R. GREGORY & P. D. HEBERT. 2002. From Pixels to Picograms A Beginners' Guide to Genome Quantification by Feulgen Image Analysis Densitometry. J. Histochem. Cytochem. 50: 735-749. [ Links ]
27. HUNZIKER, A. T. 1979. South American Solanaceae: A synoptic survey. In: HAWKES, J. G., R. N. LESTER & A. D. SKELDING (eds.), The Biology and Taxonomy of the Solanaceae, pp. 49-85. Academic Press, New York. [ Links ]
28. JUDD, W. S., C. S. CAMPBELL, E. A. KELLOGG, P. F. STEVENS & M. J. DONOGHUE. 1999. Plant systematics: a phylogenetic approach. Sinauer Associates, Sunderland. [ Links ]
29. KALENDAR, R., J. TANSKANEN, S. IMMONEN, E. NEVO & A. H. SCHULMAN. 2000. Genome evolution of wild barley (Hordeum spontaneum) by BARE-1 retrotransposon dynamics in response to sharp microclimatic divergence. Proc. Natl. Acad. Sci. USA 97: 6603-6607. [ Links ]
30. KAO, Y. Y., S. B. CHANG, T. Y. LIN, C. H. HSIEH, Y. H. CHEN, W. H. CHEN & C. C. CHEN. 2001. Differential accumulation of heterochromatin as a cause for karyotype variation in Phalaenopsis orchids. Ann. Bot. 87: 387-395. [ Links ]
31. KNIGHT, C. A. & J. M. BEAULIEU. 2008. Genome Size Scaling through Phenotype Space. Ann. Bot. 101: 759-766. [ Links ]
32. LAS PEÑAS, M. L., F. E. CHIARINI, G. BERNARDELLO & C. BENÍTEZ DE ROJAS. 2006. Karyotypes of some species of Cestrum, Sessea, and Vestia (tribe Cestreae, Solanaceae). Caryologia 59: 131-137. [ Links ]
33. LEITCH, I. J., M. W. CHASE & M. D. BENNETT. 1998. Phylogenetic analysis of DNA C-values provides evidence for a small ancestral genome size in flowering plants. Ann. Bot. 82: 85-94. [ Links ]
34. LEITCH, I. J., I. KAHANDAWALA, J. SUDA, L. HANSON, M. J. INGROUILLE, M. W. CHASE & M. F. FAY. 2009. Genome size diversity in orchids: consequences and evolution. Ann. Bot. 104: 169-481. [ Links ]
35. MCLENNAN, M. W. & W. R. KELLY. 1984. Cestrum parqui (green cestrum) poisoning in cattle. Aust. Vet. J. 61: 289-291. [ Links ]
36. MONTERO-CASTRO, J. C., A. DELGADO-SALINAS, E. DE LUNA & L. E. EGUIARTE. 2006. Phylogenetic Analysis of Cestrum Section Habrothamnus (Solanaceae) Based on Plastid and Nuclear DNA Sequences. Syst. Bot. 31: 843-850. [ Links ]
37. MOSCONE, E. A. 1992. Estudios de cromosomas meióticos en Solanaceae de Argentina. Darwiniana 31: 261-297. [ Links ]
38. OLMSTEAD, R. G., L. BOHS, H. A. MIGID, E. SANTIAGO-VALENTIN, V. F. GARCIA & S. M. COLLIER . 2008. A molecular phylogeny of the Solanaceae. Taxon 57: 1159-1181. [ Links ]
39. OLMSTEAD, R. J., J. A. SWEERE, R. E. SPANGLER, L. BOHS & J. D. PALMER. 1999. Phylogeny and provisional classification of the Solanaceae based on chloroplast DNA. In: NEE, M., D. E. SYMON, R. N. LESTER & J. P. JESSOP (eds.), Solanaceae IV, pp. 111-137. Kew: Royal Botanical Gardens. [ Links ]
40. PELLICER, J., M. F. FAY & I. J. LEITCH. 2010. The largest eukaryotic genome of them all? Bot. J. Linn. Soc. 164: 10-15. [ Links ]
41. POGGIO, L. & J. H. HUNZIKER. 1986. Nuclear DNA content variation in Bulnesia. J. Hered. 77: 43-48. [ Links ]
42. PRAÇA-FONTES, M. M., C. R. CARVALHO & W. R. CLARINDO. 2011. C-value reassessment of plant standards: an image cytometry approach. Plant Cell Rep. 30: 2303-2312. [ Links ]
43. PRINGLE, G. J. & B. G. MURRAY. 1991. Karyotype diversity and nuclear DNA variation in Cyphomandra. In: HAWKES, J. G., R. N. LESTER, M. NEE & N. ESTRADA-RAMOS (eds.), Solanaceae III: Taxonomy, Chemistry, Evolution, pp. 247-252. Royal Botanic Gardens, Kew. [ Links ]
44. PRINGLE, G. J. & B. G. MURRAY. 1993. Karyotypes and C-banding patterns in species of Cyphomandra Mart. ex Sendtner (Solanaceae). Bot. J. Linn. Soc. 111: 331-342. [ Links ]
45. RASKINA, O., J. C. BARBER, E. NEVO & A. BELYAYEV. 2008. Repetitive DNA and chromosomal rearrangements: speciation-related events in plant genomes. Cytogenet. Genome Res. 120: 351-357. [ Links ]
46. SCHULTES, R. E. & R. F. RAFFAUF. 1991. Phytochemical and Ethnopharmacological Notes on the Solanaceae of the Norhwest Amazon. In: HAWKES, J. G., R. N. LESTER, M. NEE & N. ESTRADA-RAMOS (eds.), Solanaceae III: Taxonomy, Chemistry, Evolution, pp. 25-49. The Royal Botanic Garden and The Linnean of London. [ Links ]
47. SHARMA, A. K. & A. SHARMA. 1957. Karyotype studies in Cestrum as an aid to taxonomy. Genetica 29: 83-100. [ Links ]
48. SHE, C. W., Y. C. SONG & X. H. JIANG. 2012. Comparative genomic in situ hybridization analysis on the chromosomes of five grass pecies with rice genomic DNA probe. Afr. J. Biotechnol. 11: 9508- 9515. [ Links ]
49. SLOVÁK, M., P. VÍT, T. URFUS & J. SUDA. 2009. Complex pattern of genome size variation in a polymorphic member of the Asteraceae. J. Biogeogr. 36: 372-384. [ Links ]
50. SOLTIS, D. E., P. S. SOLTIS, M. D. BENNETT & I. J. LEITCH. 2003. Evolution of genome size in the angiosperms. Am. J. Bot. 90: 1596-1603. [ Links ]
51. SYKOROVA, E., K. Y. LIM, M. W. CHASE, S. KNAPP, I. J. LEITCH, A. R. LEITCH & J. FAJKUS. 2003b. The absence of Arabidopsis-type telomeres in Cestrum and closely related genera Vestia and Sessea (Solanaceae): First evidence from eudicots. Plant J. 34: 283-291. [ Links ]
52. SYKOROVA, E., K. Y. LIM, J. FAJKUS & A. R. LEITCH. 2003a. The signature of the Cestrum genome suggests an evolutionary response to the loss of (TTTAGGG)n telomeres. Chromosoma 112: 164-172. [ Links ]
53. TSCHISCHOW, N. T. 1956. Número de cromosomas de algunas plantas chilenas. Bol. Soc. Biol. Concepción 31: 145-147. [ Links ]
54. UOZU, S., H. IKEHASHI, N. OHMIDO, H. OHTSUBO, E. OHTSUBO & K. FUKUI. 1997. Repetitive sequences: Cause for variation in genome size and chromosome morphology in the genus Oryza. Plant Mol. Biol. 35: 791-799. [ Links ]
55. URDAMPILLETA, J. D., F. CHIARINI, L. STIEFKENS & G. BERNARDELLO. 2014. Chromosomal differentiation of Tribe Cestreae (Solanaceae) by analyses of 18-5.8-26S and 5S rDNA distribution. Pl. Syst. Evol. DOI 10.1007/s00606-014-1158-x. [ Links ]
56. VENORA, G., B. OCAMPO, K. B. SINGH & F. SACCARDO. 1995. Karyotype of the Kabuli-type chickpea (Cicer arietinum L.) by image analysis system. Caryologia 48: 147-155. [ Links ]
57. VILHAR, B., J. GREILHUBER, J. D. KOCE, E. M. TEMSCH & M. DERMASTIA. 2001. Plant genome size measurement with DNA image cytometry. Ann. Bot. 87: 719-728. [ Links ] [ Links ]
Recibido el 14 de marzo de 2015,
aceptado el 12 de junio de 2015.













